阪崎克罗诺杆菌ESA_01393基因缺失及对表型的影响
2023-01-03宗文月杜欣军
李 萍,宗文月,杜欣军,王 硕
(1.食品营养与安全国家重点实验室,天津科技大学食品科学与工程学院,天津 300457;2.天津市食品科学与健康重点实验室,南开大学医学院,天津 300071)
阪崎克罗诺杆菌(Cronobacter sakazakii)是一种无芽孢、有周生鞭毛、能运动、兼性厌氧的革兰氏阴性杆菌[1],是一类能够在人和动物肠道内生存的机会致病菌,能够引起新生儿脑膜炎和坏死性小肠结肠炎[2].相关研究[3]表明,阪崎克罗诺杆菌是引起严重感染的机会病原体,尤其是在婴儿、老年人和免疫功能低下的人中.出生体质量小于2.5kg的婴儿和出生不足 28d的新生儿有更高的感染风险[4].阪崎克罗诺杆菌主要存在于婴幼儿配方奶粉及其他婴幼儿食品中[5],新生儿感染后的死亡率高达 80%[6],幸存者往往患有严重的、不可逆转的神经系统疾病.阪崎克罗诺杆菌在婴幼儿配方奶粉中的存活能力强,对婴幼儿的危害较大,引起了大众的广泛关注.
许多环境损伤和内源性过程可导致 DNA损伤,对细胞生存构成直接威胁.在大多数细菌中,DNA损伤通过应急反应(SOS反应)的转录调控过程进行处理[7-8].在大肠杆菌(Escherichia coli)中,SOS反应是细胞遇到DNA损伤因子后被诱导的一种由40多个基因组成的调控机制,其中许多基因参与了 DNA损伤的耐受和修复,如 recA、lexA、umuDC、recN、sulA、polB、uvrA、uvrB 和 uvrD[9-12].这个过程受 SOS调节因子LexA和RecA的控制.SOS反应通路是一种可诱导的 DNA损伤修复系统[13].在没有 DNA损伤的情况下,LexA阻止SOS基因的转录[14].当DNA损伤时,重组蛋白 RecA在单链 DNA上形成核微丝.RecA核蛋白丝激活 LexA自身蛋白水解,SOS反应基因的转录被打开[8,14].除了两个关键的 SOS反应调节因子 LexA和 RecA,其他一些压力源和压力反应也可以控制 SOS反应的因素[15].细菌暴露于DNA损伤剂以及其他环境和细胞内因素,这些因素会在宿主内的许多部位触发SOS反应[16].
与NCBI中大肠杆菌的yebG基因(DNA损伤诱导蛋白)序列的比对结果表明,阪崎克罗诺杆菌中的ESA_01393基因序列覆盖度为 67%,相似度为 68%.操纵子融合表明,在大肠杆菌中 yebG基因的表达受SOS反应调节因子 LexA和 RecA的调控[9,17].Uranga等[18]研究发现,yebG基因是SOS反应调控的一部分,在 DNA受到损伤后被高度诱导.丝裂霉素C处理大肠杆菌后,会诱导 yebG 基因的表达[17].因此,编码 DNA损伤诱导蛋白的 yebG基因被鉴定为大肠杆菌的一种新的 SOS反应调控基因[17],但基因的功能尚不清楚.为了进一步研究阪崎克罗诺杆菌中的 ESA_01393基因的功能,本研究在阪崎克罗诺杆菌 ATCCBAA-894中进行了基因敲除,并对其运动能力、生物膜形成能力、耐干燥能力以及 DNA损伤修复能力进行了评估,为后续阪崎克罗诺杆菌的致病性及耐干燥机制研究奠定基础.
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
阪崎克罗诺杆菌(Cronobacter sakazakii)ATCC BAA-894保存在本实验室的-80℃冰箱中.质粒pCVD442、质粒 pACYC184、大肠杆菌(Escherichia coli)S17-1λpir和大肠杆菌 DH5α由本实验室保存.质粒 pCVD442-Q-H和质粒 pACYC184-ESA_01393为本研究构建(表1).

表1 本实验所用菌株和质粒Tab.1 Bacteria strains and plasmids used in this study
1.1.2 试剂与仪器
细菌基因组DNA提取试剂盒、PCR产物纯化试剂盒、胶回收试剂盒及质粒提取试剂盒,Omega Bio-Tek公司;KOD酶、Taq酶和dNTP mix(10mmol/L),宝生物工程(大连)有限公司;氯霉素,上海麦克林生化科技有限公司;氨苄青霉素,BBI生命科学有限公司;限制性内切酶,美国 NEB公司;D2000DNA marker、1kb plus DNA marker、2×Taq PCR master mix和上样缓冲液,北京康为世纪生物科技有限公司;T4连接酶,赛默飞世尔科技公司;LB培养基,北京索莱宝科技有限公司;平板计数培养基(PCA计数培养基),青岛海博技术有限公司;其他试剂均为国产分析纯试剂.
PCR仪、电泳仪和凝胶成像仪,伯乐生命医学产品(上海)有限公司;Sunrise-Basic型酶标仪,帝肯奥地利有限责任公司;超微量分光光度计,德国Berthold公司;3-30K型台式高速冷冻离心机,德国Sigma公司.
1.2 实验方法
1.2.1 PCR引物设计
使用NCBI找到ESA_01393基因(GenBank号为WP_004386660.1),选取其上下游同源臂各700bp的相应序列,使用Premier 5设计ESA_01393基因上下游同源臂的引物ESA_01393-Q F/ESA_01393-Q R和ESA_01393-H F/ESA_01393-H R,并且分别在上下游同源臂引物的 5′端加入相应的酶切位点.ESA_01393 1-F/ESA_01393 1-R和ESA_01393 2-F/ESA_01393 2-R为基因敲除验证的引物,分别位于 ESA_01393基因上和 ESA_01393基因的两端各 700bp处.cpESA_01393 F/cpESA_01393 R为基因回补的引物,位于 ESA_01393基因上,并且在引物的 5′端加入相应的酶切位点.引物序列见表2.
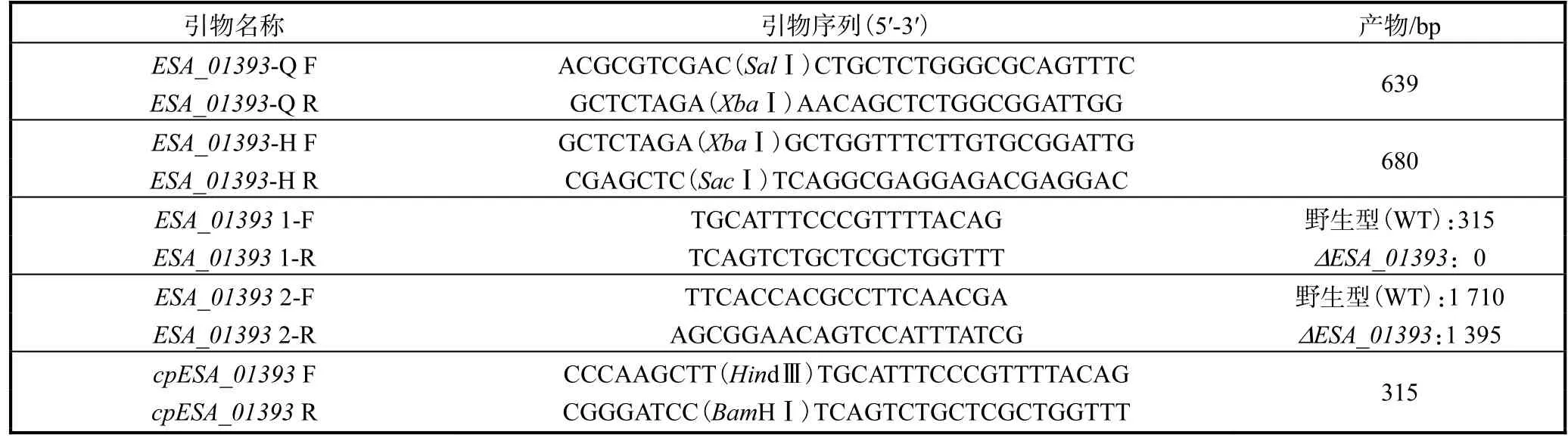
表2 引物序列Tab.2 Sequences of primers
1.2.2 基因敲除及回补株的构建
采用 Ji等[19]的方法并稍作改动,对 C.sakazakii ATCC BAA-894野生型(WT)进行基因突变.使用ESA_01393-Q F/ESA_01393-Q R和ESA_01393-H F/ESA_01393-H R两对引物从 WT的全基因组序列中扩增出待敲除基因的上下游片段(表2).将上下游片段克隆到 pCVD442自杀载体中,产生重组载体pCVD442-Q-H.将重组载体转化到大肠杆菌 S17-1λpir感受态中,通过电转化构建ΔESA_01393突变株.
使用 cpESA_01393F和 cpESA_01393R两个引物构建了含有 ESA_01393编码序列及其启动子的互补质粒 pACYC184-ESA_01393.将互补质粒克隆至ΔESA_01393突变株中,获得携带ESA_01393基因的回补株cpESA_01393.回补株引入的酶切位点分别为HindⅢ和BamHⅠ.
1.2.3 生长曲线的测定
根据文献[20]的方法测定细菌的生长曲线.将菌株 WT、ΔESA_01393和 cpESA_01393进行过夜培养,菌液以 1:100比例转接至新鲜的 LB液体培养基中,37℃、200r/min培养 14h.从转接开始,每隔1h取样,测定 600nm 处的吸光度(A600)直至平稳期,必要时稀释一定倍数.用 LB液体培养基作为空白对照,绘制生长曲线.每个样本重复3次.
1.2.4 运动性的测定
通过测定细菌软琼脂的迁移直径(0.3%)对运动性进行评估[21].将菌株 WT、ΔESA_01393和cpESA_01393分别接种于无菌的LB培养基中,37℃过夜培养.分别取 10μL菌液置于软琼脂运动板(含0.3%琼脂的 LB 琼脂培养基)上,30℃培养 16h,观察菌落大小,每个样本重复3次.
1.2.5 生物膜形成能力的测定
用结晶紫染色法测定阪崎克罗诺杆菌生物膜的形成能力[22].取吸光度介于 0.6~0.8之间的菌株WT、ΔESA_01393和 cpESA_01393的菌液制备悬浮液.每孔加入100μL悬浮液,37℃孵育48~72h;加入 200μL 99%甲醇固定生物膜,15min后弃去上清液.在室温下风干后,用 200μL 1%结晶紫染料染色30min,用无菌水洗涤3次;加入200μL 95%乙醇进行脱色.以培养基为阴性对照.使用Sunrise-Basic酶标仪测定570nm处吸光度,每个样本重复3次.
1.2.6 耐干燥性的测定
参照Farrow等[23]的方法并稍加改动进行细菌干燥耐受性的评估.将菌株 WT、ΔESA_01393和cpESA_01393培养至对数期,获得悬浮液.为了记录初始细胞数,将每种细菌悬浮液的10μL样品用PBS缓冲液稀释至 100μL,然后连续稀释并接种至 PCA计数培养基上,37℃培养过夜,计数,此时为干燥前的菌落数.吸取200μL的菌液置于96孔板中,然后将 96孔板放在无菌干燥器里,再将干燥器置于温度42℃、相对湿度45%的培养箱恒温恒湿培养,进行耐干燥性实验评估,时间为 6d.为了确定菌体干燥后的死亡率,将 100μL的 PBS置于每个干燥的样品上,在室温下孵育 5min,将菌体吹吸重悬后进行梯度稀释.使用PBS进行连续稀释并接种到PCA计数培养基上,37℃培养过夜,计数.每个样本重复3次.
1.2.7 紫外线照射
根据 Oh等[24]的方法并稍加改动,评估紫外线(UV)照射后菌体的死亡率.菌株 WT、ΔESA_01393和 cpESA_01393培养至 A600=0.6~0.8,然后将菌液分别放置在培养皿里,使用紫外灯进行照射.将分别照射了5min、10min和15min的3种菌株用生理盐水进行梯度稀释,涂布至 PCA固体培养基中,37℃培养箱培养后计数,每个样本重复3次.
1.2.8 丝裂霉素C处理
根据 Oh等[24]的方法并稍加改动,评估丝裂霉素 C处理后菌体的死亡率.菌株 WT、ΔESA_01393和 cpESA_01393培养至 A600=0.6~0.8,然后将 3种菌株的菌液分别置于离心管中,加入 0.1%的丝裂霉素 C,30℃培养箱培养.将分别培养了 10min、20min和30min的3种菌株用生理盐水进行梯度稀释,涂布至 PCA固体培养基中,37℃培养箱培养后计数,每个样本重复3次.
1.3 统计学分析
统计学分析采用 SPSS 16.0统计软件进行Student’s t检验.用 GraphPad Prism 简单单向 t-test检验或多重比较,*和**分别表示组间具有显著性差异(P<0.05)和极显著差异(P<0.01).
2 结果与分析
2.1 基因敲除结果的验证
利用同源重组技术构建了突变株ΔESA_01393及其回补株 cpESA_01393.ΔESA_01393和 cpESA_01393菌株通过 PCR验证和测序验证.分别以 WT菌株和ΔESA_01393菌株DNA为模板进行PCR.如图1(a)所示,以ESA_01393 1-F/ESA_01393 1-R为引物,WT菌株扩增出 315bp的产物,ΔESA_01393菌株没有出现 DNA条带.如图1(b)所示,以ESA_01393 2-F/ESA_01393 2-R为引物,WT菌株扩增出 1710bp的产物,ΔESA_01393菌株扩增出1395bp的产物.如图1(c)所示,以 cpESA_01393F/cpESA_01393R为引物,cpESA_01393菌株扩增的DNA条带为315bp.PCR结果表明,ΔESA_01393和cpESA_01393菌株构建成功,可以进行后续实验.
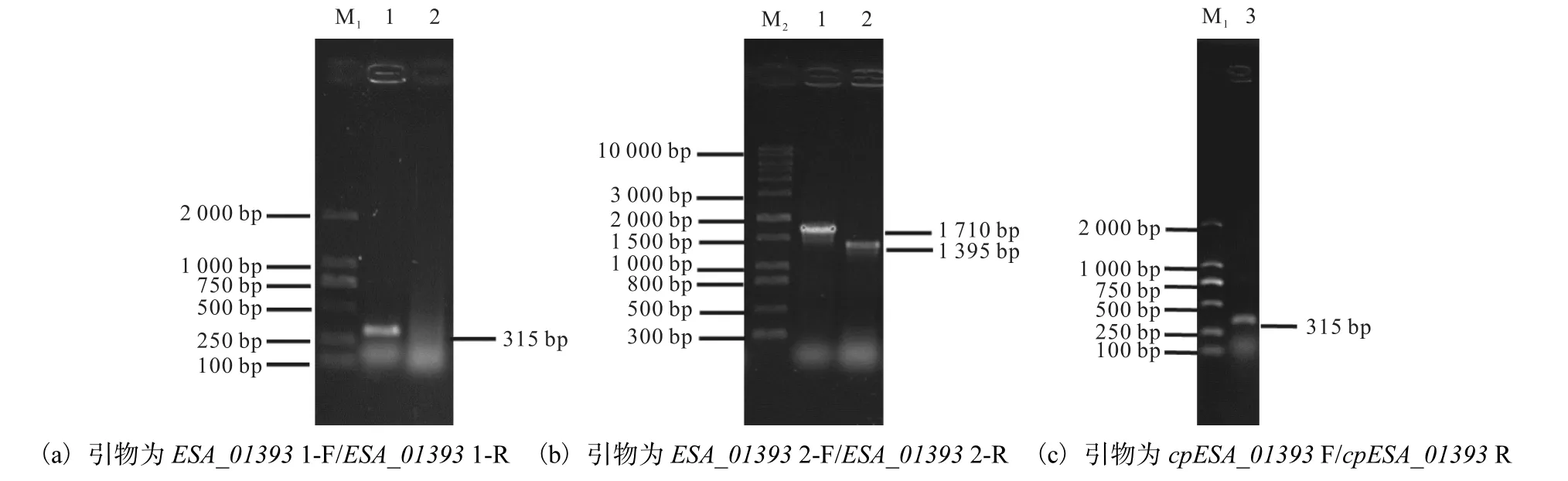

图1 阪崎克罗诺杆菌ESA_01393基因敲除和回补株构建结果的验证Fig.1 Validation of the results of ESA_01393 gene knock-out and cpESA_01393 complementary strain in C.sakazakii
2.2 ESA_01393基因对阪崎克罗诺杆菌ATCCBAA-894生长的影响
比较阪崎克罗诺杆菌野生型、ΔESA_01393突变株及 cpESA_01393回补株的生长曲线,结果如图2所示.与野生型相比,ΔESA_01393突变株显示出相似的生长速率,表明基因缺失后其生长未受影响,同时 cpESA_01393回补株的 A600没有明显变化.这说明 ESA_01393基因的缺失不影响细菌的生长,是细菌的非致死基因,对细胞的生长是非必需的,同时也排除了生长规律不同对后续研究结果的影响.

图2 阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的生长曲线比较Fig.2 Comparision of growth curves for the growth of WT,ΔESA_01393 and cpESA_01393 strains of C.sakazakii
2.3 ESA_01393基因对阪崎克罗诺杆菌ATCC BAA-894运动能力的影响
阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的运动性鉴定结果如图3所示.阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株在 0.3%的琼脂培养基上形成大小相似的运动环;与野生型比较,ΔESA_01393突变株运动能力没有较大变化,回补株运动能力也与野生型相似.运动环直径的测量结果表明,基因 ESA_01393的缺失不影响阪崎克罗诺杆菌的运动能力.

图3 阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的运动性鉴定Fig.3 Mobility identification of WT,ΔESA_01393and cpESA_01393 strain of C.sakazakii
2.4 ESA_01393基因对阪崎克罗诺杆菌ATCC BAA-894生物膜形成能力的影响
生物膜的形成取决于细菌诱导 SOS反应的能力[25].为了验证 ESA_01393基因对阪崎克罗诺杆菌ATCC BAA-894生物膜形成能力的影响,评估了野生型、突变株和回补株的生物膜形成量(图4).与野生型相比,ΔESA_01393突变株所形成的生物膜形成量略有减少,但没有显著性.cpESA_01393回补株显示出与野生型菌株相似的生物膜形成能力.生物膜的形成过程可能受到多种因子的调节,基因ESA_01393可能不是主要的调控因子.
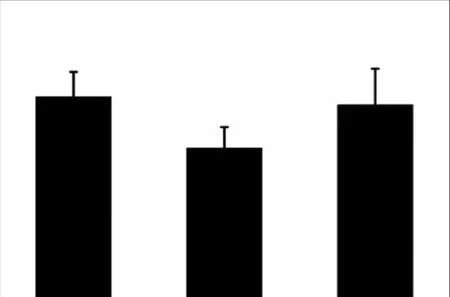
图4 阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的生物膜合成能力比较Fig.4 Comparision of biofilm formation of the WT,ΔESA_01393 and cpESA_01393 strain of C.sakazakii
2.5 ESA_01393基因对阪崎克罗诺杆菌ATCC BAA-894耐干燥性的影响
阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的干燥死亡率如图5所示.与野生型相比,ΔESA_01393突变株干燥死亡率更高,回补株表现出与野生型相似的干燥死亡率.因此,ESA_01393的缺失会降低阪崎克罗诺杆菌耐受干燥环境的能力,该基因能够在一定程度上抵抗干燥环境的压力.
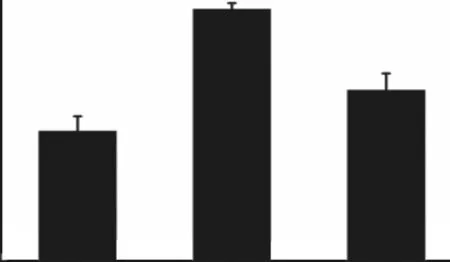
图5 阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的干燥死亡率Fig.5 Drying mortality rate of WT,ΔESA_01393and cpESA_01393 strain of C.sakazakii
阪崎克罗诺杆菌具有一些重要的毒力相关因子或应激生存因子,尤其是在各种不利环境条件下仍能存活的能力,最显著的是耐干燥和耐渗透胁迫的能力[26].细菌不断地暴露在变化和压力的环境中.全部调节系统的协调反应使细菌能够生存并适应各种环境压力[27].多因子和冗余分子机制参与了持久性和耐受性细胞的生成和生存[28-29],其中最著名的耐压机制包括应激反应、SOS反应、抗氧化能力、毒素-抗毒素系统(TA)、群体感应、能量代谢和药物外排泵[29-32].最近有研究[15]表明,虽然 SOS反应最初被认为是调节 DNA损伤修复,但其对细菌的耐受性也有着重要影响.在大肠杆菌中,SOS反应在细胞持续形成的过程中增加了细菌的抗生素耐药性[33-34].在阪崎克罗诺杆菌中,SOS反应参与了生物膜的形成、K+的积累[35]、海藻糖和甜菜碱的合成[36]等过程,因此很可能经由以上途径间接参与该菌的耐干燥调控.然而到目前为止,在阪崎克罗诺杆菌中还没有 SOS反应基因参与耐干燥机制的报道.在大肠杆菌中,yebG基因被鉴定为一种新的SOS反应调控基因[17],而阪崎克罗诺杆菌中yebG基因的同源物ESA_01393基因可能通过SOS反应参与该菌的耐干燥过程.
2.6 ESA_01393基因对阪崎克罗诺杆菌ATCC BAA-894受到UV照射和丝裂霉素C处理的影响
UV照射和丝裂霉素C处理后,比较阪崎克罗诺杆菌野生型、ΔESA_01393突变株及cpESA_01393回补株的死亡率,结果如图6所示.由图6(a)可知:在短时间内(5min和 10min),菌株的 DNA受到损伤时,与野生型相比,ΔESA_01393菌株的死亡率显著升高.由图6(b)可知:当长时间 UV照射(15min)和丝裂霉素C处理(30min)时,菌株的DNA受到严重损伤,菌株 WT、ΔESA_01393和 cpESA_01393显示出相似的菌株死亡率,且死亡率均达到 90%以上.这可能是由于 DNA损伤比较严重,引起细菌细胞大量死亡.结果表明,阪崎克罗诺杆菌ESA_01393基因参与了DNA损伤修复过程.
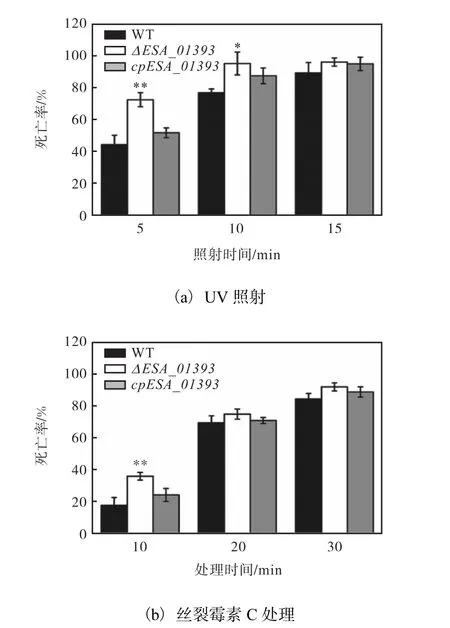
图6 UV照射和丝裂霉素C处理后,野生型、ΔESA_01393突变株及cpESA_01393回补株的死亡率比较Fig.6 Comparison of mortality rates of the WT,ΔESA_01393 and cpESA_01393 strain afterUV irradiation and treated with mitomycin C
细菌的 SOS反应是一种广泛存在的处理 DNA损伤的转录调控机制[37].SOS反应是由含有损伤的DNA 复制过程中单链 DNA 的积累引起的[38].当DNA聚合酶在 DNA损伤处停止而复制解旋酶继续解开DNA时,就会产生单链DNA[39].SOS反应需要多个基因的表达,这些基因在 DNA损伤反应中发挥多种功能,包括切除修复、同源重组、翻译DNA复制和细胞分裂停止[12].DNA损伤修复后恢复正常生理状态,DNA损伤较广泛时可能会导致细胞死亡[40].因此,当发生 DNA损伤时,细菌会激活 SOS反应,以适应 DNA损伤压力[41-42].最常见的引起 DNA损伤的因素包括了紫外线照射和复制抑制剂抗生素丝裂霉素 C暴露[43].研究[41-42]发现紫外线照射后的大肠杆菌通过 SOS反应进行 DNA损伤修复,也有研究[9]表明yebG基因受SOS反应诱导表达,但没有明确该基因的功能.本研究证实了阪崎克罗诺杆菌中yebG基因的同源物ESA_01393基因参与UV照射和丝裂霉素C处理引起的DNA损伤修复过程,并发挥了重要的作用.
3 结 论
采用同源重组的方法敲除了阪崎克罗诺杆菌ESA_01393基因,并对 ESA_01393基因的功能进行研究.比较了阪崎克罗诺杆菌野生型、ΔESA_01393突变株及 cpESA_01393回补株在生长曲线、运动性、生物膜形成、耐干燥性和 DNA损伤修复能力等方面的差异.研究发现 ESA_01393基因对阪崎克罗诺杆菌的生长、运动性和生物膜形成没有明显影响,但是可能作为细菌 SOS反应的一部分,参与了该菌耐干燥和DNA损伤修复过程.本研究为了解阪崎克罗诺杆菌 ESA_01393基因的功能及其在阪崎克罗诺杆菌中的分子机制提供了参考.
