奶牛乳脂代谢关键miRNAs的筛选及鉴定
2023-01-03刘佳敏禹保军冯小芳顾亚玲
刘佳敏,禹保军,母 童,张 迪,冯小芳,张 娟,王 影,温 万,顾亚玲*
(1.宁夏大学农学院,银川 750021;2. 宁夏回族自治区反刍动物分子细胞育种重点实验室,银川 750021;3.宁夏畜牧工作站,银川 750002)
牛乳中富含多种营养物质,蛋白质、脂肪、微量元素以及多种维生素是人体必不可少的营养来源,其中脂肪和蛋白质含量是评价乳品质的重要参考因素,产奶性状亦是奶牛育种的主要目标[1]。近年来在科学育种及饲喂下,奶牛产奶量已大幅提高,但乳脂率增长甚微。甘油三酯、胆固醇和游离脂肪酸是构成乳脂的主要成分,乳脂是乳的重要组成部分,在乳品质评价中起到决定性作用。奶牛生长发育过程中乳脂合成代谢受多因素的影响,其性状表现与遗传因素密切相关,受到多个转录因子的调节[2]。而目前对于奶牛乳脂代谢分子机制的相关研究还十分欠缺,尤其是对高低乳脂荷斯坦奶牛乳脂合成的转录后调控机制方面的研究。因此,利用分子遗传学理论和技术方法研究影响奶牛乳脂性状的调控因子,以期获得调控奶牛乳脂率的关键mRNA和miRNA并验证其生物学功能,通过遗传育种手段提高奶牛的泌乳性能,对于奶牛的育种工作具有重要意义。
非编码RNA(ncRNA)作为基因表达的一种重要调控因子,在农业生产的多个领域被广泛研究。其中,microRNAs(miRNAs)是一类长度约22 nt左右的内源性小非编码RNA,通过碱基互补配对方式与mRNA非编码区结合位点结合,调控靶基因降解或抑制转录后翻译过程,从而调控基因的表达[3]。已有研究阐明,乳脂合成的主要方式包括脂肪酸从头合成和血液中长链脂肪酸的摄取[4]。随着生物学技术的发展,已挖掘出一系列调控乳脂合成代谢的主效基因与转录因子。在调控乳脂代谢方面,催乳素作为泌乳的关键激素可以促进miRNA-23a、miRNA-27b、miRNA-103和miRNA-200a表达,而且miRNA-200a过表达后可以抑制脂滴形成相关基因的表达[5]。microRNA-193a-5p[6]和bta-miR-34b[7]均会降低甘油三酯和不饱和脂肪酸含量,证实在脂质代谢中的关键作用。在调控乳脂合成方面,过表达bta-miR-125a[8]和miR-103[9]能够促进乳脂合成相关基因的表达,增加甘油三酯的积累、脂滴的形成和不饱和脂肪酸的比例。此外,miRNA作为生物性状功能发挥重要的转录因子,可以影响乳腺上皮细胞中的脂质含量[10],也可以通过多种代谢通路的调节促进甘油三酯和胆固醇的合成,是阐明乳脂性状表现的关键特征分子[8,11]。
本研究筛选乳脂率具有极端差异的荷斯坦奶牛,基于转录组测序鉴定高低乳脂荷斯坦奶牛乳腺上皮细胞(bovine mammary epithelial cells, BMECs)中乳脂代谢的关键miRNA。通过miRNA-mRNA关联分析确定互作网络中的核心差异表达miRNA和mRNA,并验证其在高低乳脂荷斯坦奶牛乳腺上皮细胞中的表达量,分析其与乳脂率的相关性,为从转录水平探究宁夏地区荷斯坦奶牛乳脂合成代谢的调控机制提供理论依据。
1 材料与方法
1.1 试验材料
本试验选取宁夏农垦贺兰山奶业茂盛牧场的健康中国荷斯坦奶牛,饲养背景一致(表1),月龄相近,处于泌乳中后期(150~220 d)的第一胎次荷斯坦牛400头。采集每头牛早、中、晚班次的乳样进行DHI测定,筛选体细胞数在10万·mL-1以内,乳脂率具有极端差异的荷斯坦奶牛(表2)[12]各4头。采集牛奶并装入50 mL离心管中,将离心管封口并装入加有温水的保温瓶中,保温瓶温度始终保持在37 ℃,带回实验室进行乳腺上皮细胞的分离培养[12]。

表1 日粮成分及营养成分(以干物质为基础)百分比

表2 荷斯坦牛高低乳脂率和SCC
1.2 方法
1.2.1 乳腺上皮细胞总RNA的提取与质量检测 TRIzol法提取高低乳脂奶牛乳腺上皮细胞的总RNA。根据10 g·L-1琼脂糖凝胶电泳条带判断RNA有无降解和污染(图1),用分光光度计和Qubit®RNA分析试剂盒检测RNA的浓度和纯度,使用RNA Nano 6000检测试剂盒在Bioanalyzer 2100系统上检测RNA的完整性。乳腺上皮细胞总RNA的OD值(OD260nm/OD280nm比值)均在1.8~2.2之间,RIN值(完整指数)均大于8.0,说明RNA纯度和完整性都较好。

M.DNA相对分子质量标准;1~8. 依次为样本H_2190、H_2226、H_2098、H_2137、L_2046、L_2170、L_2034和L_2037M.DL2000 DNA marker; 1-8. The samples H_2190, H_2226, H_2098, H_2137, L_2046, L_2170, L_2034 and L_2037图1 提取的乳腺上皮细胞总RNA的琼脂糖凝胶电泳Fig.1 Agarose gel electrophoresis of total RNA extracted from mammary epithelial cells
1.2.2 文库构建与转录组测序 乳腺上皮细胞总RNA提取成功并检测合格后,以每个样品3 μg总RNA为起始量。由于small RNA的5′端具有完整的磷酸基团,3′端具有羟基,这个特殊结构可以直接加接头,进行反转录cDNA的合成。然后对cDNA片段纯化、PCR扩增、PAGE富集筛选目标片段、切胶回收获得cDNA文库。库检合格后,通过Illumina-Hiseq对不同样品的cDNA文库按照有效浓度及目标下机数据量的需求进行测序,由北京诺禾致源科技股份有限公司完成。
1.2.3 测序数据处理及差异miRNA筛选 测序得到的原始图像数据文件中包含reads信息及其对应测序质量情况。为了保证数据分析的质量,需对Raw data中碱基测序错误率(Qphred =-10lg e)、带接头、低质量的reads进行处理,分析Q20,Q30和GC含量,即最后获得的clean reads质量较高。测序结果质量控制之后,筛选8个样品中small RNA(sRNA)的长度,用bowtie将长度在18~35 nt的sRNA与牛(Bostaurus)参考基因组进行比对。将比对上的reads进一步匹配miRBase中已有的序列,可得到已知miRNA的二级结构和长度等信息。剩下的未匹配的reads借助miRNA前体的发夹结构,使用miREvo和miRdeep2软件预测样品中novel miRNAs。对于已知和novel sRNA中的其他各类型RNA,应当通过技术手段注释并去除。基于高乳脂和低乳脂比较组间有生物学重复,采用DESeq2分析各差异表达组合,得到各样本间基因的表达水平之后,通过相关性分析检验试验样本选择的可靠性及合理性。用TPM归一化处理已知和新预测miRNA的表达量,以P<0.05为标准筛选比较组间差异表达miRNA。
1.2.4 差异表达miRNA靶基因预测及功能富集分析 使用miRanda和RNAhybrid预测miRNA的靶基因,取其交集即为后续分析的候选靶基因集合。采用Goseq方法对差异表达miRNA的靶基因集合进行Gene Ontology分析(http://www.geneontology.org/)。P<0.05的GO条目可认为是差异基因显著富集。基于KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库(http://www.genome.jp/kegg/)中的Pathway确定候选靶基因参与的主要生化代谢途径和信号转导途径,以P<0.05为条件确定差异基因显著富集的KEGG通路。
1.2.5 miRNA-mRNA关联分析 选择高低乳脂组差异表达miRNAs靶向的差异表达mRNAs,使用Cytoscape_v3.7.2软件构建互作网络。然后用pearson相关检验计算差异表达miRNAs及靶mRNAs与乳脂率、乳蛋白率、日产奶量和体细胞数之间的相关性,进而确定调控乳脂率的关键miRNA-mRNA。
1.2.6 差异表达miRNA和mRNA的RT-qPCR验证 随机选择差异表达的9个miRNAs和6个mRNAs对测序数据进行验证。miRNA的引物采用茎环法设计,mRNA的引物用Primer6.0软件设计(表3),以U6和GAPDH分别作为miRNA和mRNA的内参。使用与转录组测序样本一致的RNA,按照反转录试剂盒PrimeScriptTMRT reagent Kit(Perfect Real Time)的说明合成cDNA,以cDNA为模板,使用QuantiNovaTMSYBR®Green试剂盒进行实时荧光定量PCR检测。反应体系:上、下游引物各0.5 μL,cDNA 2 μL,2× QuantiNova SYBR Green PCR Master Mix 10 μL,RNAase Free ddH2O补至20 μL。反应条件:95 ℃预变性2 min;95 ℃变性5 s,退火(退火温度见表3)30 s,40个循环,每个样品3个重复。利用 2-ΔΔCt计算miRNA和mRNA的相对表达量,结果表示为“平均值±标准误”。

表3 引物信息
2 结 果
2.1 测序数据评估及样品间相关性分析
荷斯坦奶牛乳腺上皮细胞样本8个small RNA文库测序获得11 976 914~16 235 680的clean reads,占总raw reads的比例均在97%以上。各个样品的测序错误率(error rate)均为0.01%,Q20和Q30的比例均在97%和91%以上,碱基GC含量基本相等,碱基组成稳定均衡(表4)。高乳脂组和低乳脂组sRNA序列与参考基因组的匹配率均在93%以上。将匹配上的reads与miBase中特定范围序列进行比对分析,可得到8个样本known miRNA共有21 346种、85 152 090个,novel miRNA分别为744、192、316、364、364、245、366和366(表5)。sRNA的序列长度主要集中在21~24 nt,其中23 nt的sRNA序列比率最高(图2)。通过定量分析并用TPM归一化处理,发现8个样本中只有个别miRNA表达量极高,多数表达较为集中(图3A)。为了排除异常样本带来的误差,计算评估样本间pearson相关性:R2>0.9(图3B)。以上结果表明测序数据质量较高,可以用于后续的差异miRNA分析。
2.2 高低乳脂组奶牛乳腺上皮细胞差异表达miRNA筛选
为了探究调节奶牛乳脂率的关键因子,在高乳脂组和低乳脂组之间共鉴定了34个差异表达miRNAs(上调16个,下调18个)(表6,图4A),其中33个是已知 miRNAs,1个新 miRNA。通过层次聚类分析,进一步确定高乳脂组和低乳脂组间差异表达miRNAs的表达水平有显著差异(图4B)。

表4 原始数据质量评估

表5 sRNA与参考基因组比对情况

图2 sRNA片段的长度分布Fig.2 Length distribution of sRNA fragments

A. miRNAs的TPM分布;B. 样品间miRNA表达量相关性A. TPM distribution of miRNAs; B. Correlation of miRNA expression between samples图3 miRNA测序数据评估Fig.3 miRNA sequencing data evaluation
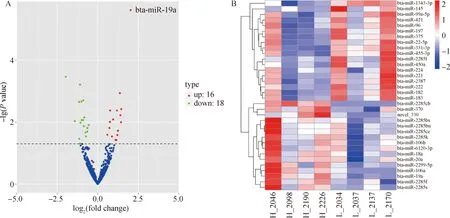
A.差异表达miRNAs火山图; B.差异miRNAs表达谱聚类热图A. Volcano map of differentially expressed miRNAs; B. Heat map of clustering of differential miRNAs expression profiles图4 差异表达miRNAs鉴定及聚类分析Fig.4 Identification and clustering analysis of differentially expressed miRNAs
2.3 差异表达miRNAs的 GO和KEGG富集分析
采用miRanda和RNAhybrid两个软件预测得到已知miRNA和新miRNA的靶基因,取其交集进行GO功能和KEGG通路注释分析。结果显示,靶基因集合显著富集的GO条目有241个。在生物学过程(biological process,BP)中富集的203个条目包括细胞内信号转导、单一生物体过程和细胞死亡等,分子功能(molecular function,MF)中富集的17个条目包括激酶结合、磷酸转移酶活性和肾上腺素受体结合等,细胞组分(cellular component,CC)中富集的21个条目包括细胞质和细胞内等(图5A)。KEGG通路富集结果显示,差异表达miRNAs的靶基因共富集到272条通路,在15条通路显著富集。其中TNF信号传导途径、MAPK信号传导途径、Ras信号传导途径、Rap1信号通路、催产素信号传导途径和GnRH信号传导途径等通路(图5B)与乳脂代谢密切相关。结合GO和KEGG分析可发现,奶牛乳脂代谢可能受到多种机制的调节,包括信号分子传导、能量代谢和激酶活性调节。
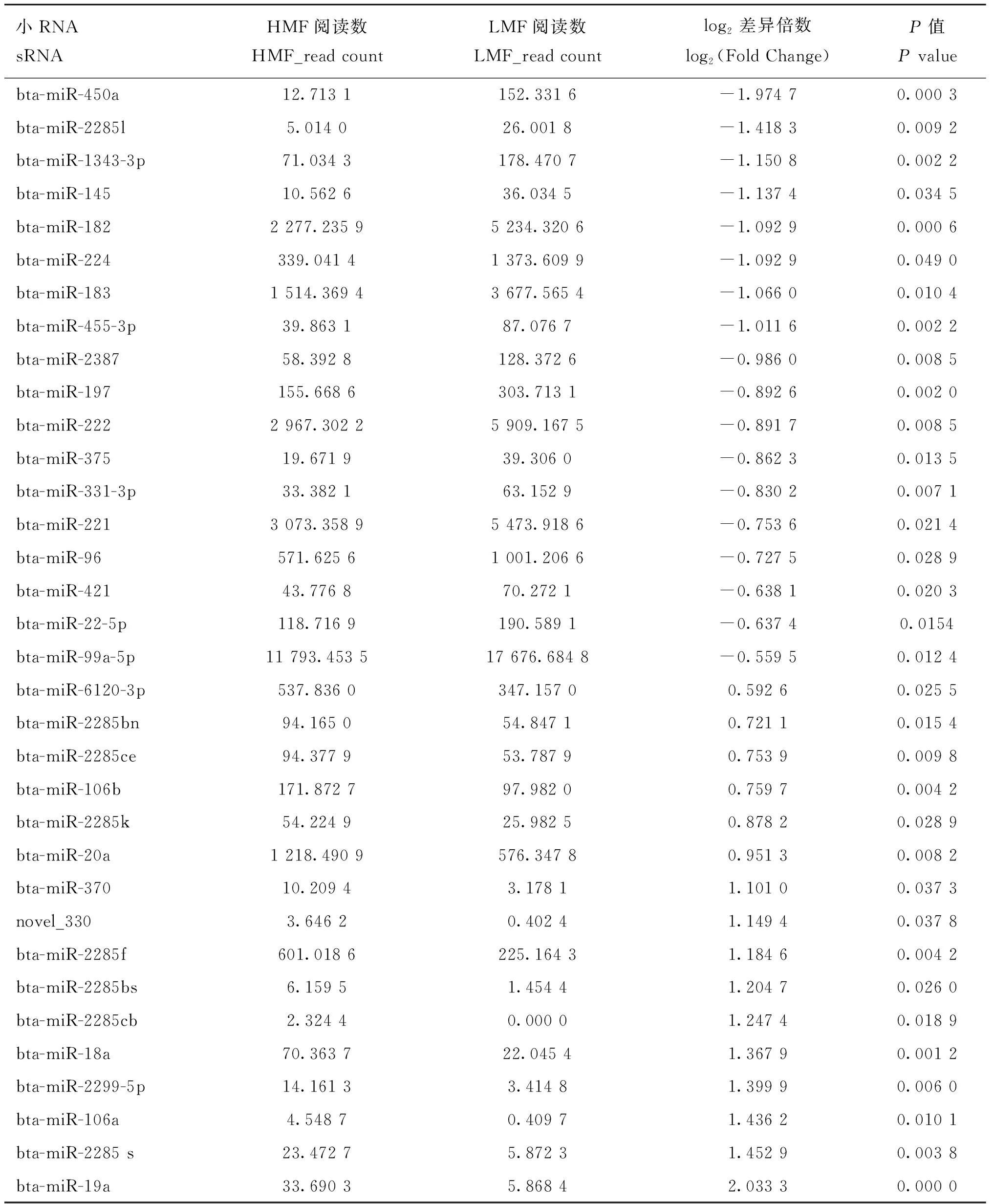
表6 差异表达miRNAs
2.4 miRNA-mRNA关联分析
为了确定与乳脂代谢相关的关键miRNA-mRNA,构建差异表达miRNA与差异表达靶mRNA的互作网络。结果共鉴定出16个miRNA-mRNA互作对(9个miRNAs和9个mRNAs),其中有8个互作对具有负调控关系(图6A)。对9个差异靶标基因进行功能分析发现,主要富集于蛋白激酶A结合、磷脂酰肌醇磷酸酯酶活性、mRNA分解代谢和胰岛素分泌的正向调节等多个生物学功能(图6B)。只有一个基因MTM1显著富集到磷酸肌醇的代谢和磷脂酰肌醇信号系统,与甘油三酯和乳脂合成密切相关。此外,将差异表达miRNA及靶标mRNA与乳脂率进行相关性分析,发现miR-1343-3p和MTM1与乳脂率显著负相关,miR-370、miR-2285cb和SRRM2与乳脂率显著正相关(图6C)。综上,可推测miR-370-MTM1、miR-1343-3p-DIS3L2、miR-2285cb-XLOC-122799和miR-2387-SRRM2是调控乳脂代谢的关键因子。

A.差异表达miRNAs与差异靶mRNAs互作网络;B.差异靶mRNAs功能分析; C.差异miRNA-mRNA与乳成分之间的相关性A. Interaction network between differentially expressed miRNAs and differential target mRNAs; B. Functional analysis of differential target mRNAs; C. Correlation between differential miRNA-mRNA and milk components图6 miRNA-mRNA关联分析Fig.6 miRNA-mRNA association analysis
2.5 差异miRNAs与mRNAs的实时荧光定量PCR验证
选择组间差异表达的9个miRNAs和6个mRNAs验证测序结果的可靠性。结果如图6所示,miR-20a、miR-18a、miR-2285f、miR-106b和miR-19a在高乳脂组乳腺上皮细胞中的表达高于低乳脂组,表现为上调,miR-445-3p、miR-1343-3p、miR-224和miR-182表现为下调。DIS3L2、AKAP13、MTM1、ANO1、CBY1和AKIP1在高乳脂组的乳腺上皮细胞中的表达低于低乳脂组,均表现为下调。RT-qPCR结果与转录组测序结果相对表达量存在差异,但其调控方向和表达模式均一致(图7、图8),说明转录组测序结果可靠,可用于后续功能试验研究。
3 讨 论
乳腺上皮细胞是主要的产乳细胞,是研究乳脂的重要材料[13-15]。乳是由乳腺上皮细胞合成并分泌的。乳脂是乳中的重要成分之一,其合成与代谢受到严格的程序性调控,在生产上受到饲养管理、营养、疾病以及遗传等多个因素的影响[16]。miRNA作为表观遗传调控研究的热点,在细胞生长[17]、增殖[18]、分化[19]和凋亡[18]等多种生物学过程中广泛研究。在产乳性状方面,miRNA能够调控乳腺的发育[20]、乳腺细胞的增殖和分化、乳脂代谢[21]及泌乳过程[22]。因此,本研究选择乳脂率极端差异的荷斯坦奶牛,从牛奶中分离培养原代乳腺上皮细胞并进行转录组测序,以miRNA与mRNA的靶向关系为基础进行分析,进而确定宁夏地区荷斯坦奶牛乳脂的遗传特性及机理。
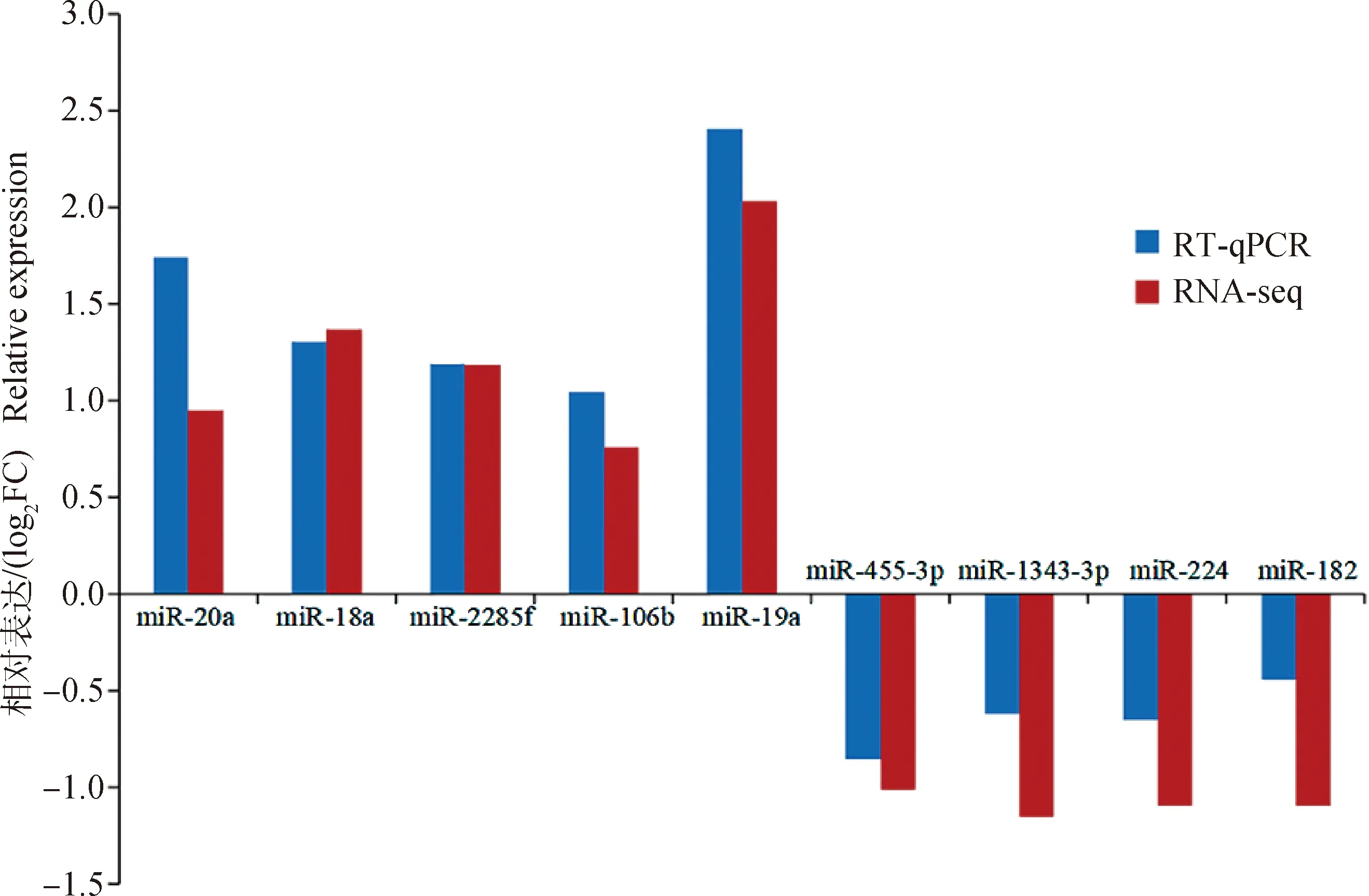
图7 差异表达miRNAs的RT-qPCR和RNA-seq定量分析比较Fig.7 Comparison of quantitative analysis of differentially expressed miRNAs by RT-qPCR and RNA-seq

图8 差异表达mRNAs的RT-qPCR和RNA-seq定量分析比较Fig.8 Comparison of quantitative analysis of differentially expressed mRNAs by RT-qPCR and RNA-seq
miRNA作为重要的小非编码调控因子,可以靶向靶基因在转录和翻译水平调节其表达,从而影响个体性状表现。miR-370作为新型的脂肪生成调节剂,在脂质代谢中起重要作用。研究发现,过表达miR-370可以显著抑制脂肪的形成和分化,导致与脂肪形成相关的基因PPARγ和aP2表达下调,进而使甘油三酯的积累量降低[23]。miR-370-3p通过靶向Mknk1对脂肪生成和脂肪酸代谢起着重要调控作用[24]。miR-197与脂肪也具有调控关系,miR-197-3p会使甘油三酯和胆固醇的水平下降,但lncRNA HCG18-miR-197-3p轴会增加脂肪积累[25]。根据miRNA-mRNA互作关系,确定可能导致相关基因表达变化的功能性miRNA[26]。本研究通过miRNA-mRNA互作分析发现,bta-miR-370、bta-miR-197与ANO1、MTM1、XLOC-122799有调控关系,可视化网络图中以上基因与bta-miR-331-3p、bta-miR-2387、bta-miR-1343-3p、bta-miR-2285 s和bta-miR-2285cb互作。因此,推测以上miRNAs可能通过靶向靶标基因ANO1、MTM1、XLOC-122799、DIS3L2、SRRM2、NDRG1、CBY1和AKIP1调控乳脂代谢过程。同时,结合miRNA-mRNA与乳成分相关性分析,发现bta-miR-1343-3p、bta-miR-2285cb、bta-miR-370、MTM1、SRRM2与乳脂率显著相关。因此,可推测miR-370-MTM1、miR-1343-3p-DIS3L2、miR-2285cb- XLOC-122799和miR-2387-SRRM2是调控乳脂代谢的关键因子。
mRNA作为重要的调控因子直接或间接的参与乳腺的生长发育[27]、泌乳的启动、乳脂的合成[28-29]。磷酸肌醇是脂质信号分子,其在脂质转运中具有重要作用[30]。本研究通过靶基因富集筛选出两条关键通路磷酸肌醇代谢和磷脂酰肌醇信号通路,有研究证实,磷酸肌醇代谢和磷脂酰肌醇信号通路可调节乳脂合成中的关键酶,参与脂类生物合成过程[31]。通过功能注释筛选出差异表达显著的基因共6个,A激酶锚定蛋白13(A-kinase anchor protein 13,AKAP13)、A 激酶相互作用蛋白1(A-kinase-interacting protein 1 isoform X1,AKIP1)、Anoctamin 1(ANO1)、chibby同源蛋白1(protein chibby homolog 1,CBY1)、DIS3 样核酸外切酶 2(DIS3-like exonuclease 2,DIS3L2)和肌管蛋白(myotubularin,MTM1)在高乳脂和低乳脂荷斯坦奶牛乳腺上皮细胞中表达量较高。通过STRING数据库分析,MTM1参与磷酸肌醇代谢和磷脂酰肌醇信号系统等多个通路,具有磷脂酰肌醇代谢过程、磷酸酶活性和脂质修饰等功能;CBY1通过与CTNNB1/β-catenin结合并通过与 TCF/LEF 转录因子的竞争抑制β-catenin介导的转录激活来抑制 Wnt/Wingless 通路,有促进脂肪细胞分化的能力。AKAP13参与MAPK、cAMP、Rap1和Wnt信号通路等多个经典通路;ANO1参与MAPK、cAMP、Rap1、PI3K-Akt 和AMPK信号通路等多个经典通路。有研究证明,PI3K-Akt、MAPK、TNF和Wnt等都是与脂代谢相关的通路[32-34],在奶牛的乳脂代谢与合成中起重要调控作用。以上结果进一步说明,MTM1、CBY1、AKAP13和ANO1可能参与调节乳脂合成代谢过程,具体调控机制有待进一步研究。
4 结 论
本研究通过RNA-seq技术描述了中国荷斯坦牛奶牛乳腺上皮细胞中的miRNA表达谱,从高乳脂和低乳脂组筛选出34个差异表达miRNAs,其中16个上调,18个下调。通过功能分析确定MAPK信号通路、Ras信号通路、Rap1信号通路、催产素信号通路和磷酸肌醇代谢等通路是乳脂代谢所必需的。通过miRNA-mRNA联合以及乳脂率相关性分析,鉴定到miR-370-MTM1、miR-1343-3p-DIS3L2、miR-2285cb- XLOC-122799和miR-2387-SRRM2可能直接或间接参与乳脂合成代谢过程,其可作为奶牛泌乳性状分子调控功能研究的候选基因。
