奶牛蹄皮炎关联微生物群落结构及多样性分析
2022-12-27李镔罡肖海侠艾柯代吐鲁洪杨学云马万鹏刘璐瑶苏战强
李镔罡 ,肖海侠 ,艾柯代·吐鲁洪 ,杨学云 ,马万鹏 ,刘璐瑶 ,苏战强 ,3*
(1.新疆农业大学动物医学学院,乌鲁木齐 830052;2. 新疆畜牧科学院兽医研究所,乌鲁木齐 830011;3. 新疆草食动物新药研究与创制重点实验室,乌鲁木齐 830052)
奶牛蹄病是各种蹄病的总称,发生在奶牛的四肢,包括蹄皮炎、蹄叶炎、白线病和蹄底溃疡等。牛蹄病是规模化牛场最常见且危害较严重的疾病之一,除了导致奶牛跛行,蹄病还会对牛只的生产、繁殖及行为造成影响,同时还会降低动物福利,继而直接或间接的造成经济损失,有调查表明每一头患蹄病的奶牛在整个泌乳期可造成0.114 ~0.320 万元的损失[1'2],有些牛场因蹄病造成的经济损失甚至超过其养殖成本的一半。奶牛蹄皮炎是指蹄后跟系部皮肤出现溃烂与炎症并在病灶周围形成白色边缘伴有长毛发状或其周边附着较厚的多毛性疣。 奶牛蹄皮炎是一种细菌性疾病,当牛患蹄皮炎时,患处表现为有腐败气味,表皮增厚和肿胀,高发于牛蹄系部于指间(趾),蹄底皮肤过度角质化,如果未能及时发现该病,患处则会破溃脱落出血,导致创面暴露于环境中,继发深层感染,且会有分泌物渗出,四肢的指(趾)间和系部均可发病,有时形成结痂[3]。发病奶牛大多体温正常,跛行程度因严重性而表现不一。奶牛蹄皮炎发病机理十分复杂,引起奶牛蹄皮炎的因素多种多样,其中包括管理因素[4'5]、生理因素[6]、行为因素[7]、遗传因素[8,9]等。
在前期的调查中新疆乌鲁木齐周边地区的奶牛蹄病以蹄皮炎为主,占蹄病的88.24%,传统的微生物分离鉴定方法存在一些局限性, 有许多的细菌在样本中含量相对较少或难以制造某些细菌的生长环境,导致无法培养或生长发育不良[10]。 如果不能将全部细菌分离鉴定,便不能全面的反应样本中的菌群分布情况,因此需借助高通量测序技术对奶牛蹄部腐肉样本中的菌群进行多样性的分析,为以后的研究提供数据参考。 本研究采集奶牛蹄皮炎样本和健康奶牛蹄样本,通过Ⅰllumina NovaSeq 二代测序技术研究其微生物的菌落结构,分析奶牛蹄皮炎样本中的菌落组成,为奶牛蹄皮炎的防控提供更全面的理论依据。
1 材料与方法
1.1 主要仪器与试剂
NanoDrop ND-1000 分光光度计,美国Thermo 公司;高速冷冻离心机,德国赛多利斯公司;PCR 仪,美国Thermo 公司。
1.2 方法
1.2.1 样品采集与处理
从新疆昌吉地区某2 个规模化奶牛场(A,B)采集健康奶牛蹄样本和奶牛蹄皮炎样本各10 份,其中5份为 A 场奶牛蹄皮炎样本(A_FR1、A_FR2、A_FR3、A_FR4、A_FR5)、5 份为 A 场健康牛蹄部样本(A_JK1、A_JK2、A_JK3、A_JK4、A_JK5)、5 份为 B 场蹄部腐肉样本(B_FR1、B_FR2、B_FR3、B_FR4、B_FR5)、5 份为B 场健康牛蹄部样本(B_JK1、B_JK2、B_JK3、B_JK4、B_JK5)。采样前使用生理盐水将患蹄表面粪污冲洗干净,使用消毒后的修蹄刀暴露患处,用消毒后的剪刀采集蹄皮炎腐肉并使用灭菌棉签蘸取健康蹄部拭子,分别放于高压处理后的5 mLEP 管低温保存,将样品标记后迅速送回实验室进行检测。
1.2.2 16S rRNA 基因 V3-4 区的 PCR 扩增
使用离心机将样品1 200 rpm/min 离心3 min,对样本使用基因组DNA 试剂盒提取基因组模板,提取步骤参照说明书。 使用分光光度计与1.2%琼脂糖凝胶电泳对提取的DNA 模板进行质量检测, 并调整DNA 浓度。 DNA 质量达标后使用引物 338F-806R (338F:5'-ACTCCTACGGGAGGCAGCA-3''806R:5'-GGACTACHVGGGTWTCTAAT-3')对细菌 16S rRNA 基因 V3-V4 区的 PCR 扩增[11]。 反应程序:98 °C,2 min +(98 °C,15 s + 55 °C,30 s + 72 °C,30 s)× 25 + 72 °C,5 min。 将合格的 PCR 产物进行回收,并使用Quant-iT PicoGreen dsDNA Assay Kit 分析试剂盒对其进行定量检测, 将PCR 产物委托上海派森诺生物技术有限公司的Illlumina NovaSeq6000 测序平台进行测序。
1.2.3 生物信息学分析
使用Qiime (1.9.1) 软件对测序获得的原始数据进行拼接、 过滤与去嵌合体, 得到有效序列。 调用UCLUST 将有效序列按97%的序列相似度归类和可操作分类单元划分,并在结果中筛选丰度较高的可操作分类单元,选为代表序列。 使用Greengenes 数据库(Release 13.8)将可操作分类单元进行查询比对,将其进行物种注释,并统计样本在不同分类水平的丰度情况。 对可操作分类单元的序列数进行稀疏曲线分析 。 使 用 Qiime 软 件 计 算 各 样 本 的 Chao1、Faith-pd、Good coverage、Shannon、Simpson、Pielou-e 和Observed species 多样性指数,并根据Bray-Curtis 距离算法使用R 语言得到Beta 多样性距离矩阵。 使用UPGMA 的加权平均法对样本中属水平丰度靠前的菌群进行聚类分析,并对其绘制热图。
2 结果与分析
2.1 Alpha 多样性分析
本研究通过计算 Chao1、Faith-pd、Good coverage、Shannon、Simpson、Pielou-e 和 Observed species 多样性指数对A 和B 两牛场中奶牛蹄皮炎样本和健康奶牛蹄样本进行Alpha 多样性分析, 结果如图1 所示,A 牛场奶牛蹄皮炎样本于健康蹄样本和B 牛场奶牛蹄皮炎样本于健康蹄样本各个指数均值差异性均显著(P < 0.05),且各个样本的Coverage 值范围为0.9430 ~0.9966,表明测序基本覆盖到了全部的OUT 数据,可反映样本的真实情况。
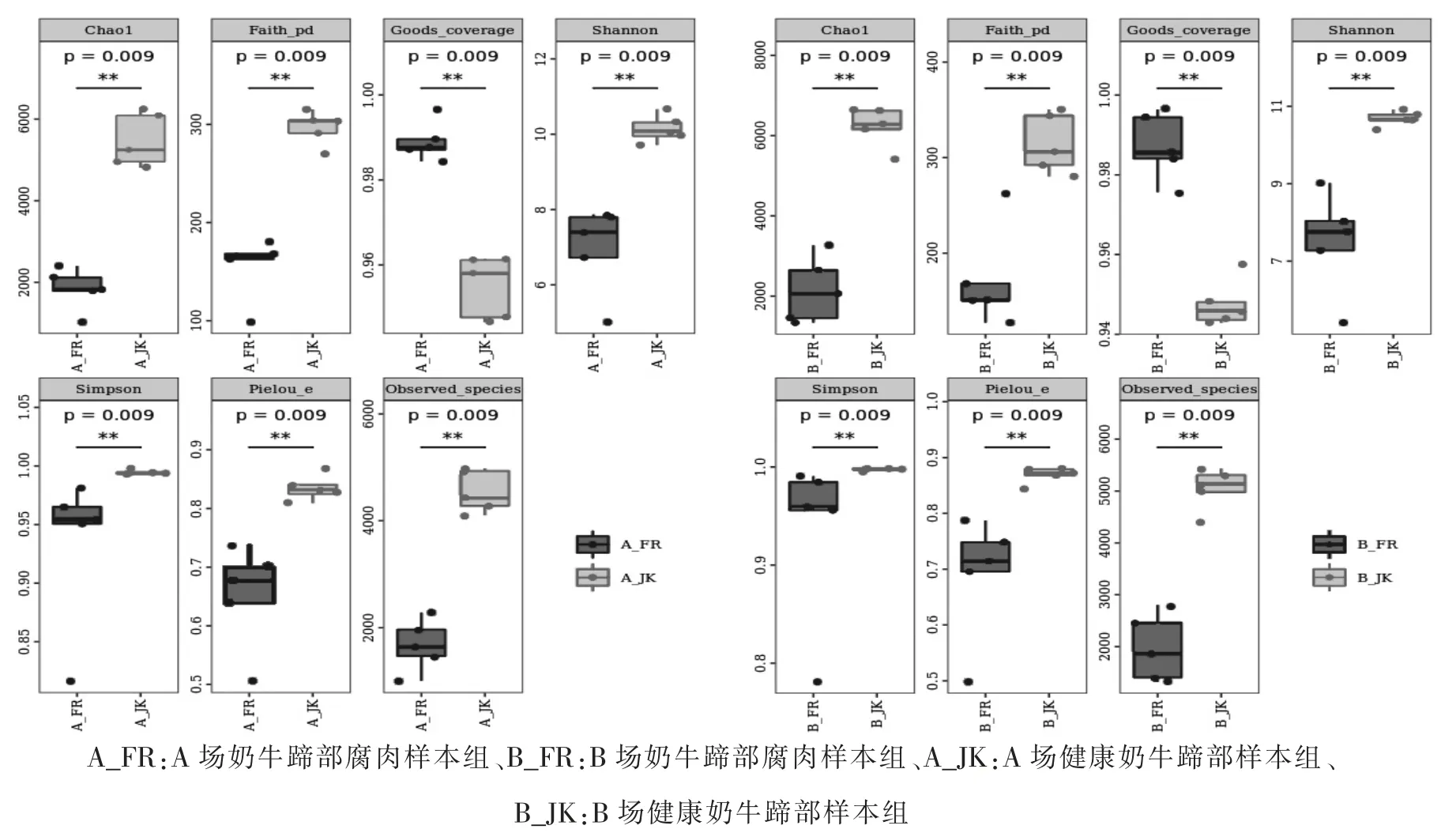
图1 A_FR、B_FR、A_JK 和 B_JK 组样本群落 Alpha 多样性指数
2.2 Beta 多样性分析
通过主坐标分析(Principal coordinate analysis,PCoA)发现A 牛场蹄皮炎样本和A 牛场健康奶牛蹄样本之间及B 牛场蹄皮炎样本和B 牛场健康奶牛蹄样本之间聚集在一起的微生物群落结构差异度较大,见图2。 PERMANOVA 差异检验分析表明A 牛场蹄皮炎样本组和A 牛场健康奶牛蹄样本组、B 牛场蹄皮炎样本组和B 牛场健康奶牛蹄样本组、A 牛场蹄皮炎样本组和B 牛场蹄皮炎样本组菌群结构差异显著(P<0.05);A 牛场健康奶牛蹄样本组和B 牛场健康奶牛蹄样本组菌群结构差异不显著(P>0.05)。
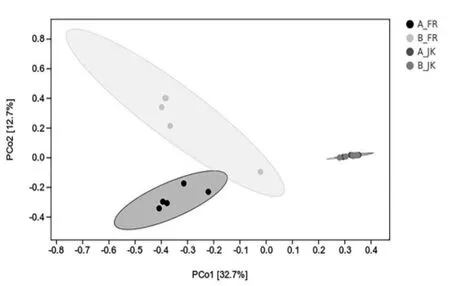
图2 A 牛场奶牛蹄皮炎样本组、B 牛场奶牛蹄皮炎样本组、A 牛场健康奶牛蹄样本组和B 牛场健康奶牛蹄样本组菌群的PCoA
2.3 在门分类水平上的细菌组成
利用QIIME 软件分析犊牛肺炎样本和健康样本在门分类水平上丰度前10 个物种。 结果表明,在奶牛蹄皮炎腐肉样本中,A 场的优势菌分别为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和梭杆菌门(Fusobacteria) 等;B 场中的优势菌分别为厚壁菌门 (Firmicutes)、 拟杆菌门 (Bacteroidetes)、 变形菌门(Proteobacteria)、螺旋体(Spirochaetes)等。 A 场和 B 场相比,梭杆菌门(Fusobacteria)在 A 场的样本中被检出,而螺旋体(Spirochaetes)仅在A 场的A_FR1、A_FR5 中检出但丰度最高仅为1.74%。 B_FR1 中拟杆菌门的丰度为75.03%,在样品中丰度值最高。 健康牛蹄部样本中优势菌群为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria);厚壁菌门(Firmicutes)在健康牛蹄部样本中丰度均高于64.10%,其次为拟杆菌门(Bacteroidetes),且丰度均高于12.90%。 蹄部腐肉样本于健康样本相比,A 场的梭杆菌门(Fusobacteria)于B 场的螺旋体(Spirochaetes)丰度均高于健康样本,见图3。
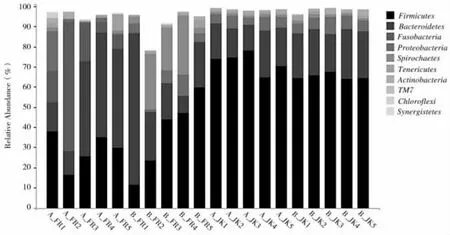
图3 样本中细菌门分类水平的比较
2.4 在属分类水平上的细菌组成
利用QIIME 软件分析奶牛蹄部腐肉样本和健康牛蹄部样本在属分类水平上丰度较高的前10 个物种。 结果如图4, 在A 牛场奶牛蹄部腐肉样本中优势菌群为梭杆菌属 (Fusobacterium)、 卟啉单胞菌属(Proteobacteria); 在B 牛场中奶牛蹄部腐肉样本中优势菌群为密螺旋体 (Treponema)、 卟啉单胞菌属(Proteobacteria);A 场和B 场奶牛蹄部腐肉样本中的主要菌群分别为梭杆菌属(Fusobacterium)、密螺旋体(Treponema)、卟啉单胞菌属(Porphyromonas)等。 在 A 场样本中,A_FR1、A_FR2、A_FR3 中丰度最高的菌群为梭杆菌属(Fusobacterium),分别占总细菌数的 15.04%、58.60%、16.93%,在 A_FR4、A_FR5 中,丰度最高的菌群为啉单胞菌属 (Proteobacteria), 分别占8.43%、9.11%。 不同于A 场,B 场中大量存在梭杆菌属(Fusobacterium),而 A 场中大量存在密螺旋体(Treponema)。
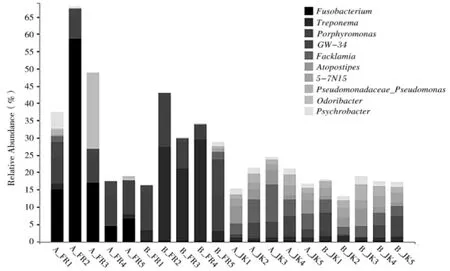
图4 细菌属分类水平的比较
2.5 物种组成热图分析
使用平均丰度前10 位的属的丰度数据绘制热图。 结果如图5 所示,热图横、纵坐标分别为不同样本编号和丰度前10 的菌属名称。 图中色块颜色越深,代表样本中该属细菌丰度高于其他样本;色块颜色越浅,代表样本中该属细菌的丰度低于其他样本。 结果显示在属水平上A 和B 两牛场奶牛蹄皮炎样本与健康牛蹄样本前10 位物种的丰度差异较大。 梭杆菌属(Fusobacterium)、密螺旋体(Treponema)与卟啉单胞菌属 (Porphyromonas) 等在奶牛蹄皮炎样本中丰度较高, 而费克蓝姆菌属 (Facklamia)、 异位线虫(Atopostipes)与厌氧球菌属(Anaerococcus)丰度较高。
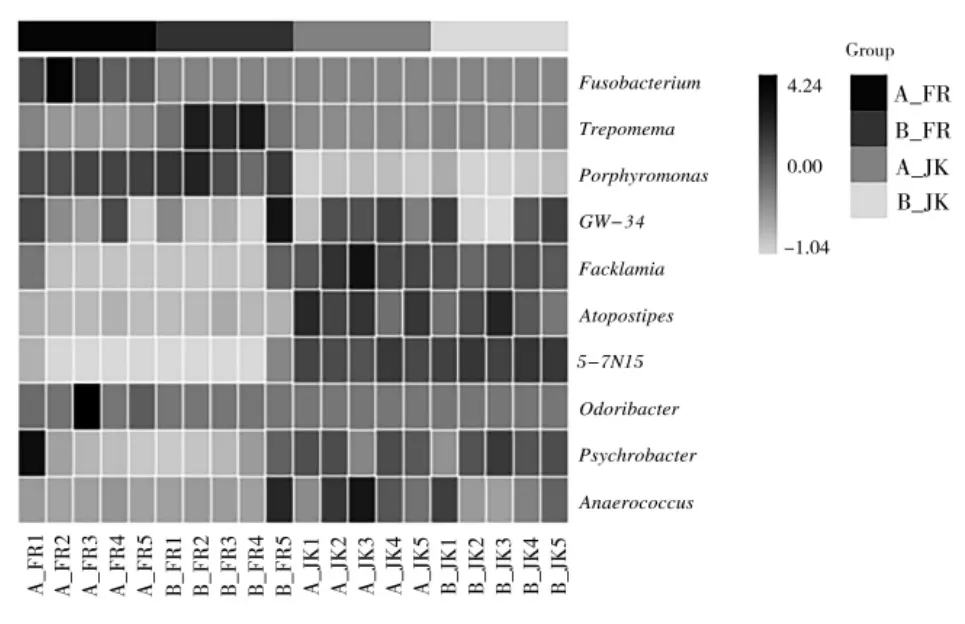
图5 物种聚类的属水平物种组成热图
3 讨 论
奶牛蹄皮炎是一种细菌病[12],可导致奶牛的跛行,引起炎症反应及皮肤损伤,使得奶牛在行走或站立时疼痛,进而使得奶牛采食量下降和泌乳量降低等情况出现,造成经济损失。 Burger 等人研究表明,奶牛27%的经济亏损都是由蹄病造成的[13]。另有调查显示,每头患蹄皮炎的奶牛损失的总成本(治疗成本、产奶量下降成本)为1990 元左右[14]。 现如今对奶牛蹄皮炎的病因及传播机制尚不明确,但随着基因测序等实验室技术的发展,奶牛蹄皮炎的相关病原菌和疾病传播机制也在慢慢被揭开。 曾有多位学者在本病样本中分离出多种细菌,尤其是密螺旋体[15]。 此外,有报道称本病相关细菌难以使用培养基培养,且难以建立感染模型[16~18]。 因此,本次研究利用高通量测序技术对奶牛蹄皮炎样本和健康奶牛蹄样本进行检测,可对“无法培养”的微生物进行测序,检测奶牛蹄病样本中微生物群落结构的多样性。资料显示,我国使用高通量测序技术对奶牛蹄病的微生物多样性检测的案例很少。 在本试验中,提供了我国新疆地区牛场中蹄皮炎腐肉样本中的数据信息,为将来对奶牛蹄病的防控措施的制定及治疗时的药物选择提供了参考。 本次试验结果表明,患蹄病的奶牛蹄部腐肉处菌群多样性在不同细菌分类水平均上升,蹄病腐肉中的菌群结构更复杂。 王德海对齐齐哈尔市某规模化牛场中奶牛蹄皮炎病原微生物多样性进行检测,结果发现样本中优势菌门为厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)等,但未发现螺旋体(Spirochaetes)的存在,这一结果可能是因为不同地区奶牛蹄皮炎的流行情况不同[19]。 本次实验结果发现,奶牛蹄皮炎中的菌门分布情况与大多数哺乳动物肠道内优势菌门相似[20'21],因此,部分奶牛蹄病的一些致病菌可能来自于奶牛肠道中的细菌。 如果及时清理牛场圈舍中的粪污,可能会减少牛蹄与致病菌的接触时间,减少发病几率。 但与肠道菌群不同的是,拟杆菌门(Bacteroidetes)的比例会上升,厚壁菌门(Firmicutes)与拟杆菌门(Bacteroidetes)的比例失调可能是引起蹄病的原因也可能是发生蹄病后的症状,还需要进一步研究。
密螺旋体一直被认为是导致奶牛患蹄皮炎的主要病原微生物, 并且在受感染的奶牛所在牛场的奶牛粪便及环境样本中检测到密螺旋体[15'22]。 本次实验中,在A、B 牛场均检出螺旋体(Spirochaetes),试验结果与之相符。 A 牛场与B 牛场采集的奶牛蹄皮炎样本中菌群分布有所差异,A 场和B 场相比,梭杆菌门(Fusobacteria)仅在A 场的样本中被检出,而螺旋体(Spirochaetes)仅在 A 场的 A_FR1、A_FR5中检出但丰度最高仅为1.74%,可能是由于牛场环境不同,导致不同牛场中奶牛蹄部患处的细菌分布也存在差异。 A、B 两牛场健康牛蹄部样本的细菌分布与丰度相似, 该结果表明两牛场中环境均分布情况相似。
奶牛蹄皮炎的发生与多种细菌有关。 在属分类水平上,A 牛场中的梭杆菌属(Fusobacterium)丰度上升,坏死梭杆菌属于梭杆菌属(Fusobacterium),该菌侵染力不强,但可以轻易的侵入受伤的皮肤组织,会使组织出现浓肿和腐臭气味,这与先前在牛场采样时患病奶牛症状相符,因此梭杆菌属(Fusobacterium)丰度值的上升可能是部分蹄病的症状之一;B 牛场中的密螺旋体(Treponema)丰度值高于该场健康蹄部样本,最高可达29.43%,有学者认为,奶牛蹄皮炎是由密螺旋体导致的[15],密螺旋体的丰度值的升高也与其相互验证。当奶牛发生蹄皮炎时,菌群的多样性降低,1 种或多种细菌的丰度增高。但在研究中发现,不同奶牛场中丰度增高的菌群存在差异,但厚壁菌属起主导作用,可能与牛场环境有关。
4 结 论
奶牛患蹄皮炎后,患处腐肉菌群多样性及丰度明显升高。 不同牛场蹄皮炎患处样本中厚壁菌门丰度较高,但不同牛场中还有其它不同菌门上升。当奶牛患蹄皮炎后患处腐肉菌群失调,菌群结构分布以及丰度变化与奶牛蹄皮炎的发生存在密切关联。
