大豆盐胁迫响应扩展蛋白基因的筛选及GmEXPA17a的克隆和表达分析
2022-12-20王玉斌张彦威刘薇李伟王彩洁戴海英徐冉张礼凤
王玉斌,张彦威,刘薇,李伟,王彩洁,戴海英,徐冉,张礼凤
(山东省农业科学院作物研究所/山东省特色作物工程实验室,山东 济南 250100)
植物在生长发育过程中经常遭遇干旱、冷害和土壤盐渍化等非生物胁迫的危害,其中盐胁迫是植物受到的主要非生物胁迫之一[1]。由于长期以来人类不合理灌溉加上气候条件的剧烈变化,我国耕地盐碱化程度不断恶化,严重威胁我国粮食安全和农业可持续发展。大豆是我国重要的粮油饲兼用作物,在保障国家粮食安全中具有重要的地位。虽然大豆属于中度耐盐作物,但盐胁迫会严重影响大豆生长,降低大豆产量与品质[2]。因此,通过育种改良培育抗盐大豆品种,实现盐碱地大豆产量与品质的协同提高,对缓解目前我国面临的粮食安全具有重大意义。
扩展蛋白(expansin)是一类参与调控细胞壁扩张和细胞生长的活性蛋白,广泛存在植物体内,参与调控众多植物生长发育与生理过程,包括种子萌发、根系发育、茎和叶的生长、气孔移动和果实软化等[3]。而且扩展蛋白通过调控细胞壁松驰在植物适应环境胁迫过程中起着关键作用[4]。通过系统进化树分析一般可以将扩展蛋白分为四个亚家族,分别为α-expansin(EXPA)、β-expansin(EXPB)、expansin-like A(EXLA)和expansinlike B(EXLB)[5]。其中EXPA 和EXPB 家族蛋白已被证明会引起细胞壁的松弛[6]。扩展蛋白一般由250~275 个氨基酸组成,典型的扩展蛋白通常包含N端结构域、C 端结构域和信号肽结构域[3]。
越来越多的证据表明扩展蛋白基因参与不同植物对盐胁迫的响应[7]。超表达AtEXP3和AtEXPB1可增加拟南芥对盐胁迫的敏感性[8];月季RhEXPA4在拟南芥中过量表达可以通过促进侧根发育提高其抗盐性[9];玉米中已报道ZmEXPA1、ZmEXPA3、ZmEXPA5、ZmEXPB1和ZmEXPB2通过调控细胞扩展减少盐胁迫对玉米造成的离子毒害[10];在烟草中超表达小麦EXPA2基因可以通过降低Na+/K+含量,提高抗氧化能力从而增加烟草抗盐性[11];OsEXPA7通过协调Na+转运、ROS 清除和细胞壁松弛,提高水稻抗盐能力[12]。大豆基因组中共有75 个扩展蛋白基因家族成员[13],第一个被发现的扩展蛋白基因是在根中表达的α-expansin 家族基因GmEXP1,其过表达可促进烟草细胞和根的生长[14]。β-expansin家族的GmEXPB2受磷胁迫诱导,主要在根中表达,其过量表达通过提高根毛长度影响根系磷的吸收[15,16]。但目前关于大豆扩展蛋白基因参与盐胁迫响应还未见报道。
本研究利用实验室前期筛选出的耐盐大豆品种齐黄34[17],对不同时间点盐胁迫处理的根系进行转录组测序分析,鉴定出7 个持续响应盐胁迫的扩展蛋白基因,并对响应盐胁迫较强的GmEXPA17a进行了克隆、生物信息学分析、组织表达分析、盐胁迫和激素应答分析,为进一步研究扩展蛋白基因在大豆盐胁迫响应中的功能奠定了基础。
1 材料与方法
1.1 试验材料与试剂
大豆材料为耐盐品种齐黄34。使用艾德莱RNA 提取试剂盒提取齐黄34 根部RNA,pLB 零背景快速克隆试剂盒和胶回收试剂盒以及菌液鉴定用到的2×TaqPCR MasterMix 购自天根生化科技(北京)有限公司,DH5α 感受态细胞购于济南雨硕生物科技有限公司,反转录试剂盒、实时荧光定量PCR 试剂和基因克隆用到的高保真酶均购自南京诺唯赞生物科技有限公司。
1.2 试验方法
1.2.1 材料培养及处理 大豆种子经10%次氯酸钠(V/V)消毒20 min 后,用蒸馏水清洗5 遍。将消毒好的种子均匀放置在湿润的滤纸上催芽。3 d 后挑选生长一致的幼苗转移至底部有网眼的塑料筐中,塑料筐放置于盛有改良1/2Hoagland营养液的塑料盆中,保持根充分浸入营养液中,并用通气泵通气。试验在山东省农业科学院作物研究所光照培养室进行,培养温度为25℃/22℃日/夜,光照时间为16 h/d,湿度控制在50%~60%。待真叶展开,挑选长势一致的幼苗转移至盛有5 L 改良Hoagland 营养液的塑料盒中进行培养,通气24 h,6 d 后用150 mmol/L NaCl 处理,分别在0、1、3、6、12 h 取根部组织,每个时间点3 次生物学重复,样品提取RNA 后由北京诺禾致源科技股份有限公司进行转录组测序分析。另外进行盐处理转录组数据验证,分别在0、1、3、6、9、12 h 取地上部和地下部,每个时间点3 次生物学重复。
激素处理试验:真叶展开后转移至5 L 改良Hoagland 营养液中,3 d 后换新的营养液并在其中分别加入10 μmol/L 乙烯合成前体1-氨基环丙烷-1-羧酸(1-aminocyclopropane-1-carboxylic acid, ACC)、 100 μmol/L 脱落酸(ABA)、 10 μmol/L 赤霉素(GA3)。在激素处理0、1、3、6、9、12 h 取根部组织,每组处理均为3 次生物学重复。
1.2.2 大豆盐胁迫响应扩展蛋白基因的鉴定对转录组进行分析,将P-adjust value<0.05 且的扩展蛋白基因定为差异表达基因,筛选4 个时间点均响应盐胁迫的大豆扩展蛋白基因。将差异扩展蛋白基因在拟南芥TAIR 数据库中进行BLAST,分值最高的拟南芥基因作为每个大豆扩展蛋白基因的同源基因。
1.2.3 大豆盐胁迫响应扩展蛋白基因上游顺式作用元件分析 利用差异表达扩展蛋白基因ID号从Phytozome(https:/ /phytozome-next.jgi.doe.gov/)数据库中提取基因起始密码子上游1 500 bp 启动子序列,使用在线软件PlantCare(http:/ /bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析上游可能存在的顺式作用元件。利用GSDS 2.0 在线网站(http:/ /gsds.cbi.pku.edu.cn/)[18]进行顺式作用元件的可视化。
1.2.4GmEXPA17a基因的克隆 提取齐黄34 根系组织RNA,并反转录成cDNA,具体操作步骤参考试剂盒说明书。从Phytozome 中下载GmEXPA17a的CDS 序列并设计克隆引物,引物由擎科生物技术有限公司合成,引物序列为GmEXPA17a-F:5′-ATGGGAAAGTTCATTTTGAG-3′和GmEXPA17a-R:5′- GAACTGGACATTGCTGGTGA-3′。以cDNA 为模板,利用诺唯赞的KOD 高保真酶对CDS 全长进行克隆。PCR 反应体系(15 μL):2×ChamQ Universal SYBR qPCR Master Mix 7.5 μL,Primer F 0.3 μL,Primer R 0.3 μL,cDNA 1.5 μL,ddH2O 5.4 μL,反应程序:95℃3 min;95℃15 s,56℃30 s,72℃1 min,共35 个循环;72℃5 min。PCR 产物经1%琼脂糖凝胶电泳检测后,将长度762 bp 的片段切胶回收,利用天根pLB 零背景快速克隆试剂盒将目的片段与pLB 载体连接后转化大肠杆菌DH5α,菌液涂布于含有氨苄青霉素的LB 固体培养基上,倒置于37℃恒温培养箱过夜培养,挑取单克隆进行菌液PCR 扩增鉴定,阳性克隆摇菌后送往青岛擎科生物科技有限公司进行测序,测序成功后利用DNAMAN 软件进行序列比对。
1.2.5 GmEXPA17a 的生物信息学分析 采用在线软件GSDS 2.0 进行GmEXPA17a基因结构的绘制;在NCBI 中(https:/ /www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行GmEXPA17a 保守结构域分析;利用在线分析软件ExPASy(https:/ /web.expasy.org/compute_pi/)计算GmEXPA17a 蛋白的分子量和等电点;利用SOMPA 软件(https:/ /npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page =npsa_sopma.html)预测其二级结构;使用NetPhos 3.1(https:/ /services.healthtech.dtu.dk/service.php? NetPhos-3.1)软件进行磷酸化位点预测;利用TMHMM Server v.2.0(http:/ /www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构域预测;氨基酸序列在NCBI 中进行BLAST,选取分值较高的氨基酸采用DNAMAN 软件与GmEXPA17a进行序列比对。
1.2.6 实时荧光定量PCR(qRT-PCR) 利用NCBI 网站中的Primer-Blast 在线软件(https:/ /www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi? LINK_LOC =BlastHome)设计用于检测基因GmEXPA17a表达量的特异性引物,引物序列为qGmEXPA17a-F:5′-TCAGGGGCACTTCCATTGTT-3′和qGmEXPA17a-R:5′- ACACCAGCCACCATTGTCATT-3′。以大豆GmActin为内参基因,引物序列为qGmActin-F:5′-CGGTGGTTCTATCTTGGCATC- 3′和qGmActin-R:5′-GTCTTTCGCTTCAATAACCCTA-3′。提取不同组织部位、盐胁迫处理、激素处理和对照样品的RNA 并反转录成cDNA。以稀释10 倍的cDNA 为模板,利用诺唯赞生物科技有限公司的TB green premix ExTaq试剂盒进行荧光定量PCR 体系的配置,在罗氏480 荧光定量PCR 仪进行实时荧光定量PCR 检测,每个样品进行3 次重复。反应程序:95℃30 s;95℃5 s,60℃30 s, 40 个循环。利用2-ΔΔCt方法计算基因的相对表达量。
1.3 数据分析
利用Microsoft Excel 2010 进行数据处理和绘图。用SAS 9.0(SAS Institute Ltd., USA)进行显著性统计分析。差异显著性利用Fisher’s LSD(P<0.05)进行检验。
2 结果与分析
2.1 大豆盐胁迫响应扩展蛋白基因的筛选
对耐盐品种齐黄34 根系进行盐胁迫处理,以未经盐处理的根系样品为对照进行转录组测序,通过分析共筛选到7 个扩展蛋白基因在4 个处理时间点表达量发生显著变化。如表1 所示,盐胁迫处理显著诱导GmEXPA17a、GmEXPA17b、GmEXPA17c、GmEXLB1a和GmEXLB1b五个基因的表达,其中GmEXPA17a和GmEXLB1a响应程度较大。GmEXPA10和GmEXPA12基因的表达在盐处理后呈下降趋势。

表1 大豆盐胁迫响应扩展蛋白基因
2.2 盐胁迫响应扩展蛋白基因启动子分析
在Phytozome 上下载7 个持续响应盐胁迫的大豆扩展蛋白基因起始密码子前1 500 bp 的启动子序列,输入到PlantCare 在线软件中对启动子进行顺式作用元件分析。结果发现这些基因的启动子上存在多个与非生物胁迫及植物激素应答元件相关的顺式作用元件,包括干旱诱导响应元件(MBS)、胁迫响应(TC-rich)、脱落酸(ABRE)、乙烯(ERE)、赤霉素(P-box)和生长素响应(TGAelement)元件(图1)。统计发现,7 个扩展蛋白基因启动子上均含有脱落酸响应元件。
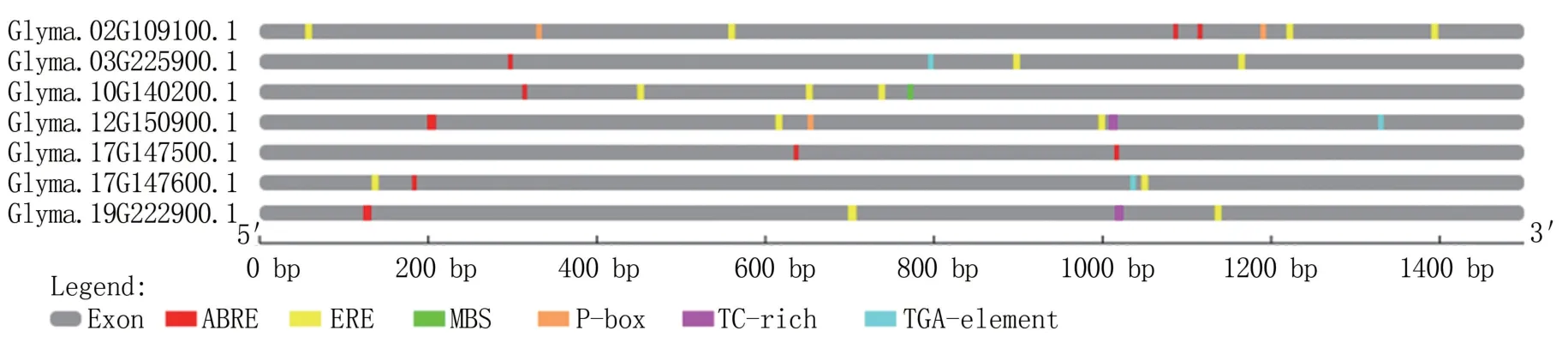
图1 大豆盐胁迫响应扩展蛋白基因启动子顺式作用元件示意图
2.3 GmEXPA17a 的克隆和生物信息学分析
本研究对α-expansin 家族响应盐胁迫程度最大的基因Glyma.19G222900.1(GmEXPA17a)进行了分析。该基因位于第19 号染色体,基因序列全长1 298 bp,其中开放阅读框762 bp,含有3 个外显子和2 个内含子(图2A)。从齐黄34 的根部克隆了GmEXPA17a基因的CDS 序列全长,并连接到pLB 载体上进行转化与测序,结果发现齐黄34 中GmEXPA17a的开放阅读框序列与大豆威廉姆斯82 的参考基因组序列完全相同。该基因编码254 个氨基酸,CDD 结构域分析发现,GmEXPA17a 的第1~254个氨基酸为PLN00193 结构域(图2B)。生物信息学分析结果表明,其蛋白质平均分子量和等电点分别为27 921.83 Da 和9.72。蛋白质二级结构预测结果表明,GmEXPA17a 蛋白由10.24%的α 螺旋、53.15%的无规则卷曲、28.74%的延伸链和7.87% 的β 转角构成(图2C)。磷酸化位点预测分析发现,GmEXPA17a 蛋白共有磷酸化位点Tyr 2 个、Ser 15 个、Thr 4 个(图2D)。GmEXPA17a 蛋白含有典型的跨膜结构域,推测为膜定位蛋白(图2E)。
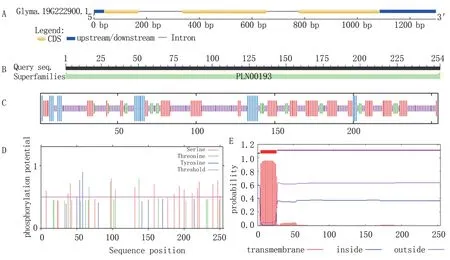
图2 GmEXPA17a 的生物信息学分析
将GmEXPA17a 的氨基酸序列在NCBI BLAST 中进行查找比对,其与刺毛黧豆、野生大豆、木豆、豇豆和赤豆等豆科作物的扩展蛋白相似度较高。对其进行氨基酸序列相似性比对,发现GmEXPA17a 与野生大豆(XP_028217735.1)的扩展蛋白相似度最高,相似度达到99.61%。各个扩展蛋白基因的氨基酸序列在氨基端和羧基端的保守性较高(图3)。

图3 GmEXPA17a 氨基酸与其它豆科植物扩展蛋白氨基酸序列的比对
2.4 GmEXPA17a 对盐胁迫处理的响应
为了验证转录组试验数据的准确性,重新对齐黄34 进行不同时间盐胁迫处理,然后取地上部和地下部样品进行GmEXPA17a表达量检测,以未添加NaCl 处理的0 h 作为对照。结果如图4所示,盐处理显著诱导不同时间点大豆地上部和地下部GmEXPA17a基因的表达,无论在地上部还是地下部,GmEXPA17a对盐胁迫响应较快,处理1 h 后显著上调。与地上部相比,盐处理对地下部GmEXPA17a诱导量较大,盐处理3 h 后,地上部GmEXPA17a表达量变化较小(图4)。
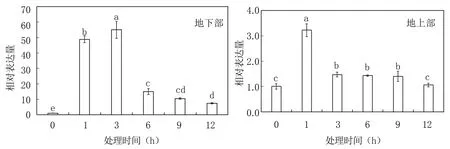
图4 盐处理后地上部和地下部GmEXPA17a 基因的相对表达量
2.5 GmEXPA17a 表达模式分析及对不同激素处理的响应
取齐黄34 不同部位,利用RT-qPCR 检测GmEXPA17a基因的表达模式,结果如图5 所示。GmEXPA17a在大豆多个组织中均有表达,其中在根中的表达量最高。

图5 GmEXPA17a 基因在不同组织部位的相对表达量
用不同植物激素处理齐黄34 幼苗,取根部进行GmEXPA17a表达量检测,结果如图6 所示。脱落酸(ABA)处理条件下,GmEXPA17a的表达量在1、3、6、9、12 h 均显著上调,处理9 h 时达到最高;与ABA 处理相似,乙烯合成前体(ACC)处理同样显著诱导GmEXPA17a表达,ACC 处理6 h时GmEXPA17a表达量最高,之后逐渐下降;GmEXPA17a对赤霉素(GA3)响应较快,处理3 h达到最高值,然后逐渐下降。这些结果表明GmEXPA17a的启动子上含有脱落酸、乙烯和赤霉素响应元件,能够调控其表达。

图6 不同激素处理后GmEXPA17a 基因的相对表达量
3 讨论与结论
土壤盐渍化严重影响大豆的生长和干物质积累,造成产量的下降。植物细胞壁对抵御生物和非生物胁迫起着重要的屏障作用[7,19]。扩展蛋白作为一种细胞壁蛋白,在植物响应盐胁迫和干旱过程中发挥重要作用。目前已报道大豆中扩展蛋白通过调控根系发育参与磷胁迫和干旱胁迫响应[15,20]。然而,扩展蛋白在大豆盐胁迫响应中的功能未见报道。
本研究通过对齐黄34 大豆根系盐处理后不同时间点的转录组进行分析,鉴定出7 个持续响应盐胁迫的扩展蛋白基因。启动子分析发现,这7 个基因的启动子上均含有植物激素或胁迫应答元件。植物激素通过信号转导途径调控下游盐胁迫响应基因的表达,广泛参与到渗透胁迫、ROS平衡和Na+转运等植物耐盐过程中,其中研究较多的是植物激素ABA[21]。本研究结果表明,这7个持续响应盐胁迫的扩展蛋白启动子上均含有ABA 应答元件(ABRE),推测这些基因可能通过ABA 信号参与调控盐胁迫响应。此外,这些扩展蛋白基因的启动子上均至少含有乙烯和赤霉素信号元件,这两种激素在调控植物根系发育中均发挥重要作用[22],且目前已报道的大豆扩展蛋白主要通过调控根系发育参与到非生物胁迫反应中[15]。因此,我们推测大豆扩展蛋白可能通过植物乙烯和赤霉素信号调控根系构型参与到盐胁迫响应中。
本研究从耐盐品种齐黄34 中克隆了α 家族中对盐胁迫响应程度最大的大豆扩展蛋白基因GmEXPA17a,该基因的表达在地上部和地下部均受到盐胁迫的显著诱导,而且地下部该基因对盐胁迫响应较强烈。有研究表明,拟南芥扩展蛋白基因AtEXPA17(AT4G01630.1)正调控侧根发育,AtEXPA17敲除会显著抑制拟南芥侧根的发育[14]。前人研究表明GmEXLB2通过调控大豆根系构型参与营养胁迫和干旱胁迫响应[15]。本研究中克隆得到的GmEXPA17a为拟南芥AtEXPA17的同源基因。因此,GmEXPA17a可能通过调控大豆侧根的发育参与大豆盐胁迫响应。通过对齐黄34 进行淹水处理后进行转录组测序筛选到了大量差异表达基因,发现GmEXPA17a在淹水处理的4 个时间点均显著上调[23]。因此我们推测GmEXPA17a可同时响应盐胁迫和涝渍胁迫,是研究大豆逆境胁迫响应的重要候选基因。组织表达分析发现GmEXPA17a基因在根、茎、叶和花中均有表达,在根中表达量最高。Zhu 等[13]通过转录组数据库分析大豆扩展蛋白在不同组织器官中的表达发现,31%的大豆扩展蛋白基因属于组织型表达,即在不同组织中表现出偏好性,只在特定的组织或器官中出现表达高峰,因此推测,GmEXPA17a 也属于偏好性蛋白。
GmEXPA17a的启动子中含有脱落酸(ABA)、乙烯合成前体ACC 和赤霉素(GA3)应答元件。实时荧光定量PCR 证明该基因的表达量在3 种激素处理后不同时间点均显著上升,表明该基因受到激素的调控,可能参与了ABA、ACC和GA3的信号转导途径。高浓度盐处理可以显著诱导内源脱落酸含量的增加[24,25],内源ABA 含量的升高可以提高水稻[26]、玉米[27]和菜豆[28]的耐盐性。本研究中ABA 处理可以显著诱导GmEXPA17a基因的表达,因此,GmEXPA17a对盐胁迫的强烈响应很有可能是通过对脱落酸信号改变的感知而实现的。
本研究通过转录组测序筛选到7 个持续响应盐胁迫的扩展蛋白,为今后研究大豆扩展蛋白基因家族在逆境胁迫响应过程中的功能提供了资源。同时,GmEXPA17a的克隆、生物信息学分析和激素应答分析为下一步研究其功能提供了理论基础。
