向日葵黑茎病菌RPA/CRISPR-Cas12a快速检测方法的建立
2022-12-19邝瑞瑞段维军李雪莲范在丰吴品珊
邝瑞瑞,雷 荣,江 丽,段维军,李雪莲,符 娜,范在丰,李 远,吴品珊*
(1.中国检验检疫科学研究院,北京 100176;2.中国农业大学植物保护学院,北京 100093;3.重庆医科大学永川附属医院,重庆 402177;4.宁波检验检疫科学技术研究院,宁波 315012;5.伊犁师范大学,伊犁 835000)
向日葵是我国重要的油料作物之一,栽培面积仅次于大豆和油菜,不仅具有重要的经济价值[1],还有观赏价值、药用价值[2]。根据中国海关统计数据,我国每年需要进口15万t左右的向日葵种子,为防止检疫性有害生物的传入及扩散,需持续加强进境向日葵种子检疫检测和田间种植监测,为此需不断提升检测技术手段和开发新技术。
向日葵黑茎病菌Leptosphaerialindquistii(Frizzi) Gruyter,Aveskamp &Verkley,无性阶段为PhomamacdonaldiiBoerema[3],是向日葵上的一种毁灭性有害生物,2010年由原农业部和原国家质量监督检验检疫总局(第1472公告,关于将向日葵黑茎病列为进境检疫性有害生物的公告)列为进境检疫性真菌。该病原菌1964年在加拿大被首次发现后[4],相继扩展到法国、美国、阿根廷等国家和地区[3,5-6],引发的病害发生率高,产量损失大[7]。2007年我国新疆伊犁地区首次报道了该病原菌的局部分布[8],随后内蒙古等地也报道了其分布[9]。向日葵黑茎病传播速度快,严重降低向日葵抗逆性,造成巨大的经济损失[10-11]。向日葵黑茎病初期危害植株叶柄基部,然后扩展到茎秆形成椭圆形、边缘清晰的黑色病斑,严重时环绕茎秆造成植株倒伏,叶片枯萎、花盘瘦弱,减产严重[12-13]。
目前对向日葵黑茎病菌的检疫鉴定,按照出入境检验检疫行业标准《向日葵黑茎病菌检疫鉴定方法》(SN/T 3174-2012)[14]进行,包括分检、挑选带病组织进行分离培养、形态学观察、核酸提取、利用通用引物ITS1/ITS4 或ActF1/ActR1进行扩增、测序及序列比对。其他已报道的检测技术包括根据向日葵黑茎病菌ITS序列特异位点设计的特异引物320FOR/320REV[15]和LEPB/LEPF[16],以及基于Actin基因设计的LLF/LLR[17]引物进行PCR检测等。实时荧光PCR具有不易污染、操作简单、灵敏度高、特异性强等优点[18],但是需要专门的仪器,耗时较长,不适于田间野外的现场检测。环介导等温扩增(loop-mediated isothermal amplification,LAMP)在60~65℃等温条件下反应,可与比色法结合,检测向日葵黑茎病菌的灵敏度为10个分生孢子/g向日葵种子[19],但是反应温度较高,需要多对引物,引物设计复杂,要求高。
重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)是一种仿照T4噬菌体的体外核酸等温扩增方法。该技术无需造价高昂的升降温仪器,仅需一对引物,即可在37~42℃恒温条件下,对目标DNA进行快速的指数级扩增[27],降低了设备需求,反应速度更快,灵敏度高和特异性强,是现场快速检测优选的核酸扩增技术。CRISPR-Cas系统由成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)及CRISPR相关蛋白(CRISPR associated protein,Cas)构成[20],已被广泛用于病原物检测[21],但在植物真菌病害上的应用较少[22-24]。Cas12a是第二类Ⅴ型CRISPR RNA (CvRNA) 引导的一种核酸内切酶[25],在crRNA引导下识别原始间隔区相邻基序(protospacer adjacent motif,PAM)(TTTN)序列下游18~25 nt的靶标DNA,当Cas12a/crRNA复合体靶向结合到核酸底物后,会激活非特异性的ssDNA的反式切割活性。基于这一性质,Chen等[26]开发了一种新的DNA 检测方法—DETECTR(DNA endonuclease targeted CRISPR trans reporter),为病害的分子诊断提供了一种新的策略。
本研究拟将RPA技术与CRISPR-Cas12a系统相结合(RPA/CRISPR-Cas12a),在RPA的基础上进一步增加选择性和灵敏度[28-30]。采用CRISPR-Cas12a荧光检测和侧流向试纸条检测两种方法,通过优化检测条件,建立一种快速现场检测向日葵黑茎病菌的分子生物学方法,为口岸检疫以及田间早期诊断提供新技术。
1 材料与方法
1.1 材料
1.1.1供试菌株
本研究所用向日葵黑茎病菌菌株来源于从美国进口的向日葵种子中截获的向日葵黑茎病菌以及向日葵黑茎病菌的近似种、向日葵上发生的其他真菌和一些常见真菌(表1)。

表1 本研究所用的真菌信息Table 1 Fungal isolates used in this study
1.1.2主要试剂
RPA基础扩增试剂盒,英国TwistDX公司;侧向流试纸条检测试剂盒,北京宝盈同汇生物技术有限公司;DNA浓度测量试剂盒、Qubit®2.0荧光仪、Qubit®dsDNA HS Assay Kit,美国ThermoFisher Scientific公司;磁珠法植物基因组DNA提取试剂盒(DP342)、无酶无菌水、RNA酶抑制剂(40 U/μL,NG209),天根生化科技(北京)有限公司;Cas12a,美国 NEB 公司;PerfectStart®Ⅱ Probe qPCR SuperMix,北京全式金生物技术股份有限公司。超纯水取自 Milli-Q纯水系统(德国 Millipore 公司);LightCycler480 实时荧光 PCR 仪,瑞士罗氏公司。便携式加热装置为自主研发设计,委托重庆初生犊科技有限公司制造 (型号为GeneFASD-37)。所用引物序列见表2。
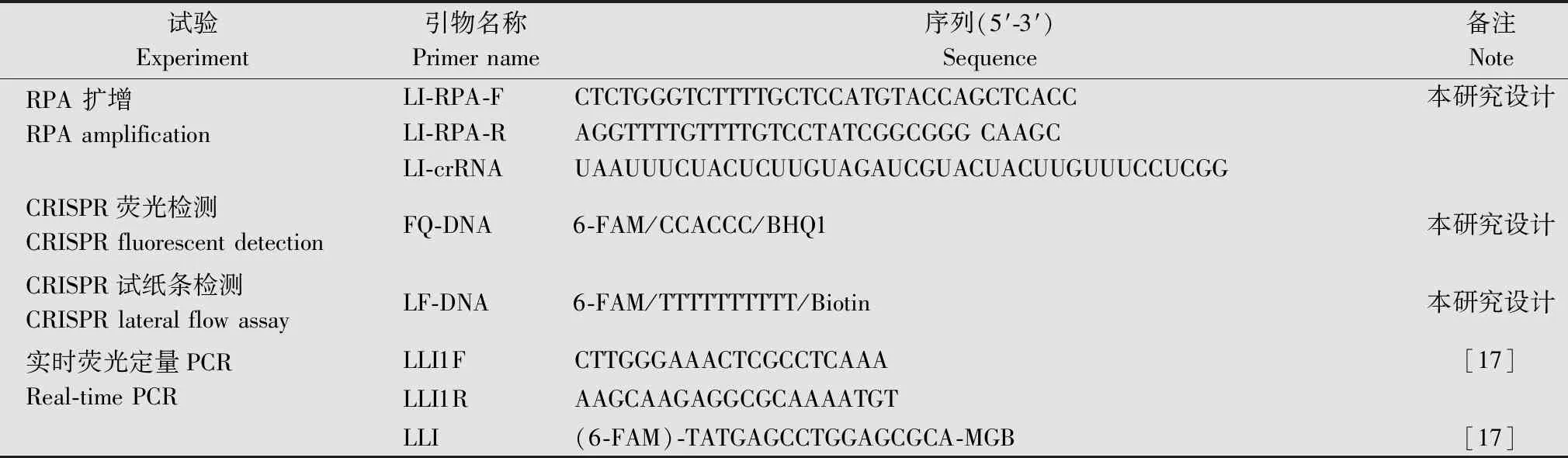
表2 本研究所用的引物、报告分子和探针序列Table 2 Primer,reporter and probe sequences used in this study
1.2 方法
1.2.1菌株培养及其基因组DNA提取
将供试菌株接种到PDA培养基上,25℃,L∥D=12 h∥12 h条件下培养5 d,收集菌丝和孢子,液氮研磨后使用植物基因组DNA提取试剂盒提取基因组DNA。用Qubit®2.0荧光仪和Qubit®dsDNA HS检测试剂盒对提取DNA进行定量。
1.2.2RPA引物设计
从NCBI数据库中下载向日葵黑茎病菌及其近似种的ITS序列,选择特异性序列设计引物LI-RPA-F/LI-RPA-R和LI-crRNA(表2)。引物和LI-crRNA由通用生物(安徽)股份有限公司合成。
1.2.3RPA/CRISPR-Cas检测方法
RPA反应扩增体系(50 μL):每个反应管中加入29.5 μL 水化液(rehydration buffer),11.2 μL的RNAse free去离子水,10 μmol/L上游、下游引物各2.4 μL,2 μLDNA,280 mmol/L醋酸镁2.5 μL,充分混匀后,将反应管转移到便携式加热装置中,37℃加热30 min。
CRISPR-Cas12a检测采用荧光和试纸条两种方式。荧光检测法体系:DEPC-H2O 13.4 μL,NEBuffer 3.1 (10×)2 μL,5 μmol/L Cas12a 0.4 μL,40 U/μL RNase inhibitor 0.5 μL,0.1 mmol/L DTT 0.5 μL,5 μmol/L荧光报告分子FQ-DNA 0.8 μL,0.01 mmol/L LI-crRNA 0.4 μL,将上述溶液加到反应管中充分混合后,取2 μL RPA扩增产物加至反应管的盖子内侧,振荡离心后,迅速将反应管转移到实时荧光PCR仪中,37℃加热30 min进行荧光检测。
侧向流层析试纸条检测体系:DEPC-H2O 12.2 μL,NEBuffer 3.1 (10×)2 μL,5 μmol/L Cas12a 0.4 μL,40 U/μL RNase inhibitor 0.5 μL,0.1 mmol/L DTT 0.5 μL,5 μmol/L侧向流层析试纸条报告分子LF-DNA 2 μL,0.01 mmol/L LI-crRNA 0.4 μL,上述溶液加到反应管中充分混合后,加入2 μL RPA扩增产物,振荡离心后,将反应管转移到便携式加热装置中,37℃加热20 min。反应完成后加入80 μL去离子水,混合均匀,将试纸条的样品端插入离心管中观察结果。
1.2.4RPA/CRISPR-Cas检测体系的特异性
根据上述 RPA/CRISPR-Cas扩增反应体系,以无菌水为阴性对照,采用引物对LI-RPA-F/LI-RPA-R对真菌DNA(表1)进行RPA扩增,其扩增产物首先用CRISPR-Cas12a荧光法检测。再对RPA扩增产物采用侧向流试纸条进行检测,每个样品设置3个重复,根据检测结果判断检测方法是否具有特异性。
1.2.5RPA/CRISPR-Cas检测体系优化
RPA扩增体系中,设置5个反应时间,分别为10、20、25、30 min和40 min,其他条件不变。以无菌水为阴性对照,采用CRISPR-Cas12a荧光检测,观察不同条件下荧光信号强度。
CRISPR-Cas12a荧光检测体系中,对荧光报告分子浓度进行优化,设置最终报告分子浓度为50、100、200、400,800 nmol/L等5个浓度,其他条件不变。以无菌水为阴性对照,观察不同条件下荧光信号强度。
CRISPR-Cas12a试纸条检测体系中,对试纸条报告分子浓度,反应时间进行优化。在最终100 μL反应体系中设置5个报告分子浓度:50、100、200、400,800 nmol/L。反应时间设置5个梯度:5、10、15、20 min和30 min。保持其他条件不变,以无菌水为阴性对照,观察不同条件下试纸条变化情况。
1.2.6RPA/CRISPR-Cas检测体系灵敏度
将向日葵黑茎病菌DNA进行10倍梯度稀释为 1 ng、0.1 ng、10 pg、1 pg、0.1 pg、0.01 pg,每个样品设置3个重复,按照优化后的反应体系进行RPA/CRISPR-Cas扩增和检测,并与实时荧光PCR方法进行比较。
实时荧光PCR检测体系采用段维军等[18]设计的特异性引物LLI1F/LLI1R和探针 LLI(表2)。引物由擎科生物合成,荧光探针和LI-crRNA由通用生物(安徽)股份有限公司合成。探针5′端含有 FAM 报告荧光染料,3′端含有不发荧光的淬灭基团并具有 MGB 分子。反应体系:2xTaqMan PCR Master Mix 10 μL,10 μmol/L上下游引物各0.4 μL,10 μmol/L探针0.4 μL,DNA 1 μL,去离子水补充至20 μL。反应条件:95℃ 30 s;94℃ 5 s,60℃ 1 min,循环40次。
1.2.7实际样品检测
抽取宁波口岸进口的来自美国、法国、哈萨克斯坦、意大利和加拿大的向日葵种子以及采自新疆伊犁的向日葵茎秆进行实际样品检测,将种子样品倒入洁净白瓷盘内,尽量挑选干瘪、弱小、畸形的疑似感病种子,液氮研磨后使用植物基因组DNA提取试剂盒提取基因组DNA。采用本研究优化后的RPA/CRISPR-Cas体系进行向日葵黑茎病菌检测。
2 结果与分析
2.1 RPA/CRISPR-Cas12检测向日葵黑茎病菌的特异性分析
根据向日葵黑茎病菌及其近似种的ITS序列,用DNAMAN进行多序列比对,选择差异性大的序列设计了引物LI-RPA-F和LI-RPA-R,通过NCBI BLAST进一步验证扩增产物的特异性,扩增产物总长为114 bp。再结合CRISPR-Cas识别位点PAM,设计了LI-crRNA。
采用LI-RPA-F/LI-RPA-R对所有供试真菌的基因组DNA(表1)进行RPA扩增,其扩增产物首先用CRISPR-Cas12a荧光法检测,结果(图1a)显示,向日葵黑茎病菌DNA样品出现明显的荧光信号,而其他真菌DNA的荧光信号很低。CRISPR-Cas12a试纸条测试结果(图1b)显示,向日葵黑茎病菌DNA样品出现了明显的质控线(C)和检测线(T),而空白对照及其他真菌DNA样品仅有质控线,无检测线。对扩增产物进行测序,核苷酸序列与向日葵黑茎病菌的ITS序列一致,说明RPA/CRISPR-Cas12a的RPA引物和LI-crRNA能特异性检测向日葵黑茎病菌,可以做进一步试验。

图1 向日葵黑茎病菌RPA/CRISPR-Cas12a检测体系的特异性Fig.1 The specificity of RPA/CRISPR-Cas12a detection system for Leptosphaeria lindquistii
2.2 RPA/CRISPR-Cas检测体系优化
2.2.1RPA反应时间优化
取RPA扩增时间分别为10,20,25,30,40 min后的扩增产物,进行CRISPR-Cas12a荧光检测。结果表明,CRISPR-Cas12a荧光强度随RPA扩增时间增加而增强,而空白对照均无明显荧光信号(图2a)。反应时间为30、40 min 时,CRISPR-Cas12a荧光检测强度较高,为了满足快速检测的需求,采用RPA扩增时间30 min作为后续的试验条件。
2.2.2CRISPR-Cas12a荧光检测报告分子浓度优化
报告分子浓度优化结果表明,CRISPR-Cas12a荧光报告分子浓度越高,荧光强度越强,当报告分子终浓度达到200 nmol/L以上时,可以看到明显的荧光信号,报告分子终浓度为800 nmol/L时,荧光强度极为明显(图2b)。考虑成本,故采用CRISPR-Cas12a荧光报告分子最终浓度200 nmol/L为最优选择。
2.2.3CRISPR-Cas12a试纸条检测反应时间优化
CRISPR-Cas12a试纸条检测反应时间优化结果表明,CRISPR/Cas12a反应5 min,即可产生可检测到的产物,显示弱的检测线(T线);随CRISPR反应时间增加,检测线随之加深,反应20 min即出现明显的检测线(图3a)。故可选择20 min作为CRISPR/Cas12a的反应时间。

图2 向日葵黑茎病菌RPA/CRISPR-Cas12a荧光检测体系的优化Fig.2 Optimization of the RPA/CRISPR-Cas12a fluorescent detection system for Leptosphaeria lindquistii

图3 向日葵黑茎病菌CRISPR-Cas12a试纸条检测优化Fig.3 Optimization of CRISPR-Cas12a lateral flow assay for Leptosphaeria lindquistii
2.2.4CRISPR-Cas12a试纸条检测报告分子浓度优化
在100 μL试纸条检测体系中的LF报告分子浓度为50 nmol/L时,空白对照的检测线有微弱红色条带,表现出假阳性。LF报告分子浓度为100 nmol/L时,空白对照的检测限处隐约有点条带,说明LF报告分子浓度低,容易产生假阳性(图3b)。报告分子浓度高于200 nmol/L,空白对照无假阳性现象。可采用CRISPR-Cas12a试纸条报告分子最终浓度200 nmol/L进行后续的试验。
2.3 RPA/CRISPR-Cas检测体系灵敏度
将提取的向日葵黑茎病菌的基因组DNA 10倍稀释,以引物LLI1F/LLI1R和探针LLI进行实时荧光定量PCR检测,结果(图4a)表明,DNA含量为1 ng、0.1 ng、10 pg、1.0 pg、0.1 pg时,都能检测到荧光信号,表现为阳性扩增,3次重复试验的平均Ct值分别为17.95,22.18,25.49,30.26和34.24。虽然0.01 pg基因组DNA产生弱的荧光信号,但是其Ct值为35。表明实时荧光定量PCR方法的灵敏度为0.1 pg,与文献报道的灵敏度一致。

图4 两种方法检测向日葵黑茎病菌DNA的灵敏度Fig.4 Sensitivity of two methods for detection of Leptosphaeria lindquistii DNA
利用优化的RPA/CRISPR-Ca12a荧光法检测相同的DNA样品,结果表明,1 pg向日葵黑茎病菌基因组DNA产生明显的荧光信号,0.1 pg DNA产生可被检测的弱荧光信号(图4b)。表明CRISPR-Cas12a荧光法的灵敏度为0.1 pg,与实时荧光PCR的灵敏度相当。利用优化的RPA/CRISPR-Cas试纸条检测10倍梯度稀释的向日葵黑茎病菌基因组DNA,结果表明,1 pg DNA产生弱的检测信号,而0.1 pg DNA不能被检测到。表明CRISPR-Cas12a试纸条的灵敏度为1 pg,低于实时荧光定量PCR和RPA/CRISPR-Cas12a荧光法。
2.4 向日葵种子样品的检测
采用优化后的RPA/CRISPR-Cas体系检测了10份从宁波口岸进口的向日葵种子,其中有2份哈萨克斯坦进口的向日葵种子,荧光法和试纸条检测都为阳性,与实时荧光定量PCR结果相同。证明该扩增体系可以检测来自不同地理来源的向日葵黑茎病菌,适用于口岸向日葵黑茎病菌的快速检测(图5)。

图5 向日葵黑茎病菌实际样品的检测Fig.5 Detection of actual samples for Leptosphaeria lindquistii
3 结论与讨论
快速精准的检测技术为防止检疫性有害生物的传入及扩散提供强有力的技术保障。为了有效防控向日葵黑茎病菌的传播及危害[6],已建立了形态学鉴定[30-31]和分子生物学检测技术[15,17-18,32-35]。当前采用的分子生物学技术,以普通PCR[15-17]和TaqMan实时荧光定量PCR[18]为主,虽然灵敏度高[33],但均需要专门的热循环设备,不适合用于田间和口岸的现场检测。重组酶聚合酶等温扩增技术利用一对引物在37℃即可对核酸进行指数扩增,整个反应过程约20 min[36]。本研究建立的CRISPR-Cas12a检测体系也是在37℃下进行,并且在crRNA的引导下,识别到目标DNA后,发挥其切割功能,因此两者的结合,可以极大地提高RPA扩增的特异性和灵敏度。在Cas12a/crRNA反应体系中,加入荧光报告分子(FAM-CCACCC-BHQ1),如果存在目标序列,荧光报告分子被切割出FAM基团,就可被488 nm光源激发产生绿色荧光;如果不存在目标序列,则FAM基因的荧光会被BHQ1淬灭而不产生绿色荧光。荧光强度大小与目标DNA浓度正相关。由于RPA/CRISPR-Cas12a检测体系包含RPA的指数扩增反应和Cas12a/crRNA的酶-核酸反应,荧光强度与原始DNA模板的函数关系研究尚无报道,还需要进一步试验。
RPA/CRISPR-Cas12a荧光检测法灵敏度高,与TaqMan探针实时荧光定量PCR的灵敏度相当[17],但是因为需要激发光源,易受干扰,对环境要求高,不适于野外田间检测。侧流向试纸条检测法不需要任何仪器设备,在常温下即可进行。在CRISPR-Cas12a反应体系中加入试纸条报告分子(FAM-T10-Biotin),可采用金纳米核酸试纸条进行检测。当Cas12a/crRNA识别到目标序列时,报告分子被切割后,产生FAM与金纳米(AuNPs)上的FAM抗体结合,在检测线处产生条带;否则AuNPs-FAM-Biotin只留在控制线处产生条带。由于试纸条检测在常温20 min内可完成,检测结果用肉眼即可观察,更适合用于野外田间检测。
为了更好的特异性检测,本研究采用RPA与CRISPR-Cas12a联用的方式,先用RPA对目的基因进行指数扩增,再用Cas12a/crRNA核酶复合物进行进一步的目标基因识别,从而极大地提高了特异性。RPA的指数扩增,同时也增加了检测灵敏度。试验条件优化结果表明,RPA反应时间越长,产生的扩增产物越多,最终的信号越强。但同时也可观察到,RPA反应时间为10 min时,就可检测到产物。而CRISPR-Cas12a反应15 min,就能检测到扩增产物,说明RPA和CRISPR-Cas12a均为快速检测技术,可以用于病原菌的快速检测方法开发。RPA/CRISPR-Cas12a荧光检测操作相对复杂,且需要特定的仪器,但灵敏度高,适用于实验室内检测。RPA/CRISPR-Cas12a试纸条检测体系对环境的要求低,仅需一台便携式升温仪器,恒温条件37℃下进行反应,不需要繁琐的琼脂糖凝胶电泳检测,更加简便快捷,操作易上手,比起传统的分子检测,更适用于向日葵黑茎病菌的快速现场检测、口岸检疫等需求。
