基于环境DNA宏条形码的柴河浮游藻类研究
2022-12-10吕嘉诚林渊源李爱军
吕嘉诚,林渊源,李爱军,赵 峥
(1.昆明学院,昆明市河湖健康评估与修复院士工作站,云南省高校河湖健康评估与修复重点实验室,云南 昆明 650214;2.云南省生态环境监测中心,云南 昆明 650034)
0 引言
浮游藻类是水生态系统关键类群之一[1]。浮游藻类多样性可反映水生态系统健康状况,对指导受损水生态系统的修复有重要作用,广泛应用于河湖生态健康评估和修复[2]。近年来滇池浮游藻类群落结构引起人们的广泛关注,学者们探寻其群落结构的变化特征[3],研究影响群落结构的环境因子[4],为滇池的治理和修复提供了诸多基础信息。柴河是滇池的主要入湖河流,由于河流周边持续的农业活动,成为滇池流域典型的农业面源污染河道[4]。研究发现柴河从上游到下游浮游藻类检出率、藻密度与河流位置无关,与采样点具体环境有关,石门坎水库未能检出浮游藻类,且在甸头人民坝发生栅藻(Scenedesmus)水华现象[5]。
环境DNA宏条形码技术的应用,为分析浮游藻类多样性提供了新途径[6]。许多研究已经证明了环境DNA宏条形码技术在监测浮游藻类方面的出色效果[7]。有研究表明环境DNA宏条形码技术与传统方法相比在显示浮游藻类丰度方面有显著提升,同时能监测到更为丰富的浮游藻类多样性[8]。在评估水生生物多样性方面结果可靠,并在描述水生生态景观差异时展现出很大潜力[9]。已有研究人员使用环境DNA高通量测序方法对太湖等典型湖泊藻类进行了监测,证明环境DNA高通量测序是一种潜在代替传统监测方法的优秀工具[10-13]。
基于传统形态学鉴定的浮游藻类研究,对专业要求较高且费时费力。为了高效快速地监测柴河浮游藻类状况,本研究于2021年7月,在柴河水库及水库上下游的三个采样点,通过环境DNA宏条形码技术及显微鉴定计数法调查柴河浮游藻类的群落组成和分布,旨在探讨环境DNA宏条形码对柴河浮游藻类研究的适用性,并科学评价其水质,为柴河水资源保护提供基础资料。
1 实验方法
1.1 采样点布设和环境样品采集
柴河位于昆明市晋宁区。作为滇池南岸主要入湖河流,柴河承担着防洪、灌溉等任务。柴河流域以第一产业为主,农药化肥使用量较大,流域内农田污染物随降雨进入河道,是滇池湖泊富营养化的驱动力之一[14]。在雨季(2021年7月)对柴河水进行环境DNA测序及显微镜鉴定计数进行浮游藻类多样性分析。研究中,在柴河选择了柴河水库及其上下游三个采样点位(24°31′~24°37′N,102°40′~102°41′E),点位海拔(1955~1916 m),这些地点包括了位于六街镇的柴河源头和承担生活用水的柴河水库以及段七村旁的柴河主河道(图1)。
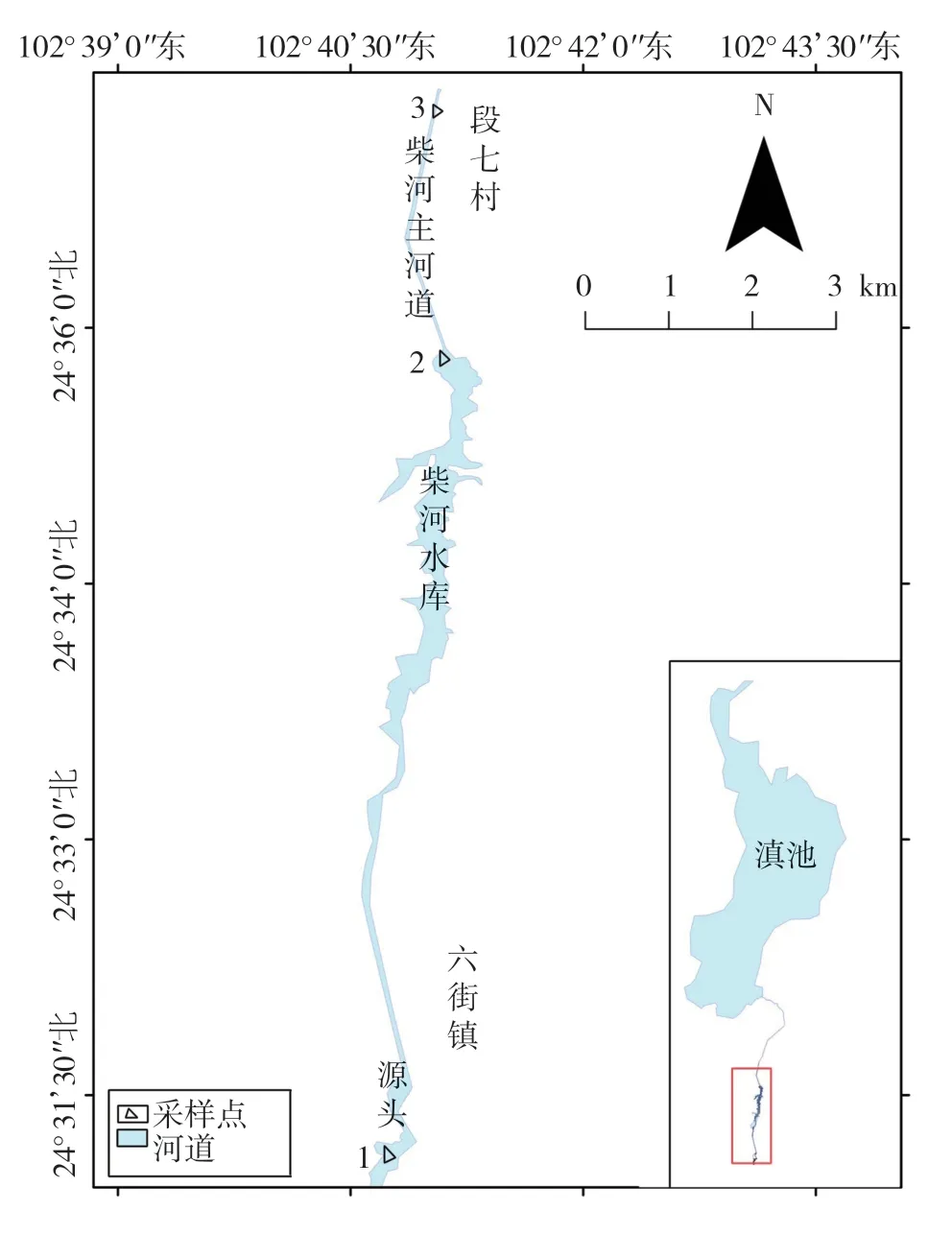
图1 柴河采样点位
eDNA方法和显微鉴定计数法在相同点位采样,两种方法在水体表层分别采集水样2 L。采样时对现场进行观察并做好采样记录。样品采集好后带回实验室,放置在4℃冰箱中保存至过滤前。过滤前将水样混匀,然后使用真空过滤泵经0.45 mm滤膜得到三组重复样品,三个重复的样本来自同一采样点。在每个采样点使用过滤相同体积去离子水的方法,建立空白对照,过滤后滤膜储存在-80℃冰箱内至分子生物学实验前。
1.2 浮游藻类多样性分析的传统方法
研究中鉴定浮游藻类使用的传统方法是显微鉴定计数法,每个采样点用采水器采集2 L水样,水样放入棕色瓶并加入30 mL鲁哥氏液固定,使用当天采集的水样获取采样点浮游藻类数据。这种方法确保了两种方法之间确定的浮游藻类数据差异不是由外在水质变化等因素造成的。在采样点采集样品时,将同一点位采集的样本混匀,最大限度提高了检测浮游植物的准确性和概率。采样后将采样瓶放入冷冻保温箱保存,并及时运回实验室,具体操作根据《SC/T 9402-2010 淡水浮游生物调查技术规范》进行[15],浮游藻类形态学鉴定由云南省生态环境监测中心专家进行鉴定。
1.3 DNA宏条形码技术监测分析方法
1.3.1 DNA提取与PCR扩增
水环境DNA提取实验在“昆明市河湖健康评估与修复院士工作站” “云南省高校河湖健康评估与修复重点实验室”进行,实验室具备完成本项目研究的仪器设备和工作条件。按照提取试剂盒的操作步骤,使用PowerWater水样DNA提取试剂盒提取DNA,但根据实际情况需稍加修改使用。每个采样点水样摇匀后取500 mL经0.45 μm滤膜过滤,将滤膜放入到研磨管中,在SP buffer和Lysis S Buffer作用下,通过剧烈震荡,基因组DNA被释放出来。通过离心,去除大部分杂质。加入沉淀腐殖酸、蛋白质等杂质的DA buffer,然后加入有适当盐分及pH值的Bingding Buffer,DNA被特异性吸附在Biospin离心柱膜上。通过洗涤,将金属离子等残留的杂质去除。最后使用Elution Buffer将DNA从滤膜上洗脱下来,获得基因组DNA。过滤的空白对照和阴性对照与样品一起提取,并与样品采用相同的方案。经提取的环境DNA使用超微量紫外分光光度计测定DNA浓度,并同时在1%琼脂糖凝胶中检测。使用超微量紫外分光光度计测定过滤空白和阴性对照的环境DNA浓度,空白和阴性对照结果应显示为空白及阴性。
使用引物序列为“TCCCTGCCHTTTGTACAC AC”的正向引物,序列为“CCTTCYGCAGGTTC ACCTAC”的反向引物扩增真核浮游藻类的18S rRNA的V9区。采用序列为“ACCTACGGGRSGC WGCAG”的正向引物,序列为 “TTACCGCGGC KGCTGG”的反向引物扩增原核浮游藻类的基因,以识别原核浮游藻类种类,该引物针对核糖体小亚基16S rRNA的V3区[16-17]。DNA扩增再在Applied Biosystems VeritiPro PCR平台进行,对每个样本做三次PCR重复。PCR反应体系为 30 μL,包括15 μL 2×Rapid Taq Master Mix, 12 μL ddH2O,1.5 μL正向引物,1.5 μL反向引物以及1 μL的DNA模板(环境样品、过滤空白和阴性对照)。对于所有样本反应程序如下:95℃预变性3 min,然后对反应进行33个循环(95℃保持 15 s,58℃保持15 s,72℃保持3 s)最后72℃保持5 min以达到彻底延伸。PCR扩增后,在2%的琼脂糖凝胶中检测PCR产物,并保证过滤空白和阴性对照都没有扩增迹象。
1.3.2 文库构建及高通量测序
使用胶回收试剂盒切胶回收PCR产物,并用TE缓冲液洗脱回收目标DNA片段。使用IIIumina公司TruSeq DNA PCR-Free Sample Prep Kit(FC-121-30001/3003)。
测序采用PE300测序方式,测序试剂盒使用IIIumina公司MiSeq Reagent Kit v3(600 Cycle)。
1.3.3 生物信息学分析
使用QIIME25软件完成生物信息操作。为了获得高质量数据,对测序得到的原始数据进行序列拼接,并根据Barcode区分样品,初步筛选过滤,消除低质量数据,使用基于OTU的方法构建特征表,并去除低频率OTU以降低假阳性结果[18]。将OTU序列通过国家生物技术信息中心(NCBI)GenBank数据库下载的数据库进行比对,如果序列存在,则认为属于一个物种。
1.3.4 统计分析
计算eDNA的生物多样性指数时,使用R语言的R studio操作界面加载vegan包后进行计算[19]。测序数据与显微镜直接计数的数据一起输入EXCEL软件。将物种序列数丰富度数据与显微镜直接计数法数据分别计算,后将数据进行比较,物种的相对丰度根据OTU丰度表和物种计数表计算。使用SPSS19软件计算以确定样本组之间存在的差异[20]。平行间交叉情况计算公式为[7]:

1.3.5 基于浮游植物多样性指数的水质生物学评估
Shannon-Wiener多样性指数H'在0~1代表重污染,H'在1~2代表中污染,H'在2~3代表轻污染,H'>3代表清洁。[21]
Pielou均匀度指数J在0~0.3代表重污染,J在0.3~0.5代表中污染,J>0.5代表轻污染或无污染[21]。
2 结果分析
2.1 柴河浮游藻类多样性监测
2.1.1 柴河浮游藻类门水平群落组成
用显微鉴定计数法对3个点位采集的水样进行藻类鉴定分析,鉴定了6门16目18科24属的浮游藻类(表1)。在显微鉴定计数法观察到的浮游植物中,以细胞数区分优势门类,优势门中,蓝藻门(Cyanophyta)占95.35%、硅藻门(Bacillariophyta)占1.4%、隐藻门(Cryptophyta)占1.13%、绿藻门(Chlorophyta)占1.06%(图2a)。在显微鉴定计数法发现的24属浮游藻类中,蓝藻门假鱼腥藻属(Pseudanabaena)占76.84%、蓝纤藻属(Dactylococcopsis)占9.36%、尖头藻属(Raphidiopsis)占4.06%。
通过eDNA宏条形码技术在3个点位鉴定出了来自9门25目33科43属的浮游藻类(表1)。在eDNA高通量测序得到的有关浮游藻类的124个OTU中,最具代表性的门类,蓝藻门(Cyanophyta)占31.39%、绿藻门(Chlorophyta)占23.81%、黄藻门(Xanthophyta)占21.06%、硅藻门(Bacillariophyta)占8.69%(图2b)。环境DNA高通量测序平均每个点位发现浮游藻类32属,监测到丰度最高的藻类同样是蓝藻门假鱼腥藻属。从发生频率来看,前三位中,蓝藻门假鱼腥藻属(Pseudanabaena)占22.68%、黄藻门周泡藻属(Vacuolaria)占21.06%、绿藻门肾形藻属(Nephroselmis)占20.05%。
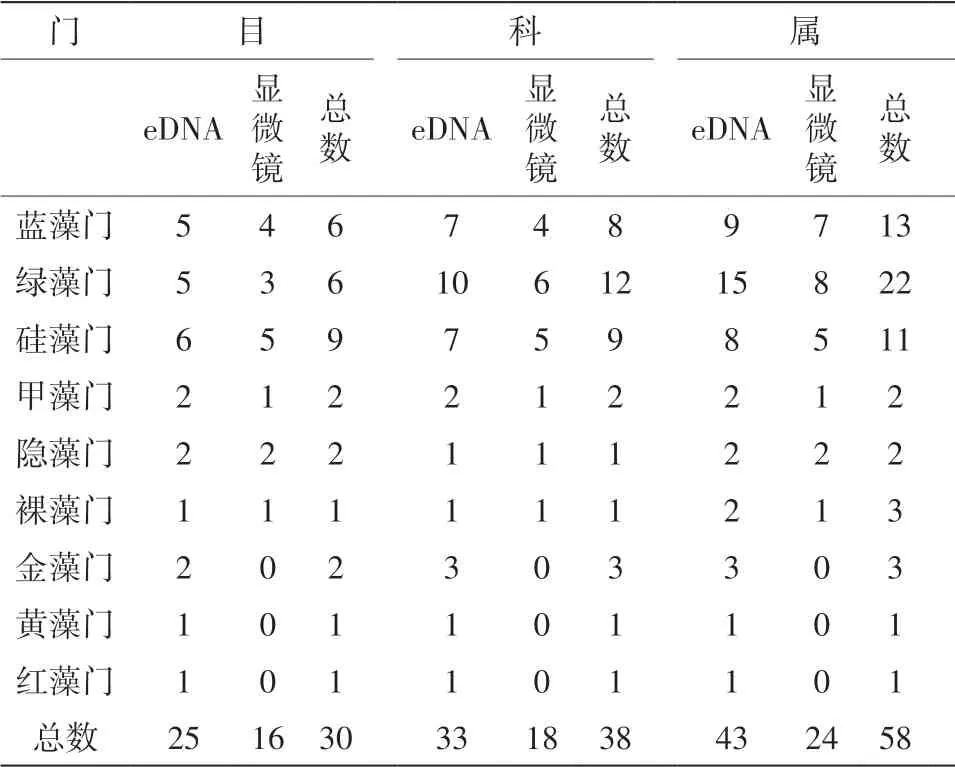
表1 eDNA宏条形码技术和显微鉴定计数法在柴河检测到的浮游藻类类群数量
结合显微鉴定计数法和eDNA高通量测序数据显示优势门为:蓝藻门(Cyanophyta)占63.37%、绿藻门(Chlorophyta)占12.44%、黄藻门(Xanthophyta)占10.53%、硅藻门(Bacillariophyta)占5.03%。通过这两种方法共同发现了蓝藻门(Cyanophyta)、绿藻门(Chlorophyta)、硅藻门(Bacillariophyta)、甲藻门(Dinophyta)、隐藻门(Cryptophyta)、裸藻门(Euglenophyta)(图2a、2b、2c)。
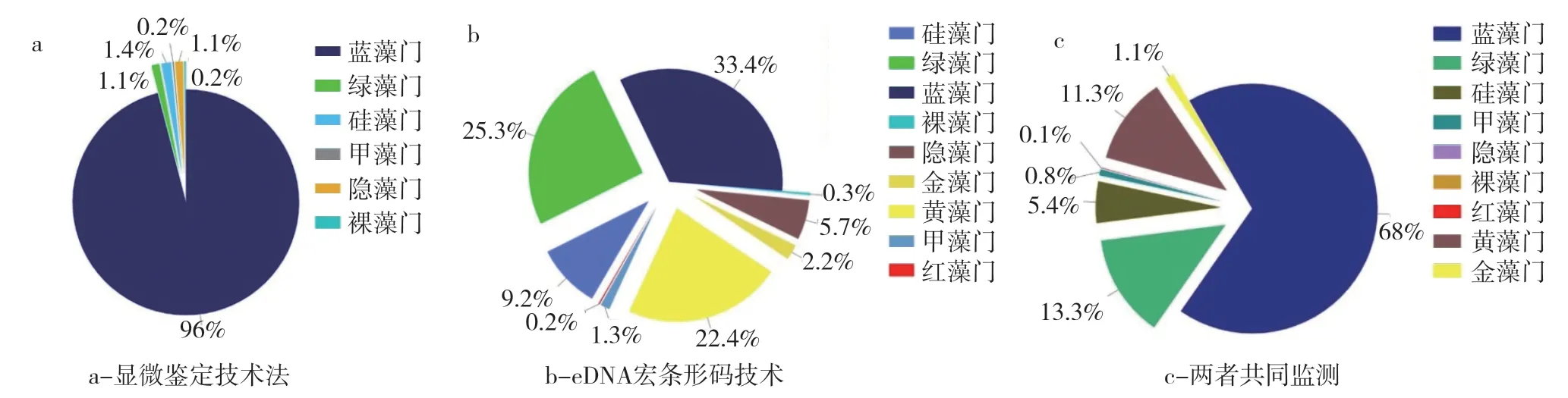
图2 两种方法监测到的柴河浮游植物门类组成
2.1.2 柴河浮游藻类属水平分布
通过eDNA宏条形码技术监测结果可以发现,实验选取的3个采样点水平浮游藻类相对丰度差异很大。点位一位于人类活动相对较少的六街镇杨树种植区,样品中衣藻属(Chlamydomonas)相对丰度最高,其次是隐藻属(Cryptomonas)、菱形藻属(Nitzschia)、钟罩藻属(Dinobryon)。点位二地处柴河水库,样品中假鱼腥藻属相对丰度最高,其次是周泡藻属(Vacuolaria)、微囊藻属(Microcystis)。点位三位于段七村,样品中菱形藻属(Nitzschia)、刚毛藻属、假鱼腥藻属是优势藻属(图4)。
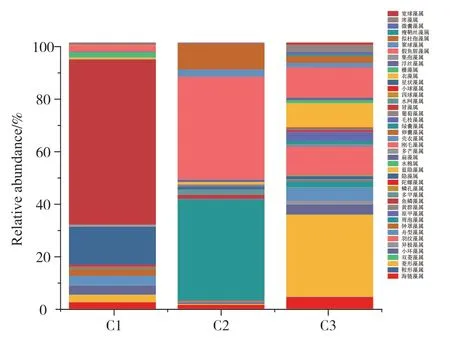
图4 eDNA宏条形码各位点相对丰度
显微镜直接计数法监测到点位一水样中隐藻属、小环藻藻属(Cyclotella)、栅藻属(Scenedesmus)为优势藻属。点位二中假鱼腥藻属、尖头藻属(Raphidiopsis)是优势藻属。点位三优势藻门为硅藻门,但相应藻属未辨别出名称(图3)。
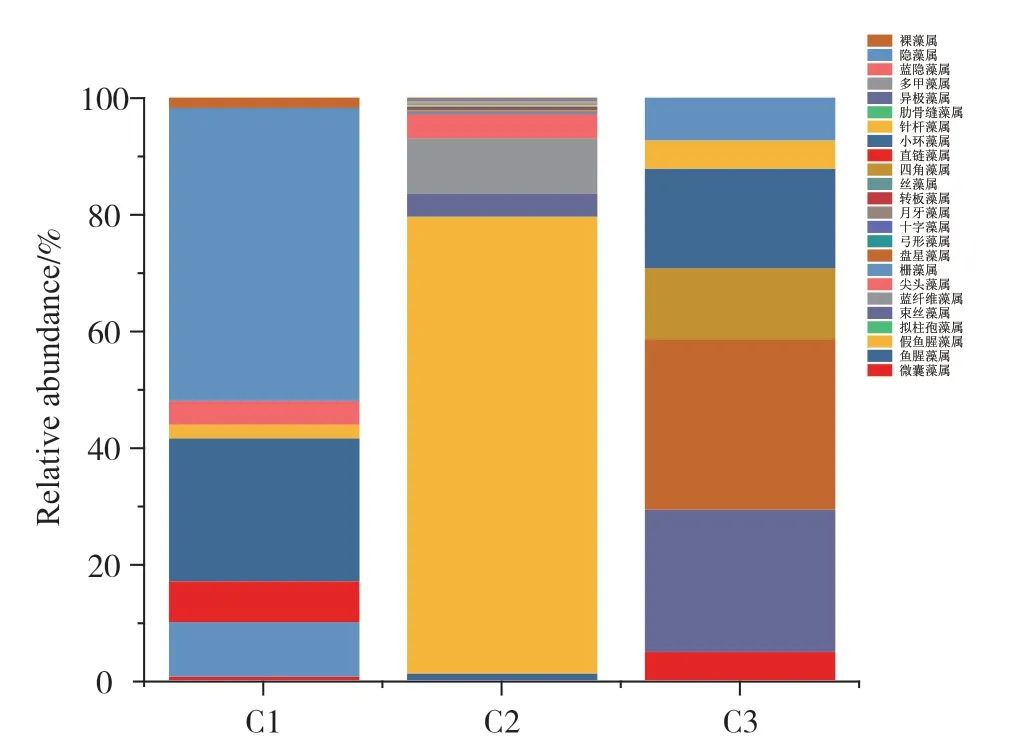
图3 显微鉴定计数法各位点相对丰度
2.2 环境DNA宏条形码监测的精准性
2.2.1 平行样本间监测情况
eDNA高通量测序从9个送检样本中共检出真核浮游藻类13051条18srDNA序列,原核浮游藻类7689条16srDNA序列。最终有124个OUT被注释到43属浮游藻类上,涵盖了蓝藻门、绿藻门、硅藻门、甲藻门、隐藻门、裸藻门、金藻门、红藻门、黄藻门等主要浮游藻类类群。在准确性方面,检测到目标类群的平行样本中,3个平行样品的交叉率达到34.96%,至少出现在两个平行样品的属所占比例为55.01%(表2)。

表2 eDNA宏条形码监测数据的精确性评价结果 (%)
2.2.2 环境DNA宏条形码技术与传统方法的交叉情况
检测出的浮游藻类中有9个属是由eDNA和显微镜直接观察法共同检测出来的,有34属和15属分别由eDNA和显微镜直接观察法单独检测出。通过两种方法共同检测到的科有色球藻科(Chroococcaceae)、念珠藻科(Nostocaceae)、假鱼腥藻科(Pseudanabaenaceae)、栅藻科(Sc-enedesmaceae)、水网藻科(Hydrodictyaceae)、小球藻科(Chlorellaceae)、双星藻科(Zygnemataceae)、圆筛藻科(Cosscinodiscaceae)、舟形藻科(Naviculaceae)、异极藻科(Gomphonemaceae)、多甲藻科(Peridiniaceae)、隐鞭藻科(Cryptomonadaceae)、裸藻科,占显微鉴定计数法的72.2%(图5a)。共同检测到的属有假鱼腥藻属、微囊藻属、席藻属、栅藻属,小环藻属、异极藻属、多甲藻属、隐藻属、裸藻属,占显微鉴定计数法的37.5%(图5b)。
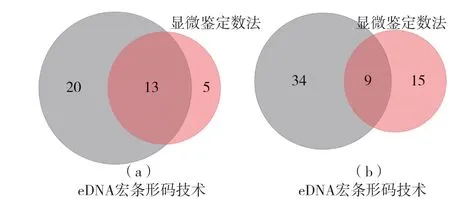
图5 通过显微鉴定计数法与eDNA宏条形码技术监测的浮游藻类科(a)、属(b)交叉情况
2.3 柴河各位点浮游藻类多样性分析
α多样性指数(Alpha Diversity)常用于衡量样品内的生物群落多样性。其中Pielou均匀度指数与物种累积数越大,表明样本的生物种类越多,丰富度越高;Shannon指数综合考虑物种数及物种频度分布的均匀性,其值越高,说明群落多样性越高,物种分布越均匀。基于18S V9及16S V3引物的宏条形码测序数据所注释的OTUs及其序列数,分析使用R语言的R studio操作界面加载vegan包后进行计算,得到各样品中的生物群落多样性指数见表2。C3点位样品的Shannon指数、Pielou均匀度指数都显著高于C1和C2点位样品,C3点位生物多样性显著高于C1和C2。C2点位样品物种累计数更高,说明点位三群落均匀度更高,物种相较于一、二点位更丰富(表3)。生物多样性指数结果与起初设想的景观差异有些许出入,但与采样时所观察的水质理化因素密切相关。
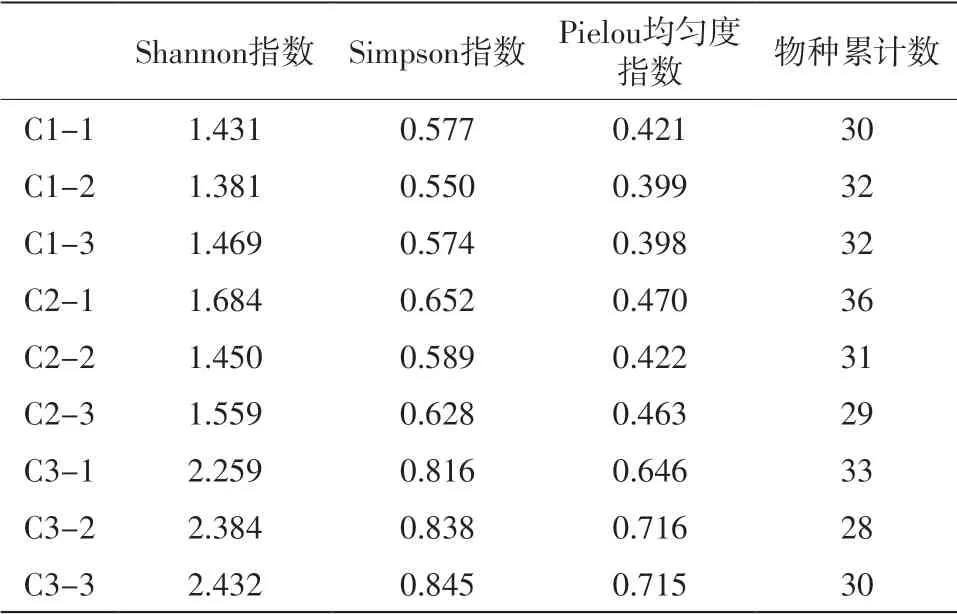
表3 Alpha多样性指数统计
2.4 基于浮游植物多样性指数的水质生物学评价
通过常用的浮游藻类评价水体水质的方法[21],根据分子生物学方法得到的浮游植物多样性指数,柴河Shannon-Wiener多样性指数(H')对源头评价为中污染,柴河水库为中污染,柴河主河道为轻污染,Pielou均匀度指数(J)对源头评价为中污染,柴河水库为中污染,柴河主河道为轻污染(表4)。

表4 柴河水质生物学评价结果
3 讨论
3.1 环境DNA宏条形码技术表征柴河浮游藻类多样性
基于环境DNA宏条形码技术在柴河共鉴定出9门25目33科43属浮游藻类单元。水库上游优势藻属为衣藻属,水库段为假鱼腥藻属,水库下游为菱形藻属。这一监测结果与显微鉴定计数法接近,eDNA宏条形码技术在科、属水平鉴定的浮游植物种类分别占显微鉴定计数法的72.2%,37.5%。资料显示柴河历史共出现67属浮游藻类[22],eDNA宏条形码监测技术方法与历史记录的浮游植物重合度高,极大程度地贴合了柴河浮游藻类的状态。对水生生物群落结构沿城市化发展进程的响应做了传统方法以及eDNA方法监测,发现eDNA方法比传统方法更加准确,且eDNA方法与水质关系更紧密[23]。运用分子生物学方法可以得到描述群落多样性的物种丰富度和均匀度指数,并将其运用于水质生物学评估[24]。
3.2 柴河不同点位浮游藻类生物多样性差异
水库上游浮游藻类Shannon-Wiener指数为1.426,Pielou均匀度指数为0.406,生物学水质评价结果为中污染。结合现场采样时对取样点周边环境记录观察发现,该采样点为天然河段,采样点周边没有明显污染源,河水浑浊,水体流速快,河道中几乎没有沉水植物,对水质净化能力差。柴河水库浮游藻类Shannon-Wiener指数为1.564,Pielou均匀度指数为0.452,水库内假鱼腥藻属占绝对优势,生物学水质评价结果为中污染。水库下游位于段七村旁主河道,浮游藻类Shannon-Wiener指数为2.358,Pielou均匀度指数为0.692,水质生物学评价结果为轻污染。采样时水库分水闸没有放水,水库水对点位三水样影响不大,且河道内有大量沉水植物,起到了净化水质的作用,是水库下游浮游藻类生物多样性优于前两个点位的原因。
4 结论与展望
环境DNA宏条形码技术在柴河浮游藻类监测物种识别方面与显微鉴定计数法具有一致性。两种方法识别出的优势藻属相同。基于环境DNA宏条形码技术的浮游藻类监测可反映不同河段水质差异。作为一种新方法,eDNA宏条形码技术可监测评估柴河浮游藻类多样性,为柴河水生态健康评估和保护提供依据。
未来,还要进一步完善统一环境DNA的操作流程,并完善本土物种DNA条形码数据库,使测序数据的可重复性、准确性提升。环境DNA宏条形码技术在监测浮游藻类多样性及其相关工作具有较高可靠性,有望提升流域内水生藻类监测工作的水平。
