鲢微卫星标记与生长性状的相关分析
2022-11-23罗相忠李晓晖梁宏伟邹桂伟
罗相忠,时 乐,沙 航,李晓晖,梁宏伟,邹桂伟,崔 峰
(1.中国水产科学研究院长江水产研究所,武汉 430223;2.安徽科技学院生命与健康科学学院,安徽凤阳 233100)
微卫星DNA是广泛存在于真核生物和原核生物基因组中的1-6个简单重复序列[1]。微卫星标记在基因组中不同区域分布差异较大,大多数富集在非编码区[2]。由于微卫星遗传标记具有高度多态性、信息含量丰富、共显性等优势,被广泛用于生物物种数量性状QTL定位、遗传连锁图谱构建和DNA指纹分析[3]。微卫星标记已经发展成为水产动物育种领域中的常用分子标记之一。
微卫星标记已经在众多水产动物中开展了与生长等性状的关联分析,取得了良好的结果。如李燕等[4]通过分析15个微卫星标记与96尾杂交鳜(Sinipercachuatsi×S.scherzeri)的体长、体质量、体高性状的相关性,获得cD90、MDJ821、PY21、PY55和72217标记分别与上述3种生长性状显著相关。余成晨等[5]研究选育草鱼(Ctenopharyngodonidella)群体生长性状与微卫星标记的关系,发现CID1512、CID973_1 和CID254_1 在雌性或雄性个体中与体质量和体长等生长性状显著相关,而CID391_2 仅在雄性个体中显著相关。刘士力等[6]对翘嘴鲌(Erythroculterilishaeformis)的2个生长激素受体GHR1 和GHR2基因的微卫星序列进行检测,发现GHR2基因中的4个微卫星位点均与体长和体质量等生长性状相关。这些为开展鲢(Hypophthalmichthysmolitrix)微卫星标记遗传多样性与生长性状的相关分析提供了借鉴。
鲢属于鲤形目鲤科鲢亚科鲢属,是我国重要养殖鱼类,为淡水养殖四大家鱼之一,其营养丰富,为人类的动物蛋白来源之一[7]。同时其以水体中浮游植物为主要饵料,属于淡水鱼类中为数不多的不投饵的滤食性鱼类,为典型的碳汇渔业对象,具有重要的生态价值[8]。鲢食物链短,易于养殖,深受养殖者和大众消费者青睐,在我国养殖产量仅次于草鱼。长丰鲢(H.molitrix)是中国水产科学研究院长江水产研究所利用综合育种技术培育的鲢新品种,具有生长速度快、体型好和适应性强等优点[9]。基于研究鲢的遗传多样性以及筛选与其生长性状相关的微卫星标记,本研究以湘江鲢(H.molitrix)和新品种长丰鲢作为研究对象,旨在筛选与鲢生长性状相关的微卫星分子标记,为今后分子标记辅助育种提供技术支撑。
1 材料和方法
1.1 样本采集
实验用2龄长丰鲢和2龄湘江鲢均采自于农业农村部鲢遗传育种中心,其中长丰鲢60尾,湘江鲢120尾。采集其胸鳍组织,置于无水乙醇的离心管中,带回实验室于-20 ℃冰箱中保存。
1.2 生长性状测定
对长丰鲢和湘江鲢依次进行框架数据X1、X2、X3、X4、X5、X6、X7、X8的测量(图1和表1)和全长(X9)、体长(X10)、头长(X11)、体高(X12)、尾柄高(X13)、眼径长(X14)、尾柄长(X15)、躯干长(X16)的测量,测量工具分别为直尺和游标卡尺。体质量(X17)使用电子天平测定。共计17个可量性状。采用SPSS 22.0软件(由美国SPSS公司开发)进行单个样本的Kolmogorov-Smirnov(K-S),检验所取样本的生长性状是否符合正态分布(P>0.05)。

图1 鲢框架结构示意图

表1 鲢形态测量表
1.3 基因组DNA的提取
鳍条经超纯水漂洗后,剪取少量鳍条组织,采用高盐法提取样本的基因组DNA。提取后,将DNA溶液经1%琼脂糖凝胶电泳和紫外凝胶成像系统检测后,加入超纯水稀释至50 ng/μL,-20 ℃冰箱中保存备用。
1.4 微卫星引物的筛选
基于本实验室鲢基因组数据(未发表),采用Primer5.0(加拿大Primer生物公司)设计96对微卫星引物,其序列和退火温度见表2,引物由武汉天一辉远生物科技有限公司合成。选用湘江鲢和长丰鲢各3尾对所有引物进行初步筛选,用1.0%的琼脂糖凝胶电泳进行检测后毛细管电泳,进而开发多态微卫星标记。

表2 鲢13个微卫星引物序列信息
1.5 PCR扩增和条带的检测
对基因组DNA进行PCR扩增,总反应体系为10 μL,其中2×Taq PCR Master Mix 5 μL,基因组DNA 1 μL,上下游引物各0.5 μL,ddH2O 3 μL。PCR扩增程序为:95 ℃预变性5 min;95 ℃变性,62℃退火30 s,72 ℃延伸30 s,以后每个循环下降1 ℃,直至52 ℃,共10个循环;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,共25个循环;72 ℃延伸20 min。扩增的PCR产物在ABI-3137XL基因分析仪(美国ABI生物公司)上利用毛细管电泳法(CE)检测。每个CE样品中含有1 μL PCR产物、0.4 μL Genescan-500分子质量标准品和20 μL去离子甲酰胺(美国ABI生物公司)。在毛细管电泳过程中,PCR产物与Genescan-500分子质量标准品的荧光信号均由基因分析仪自动保存。
1.6 数据统计及分析
利用SPSS 22.0软件对湘江鲢和长丰鲢两个群体的生长性状进行数据整理和正态分布的检验。利用Cervus 3.0软件(Cervus公司)统计两群体不同位点的等位基因数(k)、期望杂合度(He)、观测杂合度(Ho)和多态信息含量(PIC)。利用SPSS 22.0软件的线性回归对两个鲢群体的不同微卫星位点与生长性状进行关联分析,初步确定与生长性状存在显著差异的位点,再用一般线性模型(GLM)对初步确定的位点进行单变量的观测平均值的多重比较,确定同一微卫星位点不同基因型间是否存在显著差异,P<0.05为差异显著。为了排除偶然性,在利用SPSS 22.0软件分析时,每种基因型至少具有3次观察值才被统计。
2 结果与分析
2.1 长丰鲢和湘江鲢生长性状的统计
长丰鲢中体质量的变异系数最大为15.67%,湘江鲢中体质量的变异系数最大为15.68%(表3),体质量在鲢群体中的变异系数均较大,相对于其他生长性状,体质量具有更大的选择潜力。湘江鲢和长丰鲢的生长性状均服从正态分布,测量的所有性状都具有连续变异的特点,可进行后续的关联分析。
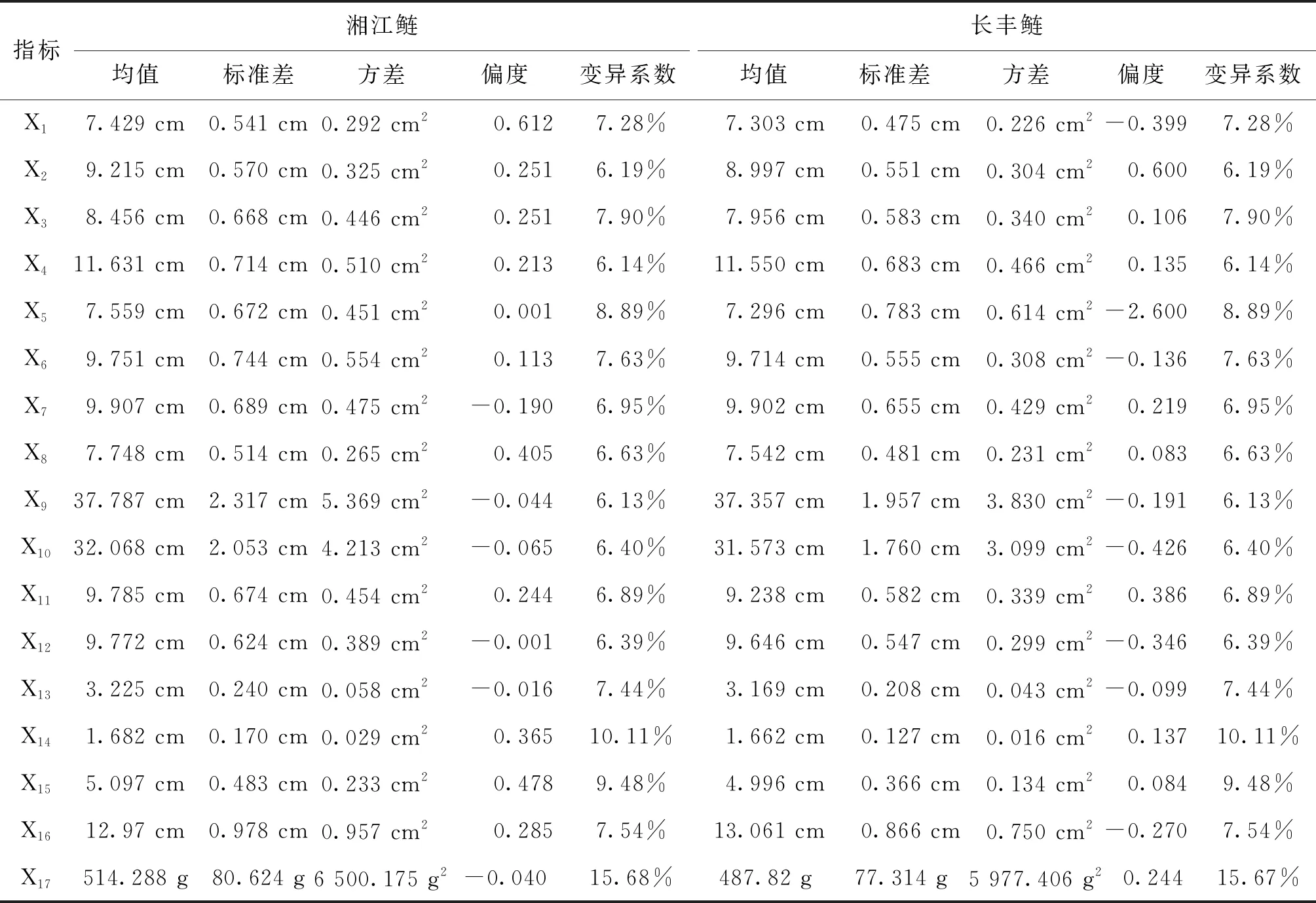
表3 长丰鲢和湘江鲢各生长性状统计
2.2 微卫星多态性标记分析
96个微卫星位点中有13个多态性位点均能在长丰鲢和湘江鲢中稳定扩增,这13个多态性位点具体分布为:位点P058位于染色体LG1上,P030、P036位于LG2上,P086位于LG5,P034位于LG9上,P021、P062位于LG12上,P056位于LG13上, P037、P043位于LG16上,P054位于LG17上,P041位于LG23上,P020位于LG24上。长丰鲢和湘江鲢两个群体的遗传多样性信息见表4。长丰鲢共检测到56个等位基因,平均k为4.31个,Ho为0.017~0.683,He为0.049~0.800,PIC范围为0.048~0.768。湘江鲢群体共检测到60个等位基因,平均k为4.62个,平均Ho为0.050~0.867,平均He为0.126~0.822,PIC为0.177~0.796。

表4 鲢生长性状有关的SSR多态位点信息
2.3 微卫星标记与鲢生长性状的相关性分析
13个微卫星位点中10个微卫星位点与生长性状显著相关,见表5。对具有关联性的9个生长性状进行不同位点的均值或多重比较。其中,筛选出湘江鲢8个与生长性状显著相关的位点(表5),长丰鲢2个显著相关位点(表6)。
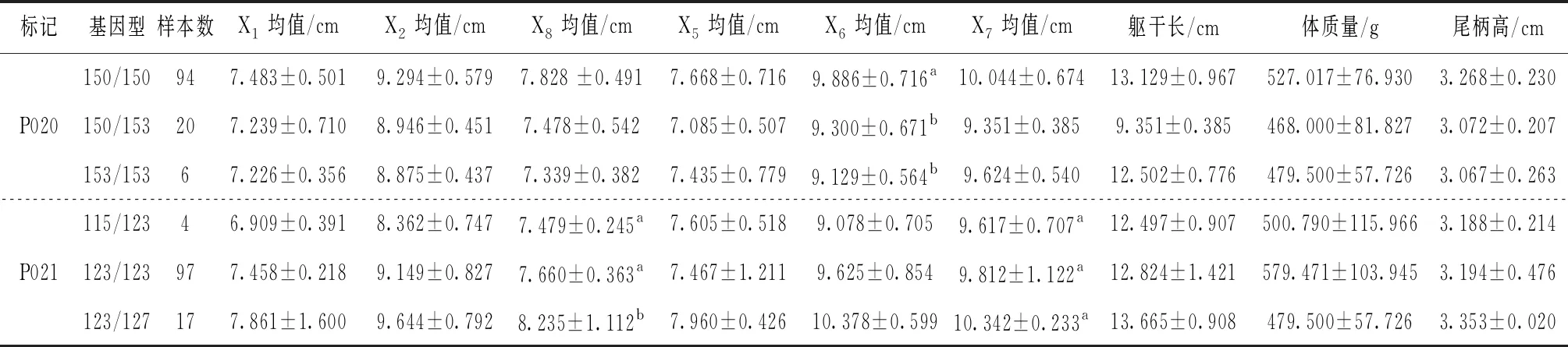
表5 湘江鲢8个微卫星位点各生长性状的多重比较
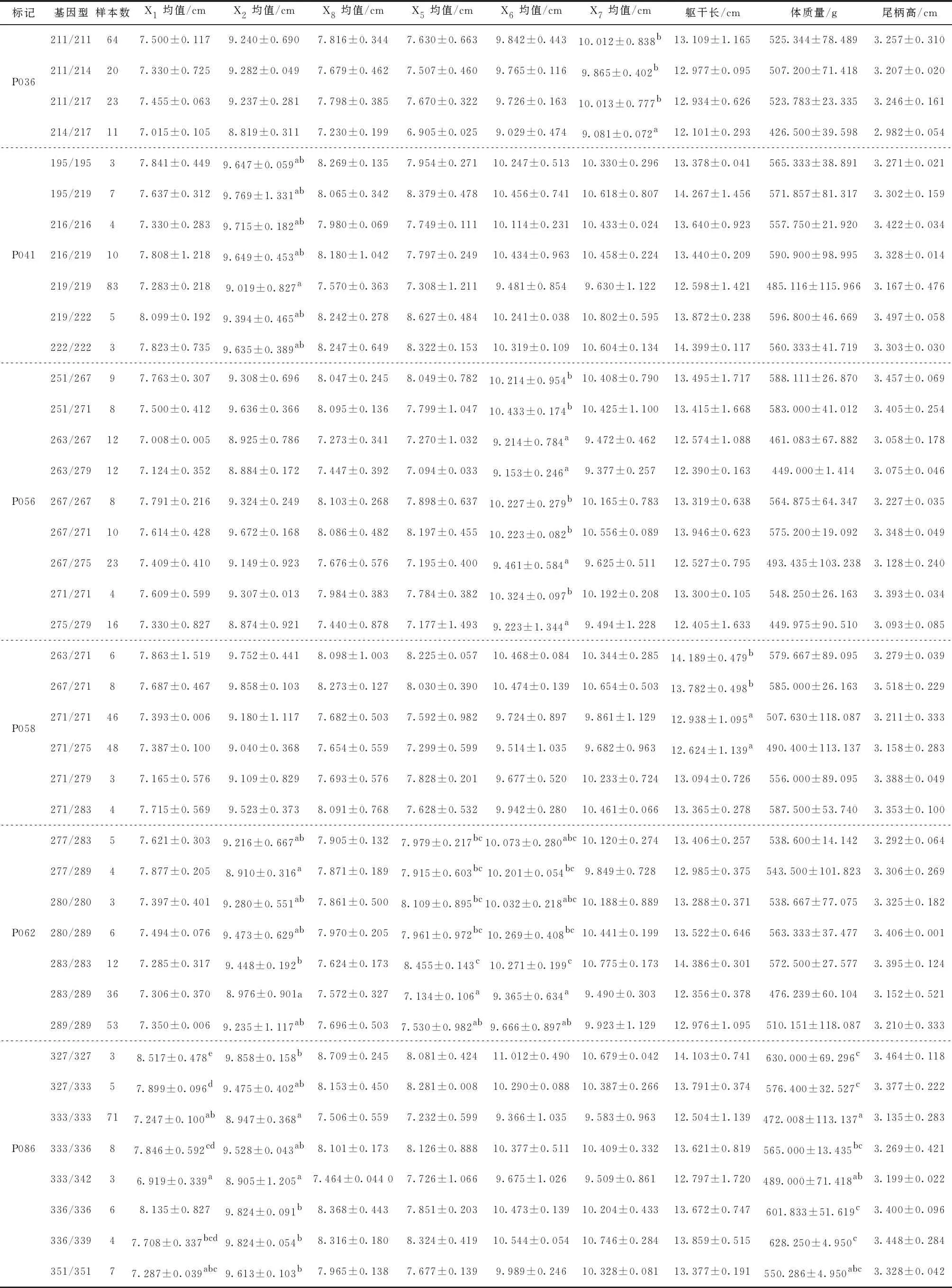
续表5

表6 长丰鲢2个微卫星位点各生长性状的多重比较
2.3.1 微卫星位点与湘江鲢生长性状的相关性分析
湘江鲢群体中,位点P020、P056、P062均与框架数据X6显著相关;位点P041、P062、P086均与框架数据X2显著相关。位点P021与框架数据X8均值显著相关。位点P058与躯干长显著相关。
对不同基因型的不同性状进行多重比较。位点P020中,150/150基因型个体各生长性状均值均高于其他基因型;个体150/150基因型的框架数据X6的均值显著高于150/153、153/153基因型,而基因型150/153与153/153个体各生长性状之间差异不显著,由此得出150/150基因型是生长性状的优势基因型。在位点P021中,基因型123/127个体各生长性状均值均优于其他两种基因型,框架数据X8上123/127基因型个体显著高于115/123、123/123基因型。位点P036,共检测到4种基因型,基因型214/217各生长性状均值均低于其他基因型,且框架数据X7显著低于211/211、211/214、211/217和217/217基因型,由此证实214/217基因型是生长性状的劣势基因型。位点P041中,基因型219/219个体各生长性状均值均低于其他基因型,且框架数据X2均值显著低于其他基因型,该基因型也被证实为生长性状的劣势基因型。在位点P058中共检测到6种基因型,基因型263/271、267/271个体的躯干长均值分别为14.189 cm和13.782 cm,显著高于基因型271/271和271/275,但基因型263/271和基因型267/271的躯干长均值没有明显差异。位点P062,283/283基因型个体的框架数据X2、X5和X6显著高于其他基因型,283/289框架数据X5显著低于基因型277/283、280/289、283/283,X6显著低于280/289、283/283基因型。位点P086中共检测到8种基因型,个体327/327基因型的框架数据X1均值显著高于基因型333/333、333/336/、333/342、336/339、351/351,高于基因型327/333和336/336但无显著差异,327/327基因型为框架数据X1的优势基因型。
2.3.2 微卫星位点与长丰鲢生长性状的相关分析
长丰鲢群体中,位点P041与尾柄高显著相关,位点P041共检测到7种基因型,基因型195/219、210/219个体的尾柄高均值分别为2.978 cm和2.928 cm,均低于基因型219/219、222/222、216/219的个体,差异显著,但基因型195/219和210/219之间无明显差异。个体222/222基因型的尾柄高均值为3.339 cm,显著高于基因型219/222、216/216、210/219的个体,高于其他基因型但无显著差异。说明基因型222/222为尾柄高的优势基因型,222 bp片段的等位基因与尾柄高呈正相关。
长丰鲢群体中,位点P086中,检测到9种基因型,基因型351/351个体的框架数据X5均值为6.080 cm,显著低于基因型333/339、333/342、336/336、327/327、318/339、333/339、333/336,低于339/339基因型但差异不明显;基因型351/351为框架数据X5的劣势基因型。
2.4 微卫星标记在鲢群体的适用性分析
2.4.1 微卫星标记P041与湘江鲢、长丰鲢生长性状的相关分析
P041位点在两群体中共检测到8种基因型,湘江鲢和长丰鲢中均检测到7种基因型,两群体共有基因型6种,分别是基因型195/291、216/216、216/219、219/219、222/222、219/222。其中,基因型222/222个体在湘江鲢、长丰鲢中的躯干长均值分别为14.399 cm和13.789 cm,均高于其群体的其他基因型,但无显著差异,说明222 bp片段的等位基因个体与躯干长呈正相关。
2.4.2 微卫星标记P086与湘江鲢、长丰鲢生长性状的相关分析
P086位点在湘江鲢中检测到8种基因型,在长丰鲢中检测到9种基因型,其中6种基因型为两群体共有:327/327、333/333、333/336、333/342、336/336、351/351。湘江鲢中333/333基因型个体的各生长性状均值均低于333/342外的其他基因型个体但无显著差异;长丰鲢中基因型333/333个体各生长性状的均值低于其他基因型,除基因型339/339、351/351外,但无显著差异,333 bp片段的等位基因与生长性状呈负相关。
3 讨论
3.1 长丰鲢与湘江鲢的遗传多样性
遗传多样性是物种多样性的基础,也是生命适应环境和进化的基础[10]。遗传杂合度是衡量群体遗传变异的参数之一,种群遗传多样性越丰富,该物种对环境的适应能力越强[11]。本实验中筛选得到的13个微卫星标记,在湘江鲢、长丰鲢中的平均观测杂合度分别为0.413、0.392;在湘江鲢、长丰鲢中的平均期望杂合度分别为0.467、0.484。两群体的杂合度都处于中度水平,遗传多样性处于中等多态水平。用于基因变异程度高低的多态信息含量(PIC)>0.5,则该位点是高度多态的;0.25 分子标记辅助育种,就是通过与基因连锁的分子标记,直接筛选目标区域的DNA[13]。其可以避免环境的影响,提高选择的准确性,因而广泛运用于许多水产生物的遗传连锁图谱构建、性状关联分析和改良品种的辅助育种等研究。湘江鲢群体中,位点P020、P056、P062均与框架数据X6显著相关(P<0.05);位点P041、P062、P086均与框架数据X2显著相关,这些位点中存在着“多因一效”的现象。同时发现,一个标记位点也可能与多个性状密切相关,位点P062与湘江鲢的框架数据X2、X5、X6显著相关;位点P086与湘江鲢的框架数据X1、X2和体质量显著相关,位点中存在着“一因多效”的现象,在对其他一些水产动物群体的遗传多样性研究也发现有类似的现象[14-15]。 近年来,分子标记与性状间的关联分析已经成为当前水产动物遗传育种的研究热点之一。不同位点的基因型与性状的关联分析,可直接通过对基因型和表型变异程度的差异分析,确定标记与数量性状之间的相关性,得到数量性状与一个或多个标记的遗传相关,从而筛选出控制数量性状的基因型[16]。但是,目前国内外文献有关鲢群体的研究主要集中在人工繁育和遗传多样性[9,17-18],有限的研究报道了有关鲢基因组上的QTL分析[19-20]。WANG等[21]对鲢的生长性状与微卫星标记的研究,鉴定了20个生长性状QTL相关标记,发现两个标记ref-16553和ref-46998_8均与体重体长等多个生长性状显著相关。本研究中标记P041在湘江鲢和长丰鲢两个群体中均存在222 bp片段的等位基因,且该位点在湘江鲢、长丰鲢中分别与框架数据X2、尾柄高有显著相关性。同一标记在长丰鲢和湘江鲢上关联的生长性状不同,可能是由于长丰鲢和普通鲢的形态略有不同[22],显著相关的位点在两群体中所表现的性状有所差异。两群体在标记P086上的333/333基因型个体各生长性状均值相对于其他基因型普遍偏低,该基因型在鲢群体中可能对生长性状有负面影响。由于样品量有限,本研究得到的结果尚需大群体样本的进一步验证。这些位点的某些基因型具有显著的生长优势,这些基因型可在未来的选育工作中有意识的富集[23],从而用于后续的分子标记辅助育种工作。 在水产生物育种研究中,关于鲢群体的生长性状与标记辅助育种的基因的连锁分析报道很少,本研究获得的与生长性状相关的标记能否在湘江鲢和长丰鲢的标记辅助选择实践中得到应用,仍需进一步深入研究与验证,以便达到其能合理地应用该标记指导育种实践,筛选具有优势生长性状的良种,提高育种速度,改良物种重要经济性状的目标。3.2 分子标记与性状之间的关联效果
