基于APE1蛋白质的小分子抑制剂的虚拟筛选
2022-10-26陈柯瑾徐梓涵殷瑜华祁艳艳吴海燕王芳李干鹏
陈柯瑾,徐梓涵,殷瑜华,祁艳艳,吴海燕,王芳,李干鹏
(云南民族大学 民族医药学院,云南 昆明 650500)
脱嘌呤脱嘧啶核酸内切酶1/氧化还原因子1(apurinic/apyrimidinic endonuclease1/redox factor1,APE1/Ref-1,简称APE1)具有DNA修复,氧化还原,参与RNA代谢等活性,同时它是氧化应激引起细胞反应的主要调控蛋白,在细胞DNA损伤修复,细胞增殖,凋亡和放化疗效应等多个细胞生物学过程中发挥重要的作用[1].在由不同因素造成的DNA损伤中,AP位点的形成是其中最常见的损伤之一,AP位点的形成阻碍了DNA的复制,导致了基因突变和遗传的不稳定性,具有很强的诱变性和细胞毒性[2].AP位点主要由碱基切除进行修复(base excise repair,BER),而这一修复过程由APE1启动.APE1作为AP核酸内切酶是BER主要限速酶之一,它的碱基切除修复活性能保护细胞免受外源性和内源性不断积聚的AP位点造成的细胞毒性,维持基因组的稳定[3].
越来越多的研究表明APE1在多种恶性肿瘤组织中显著高表达并具有异位性,临床研究表明肿瘤组织中的APE1高表达和异位表达与患者预后不良密切相关,因此APE1被认为是恶性肿瘤的重要标志物之一[4].而目前大多数晚期恶性肿瘤的治疗仍然是基于传统放化疗的综合治疗模式.然而,最初对于放化疗有效的肿瘤细胞最终会因治疗抵抗而导致肿瘤进展,这是肿瘤治疗的瓶颈和难点.DNA损伤是放化疗致伤肿瘤细胞的主要机制,而肿瘤细胞中有完善的DNA修复机制,可以快速修复放化疗所致的DNA损伤,产生放化疗抵抗[5].因此,DNA修复基因作为抗肿瘤的新靶点是目前研究的热点.经过前期大量的筛选和验证工作,目前已经有针对APE1不同活性的小分子抑制剂,包括针对其DNA修复活性的CRT0044876、InhibitorⅢ等[6-7],以及针对其转录调控活性的小分子抑制剂E3330[8-9].
自2009年以来,关于APE1新型抑制剂的报道逐渐增多,但大部分工作集中于基于RNAi技术,抑制作用仅位于基因转录层面,无法对实际生物功能单位——蛋白来进行调控.部分新的工作基于已知的X线晶体结构(1BIX)来判断APE1与其DNA底物之间可能的相互作用,并使用基于结构的药物设计工具来进行蛋白质-配体分子对接,进而对小分子药物进行虚拟筛选.然而,高精度的分子对接模拟往往需要大量人工调试,并且计算复杂度大,对计算资源要求高.快速的对接模拟则容易出现偏差,降低后期药理评估的成功率,带来不必要的实验成本.得益于深度学习技术在生物医学领域取得的最新进展,基于深度学习的蛋白质-配体结合亲和力预测技术来实现快速,高精准度的药物筛选逐渐成为可能.
为了对APE1进行基于结构的大规模的虚拟药物筛选,本论文拟从以下几方面进行研究:前期针对大规模小分子数据库(specs)运用基于深度学习的蛋白质-配体结合亲和力预测技术分别针对APE1DNA损伤修复功能域药物活性位点,对该库中的210 070个化合物与APE1结合能力进行快速虚拟筛选,获得少数对APE1由潜在抑制活性的候选小分子抑制物;运用高精度的分子对接模拟软件对候选的小分子抑制物进行2次筛选;最终获得1~10种能够与APE1活性区域特异性结合的小分子化合物,为进一步的针对目标化合物的药理研究和应用提供参考和指导.
1 材料与方法
1.1 虚拟筛选软件及材料
本研究使用的软件有AutodockVina,PyMol,GOLD,DiscoveryStudio 2019.APE1的蛋白质晶体结构PDB文件来自Protein Data Bank(https://www.rcsb.org/),小分子化合物及其结构来自Specs数据库(https://www.specs.net/).
1.2 药品试剂及仪器
筛选小分子化合物单体由荷兰Specs公司购入,化合物编号由Specs公司自主编录(ID Number);DMEM高糖培养基,CORNING公司;0.25% 胰酶-EDTA和胎牛血清(fetal bovine serum,FBS),Gibco公司;细胞增殖及细胞毒性检测试剂盒(cell counting kit-8,CCK-8),北京索莱宝科技有限公司;Annexin V-FITC/PI Apoptosis Detection Kit细胞凋亡试剂检测盒,YEASEN公司.MultiskanGO酶标仪(美国Thermo Fisher公司); SC-04 低速离心机(ZONKIA 中佳公司);CytoFELX流式细胞仪(Beckman Coulter公司)
1.3 蛋白质模型和配体处理及优化
APE1是由2个结构域组成的球形α/β蛋白(结构域1:残基44~136和295~318;结构域2:137~260和282~294),整体尺寸为 4 nm.本虚拟筛选建立在1BIX的三维结构的基础上,APE1同源二聚体的射线晶体衍射结构(PDB ID:1BIX)是目前已报道的APE1晶体结构中分辨率最高的,该结构的分辨率为 0.22 nm,包含了275个氨基酸残基[10].从PDB数据库中下载1BIX蛋白质晶体的三维结构之后应用Discovery studio软件的Protein Preparation Wizard模块对蛋白结构进行预处理.处理过程包括在1BIX晶体结构中加入氢原子,删除水分子,删除杂基团,进行氢键网络系统优化.
APE1抑制剂的虚拟筛选是针对小分子数据库(SPECS)进行的,该数据库包含 210 070 个化合物分子.配体处理用Minimize Ligands功能完成,使用 Small Molecules|Prepare or Filter Ligands|Prepare Ligands对小分子进行预处理,并使用 Minimize Ligands进行结构优化;包括3D结构转换,加氢,中和带电基团,产生生理条件下的离子化状态,产生异构体等.
1.4 确定APE1对接位点和活性参数
根据现有研究表明,APE1的修复活性位点位于其晶体结构α/β夹层的顶部,并由环区域(loop regions)围绕,活性位点与APE1修复抑制剂CRT0044876结合较为关键的氨基酸有Asn68, Asp70, Glu96, Arg177, Asn212, Asn229, Phe266, Trp280, Leu282, Asp308, His309等[11].
通过Receptor-Ligand Interactions | Define and EditBinding Site | From Current Selection 对蛋白质进行模型构建,同时根据APE1修复活性位点的关键氨基酸残基信息得到活性位点,以 Asn68, Asp70, Glu96, Arg177, Asn212, Asn229, Phe266, Trp280, Leu282, Asp308, His309 为基础找到结合位点,结合位点坐标(XYZ,26.953,11.238,22.062),半径 14.98.
1.5 基于深度学习算法的高通量筛选
本研究首先采用基于深度学习算法对蛋白质-配体结合亲和力进行预测,并筛选出亲和力较强的 10 000 个备选小分子.选定PDBBind v2016 core set数据集,以9∶1比例将所有复合体分为训练集和测试集,对深度学习模型进行训练.如图2为基于深度学习的蛋白质-配体结合亲和力预测模型.利用深度学习模型预测蛋白质-配体结合亲和力的工作流程如下:首先使用一键编码和原子特征嵌入每个原子,然后使用图注意力神经网络将活性位点和配体的结构信息编码到它们相应的原子嵌入中. 然后通过双向变换器传递配体原子嵌入和活性位点原子嵌入的串联序列. 最后沿嵌入的整个序列采用全局平均池化,然后将生成的矢量传递给多层感知器回归变量,以获得结合亲和力预测.模型采用2个图注意力网络作为编码器分别对复合体中的配体和活性位点进行编码.编码器会基于给定结构的空间信息对结构内每个原子分配嵌入向量(embedding).编码后将配体和活性位点的嵌入向量拼接成序列后输入双向Transformer模型进行处理,最终将输出特征用多层感知机处理后输出对复合体解离常数(pKa)的预测.训练过程中模型端到端地采用梯度下降进行优化.训练结束后模型在测试集上进行pKa值的预测,达到了1.278的均方根误差.

图1 PyMol可视化的活性位点,绿色部分代表APE1蛋白结构,红色球形代表由氨基酸残基组成的活性位点
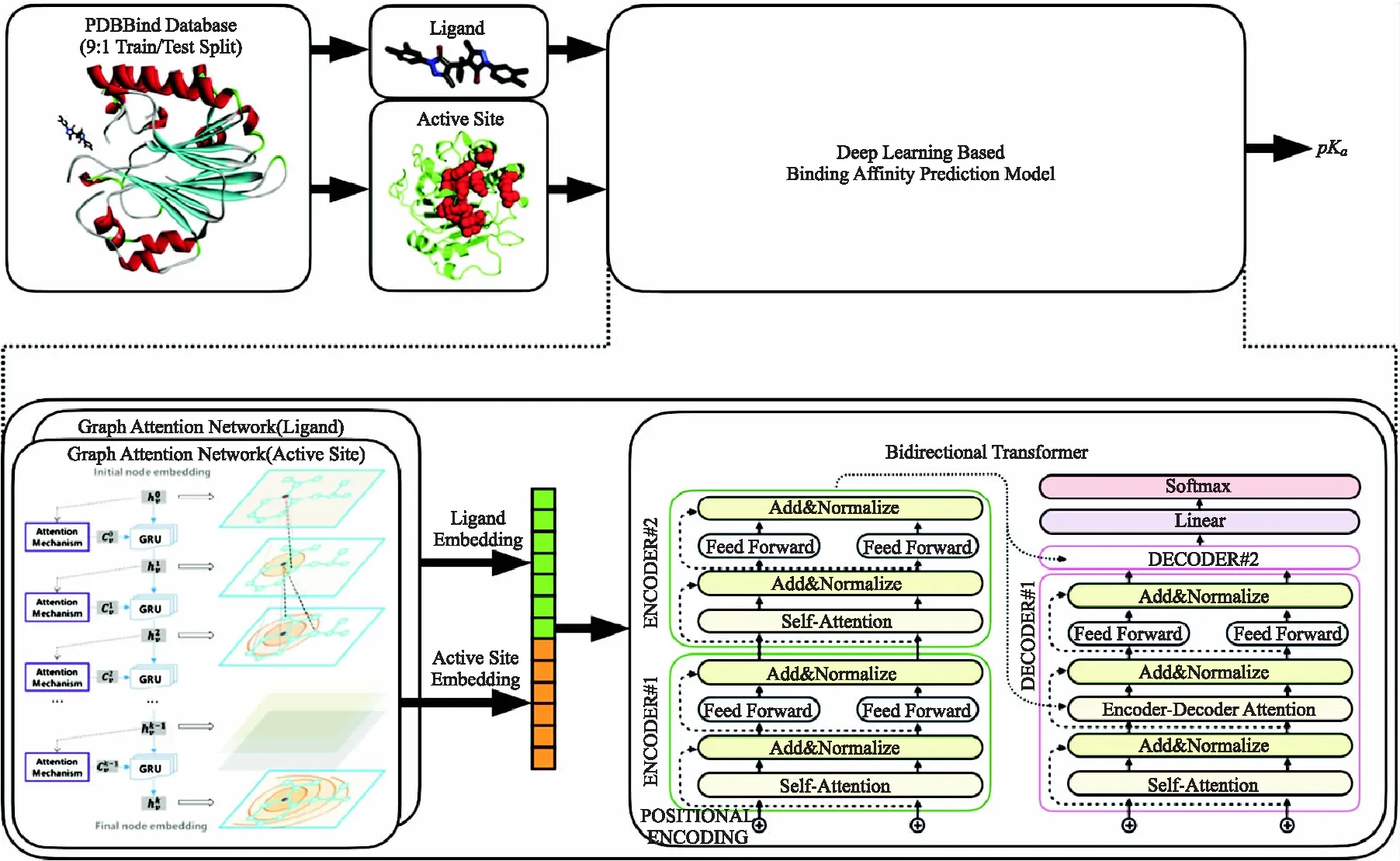
图2 基于深度学习的蛋白质-配体结合亲和力预测模型
1.6 AutoDock Vina标准精度和GOLD高精度筛选
主虚拟筛选是根据AutoDock Vina对深度学习算法筛选后所得的 10 000 个备选小分子根据半经验自由能函数,对蛋白质-配体复合物的结合亲和力分子以及多样性进行评估.Autodock Vina使用迭代局部搜索算法进行构型搜索.在这个过程中,使用AutoDockTools来处理PDBQT文件并计算受体网格.对接中使用的网格参数为center_x= 26.953, center_y= -11.238,center_z= 22.062,size_x= 40,size_y= 56, size_z= 64,exhaustiveness= 8.其余参数均采用默认值.
将得分最高的 2 000 个小分子使用FAFDrugs软件的Drug-Like Soft、PAINS、ADMET规则对小分子进行类药性筛选,将备选小分子根据筛选规则筛选出小分子化合物中分子量小于600,logP在1~5之间,同时化合物满足氢键受体数量不超过10,氢键给体数量不超过5,对化合物检测RO5、ADMET以及是否为假阳性等指标后筛选出147个化合物.最终,使用GOLD软件将保留小分子按照对接精度从低到高进行虚拟筛选,最终综合CHEMPLP的评分及AutoDock Vina对接能量结果得到活性最佳的20个化合物.
1.7 CCK-8细胞活性筛选实验
采用CCK-8检测方法,在HeLa细胞中进行20个化合物的细胞抑制率检测.将HeLa细胞吹打成细胞悬液,以每孔不低于 3 000 个/100 μL,在96孔板中接种,对细胞进行24~48 h 的培养后,将20个小分子化合物配置成(10,20,40,80,160 μmol/L)等5个浓度梯度加入到96孔板中,放入到恒温箱中培养 24 h 后按每孔 10 μL 的量加入CCK-8试剂液,在恒温箱中孵育1~4 h,待显色时间到后,在 450 nm 处用酶标仪测定每孔吸光度,检测时间不超过 24 h.对数据结果绘制量化曲线并计算其IC50值.
1.8 流式细胞术检测HeLa细胞在APE1靶向化合物作用下的凋亡
采用Annexin V-FITC/PI Apoptosis Detection Kit双标记流式细胞数检测活性最优的3个小分子作用于HeLa细胞的凋亡率.给药 24 h 后收集HeLa细胞悬液,预冷PBS洗涤细胞后离心,将Binding Buffer、Annexin V-FITC、PI Staining Solution按照 20∶1∶2 的比例配置后加入收集的细胞中,避光孵育 15 min,在流式细胞仪下进行凋亡检测.
1.9 PLIP配体相互作用分析
为得到配体和蛋白质所具有的结合亲和力,使用蛋白质-配体相互作用分析仪(PLIP)进行全自动检测和可视化3D结构中的非共价蛋白-配体的相互作用结果,并对可视化结果进行蛋白质-配体的相互作用分析.
1.10 统计学处理
各项实验至少进行3次平行独立实验,结果均以平均值±标准差表示,数据分析和作图采用GraphPad Prism 8.0统计软件进行.多组间比较采用单因素方差分析进行,P<0.05认为差异具有统计学差异.
2 结果
2.1 小分子对接能量
表1中的数据,AutoDock Vina分数表示由AutoDock Vina提供的结合亲和力预测.氢键的数目表示存由GOLD产生的蛋白质-配体复合物中的氢键的数目. GOLD Score表示由GOLD分配给蛋白质-配体复合物的ChemPLP适应性得分.IC50表示通过细胞活性筛选测得的每种化合物的半数最大抑制浓度.结果可以看出,20个小分子化合物与APE1结合能(-10.6~-15.7 kcal/mol)较CRT0044876抑制剂与APE1的结合能更好且GOLD评分更高(115.328~49.635),故筛选结果具有一定的可靠性以及提供药理实验的依据.
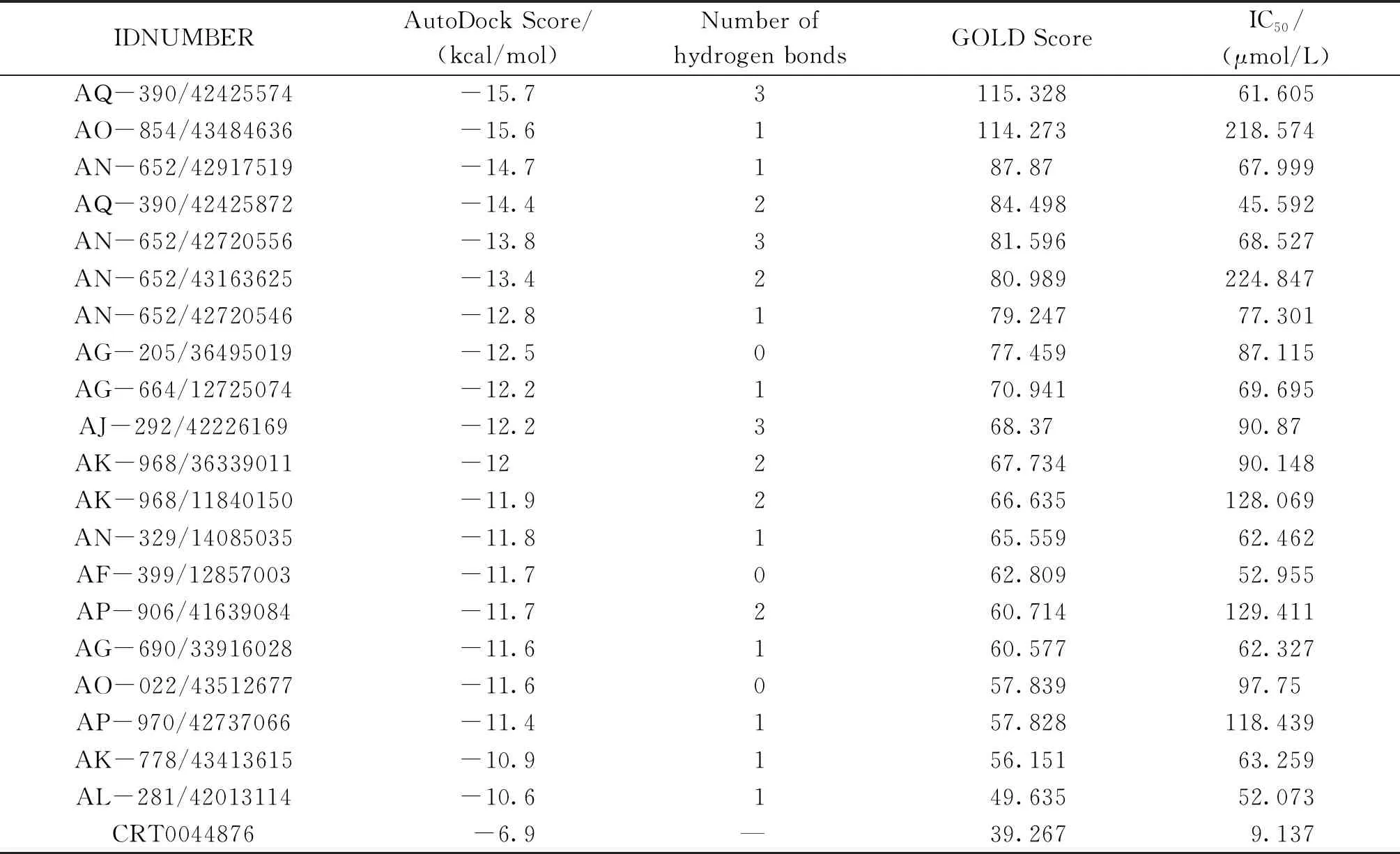
表1 CRT0044876抑制剂(阳性对照)和小分子化合物的AutoDock Vina氢键和对接能结果
2.2 CCK-8细胞活性筛选结果
经过3次平行实验得到20种化合物的IC50值,如表1所示.综合小分子对接能量结果,最终得到AQ-390/42425574,AQ-390/42425872,AK-778/43413615 3个结合能较低,IC50值较低,活性最好的小分子化合物结构见图 3,经过3次平行实验证明3个化合物对HeLa细胞的抑制活性最佳且成一定的浓度依赖性,与顺铂作为阳性对照组相比,在 160 μmol/L 的浓度时其抑制活性与阳性药接近,并绘制3种化合物的量化曲线图(图4).

图3 筛选后在Specs数据库中具有最佳对接活性的3个化合物

图4 浓度为10、40、160 μmol/L的小分子化合物和阳性药物作用于HeLa细胞(A~D)小分子化合物在10~160 μmol/L的浓度梯度下作用于HeLa细胞12~36 h(E~G)
为了进一步探究这3个小分子化合物对HeLa细胞的抑制是否与时间有关,测定HeLa细胞在160,80,40,20和 10 μ mol/L 浓度梯度的小分子化合物的作用下经过12、24、36和 48 h 后的抑制活性,结果发现小分子对HeLa细胞的抑制作用随着时间的增强不断提高,3个化合物在 36 h 时对HeLa细胞的抑制活性最强,抑制率达到40%~80%.其中化合物AQ390-42425574对HeLa细胞的抑制率具有显著性差异(P<0.01).推测AQ390-42425574 对HeLa细胞增殖的抑制具有良好的作用.
2.3 HeLa细胞在APE1靶向化合物作用下的凋亡
根据AQ-390/42425574,AQ-390/42425872,AK-778/43413615的结果可知,以空白组(凋亡率:0.29%±0.23%)和顺铂组(凋亡率:2.63%±8.43%)为对照,化合物在 160 μmol/L 浓度作用下,具有促进细胞早期凋亡(Q1-LR)的趋势,早期凋亡细胞明显增加,晚期凋亡细胞(Q1-UR)对比空白组有增加,且较于阳性药更优.
使用GraphPad prism8软件对结果进行分析,根据结果可以发现其中AK-778/43413615化合物有明显促进HeLa细胞凋亡的作用(AK-778/43413615凋亡率:9.93%±17.4%;AQ-390/42425574凋亡率:4.00%±4.82%;AQ-390/42425872凋亡率:5.49%±4.32%).

图5 流式细胞术分析HeLa细胞凋亡; AK-778 / 43413615,AQ-390 / 42425574,AQ-390 / 42425872与控制组比较*P<0.05,**P<0.01
2.4 APE1蛋白-小分子复合体筛选结果
通过Discovery studio软件中的Structure | Surface 模块对APE1蛋白模型进行表面可视化后,得到3个小分子化合物与活性位点对接示意图,使用Discovery Studio对活性位点进行可视化如图6所示.
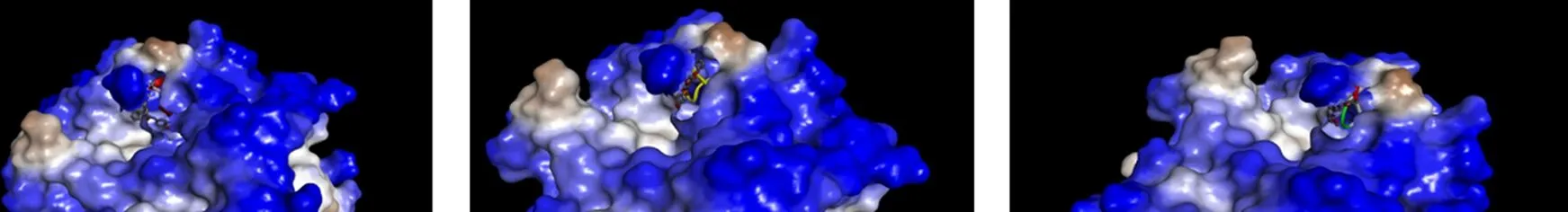
(a)AK-778/43413615 (b)AQ-390/42425574 (c)AQ-390/42425872
2.5 虚拟筛选小分子与APE1蛋白质的相互作用模型分析
为了进一步了解实验中虚拟筛选的小分子与APE1结合的相互作用模式,使用Discovery Studio软件对其蛋白质复合物的结合方式进行二维可视化,结合PLIP配体相互作用分析结果,对小分子和蛋白质结合方式进行分析.小分子AQ-390/42425872和APE1的结合图(图7 和图8)中可以发现该小分子和APE1的A亚基的ARG-177形成氢键作用,并与VAL-180形成π-烷基的疏水作用,大大稳定了蛋白质复合体的构象;并且该小分子的芳香体系与ARG-177产生了π-阳离子相互作用,对稳定复合体,药物靶点的结合起到了重要用,图中深绿色虚线为氢键,粉色虚线为π-烷基作用力,橘色线为π-阳离子作用力.对比小分子AQ-390/42425574的结合图(图9 和图10)可以看出小分子与APE1的A亚基的ARG-177形成3个氢键,并且与VAL-180,GLY-176,ARG-181等多个残基产生了强烈的疏水作用,使得疏水基团进入疏水性的蛋白口袋,使整体的疏水性基团尽可能少的暴露在水溶液中,使蛋白质复合体更加稳定从而大大提高了活性.故结合小分子的结合能可以得出小分子AQ-390/42425872的结合能量不如AQ-390/42425574,应该是与同母核的不同基团所产生的疏水作用以及形成的氢键数量不同有关.从AK-778/43413615的结合图(图11 和图12)可以看出该小分子与A亚基的ARG-177,VAL-180产生了疏水作用,并只形成1个氢键,故其结合能量远不如其他2个化合物.将3个筛选化合物与 CRT0044876抑制剂做对比,由结合图(图13和图14)可以看出该抑制剂与APE1的A亚基的ARG-177形成3个氢键,同时产生了疏水作用,但其结合能不如筛选小分子应该是由于疏水作用较弱所产生的结果.
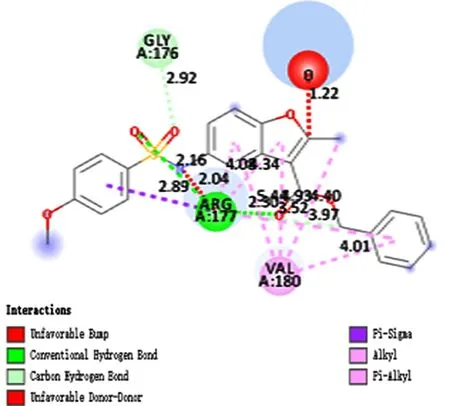
图7 AQ-390/42425872分子与APE1对接作用力示意图

图8 AQ-390/42425872分子与APE1(PLIP)相互作用图
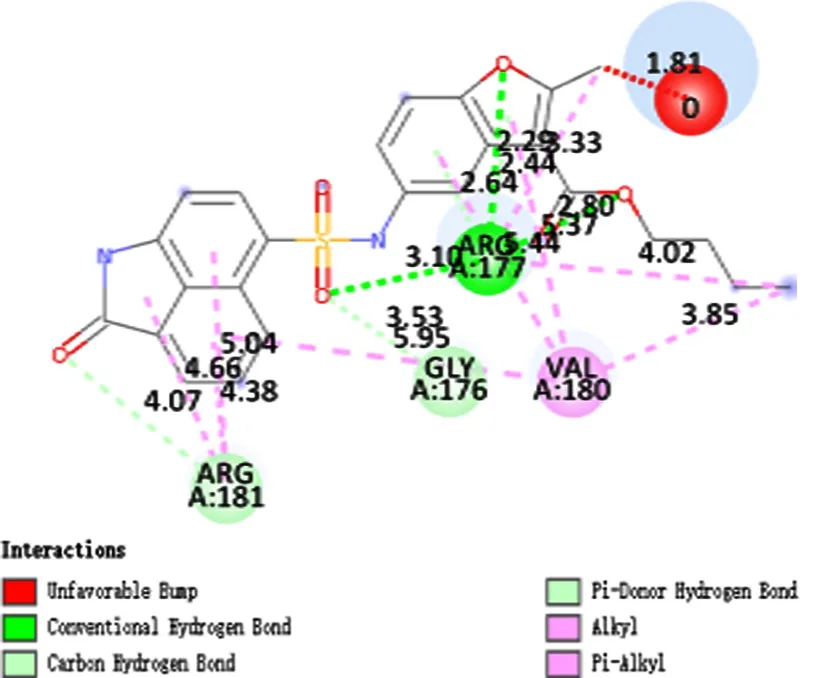
图9 AQ-390/42425574分子与APE1对接作用力示意图

图10 AQ-390/42425574分子与APE1(PLIP)相互作用图
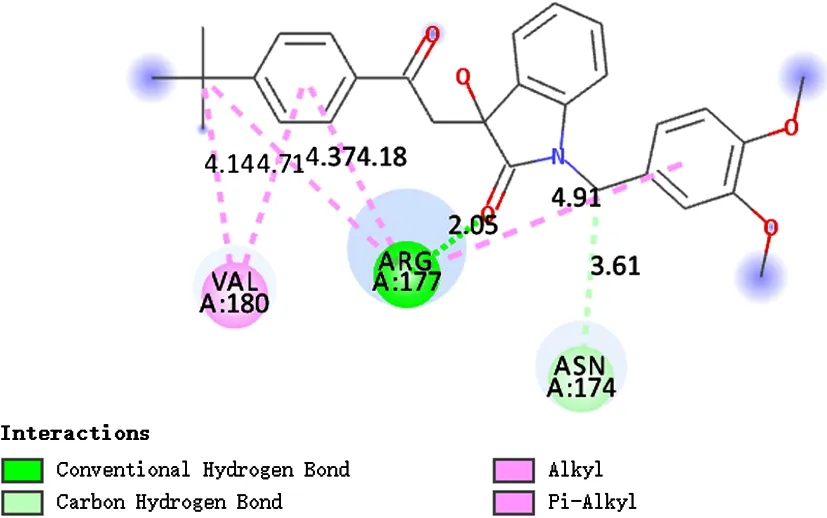
图11 AK-778/43413615分子与APE1对接作用力示意图
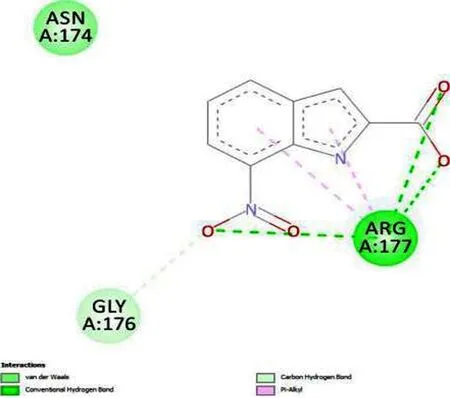
图13 CRT0044876抑制剂与APE1对接作用力示意图
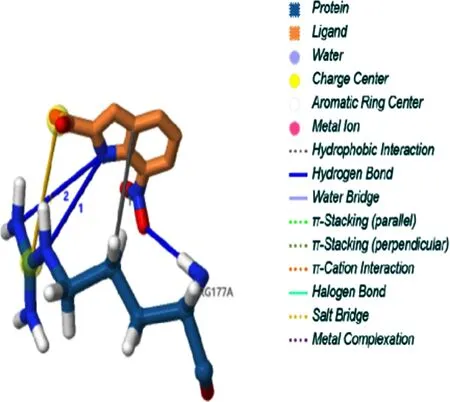
图14 CRT0044876抑制剂与APE1(PLIP)相互作用图
3 结语
近年来以APE1为靶点开发的药物已经有了很多成功的例子,主要以修复抑制剂的研究较深入,目前获得的修复活性抑制剂包括甲氧胺(MX),CRT0044876,杨梅素(Myricetin),6-羟基-左旋多巴和活性蓝2(Reactive Blue2)等[6].其中其受体抑制剂的主要寻找方向是各个数据库中的小分子化合物.通过计算机虚拟筛选技术发现其中3个化合物可以与APE1形成较好的蛋白质复合体,并通过细胞药理实验验证筛选小分子对HeLa细胞增殖具有较好的抑制作用,之后可再通过更深入的药理实验进一步验证虚拟筛选的结果是否具有较好的药物效果以及化合物是否对APE1有较强的体内抑制作用.本研究从Specs数据库的 210 070个化合物中发现了可以与APE1结合较好的化合物.说明实验使用的虚拟筛选技术能够深入挖掘小分子化合物与相应受体的结合可能性,且实验结果还可为继续研究这些化合物的潜在药性,活性中心,开发新的化合物等研究热点提供基础资料.
