基于人工智能算法的小麦全基因组选择育种技术研究
2022-10-14张树馨范钧玮许雪凌杜梦涵狄玉洁刘广臣
张树馨,范钧玮,许雪凌,杜梦涵,狄玉洁,刘广臣*
(1.鲁东大学 数学与统计科学学院,山东 烟台 264000;2.鲁东大学 信息与电气工程学院,山东 烟台 264000)
小麦是我国重要的粮食作物之一,是我国人民主要食用的细粮。为了国家农业生产发展,在中国现阶段面临人口多,耕地少的问题下,挑选出影响小麦性状的关键基因序列,提高小麦产量,有着重要的实际意义。
小麦育种关键在于选择,传统育种是以亲本以及杂交后代的表型为基础,同时结合育种材料来对优良品种进行选择[1]。但是通过表型选择耗费的周期较长,而且表型受到环境与基因的共同影响,无法准确反映其遗传特征,选择过程存在不稳定性。
随着育种技术的发展及大数据时代的到来,人们对育种的研究不断深入,研究重点也从表型层面转入到分子层面。全基因组选择(Genomic Selection,GS)是Meuwissen[2]提出的一种选择育种方法[2],是指利用覆盖整个基因组的遗传标记信息来对未知表型的个体育种值进行的估计,其作为一种高效育种方法,通过早期选择缩短世代间隔,提高育种值估计准确性,加快遗传进展,逐渐替代了传统育种方式。
现阶段用于GS 的研究方法主要有BLUP 法,Bayes法,机器学习等。BLUP 系列模型方面,Henderson 提出基于系谱矩阵(A 矩阵)的BLUP(Best Linear Unbiased Prediction)模型,其通过对育种值进行估计,明显提升了遗传进展。VanRaden 提出的基于全基因组标记的GBLUP(Genomic Best Linear Unbiased Prediction)模型[3],在大部分情况下,准确性优于传统BLUP 方法[4]。Zhang[5]提出TABLUP 模型,加入权重进行计算,从而减少了无效标记所带来的影响,增加有效标记的重要性。Edwards 等[6]提出GFBLUP(Genomic feature BLUP)模型,将GBLUP 中随机效应增加到2 个,使模型更加灵活。Bayes 系列模型方面,Meuwissen 等[7]提出BayesA 和BayesB 模型,二者标记均服从先验分布。Park 等[8]提出Bayesian LASSO 模型,Bayesian LASSO 准确性高,但迭代次数多,耗时较长[9]。Verbyla 提出BayesC 模型,在BayesB 模型上对其中的pi进行改进。Habier 等[10]在BayesC 基础上对pi 进一步优化,提出BayesCpi,BayesDpi 模型,具有更强的灵敏性。
机器学习(Machine Learning)模型方面,目前支持GS 的机器学习方法主要有随机森林(Random Forest,RF)模型[11],支持向量机(Support Vector Machine,SVM)模型[12]等。与传统的全基因组选择方法相比,机器学习算法能够提高计算效率,提供较高的预测精度。对于“大p 小n”问题,机器学习也可通过优化算法来解决,整个过程计算效率较高;同时在选取模型时运用交叉验证,充分利用样本信息,提高预测的准确性。
1 数据来源和预处理
1.1 数据来源
本研究所用的小麦基因组数据来源于文献[13](http://www.isbreeding.net/wheatGS/),所研究的小麦群体一共包含166 份材料。研究了6 个相关性状,分别是籽粒产量(Grain Yield,GY)、抽穗天数(Heading Date,HD)、株高(Plant Height,PH)、穗长(Spike Length,SL)、千粒重(Thousand Kerner Weight,TKW)和每平方穗数(Spike Number,SN)。通过提取每一个品系的单株DNA进行基因型鉴定,一共获得81 587 个SNP 标记[13]。
1.2 数据预处理
对上述小麦数据集的基因型数据进行编码,对每一个位点的基因做如下处理:若存在缺失且缺失数量大于总样本数的10%,则将该列删除;若存在缺失但缺失数量小于总样本数的10%,则以该列众数对缺失基因型信息的位点进行替换。
2 全基因组选择模型
2.1 传统育种模型原理简介
(1)GBLUP 模型。GBLUP 是一种线性混合模型,通过群体标记信息构建的亲缘关系矩阵,以及估计方差组分,对个体育种值直接进行预测,又称为直接法。GBLUP的先验假设适合由多微效基因控制的性状,对于由少数大效应标记控制的性状,预测准确性较差。
(2)BayesA 模型。BayesA 的标记效应估计模型如下
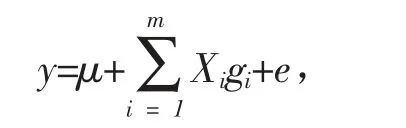
式中:y 是表型值;μ 是总体均值;X 是标记效应的设计矩阵;gi是第i 个标记的效应;gi~N(),其中效应方差服从卡方分布;m 是总标记的数量;e 表示残差向量。
2.2 机器学习模型原理
(1)RBF-SVR 模型,SVR(Support Vector Regression)支持向量回归,是支持向量机(SVM)的重要应用分支。使用SVR 作回归分析,要找出一个最佳的条状区域,再对区域外的点进行回归。与SVM 一样,需要利用核函数将低维空间映射到高维空间,这里选择高斯径向基函数(RBF)。
(2)XGBoost 模型,XGBoost(eXtreme Graident Boosting)极致梯度提升,是基于GBDT 的一种算法。XGBoost 进行许多优化,比如:利用二阶泰勒公式展开,优化损失函数,提高计算精确度;利用正则项简化模型,避免过拟合;采用Blocks 存储结构,可以并行计算等。
(3)LightGBM 模型,LightGBM(Light Gradient Boosting Machine)是一个实现GBDT 算法的框架,可以快速处理海量数据。LightGBM 方法采用histogram 算法,占用内存低,数据分割的复杂度更低;采用leaf-wise 生长策略,循环迭代,同时引入了一个阈值进行限制,防止过拟合。
(4)Linear-SVR 模型,Linear-SVR 可以有效捕捉样本的局部变化趋势,从而提高模型的预测精度。其选取每个测试样本的K 个相邻的样本,对这K 个样本使用SVR进行回归建模,利用所建立的模型对其进行预测,每个测试样本均执行上述步骤,直到所有样本预测完成。
(5)Ridge 模型,岭回归是一种用于回归的线性模型,该模型可以写为

式中:y 是表型值;X 是固定效应的设计矩阵;β 是标记固定效应的向量;Z 是随机效应设计的矩阵;μ 是随机效应的向量;ε 是随机残差。
2.3 模型训练和调参
在基于人工智能算法的小麦全基因组选择育种模型的训练过程中,为了提高模型预测的准确性和时效性,笔者通过随机搜索对配置的参数进行调整,在此过程中,关注的主要参数以及对应6 个性状采用的参数最优值见表1。
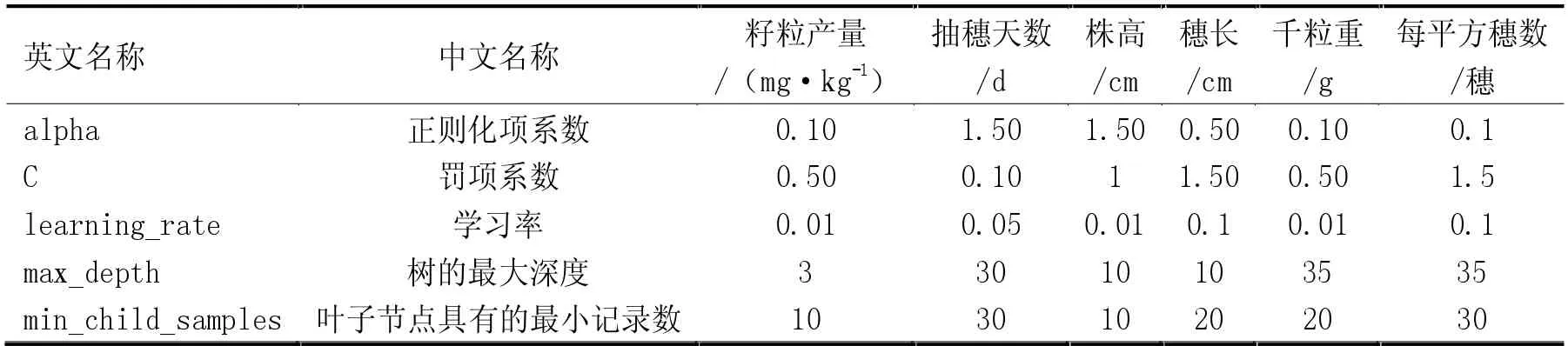
表1 全基因组选择模型参数表
3 模型结果
本研究评估了Linear-SVR,RBF-SVR,Ridge,Light-GBM,XGBoost,GBLUP,BayesA 7 个模型对小麦基因组预测准确性比较。研究随机抽取90%的样本作为测试集,10%的样本作为验证集,同时考虑到计算的准确性及效率,采用十轮十折交叉验证。对于5 个机器学习模型及2 个传统育种模型对小麦6 个性状的预测精度见表2。

表2 多性状的预测表现
从表2 中可看出,6 个性状的最佳模型分别为Ridge,GBLUP,Ridge,GBLUP,Ridge,Linear-SVR。其中TKW 的Ridge 模型预测准确性最高,达到0.693。除去SN 以外,所有性状的最高预测准确性均达到0.6 以上。
4 结束语
为积极响应国家号召保障粮食安全,提高小麦产量,进一步提升育种技术,本文通过冬小麦的6 个不同性状对5 种机器学习模型与2 种传统模型进行了对比。考虑到不同方法之的间比较,GBLUP 的预测准确性最高,其次为Ridge。考虑到每个性状的前3 个精度,Ridge 的表现优于GBLUP,有着更强的稳定性;其次传统育种模型与机器学习模型在不同的性状上,所呈现出的优势并不相同,故现阶段对于小麦不同性状而言并没有较为固定的单一模型,不同模型对不同性状的预测表现具有一定程度差异。
