莱鲍迪甙C高效转化细菌Paenarthrobacter ilicis CR5301全基因组测序及关键糖苷酶分析
2022-10-09李洪飞孙大庆曹龙奎
李洪飞,孙大庆,曹龙奎,*
(1.黑龙江八一农垦大学 国家杂粮工程技术中心,黑龙江 大庆 163319;2.东北石油大学化学化工学院,黑龙江 大庆 163318)
甜菊糖甙是从甜叶菊中提取的无热量、高甜度的四环二萜类化合物,甜度是蔗糖的20~300 倍,热量仅为蔗糖的1/300,安全无毒,具有预防动脉硬化、肥胖、龋齿症等疾病,以及抗高血糖、抗炎症、抗肿瘤、利尿和免疫调节等功效,是天然、绿色且健康的第3代甜味剂,目前已在食品、医药、日化用品等行业广泛应用。目前从甜叶菊中已检测到的甜菊糖甙超过300 种。甜菊糖甙以甜菊醇为苷元,只是在C13和C19位连接的糖基数量和种类不同,其中莱鲍迪甙C(rebaudioside C,RC)含量位列第3,甜度低、后苦味重,因此严重影响了它在食品工业中的应用。
甜菊醇作为甜菊糖甙分解代谢的终产物,已被证明是甜菊糖甙发挥生理功能的主要结构基础,也是许多药物的重要原料或合成前体。然而,它在甜叶菊中含量很低,不足0.1%,因此通过直接提取法生产甜菊醇产量很低。目前甜菊醇的制备方法有化学合成法、酶催化法和微生物转化法,其中微生物转化法以产量高、反应条件温和、收率高和成本低等特点受到研究者的青睐。
目前报道以RC为底物制备甜菊醇的微生物有霉菌、和细菌,以及人体肠道中的某些微生物。马迎迎报道显示,固体培养浸提酶液在45 ℃转化1%甜菊糖甙,50 h后底物中RC转化率达到100%,转化产物为甜菊醇,该研究者还利用液体培养菌丝将RC转化为甜茶苷,4 d后RC转化率为97.9%。Jiang Huiling等采用对甜菊糖甙中RC进行转化,甜菊糖甙底物质量分数1%,37 ℃、220 r/min培养144 h后,RC全部转化为甜菊醇。Koyama等利用人体肠道菌群孵育甜菊糖甙,观察到甜菊糖甙中RC明显减少,孵育24 h后RC剩余7%,转化产物为甜菊醇。整体而言,完全转化RC时间最短,转化效率最高。尽管人体肠道菌群可以更快地转化RC,但24 h后RC并没有完全转化,而且报道中没有证明具体哪种微生物具有RC转化能力。
本课题组前期从甜叶菊种植土壤中分离筛选到1 株可以高效转化RC的细菌,该菌株可以将1 mg/mL高纯度RC和1 mg/mL甜菊糖甙分别在8 h和10 h完全转化为甜菊醇。经16S rRNA(GenBank ID:MW926547)物种鉴定,该菌株命名为CR5301。以前称为,Busse对菌属进行了重新分类,增列了。目前菌株转化RC的研究以往鲜见报道。NCBI数据库显示,截至2021年11月,全基因组测序菌株为31 株,其中6 个基因组为完成图序列,物种收录2 个基因组序列,但均为没有组装完整的草图序列。为全面了解CR5301的遗传背景,深入解析CR5301转化RC的代谢途径和关键酶,本研究采用二代Illumina HiSeq和三代Nanopore相结合的测序方式对CR5301进行基因组完成图测序,以期得到物种第1个完成图基因组序列,之后利用生物信息学软件和公开数据库对该基因组进行全面、深入的基因功能注释、分类和预测分析,旨在为今后CR5301的RC转化代谢机制研究提供清晰的遗传背景和关键的候选酶基因信息。此外,CR5301完整基因组序列测定和解析将为今后物种的遗传、进化、生理等广泛生物学研究提供重要的遗传信息基础。
1 材料与方法
1.1 材料与试剂
CR5301由本实验室从甜叶菊种植土壤中分离、筛选获得,该菌株已保藏于中国典型培养物保藏中心(CCTCC NO:M2021851)。
察氏培养基和察氏不含蔗糖培养基 青岛高科技工业园海博生物技术有限公司;细菌基因组抽提试剂盒WizardGenomic DNA Purification Kit 美国Promega公司;测序文库构建试剂盒NEXTflexRapid DNA-Seq Kit 美国Bioo Scientific公司;其他试剂均为分析纯或生化试剂。
1.2 仪器与设备
HZQ-Q型全温振荡器 哈尔滨市东联电子技术开发有限公司;MLS-3751L-PC型高压蒸汽灭菌器 松下健康医疗株式会社;H1850R型台式高速冷冻离心机 湖南湘仪实验室仪器开发有限公司;GeneAmp9700型聚合酶链式反应(polymerase chain reaction,PCR)仪美国ABI公司;NanoDrop ONE型微量紫外-可见分光光度计 美国Thermo公司;M220型Covaris超声波破碎仪基因有限公司;TBS-380荧光仪、Illumina HiSeq测序仪美国Illumina公司;Nanopore测序仪 英国Oxford公司。
1.3 方法
1.3.1 菌株活化与培养条件
从-80 ℃冰箱取出冻存的CR5301菌株,在察氏固体培养基平板上划线,28 ℃恒温培养箱中培养48 h,挑取单菌落接种于察氏液体培养基中,28 ℃、135 r/min条件下培养24 h。取2 次活化培养菌液,2%(/)接种于新鲜察氏液体培养基中,培养至对数末期,取样用于后续实验。
1.3.2 菌株基因组DNA的提取
取活化后菌液2 mL,按照Wizard细菌基因组抽提试剂盒说明书进行基因组DNA的提取。基因组DNA样品利用微量紫外-可见分光光度计和荧光仪进行纯度和浓度测定。DNA满足质量浓度≥20 ng/μL和纯度OD/OD=1.8~2.0的样品用于后续建库测序。
1.3.3 测序文库的构建
Illumina测序文库制备:取至少1 μg基因组DNA,利用超声破碎仪Covaris进行基因组DNA片段化,将DNA样本剪切成约400 bp的片段,按照NEXTflexRapid DNASeq试剂盒说明书进行Illumina测序文库制备。Nanopore测序文库制备:取至少15 μg基因组DNA,利用Covaris将基因组DNA处理成约10 kb的片段,然后进行片段纯化,末端补平,两端分别连接Nanopore测序接头。
1.3.4 全基因组完成图测序、组装及数据质控分析
采用细菌基因组测序方法,利用二代Illumina HiSeq和三代Nanopore相结合的测序方式对CR5301基因组进行完成图测序。制备的二代测序文库在Illumina HiSeq×10测序仪上进行双末端测序(2×150 bp)。制备的三代测序文库在Nanopore测序仪上进行纳米孔测序。二代测序数据原始数据以fastq格式储存,为了使后续的组装更加准确,会对原始数据进行质量剪切,去除测序质量较低、含N比例较高及质量修剪后长度较小的reads,得到高质量的clean data。利用Canu及HGAP软件进行Nanopore数据组装,将reads组装成contigs,然后判断是否成环,得到完整的染色体和质粒基因组。最后利用Illumina测序数据对组装结果进行校正,并判断环状基因组的起始位点。基因组圈图利用CGView软件绘制。本次测序委托上海美吉生物医药科技有限公司完成。
1.3.5 基因预测及功能注释
利用Glimmer对基因组中的基因进行预测,质粒基因采用GeneMarkS软件预测,tRNAscan-SE进行tRNA预测,Barrnap进行rRNA预测,使用Tandem Repeats Finder软件进行串联重复序列预测。利用BLASTP、Diamond、HMMER等序列比对工具,从非冗余蛋白(Non-Redundant,NR)数据库、欧洲蛋白质数据库Swiss-Prot、蛋白质家族数据库Pfam、基因本体论(Gene Ontology,GO)、直系同源聚类群(Clusters of Orthologous Groups,COG)、京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)、碳水化合物活性酶(carbohydrate-active enzymes,CAZy)数据库中对预测到的基因进行功能注释。所有数据库中序列比对阈值设置-value≤10。
1.3.6 基因组共线性和进化分析
检索并下载NCBI下Genome数据库(https://www.ncbi.nlm.nih.gov/genome/)中所有物种公布的完整基因组序列数据。利用Mauve 2.4软件,将CR5301和所有已知基因组序列进行共线性比对分析。种子序列为默认值15 bp。Mauve计算获得的基因组系统进化树采用MEGA 7.0软件进行可视化。
检索并下载NCBI下RefSeq数据库(https://www.ncbi.nlm.nih.gov/refseq/)中所有物种全长16S rRNA基因序列。利用MEGA 7.0软件进行多序列比对,比对结果利用邻接法构建CR5301和所有已知物种16S rRNA基因序列的系统进化树。同为革兰氏阳性菌且基因组高GC含量的长双歧杆菌模式菌株ATCC 15707的16S rRNA基因(NR_044691.2)作为进化树的外群对照。
1.3.7 关键糖苷酶预测、基本性质和结构分析
分 别 利 用 ProtParam、 SignalP 5.0、TMHMM 2.0、SOPMA软件预测蛋白质的基本理化性质、信号肽序列、跨膜结构域和二级结构。
1.4 数据统计分析
所有数据使用Excel 2019进行整理、筛选和统计分析。利用Prism 8软件绘制柱状图。使用CGView(Version 2)绘制基因组圈图。
2 结果与分析
2.1 P. ilicis CR5301基因组组装结果和基本特征
经二代和三代测序数据联合组装,CR5301基因组获得1 个完整闭合的染色体基因组序列,没有获得质粒序列。CR5301基因组序列全长4 748 281 bp,GC含量62.92%,共含有4 458 个编码基因,包括54 个tRNA和18 个rRNA编码基因,测序深度310.36 倍。CR5301基因组测序和基因预测详细结果如表1所示。为了全面和直观地展示CR5301全基因组的基本特征,通过CGView软件绘制基因组圈图,如图1所示。
目前,GenBank数据库中物种只有2 个菌株DSM 20138(3.97 Mb,Accession No. NZ_JAFBCD010000001.1)和CECT 4207(4.09 Mb,Accession No. NZ_JAAOZD010000001.1)基因组序列公布,但它们都没有组装完整,从基因组大小看,它们仍然缺失较多的遗传信息。因此CR5301基因组完成图的测定,不仅为CR5301的RC转化功能研究提供了清晰、完整的遗传信息,并且为物种的遗传、进化、生理、代谢研究首次提供了完整、可靠的参考基因组序列,这对今后广泛的生物学研究具有重要参考价值和普遍借鉴意义。

表1 P. ilicis CR5301基因组基本特征Table 1 General characteristics of P. ilicis CR5301’s genome
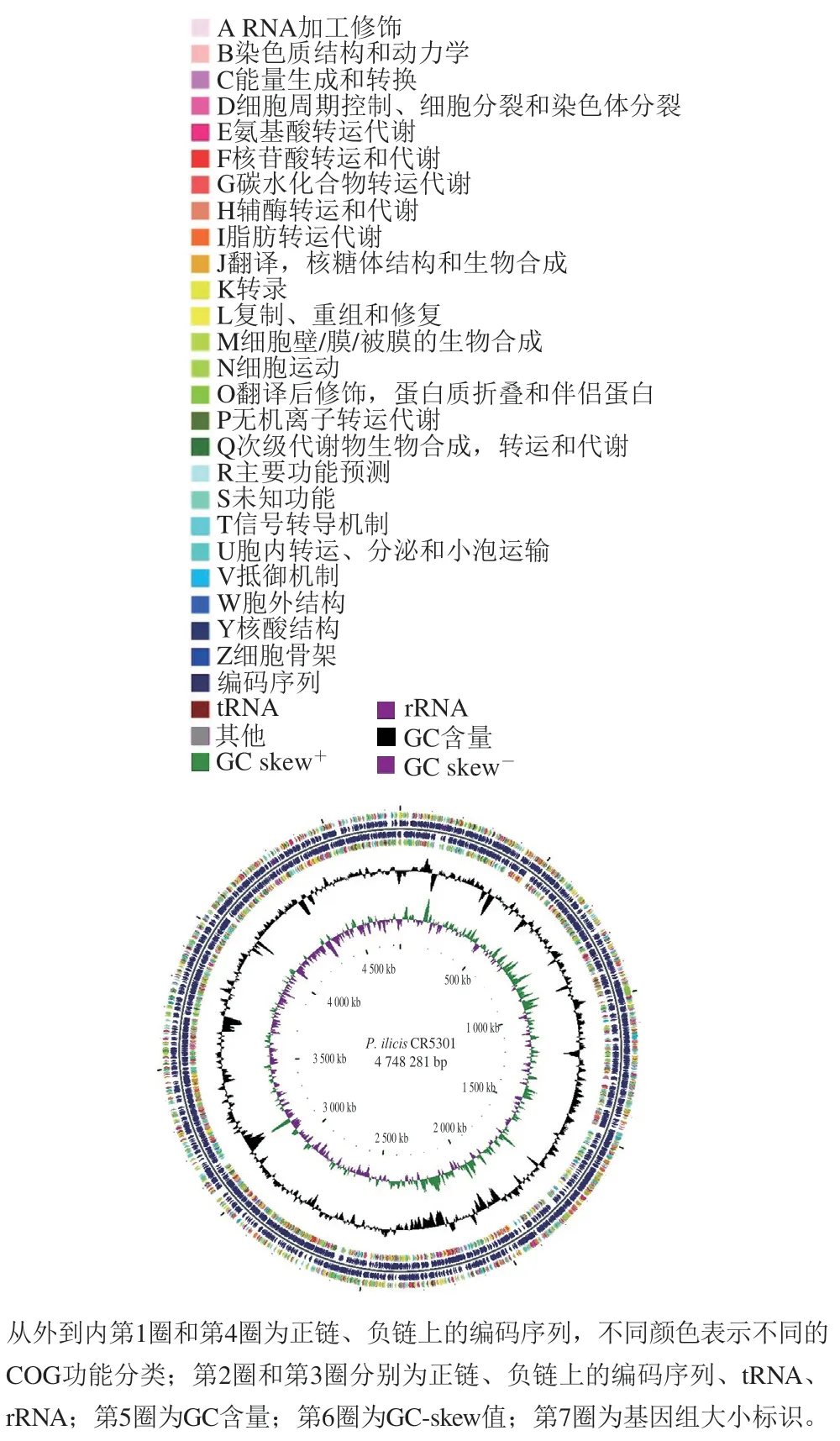
图1 P. ilicis CR5301基因组圈图Fig. 1 Graphical representation of P. ilicis CR5301’s genome
2.2 P. ilicis CR5301基因注释和功能分类
2.2.1 COG注释分析
COG是进行蛋白质直系同源分类的数据库。将测序基因的氨基酸序列与eggNOG数据库进行比对,从而预测这些蛋白质的功能并进行功能分类统计。经COG注释分析,CR5301共有21 类3 749 个基因得到了COG注释,占基因总数84.1%,结果如图2所示。未知功能的基因数量最多,共1 120 个,占注释基因总数的29.87%。其次为碳水化合物转运和代谢、氨基酸转运和代谢以及转录相关的基因,分别为379、372 个和355 个,分别占注释基因总数的10.11%、9.92%和9.47%。与无机离子转运和代谢、能量产生与转化功能相关的基因也得到较多的注释,分别为208 个和204 个。
由于CR5301的RC转化功能与碳水化合物代谢功能相关,因此对注释为碳水化合物转运和代谢的COG基因进行了分析。总共有379 个基因被注释到该功能相关的169 个COG分类中,其中基因最多的是COG0395(依赖内膜转运系统的结合蛋白,22 个)和ENOG410XP7I(转运蛋白,21 个),基因不少于10的有COG1653(转运蛋白活性,15 个)、COG1082(TIM桶结构域木糖异构酶,12 个)、COG1472(水解酶家族3,11 个)、COG1940(ROK家族蛋白,11 个)、COG0477(主要激活剂超家族蛋白,10 个)、COG0524(pfkb结构域蛋白,10 个)、COG1621(水解酶,10 个),唯一基因的COG有101 个。碳水化合物转运和代谢功能高度多样性表明,CR5301具有强大的碳水化合物代谢能力。
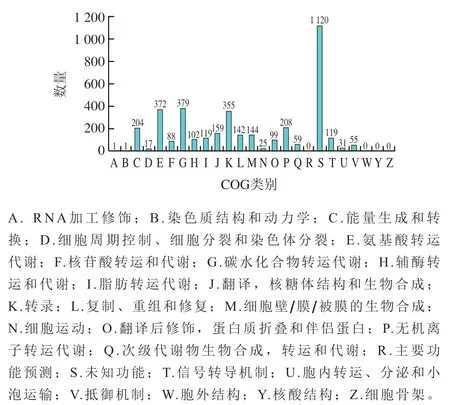
图2 P. ilicis CR5301基因的COG注释和分类Fig. 2 COG annotation and classification of P. ilicis CR5301’s genes
2.2.2 GO注释分析
CR5301在GO数据库中注释到2 439 个基因,占基因总数54.71%。三大分类中,注释到分子功能相关的基因最多,有1 987 个,注释到细胞组成和生物过程相关的基因数相近,分别为1 051 个和1 044 个,GO注释分析详细结果见图3。在生物学过程分类中,跨膜转运(GO:0022857,125 个)、DNA模板的转录调控(GO:0006355,77 个)、碳水化合物代谢(GO:0005975,60 个)和翻译(GO:0006412,58 个)基因数最多。在细胞成分分类中,膜成分(GO:0016021,692 个)、细胞质(GO:0005737,204 个)和(细胞膜GO:0005886,156 个)基因数明显高于其他GO分类。在分子功能分类中,DNA结合(GO:0003677,272 个)和ATP结合(GO:0005524,257 个)基因数也明显高于其他分类。此外,重点分析了可能参与RC生物转化过程的基因,这些基因主要涉及水解酶活性(GO:0016787,99 个)、碳水化合物代谢(GO:0005975,60 个)、水解酶活性、水解-糖基化合物(GO:0004553,20 个)、碳水化合物结合(GO:0030246,11 个)等,去掉重叠,共发现163 个基因。
2.2.3 KEGG注释分析
KEGG是系统分析基因产物在细胞网络代谢通路以及这些基因产物功能的数据库,利用KEGG 可以进一步解析基因产物在生物学上的复杂行为。经KEGG注释分析,CR5301共有1 975 个基因得到了KEGG注释,注释基因占基因总数44.30%,如图4所示。代谢通路第一层级共有6 个分类,获得注释基因由多到少的顺序为:代谢(834 个,占注释基因总数42.23%)、遗传信息处理(165 个,8.35%)、环境信息处理(138 个,6.99%)、细胞过程(102 个,5.16%)、人类疾病(7 个,0.35%)和生物体系统(5 个,0.25%)。在第二层级中,除了全局和概览图,与碳水化合物代谢通路相关的注释基因最多,注释基因301 个,占注释基因总数15.24%,明显高于其他代谢通路。这一结果进一步表明,CR5301具有碳水化合物代谢高度多样性的功能基因和代谢通路,从而对外界复杂碳水化合物的降解、转化和利用提供了巨大潜力和可能性。
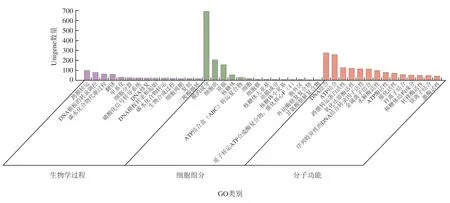
图3 P. ilicis CR5301基因的GO注释和分类Fig. 3 GO annotation and classification of P. ilicis CR5301’s genes
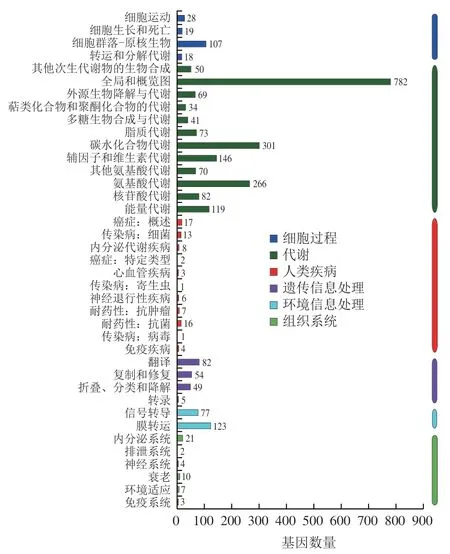
图4 P. ilicis CR5301基因的Pathway注释和分类Fig. 4 Pathway annotation and classification of P. ilicis CR5301’s genes
2.3 P. ilicis CR5301的可移动元件
在漫长的进化过程中,为适应环境的变化或提高自身的生存竞争力,细菌基因组往往会摄入一些外源基因片段,并将其整合进自己的基因组中,这些片段上一般都含有某些特定功能的编码基因,比如毒力基因、耐药基因、代谢基因等,从而改变细菌的表型,协助细菌度过“困境”或占据优势生态位,这些可在基因组内或基因组之间转移遗传信息的DNA片段统称为可移动遗传元件。细菌中可移动遗传元件主要包括质粒、基因岛、前噬菌体、CRISPR-Cas等。经分析,CR5301不含有质粒,含有4 个基因岛、1 个前噬菌体和14 个CRISPR-Cas编码序列,详细信息见表2。
基因功能分析显示,基因岛GI01和GI03分别含有1 个和4 个噬菌体同源基因,推测它们由噬菌体整合而来。基因岛GI04仅含有1 个重组酶基因,无噬菌体和质粒特有基因,无法确定其来源。3 个基因岛均不含有碳水化合物代谢酶基因。但分析发现,最大基因岛GI02不仅含有多个碳水化合物代谢酶基因,而且含有4 个-葡萄糖苷酶基因(gene1874、gene1875、gene1884、gene1887),它们是CR5301水解RC葡萄糖基侧链的候选基因,也可能是CR5301将RC转化为甜菊醇的关键酶基因。同时,基因岛GI02编码1 个释放酶基因(gene1870),而该基因是接合质粒特有基因,因此推测GI02由1 个接合质粒整合而来。前噬菌体Ph01不含有碳水化合物代谢酶基因,与分枝杆菌噬菌体D29含有多个高度同源蛋白,因此推测Ph01可能来自分枝杆菌噬菌体D29。从基因组中定位看,前噬菌体Ph01与基因岛GI03大部分区域重叠,这表明基因岛GI03很可能也来自分枝杆菌噬菌体D29。CRISPR-Cas系统是一种原核生物的免疫系统,用来抵抗外源遗传物质的入侵,如噬菌体、病毒和外源质粒。它可以识别出外源DNA,并且沉默外源基因的表达。注释分析发现,CR5301基因组含有14 个CRISPR-Cas编码序列,重复序列、间隔序列和重复次数具有丰富的多性样,这表明它在演化过程中经受过复杂的外源质粒和噬菌体入侵,已经进化出比较健全的先天免疫机制。
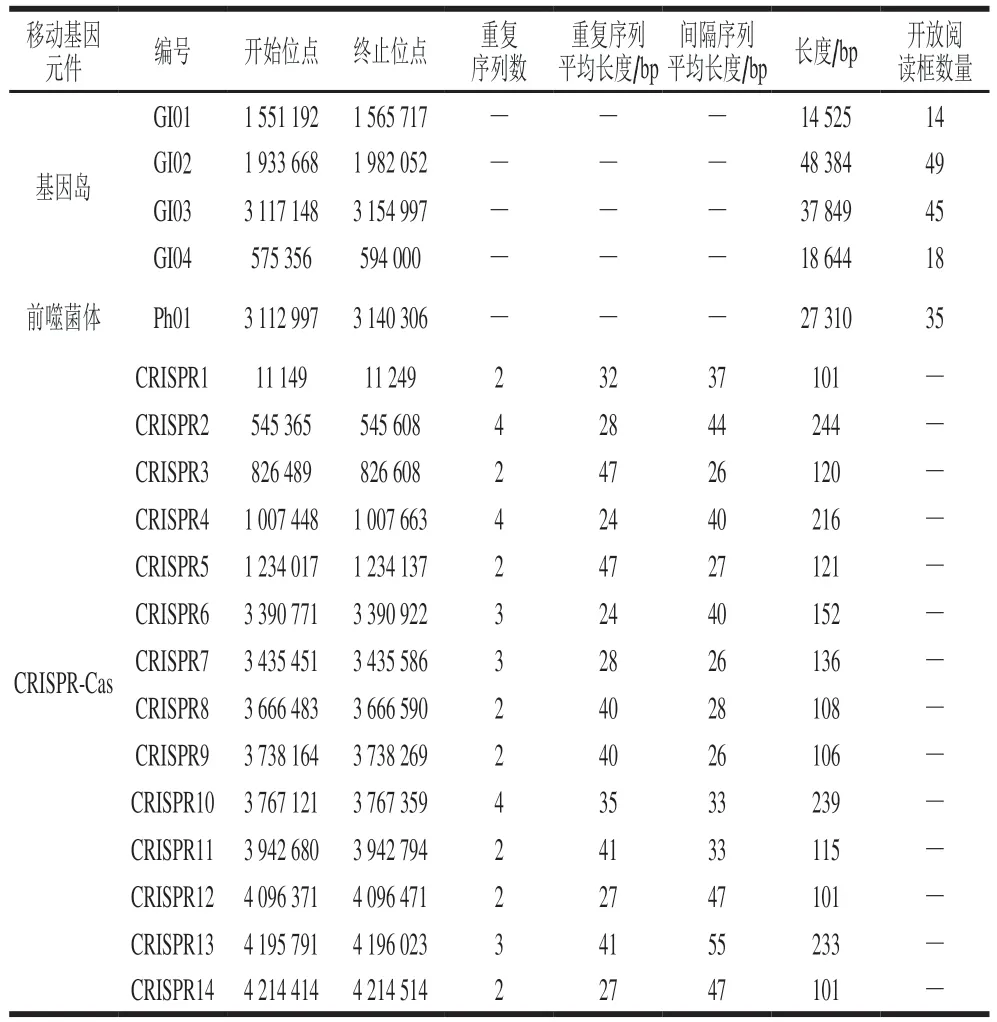
表2 P. ilicis CR5301基因组中可移动元件Table 2 Mobile genetic elements in P. ilicis CR530’s genome
2.4 P. ilicis CR5301的基因组共线性和系统进化关系
经检索目前NCBI下Genome数据库中共收录31 个菌株基因组序列,分属于6 个已知物种和未知物种,其中6 个菌株为完整基因组序列。虽然收录2 个基因组序列,但2 个基因组测序均没有组装成完成图,因此没有用于基因组共线性分析。CR5301和6 个菌株基因组共线性分析结果如图5A所示。基于基因组共线性,7 个菌株全基因组系统进化树如图5B所示。
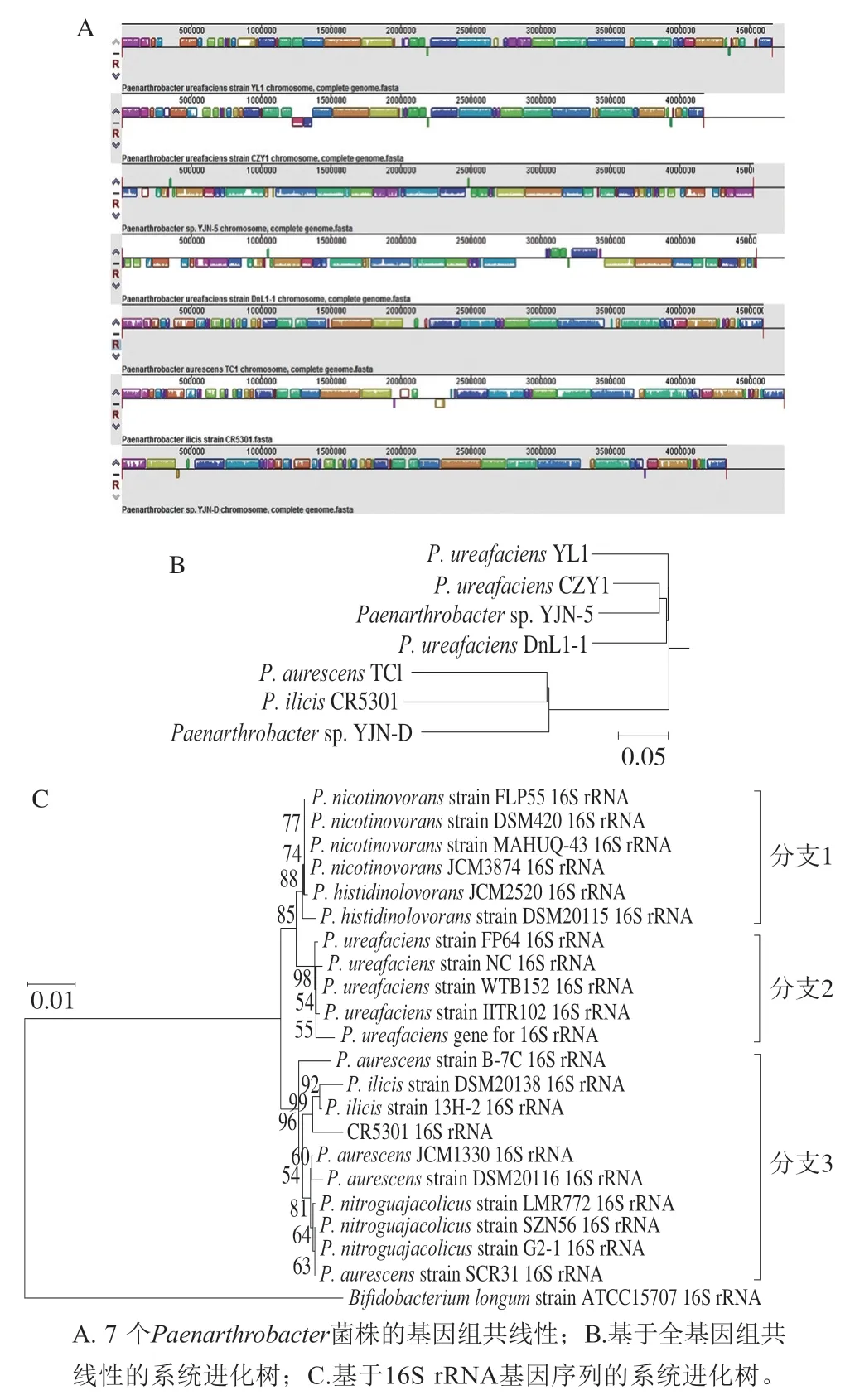
图5 P. ilicis CR5301基因组共线性和系统进化树Fig. 5 Genome collinearity and phylogenetic tree of P. ilicis CR5301
由图5A可知,CR5301基因组是所有完整测序的中基因组最大的菌株,CR5301基因组94%以上区域与其他基因组显示高度同源性和良好共线性。在7 个基因组中,CR5301和TC1基因组之间只在中间发生局部的缺失和插入突变,没有发生倒位突变,显示最高的同源性和最好的共线性。CR5301和CZY1基因组大小差异最大,表明它们之间发生最大范围的缺失和插入突变,同时两个基因组之间还发生1 个145 kb大片段(1 217 961~1 363 587 bp)和3 个小片段的倒位突变,因此这两个基因组具有最低的同源性和共线性。此外,全基因组比较分析发现,CR5301的基因岛GI01与其他6 个菌株共有,而基因岛GI02、GI03、GI04和前噬菌体Ph01均为菌株CR5301独有,这些独有的可移动元件,尤其是基因岛GI02携带的碳水化合物活性酶很可能赋予CR5301独特的碳水化合物代谢能力。图5B可以更加直观、清晰显示7 个菌株基因组的系统进化关系,CR5301和TCI、YJN-D明显聚类为一个分支,3 个菌株和YJN-5聚类为另一个分支,这表明在全基因组水平上CR5301与具有更近的亲缘关系,而与的亲缘关系较远。
另一方面,CR5301和所有已知物种16S rRNA基因的系统进化树(图5C)分析显示,所有物种形成3 个明显聚类的进化分支,分支1由和聚类形成,分支2由单独形成,分支3由、和聚类而成,CR5301明显与的2 个菌株聚类。这些结果表明,在中,CR5301与亲缘关系较近,与亲缘关系较远,这与全基因组系统进化树(图5B)分析结果一致,与Busse对菌属分类定义时的研究结果一致。这进一步证明了基因组共线性分析结果的有效性和准确性,为CR5301的系统进化、分类鉴定和比较基因组学研究提供了可靠的佐证和依据。
2.5 P. ilicis CR5301碳水化合物活性酶
CAZy数据库是碳水化合物活性酶类专业数据库。根据蛋白质结构域中氨基酸序列的相似性,可将不同来源的碳水化合物活性酶分成6大类。经CAZy注释分析,CR5301共编码174 个碳水化合物活性酶基因,这些基因的功能分类信息见图6。综合COG、GO、KEGG和CAZy数据库注释分析,分别找到379、163、301 个和174 个碳水化合物活性酶基因,去冗余分析后,CR5301基因组共含有523 个碳水化合物活性酶基因。

图6 P. ilicis CR5301基因的CAZy注释和分类Fig. 6 CAZy annotation and classification of P. ilicis CR5301’s genes
2.6 P. ilicis CR5301转化RC关键糖苷酶分析
为进一步筛选和挖掘CR5301转化RC功能的关键酶基因,对底物RC和终产物甜菊醇的分子结构进行比较分析。由图7可知,RC转化为终产物甜菊醇涉及4 个化学键的水解,包括C19位的-葡萄糖基酯键、C13位与槐糖基连接的糖苷键、-1,3-葡萄糖苷键和-1,2-鼠李糖苷键,因此可以水解这些化学键的糖苷酶很可能是CR5301转化RC的关键酶。以往研究表明,少数-半乳糖苷酶具有水解甜菊糖甙C19位和C13位糖基侧链的功能,因此,除了-葡萄糖苷酶和-鼠李糖苷酶,-半乳糖苷酶也可能是RC转化的关键酶。经功能注释检索分析,综合COG、GO、KEGG和CAZy数据库注释并去冗余获得的523 个碳水化合物活性酶中,发现11 个-葡萄糖苷酶基因、5 个-半乳糖苷酶基因、1 个-鼠李糖苷酶基因和1 个同时注释两种糖苷酶活性基因(注释信息见表3)。同时利用这3 个酶关键词在NR、Swiss-Prot和Pfam数据库注释基因中进行检索,没有发现新的基因。因此,通过上述7 个数据库综合分析,发现CR5301含有18 个转化RC关键糖苷酶候选基因。
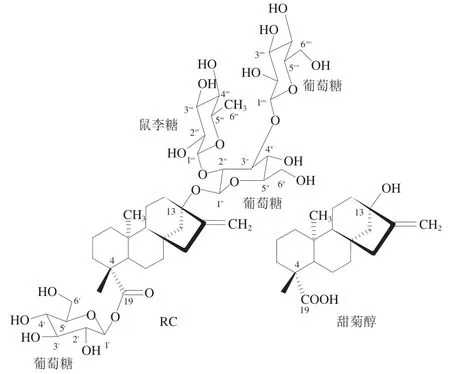
图7 RC和甜菊醇化学结构Fig. 7 Chemical structures of rebaudioside C and steviol
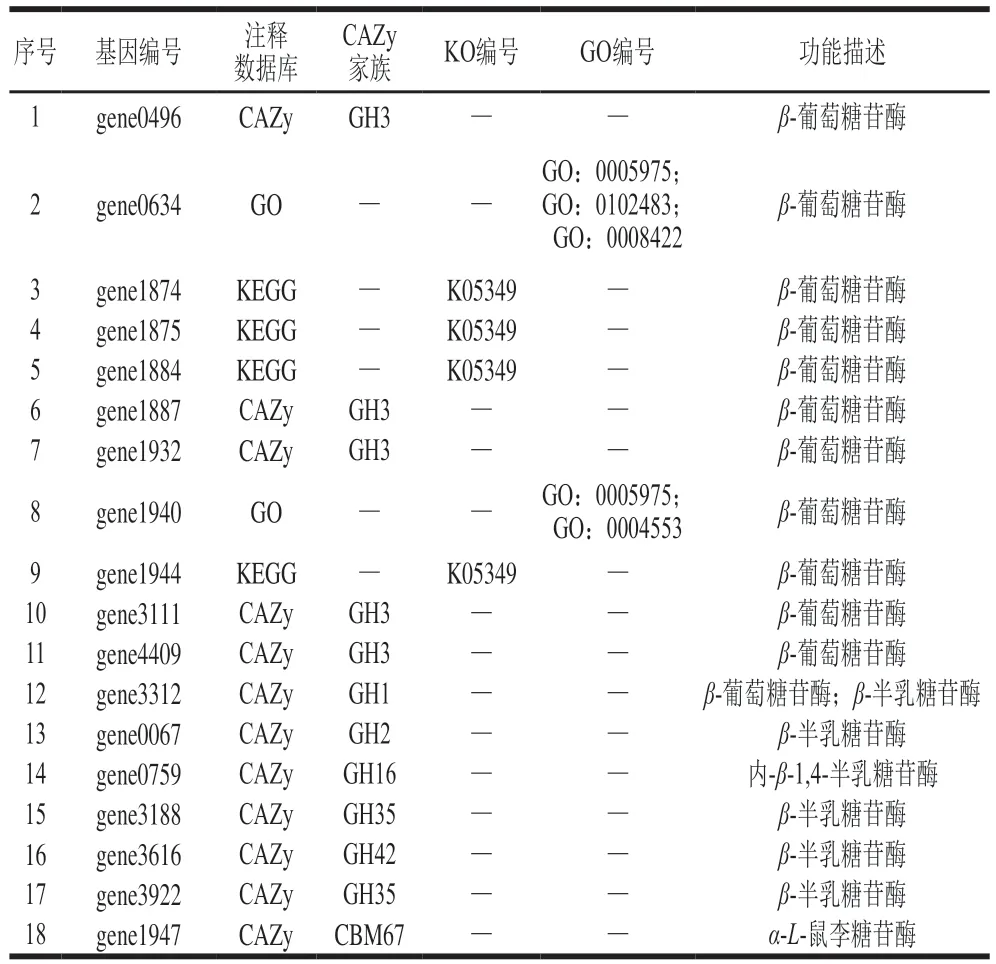
表3 P. ilicis CR5301转化RC的关键酶候选基因Table 3 Candidate genes for key enzymes of strain CR5301 for transforming RC
为了进一步了解关键糖苷酶酶学性质,通过ProtParam等软件对这些糖苷酶的物化性质和二级结构进行了预测分析,结果如表4所示。分析发现,4 个糖苷酶基因(gene0496、gene0759、gene3111、gene4409)具有信号肽序列,其中2 个基因(gene3111、gene4409)产物具有跨膜结构域,同时亲水系数预测显示只有这2 个基因产物为非水溶性蛋白,这表明gene0496和gene0759产物可能是分泌蛋白,gene3111和gene4409产物可能是膜蛋白,因此,这4 个基因对于研究CR5301胞外RC转化能力更有针对性,而其他不含有信号肽和跨膜结构域基因,很可能负责CR5301细胞质中RC转化功能。另一方面,蛋白质不稳定系数预测显示,大多数基因产物不稳定系数小于40阈值,只有4 个基因产物稍大于40,这表明这些糖苷酶大多数稳定性较好,只有4 个蛋白稳定性较差,有利于今后的工业化应用。由表4可知,这些糖苷酶二级结构中主要以无规卷曲和-螺旋为主。酶的功能部位常常位于无规卷曲构象区域,因为无规卷曲可使空间结构中的自由能达到最大而促进蛋白质结构稳定。18 个糖苷酶中,gene0759的二级结构中无规卷曲含量最高55.10%,此外不稳定系数最低23.38,表明gene0759产物结构和耐热稳定性可能最佳。
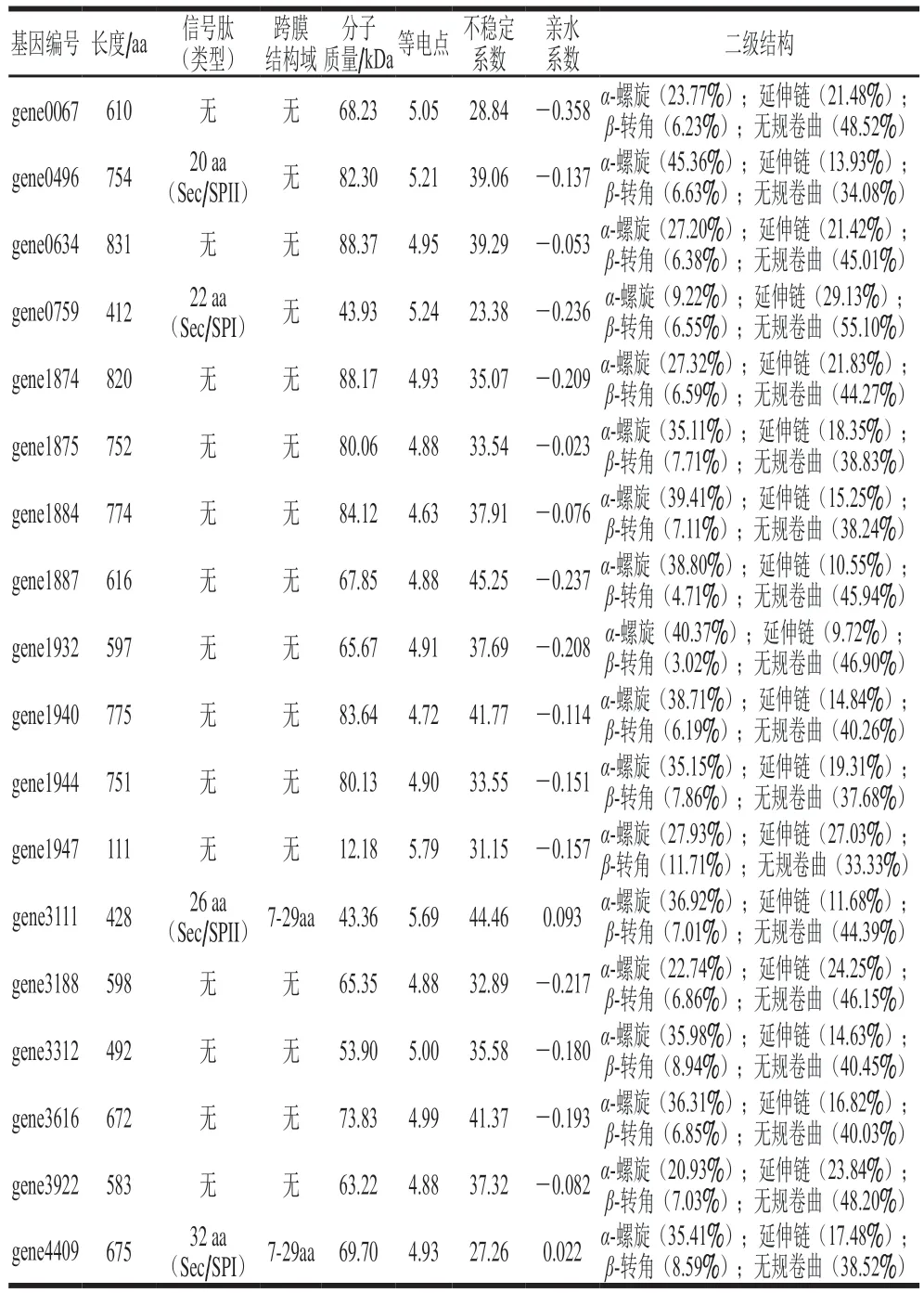
表4 关键酶基本物化性质和二级结构Table 4 General physicochemical properties and secondary structure composition of key enzymes
3 结 论
基因组装和预测分析发现,CR5301基因组为一个闭合环状染色体DNA分子,不含有质粒,染色体基因组序列全长4 748 281 bp,GC含量62.92%,共含有4 458 个编码基因,包括18 个rRNA操纵子和54 个tRNA。同时,该基因组含有4 个基因岛、1 个前噬菌体和14 个CRISPR-Cas编码序列,基因岛GI01可能来源于噬菌体,基因岛GI02可能来源于接合性质粒,前噬菌体Ph01和基因岛GI03可能起源于分枝杆菌噬菌体D29。
CR5301是物种第1个测定基因组完成图的菌株,基因组共线性分析发现,也是属已知基因组最大的菌株。全基因组系统进化分析发现,CR5301与具有更近的亲缘关系,而与的亲缘关系较远。16S rRNA基因系统进化树分析结果同样支持这个结论。
基因注释和功能分类分析发现,CR5301基因组在NR、Swiss-Prot、Pfam、COG、GO和KEGG数据库中分别注释到4 458、3 095、3 600、3 749、2 439 个和1 975 个功能基因。通过基因功能统计分类,在COG、GO、KEGG和CAZy数据库中,分别找到379、163、301 个和174 个碳水化合物活性酶基因,去冗余分析后,CR5301基因组共含有523 个碳水化合物活性酶基因。之后通过底物RC和终产物甜菊醇分子结构比较,精准定位了RC转化需要水解的化学键和对应的糖基水解酶,最终在7 个数据库注释基因中发现18 个转化RC关键糖苷酶候选基因。最后,通过生物信息学分析,预测了18 个糖苷酶的物化性质和二级结构,为今后进一步缩小CR5301转化RC的关键酶基因提供了非常重要的参考和基础。
总之,CR5301基因组完成图的测定,不仅为CR5301的RC转化机制研究提供清晰、完整的遗传信息,并且为物种的遗传、进化、生理、代谢研究首次提供了完整、可靠的参考基因组序列,这对今后的生物学研究具有重要的参考价值和普遍的借鉴意义。
