牛FoxO1基因启动子转录调控分析
2022-09-23赵毅昂魏大为张久盘宋雅萍王兴平罗仍卓么
赵毅昂 魏大为,2* 张久盘 宋雅萍 姜 超 王兴平,2 罗仍卓么,2 马 云,2
(1.宁夏大学 农学院,银川 750021; 2.宁夏大学 宁夏反刍动物分子细胞育种重点实验室,银川 750021; 3.宁夏农林科学院 动物科学研究所,银川 750021)
牛肉因其具有营养丰富、低脂肪高蛋白等特点而深受人们所喜爱。近些年来我国牛肉产量稳步提升,但仍然供不应求,存在巨大缺口。骨骼肌作为肉牛的重要的组成部分,其生长发育及与个体产肉能力和肉品质密不可分。此外,肌内脂肪(Intramuscular fat,IMF)含量与牛肉的嫩度和风味密切相关。影响骨骼肌和脂肪生成的因素有很多,大量研究发现营养、环境和遗传等是调控动物骨骼肌和脂肪生成的主要因素,但更多取决于基因调控。因此,筛选并鉴定调节骨骼肌与脂肪生长发育的功能基因对肉牛产肉力和肉质的改良有重要意义。
本研究前期通过全基因组关联分析(Genome-wide association study,GWAS)秦川牛的生长性状,筛选发现FoxO1
基因与秦川牛个体体重和体高关联密切,且Sun等研究发现叉头框转录因子1(Forkhead box,FoxO1)的多态性与秦川牛的体长显著相关。通过进一步对FoxO1
深入挖掘发现,FoxO1
作为Fox
家族17个亚群中的成员之一,受胰岛素磷脂酰肌醇-3激酶PI3K/Akt信号通路的负反馈调节,参与机体多种器细胞代谢、自噬以及DNA损伤修复等过程,在机体胰岛β细胞分化和氧化应激中也扮演着重要角色。除以上功能外,大量研究发现FoxO1
基因在动物骨骼肌发育和脂肪的生成中也发挥着重要作用。如Yang等研究发现FoxO1
的表达量和猪Ⅰ型肌纤维的含量成反比;Kamei等报道,FoxO1
基因参与骨骼肌的发育和Ⅰ型纤维正常发生,敲除FoxO1
基因的小鼠骨骼肌功能受损,全身肌肉发育不全。另一方面,FoxO1
在脂肪组织中大量表达,并参与其分化过程。研究发现,FoxO1
可以抑制脂肪生成和储存的主要调节因子PPARγ(Peroxisome proliferator-activated receptor γ),进而调控脂肪;此外,FoxO1
可通过诱导脂肪细胞中脂肪甘油三酯脂肪酶(ATGL)和溶酶体脂肪酶(LIPA)来调节脂质分解代谢。这些研究均表明FoxO1
基因在动物生长发育过程中十分重要,并有可能在牛的脂肪和肌肉中扮演着重要角色,因此,探究其在牛上的调控机制及基因功能尤为重要。启动子作为关键的转录元件,对转录因子结合进而调控基因的表达水平起到决定性作用。对FoxO1
基因启动子的探究,是揭示其调控骨骼肌和脂肪形成过程中转录调控机制的重要手段。同一基因启动子在不同物种间的核心区和转录因子结合均有差异,且目前尚未发现牛FoxO1
基因启动子转录调控机制的报道,因此,本研究以秦川牛血液基因组DNA为模板,扩增了FoxO1
基因启动子和7个逐段缺失的启动子片段,并构建双荧光素报告载体,将其转染至C2C12和3T3-L1细胞系,通过酶活检测来确定FoxO1
基因的启动子核心区域,进一步通过在线网站预测影响牛FoxO1
基因的关键转录因子,并通过定点突变技术确定核心区的关键转录因子,初步探究了牛FoxO1
启动子的转录调控作用,为研究FoxO1
基因对牛肌肉脂肪发育过程的调控作用奠定了理论基础。1 材料与方法
1.1 材料和试剂
采集24月龄秦川牛血液样本;PrimeScriptTM RT Reagent Kit with gDNA Eraser(Perfect Real Time)反转录试剂盒、总RNA提取试剂盒、总DNA提取试剂盒、T4DNA连接酶、高保真PCR扩增酶GXL、限制性内切酶QuickCutKpn
I和QuickCutHind
Ⅲ和大肠杆菌DH5α感受态细胞均购自TaKaRa公司;Lipofectamine 3000 Reagent转染试剂盒购自Invitrogen公司;pGL3-Basic 质粒、去内毒素质粒提取试剂盒、DNA胶快速回收试剂盒、双荧光素酶基因内参pRL-TK载体和Dual-Luciferase Reporter Assay System 双荧光素酶报告系统试剂盒购自Promega公司;Fast MultiSite Mutagenesis System定点突变试剂盒购自Transgen Biotech公司;OPTI-MEM培养基和胎牛血清(Fetal bovine serum,FBS)购自Gibco公司;DMEM培养基购自Hyclone公司。1.2 试验方法
1
.2
.1
牛FoxO1
基因结构及蛋白序列分析根据NCBI数据库(https:∥www.ncbi.nlm.nih.gov)中已公布的海福特牛FoxO1
基因的序列信息和转录起始位点,利用Ensembl(https:∥asia.ensembl.org/index.html)在线工具初步预测牛FoxO1
基因的启动子位置;根据GenBank数据库中已公布的牛、绵羊、山羊等物种FoxO1
基因的蛋白质序列信息,利用ProtPram 在线程序(https:∥web.expasy.org/protparam/)分析蛋白质的理化性质;使用Mega 5.0软件构建不同物种FoxO1蛋白质系统进化树。1
.2
.2
牛FoxO1
基因启动子克隆提取秦川牛血液基因组DNA,于4 ℃冰箱保存备用。参照GenBank数据库中已公布的牛FoxO1
基因序列,针对其启动子区域使用Primer 5.0软件设计引物FoxO1
-PF和FoxO1
-PR(表1),以血液基因组DNA为模板,使用Takara公司的GXL高保真酶扩增牛FoxO1
基因的启动子区(位置:-1 722/+198)。PCR 反应总体系为:上、下游引物(10 μmol/L) 各2 μL;2×PrimeSTAR GXL Buffer 25 μL;PrimeSTAR GXL DNA Polymerase 2 μL;模板50 ng;ddHO补齐至50 μL。反应条件:98 ℃ 5 min;98 ℃ 10 s,60 ℃ 15 s,68 ℃ 20 s,30个循环。使用1%琼脂糖凝胶电泳对扩增产物进行检测,对检测正确的目的条带进行切胶回收纯化,使用T4DNA连接酶将其连接于PMD19-T(Simple)载体后将其转化到大肠杆菌DH5α感受态细胞中,12 h后进行单克隆筛选后测序。1
.2
.3
牛FoxO1
基因启动子逐段缺失片段的克隆通过Primer premier 5.0软件设计7条用于启动子递减缺失片段扩增的上游引物FoxO1
-P1~FoxO1
-P7和1条固定的下游引物FoxO1-R(引物信息见表1,斜体碱基为上、下游引物5′端分别添加的Kpn
I、Hind
Ⅲ双酶切位点),来鉴定牛FoxO1
基因启动子的核心区域。使用在上一步获得的测序正确的牛FoxO1
基因启动子片段为模板进行启动子递减缺失片段的PCR扩增(扩增条件及体系同上),扩增片段测序鉴定正确后可用于下一步试验。1
.2
.4
双荧光素酶报告载体的构建使用限制性内切酶Kpn
Ⅰ和Hind
Ⅲ于37 ℃条件下分别双酶切7个牛FoxO1
基因逐段缺失启动子片段和pGL3-Basic载体2 h,用T4 DNA连接酶将双酶切后的载体和7个启动子片段在16 ℃金属浴恒温仪上逐一连接后于大肠杆菌DH5α感受态细胞中进行转化,12 h后挑选阳性单克隆菌落扩繁,小提质粒后通过双酶切鉴定。将鉴定正确的菌液大量扩繁后使用去内毒素试剂盒提取质粒,测定纯度和质量浓度。将重组质粒分别命名为:pFoxO1-1 722/+198、pFoxO1-1 330/+198、pFoxO1-1 004/+198、pFoxO1-717/+198、pFoxO1-471/+198、pFoxO1-285/+198和pFoxO1-27/+198。表1 牛基因启动子及其递减缺失片段克隆引物
Table 1 Primers were used in promoter and diminishing deletion fragment cloning of gene
用途Usage引物名称Primername引物序列(5'→3')Primersequence(5'→3')退火温度/℃Tm扩增片段长度/bpProductlength扩增区域Amplifiedregion启动子克隆PromotercloningFoxO1-PFGACGGGTGAGAATGTCAACGA601 920-1 722/+198FoxO1-PRCCTCCGGGTCCGCTGCACTGACFoxO1-P1CGGGGTACC GACGGGTGAGAATGTCAACGA601 920-1 722+198FoxO1-P2CGGGGTACC ATGGGAGGGGGCGGTGATTGAC601 528-1 330/+198FoxO1-P3CGGGGTACC TGCCTTCGGGTCTCAGGGTGC601 202-1 004/+198启动子逐段缺失片段克隆PromoterdeletionfragmentscloningFoxO1-P4CGGGGTACC ACACGCGTGTGTGCGGCCCGC60915-717/+198FoxO1-P5CGGGGTACC CGGGCTCCCGAGCGCAGGTCGA60669-471/+198FoxO1-P6CGGGGTACC ACCAGAAAATAATAACAGAGAG60483-285/+198FoxO1-P7CGGGGTACC CGGCGGCAGATCCCGTAAGTC60225-27/+198FoxO1-RCCCAAGCTT CCTCCGGGTCCGCTGCACTGAC
1
.2
.5
细胞培养、转染及酶活测定使用含10%胎牛血清和1%双抗的DMEM培养基培养小鼠成肌细胞(C2C12)系和脂肪细胞(3T3-L1)系,将其分别接种至24孔板,每孔细胞的量为1×10个,置于37 ℃、体积分数5% CO2培养箱中培养,待细胞汇合度达70%~80%时,分别将构建的0.8 μg牛FoxO1
基因启动子不同的5′端递减缺失片段重组质粒和0.02 μg内参质粒pRL-TK共转染至C2C12和3T3-L1细胞系中,转染步骤详见Lip3000转染试剂盒说明书。以pGL3-Basic 质粒为阴性对照(n
=3)。转染48 h后裂解细胞,通过检测并计算萤火虫荧光素酶活性(F
值)和海肾荧光素酶活性(R
值)的比值(F
/R
)来确定FoxO1
基因启动子核心区域。数据采用SPSS软件中2个独立样本t
检验进行方差分析。1
.2
.6
关键转录因子预测和鉴定通过在线软件Genomatix(http:∥www.Genomatix.de/cgibin∥matinspector)和JASPAR(http:∥jaspar.genereg.net/)预测启动子核心区潜在转录结合因子,设置阈值大于90%,通过将两者预测结果进行数据库比对,筛选出交集,标注牛FoxO1
基因启动子核心区转录因子结合序列。根据在线软件Genomatix和JASPAR预测的潜在转录因子,使用Fast MultiSite Mutagenesis System定点突变试剂盒进行定点突变,设计定点突变引物(表2),并进行突变片段的制备,具体操作详见说明书(目录号:FM201)。将得到的突变型和野生型分别转染C2C12细胞系后测定酶活,操作方法同上。
表2 定点突变引物信息
Table 2 Primers used in site-directed mutation
引物名称Primer name引物序列(5'→3')Primer sequence (5'→3')mMEF2A-FACAGAGAGCCAAAAATTTTAAAAAAAACCAGAGAmMEF2A-RAAAATTTTTTGGCTCTCTGTTATTATTTTCTGGTmSREBF1-FGTCAAATAGAAAAATTTTCCACTACCCCCCACCmSREBF1-RAAAATTTTTCTATTTGACATGCACACTCGCTCGCmHOXA5-FCAGGCCGGCGCCCATAAAAGGCCCGGGCGGCGCCmHOXA5-RTTTTATGGGCGCCGGCCTGGGCGGGGCTCGGTTTTmKLF4-FTGGAAAACCGAGCTTTTCCCAGGCCGGCGCCCmKLF4-RAAAAGCTCGGTTTTCCACGGGGAGGTGGCAGmKLF5-FGGGGCCGTCCGTCAGTTTTGGGCCGGCGCGCGCGmKLF5-RAAAACCCGGCCGCGCGCGCGGCGGCGGCGGCG
2 结果与分析
2.1 牛FoxO1基因结构及蛋白序列分析
查找NCBI(NC_037339.1)及Ensembl数据库公布的牛FoxO1
基因信息发现,牛FoxO1
基因位于12号染色体上,全长9 3695 bp;开放阅读框(Open reading frame,ORF)分析发现,牛FoxO1
包括3个外显子和2个内含子;确定FoxO1
转录起始位点鸟嘌呤(G)为+1位置,并向上游查找2 000 bp的序列,初步预测其启动子位置(图1)。理化性质分析发现,FoxO1
基因编码665个氨基酸,分子量(Molecular weight)为69958.93,理论pI值为6.38,不稳定性指数(Instability index)为59.53(>40.0),推测牛FoxO1蛋白为不稳定蛋白。利用GenBank数据库中公布的10个物种的蛋白质序列构建系统发育树,经过相似性比对后,结果表明(图2和表3),牛FoxO1蛋白与山羊和绵羊相似性最高分别为97.76%和97.60%,且FoxO1蛋白在除原鸡以外的不同物种间较为保守,尤其在复胃动物(绵羊、山羊和牛)之间进化保守。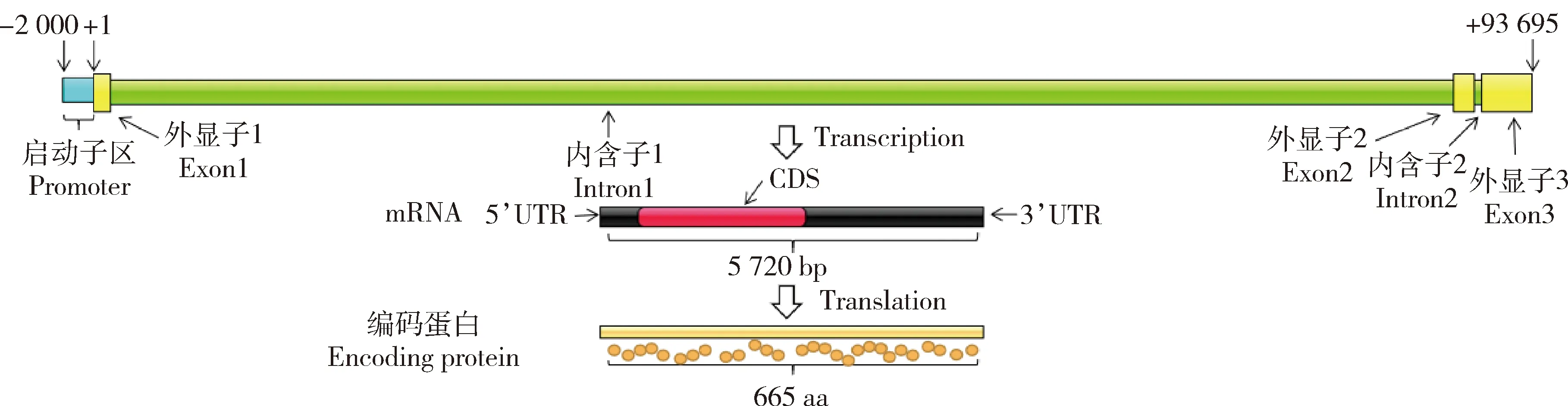
图1 牛FoxO1基因结构示意图Fig.1 Schematic diagram of bovine FoxO1 gene structure
表3 FoxO1蛋白质序列登录号及相似性比对结果
Table 3 GenBank number of the FoxO1 protein sequence and homology comparison results
物种Specie登录号GenBank No.相似性/%Similarity牛Bos taurusXP_024855821.1绵羊Ovis ariesXP_027829397.197.60犬Canis lupusXP_038290656.194.91人Homo sapiensQ12778.293.69小鼠Mus musculusQ9R1E0.290.24山羊Capra hircusXP_017912175.197.76猪Sus scrofaNP_999179.395.05家猫Felis catusXP_023106848.195.35大鼠Rattus norvegicusNP_001178775.189.34原鸡Gallus gallusNP_989659.175.04
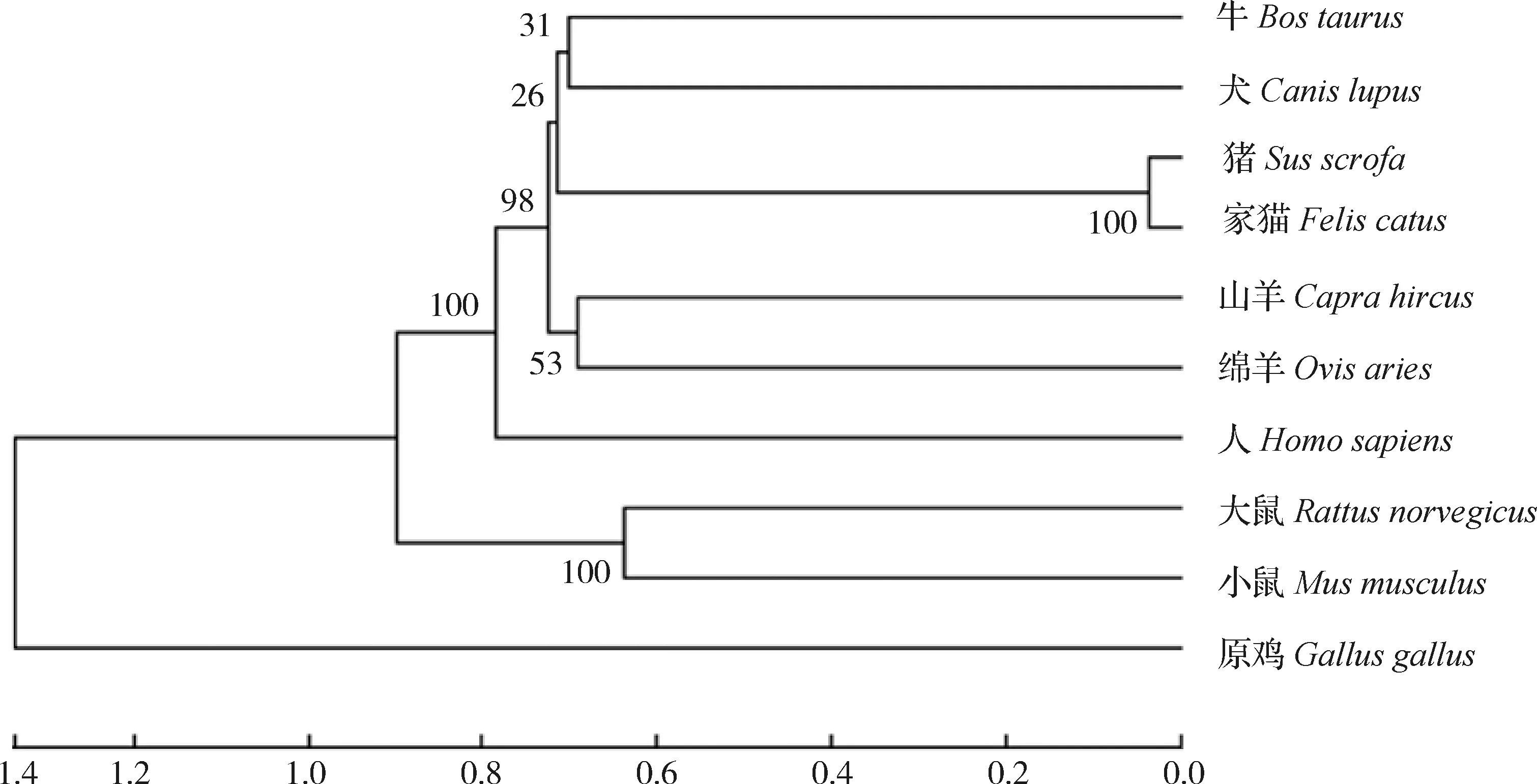
图2 牛与其他物种FoxO1蛋白质序列进化树Fig.2 FoxO1 protein sequence phylogenetic tree among Bos taurus and other species
2.2 牛FoxO1基因启动子片段的扩增
PCR扩增牛FoxO1
基因启动子序列,凝胶电泳检测结果显示(图3(a)),该条带与目的条带大小基本吻合,且条带整齐、无杂带和拖带。经测序鉴定,其长度为1 920 bp,与GenBank中已公布的序列对比完全一致,可用于后续试验。
(a)PCR扩增牛FoxO1基因的启动子片段。M,DL 4500 DNA Marker;1,启动子-1 722/+198区扩增结果;(b)牛FoxO1基因启动子逐段缺失片段扩增电泳图。M,DL 2000 DNA Marker;1~7 分别为FoxO1-P1/FoxO1-R~FoxO1-P1/FoxO1-R7扩增产物;(c)牛FoxO1基因启动子双荧光素报告重组质粒双酶切鉴定电泳图。M, DL 2000 DNA Marker;1~7,分别为Kpn Ⅰ、Hind Ⅲ双酶切pGLFoxO1-1 722/+198、pGLFoxO1-1 330/+198、pGLFoxO1-1 004/+198、pGLFoxO1-717/+198、pGLFoxO1-471/+198、pGLFoxO1-285/+198和pGLFoxO1-27/+198的结果。
2.3 牛FoxO1基因启动子核心区鉴定
2
.3
.1
启动子逐段缺失片段的扩增使用逐段缺失引物进行PCR扩增牛FoxO1
基因的逐段缺失启动子区片段,电泳检测后(图3(b))分别获得了1 920、1 528、1 202、915、669、483和225 bp的单一条带,经测序验证后与预期片段长度一致,可用于后续试验。2
.3
.2
FoxO1
基因启动子核心区的确定经过双酶切鉴定后(图3(c)),成功构建了牛FoxO1
基因逐段缺失的pGL-Basic重组质粒,将其分别命名pGLFoxO1-1 722/+198、pGLFoxO1-1 330/+198、pGLFoxO1-1 004/+198、pGLFoxO1-717/+198、pGLFoxO1-471/+198、pGLFoxO1-285/+198和pGLFoxO1-27/+198。经测序验证无误后,抽提去内毒素质粒,将其转染至C2C12和3T3-L1细胞系。通过分析逐段缺失启动子活性片段发现(图4),FoxO1
基因启动子-1 722/+198区域在两种细胞系中活性较好,进一步对缺失区域进行比对发现,缺失-285/-27片段后,双荧光素报告载体pFoxO1-27/+198在C2C12及3T3-L1细胞中的活性较pFoxO1-285/+198极显著下降(P
<0.01)。由此推测,FoxO1
基因5′端上游1.9 kb序列为具有调控基因转录活性功能的启动子区;此外缺失-285/-27 片段时酶活极显著下降,由此确定-285/-27 区域为牛FoxO1
基因启动子核心区。另一方面,研究发现FoxO1
基因在C2C12细胞系的转录活性高于3T3-L1,这一结果说明了在C2C12细胞系中更适合FoxO1
基因转录活性的测定。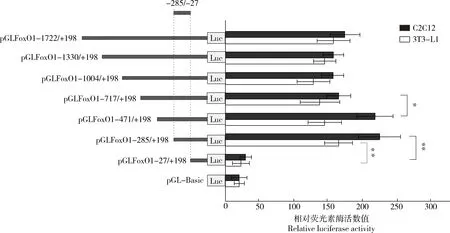
*和**分别表示差异显著(P<0.05)和极显著(P<0.01)。下同。 * and ** indicate significant difference (P<0.05) and extremely significant difference (P<0.01), respectively. The same below.图4 逐段缺失检测牛FoxO1基因启动子核心区Fig.4 Detection of core promoter region of bovine FoxO1 gene by series deletion
2.4 关键转录因子预测及鉴定
利用Genomatix和JASPAR软件对牛FoxO1
基因启动子核心区域(-285/-27片段)的关键转录因子进行预测分析。结果显示,牛FoxO1
基因启动子核心区包含肌细胞增强因子2(MEF2A)、固醇调节元件结合转录因子1(SREBF1)、同源异型盒基因5(HOXA5)、Krüppel样因子4(KLF4)和Krüppel样因子5(KLF5)等关键转录因子的结合位点(图5(a))。因此初步推测,MEF2A、SREBF1、HOXA5、KLF4和KLF5转录因子可能对FoxO1
基因的转录活性有重要的调控作用。将进一步构建的野生型载体和定点突变后构建的突变型载体分别转染C2C12细胞系中测定酶活性。结果显示,突变MRF2A位点后,酶活相比野生型降低了49.48%(P
<0.01);突变SREBF1位点后,酶活相比野生型升高了1.73%(P
>0.05);突变KLF4位点后,酶活相比野生型升高了35.15%(P
<0.01);突变HOXA5和KLF5位点后,酶活相比野生型分别降低了23.73%(P
<0.01)和33.75%(P
<0.05)(图5(b))。此结果说明,MEF2A、KLF4、HOXA5和KLF5转录因子是调控牛FoxO1
转录活性的重要因子,MEF2A和KLF4起到正向调控作用,HOXA5和KLF5起到负向调控的作用;而突变SREBF1位点后,酶活差异不显著,说明SREBF1对牛FoxO1
转录活性没有调控作用。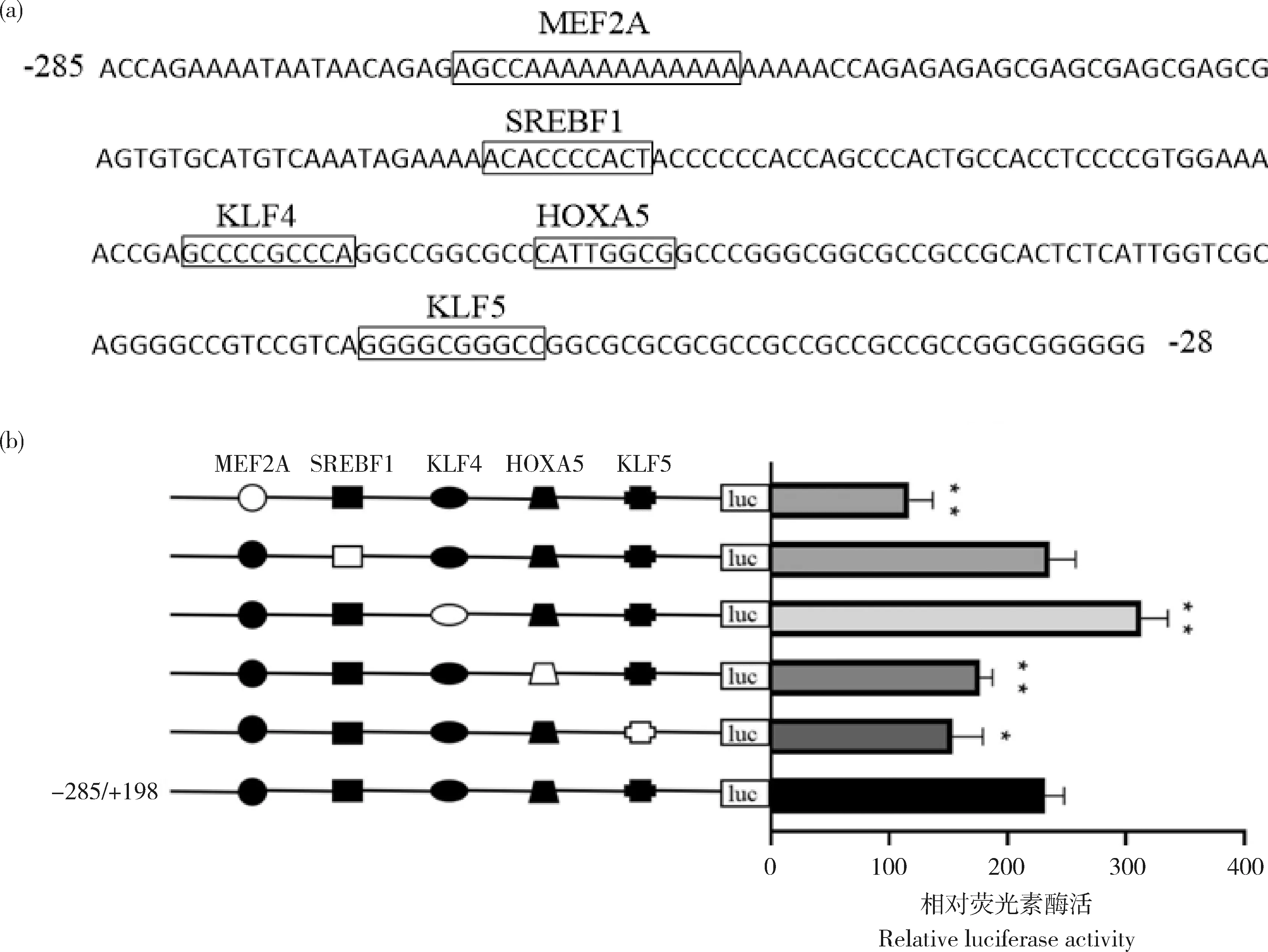
(a)牛FoxO1基因核心区域转录因子预测。方框表示相对于的转录因子结合位点。(b)定点突变关键转录因子酶活测定。纵坐标左侧为-285/+198 bp区域转录因子,白色图形为突变型,黑色图形为野生型。
3 讨论与结论
FoxO1
作为Fox
家族17个亚群中的成员之一,在脂肪细胞分化、肌纤维形成等重要生物学功能中起到重要的作用。启动子为转录调控中关键元件,其核心区存在着转录因子的结合位点,目前已有大量研究发现这些转录因子可以识别特定DNA序列,并与基因组中的调控区域结合,从而激活或抑制靶基因的转录来调节mRNA的转录水平。因此确定牛FoxO1
基因核心启动子并鉴定其转录因子结合位点对研究牛FoxO1
基因的转录调控机制至关重要。本研究通过构建逐段缺失片段,进行双荧光素酶活检测确定FoxO1
启动子的核心区为-285/-27。利用在线软件预测到FoxO1
启动子核心区域有MEF2A、SREBF1、HOXA5、KLF4和KLF5共5个转录因子的结合位点。其中,MEF2A
作为MEF2
基因家族中的一员,肌纤维稳态、对肌纤维形成、成肌细胞的分化、心肌细胞增殖、骨骼肌干细胞和再生有促进作用。研究表明,MEF2A
是骨骼肌生成过程中的重要转录调控蛋白,在成肌细胞增殖和分化中发挥重要作用,其沉默会抑制成肌细胞分化,并显著下调钙调磷酸酶肌小节结合蛋白2(MYOZ2)的表达量。HOXA5
基因作为同源框基因(Homeobox genes)家族中重要发育转录因子之一,与个体组织和器官发育密切相关。HOXA5
基因主要参与BMP4/Samd1信号通路,在脂肪组织形成和脂肪细胞分化过程中发挥着重要的调控作用。研究表明,沉默HOXA5
可有效减少3T3-L1细胞中的脂质积累。Kr
üppel
样因子(Krüppel-like factors,KLFs)家族为锌指蛋白类转录因子,具有参与调控细胞增殖和胚胎发育等生命过程的重要作用,在细胞分化和细胞周期阻滞中也扮演着重要角色。KLF4
和KLF5
均为KLF家族成员,其中KLF4作为脂肪形成的直接早期调节因子,可以诱导同为脂肪形成的早期的C/EBPβ(Enhancer binding protein β);若敲除KLF4
基因则会下调C/EBPβ水平,从而抑制脂肪生成。KLF5
因在肠上皮细胞中表达水平较高也被称为肠Krüppel样因子,有研究发现KLF5
对肌肉脂质代谢有着重要作用,如KLF5
可参与Krox20(Early growth response 2,EGR2/Krox20)诱导的C/EBP/PPARγ通路,KLF5
由C/EBPβ、C/EBPδ激活并与C/EBPα、C/EBPβ和C/EBPδ协同激活脂肪重要调节因子PPARγ2的表达。本研究通过定点突变牛FoxO1
基因启动子核心区KLF4和KLF5结合位点,发现FoxO1
转录活性分别上升和下降,这说明KLF4和KLF5对FoxO1
基因转录活性有负向和正向调控作用。综上,MEF2A、HOXA5、KLF4和KLF5均在肌肉或脂肪形成过程中起着重要的作用且其对牛FoxO1
基因转录活性具有重要调控作用,但基因的调控机制十分复杂,因此还需使用凝胶迁移阻滞试验(EMSA)和染色质免疫共沉淀(ChIP)等技术来进一步验证MEF2A、HOXA5、KLF4和KLF5转录因子是否均能结合到FoxO1
基因启动子核心区,进一步探究FoxO1
基因的转录调控机制。本研究成功克隆出牛FoxO1
基因长1 920 bp的启动子序列,确定了牛FoxO1
基因启动子的核心区为-285/-27,进一步验证出MEF2A、KLF4、HOXA5和KLF5为牛FoxO1
基因启动子核心区的关键转录因子。