北京地区番茄青枯病健康和发病植株根际细菌群落比较分析
2022-09-16马毅楠谷医林魏海雷张晓霞
马 荣,马毅楠,王 星,谷医林,魏海雷,张晓霞
(中国农业科学院农业资源与农业区划研究所/农业农村部农业微生物资源收集与保藏重点实验室,北京100081)
番茄青枯病是由茄科雷尔氏菌(Rasltonia solanacearum,简称青枯菌)引起的一种毁灭性土传病害[1],青枯菌寄主范围广泛,可侵染包括茄科植物在内的400多种植物[2,3]。青枯病在我国番茄种植区大面积发生且危害严重,目前尚未发现高效的防治方法。生物防治因其对环境友好且高效的特点而备受关注。根际促生菌(Plant Growth-Promoting Rhizobacteria,PGPR)是主要的生防微生物资源,其特点为增加土壤微生物群落多样性或增强植物对病原菌的抗性[4,5]。植物根际是植物根部与土壤之间动态而复杂的界面,根际微生物是一种特定的微生物群落,对于植株的生长、健康和抵御病原菌的侵染有着重要作用[6,7]。青枯菌的入侵能够破坏根际微生物群落组成[8],导致根际土壤中的革兰氏阳性菌(厚壁菌门和放线菌门)的失调进而促进了青枯病的发生[9]。因此,研究番茄健康与发病植株根际土壤微生物群落组成差异有助于深入了解青枯菌与其他群落成员之间的关系。此外,还可以利用构建相关性网络的方法来探讨根际微生物群落间的相互作用,在网络结构中,相互作用的微生物成员之间的连接配置和分布可以对生态系统的功能和稳定性提供强有力的预测,也可以识别出菌群中占据重要位置的特定微生物[10,11]。
番茄是北京市设施蔬菜的重要果菜,对推动农业现代化等方面起着极其重要的作用,由于青枯菌喜好高温高湿的土壤环境,青枯病的发生大多集中在我国华中、华南和华东地区,而华北地区相对较少[12],但随着气候和种植方式的变化,青枯病的发生已经有北移的趋势。此外,由于北京保护地常年连作,造成了严重的土壤生物障碍。本研究通过解析北京地区番茄青枯病发生的根际群落变化特征并结合分离培养筛选有利于番茄抗病促生的有益菌群,为青枯病的生物防控提供重要参考。
1 材料与方法
1.1 供试材料
供试病原菌:为实验室保藏的青枯菌Ralstonia solanacearumGMI1000。
番茄根际土壤样品:采自北京市昌平区、顺义区、通州区、大兴区,具体采样点见表1,各个采样地点分别采集散土、健康和发病植株根际土壤,挖出整个根系,轻抖根系,收集紧密附着根上的土壤,即为根际土壤[13]。

表1 北京市番茄根际土壤采样统计表Table 1 Area of sampling collection statistical of tomato rhizosphere soil in Beijing region
1.2 土壤理化性质的测定
土壤理化性质测定参照《土壤农化分析》,土壤pH利用复合玻璃电极仪测定,土水比为1:2.5;利用电导率仪测定土壤电导率,土水比为1:5;有机质采用重铬酸钾容量法-外加热法;土壤全氮采用半微量开氏法;碱解氮采用碱解扩散法;土壤全磷和有效磷采用钼锑抗比色法;土壤全钾和速效钾采用火焰光度法;利用流动分析仪测定土壤悬液的铵态氮和硝态氮含量[14]。
1.3 番茄根际土壤基因组的提取与检测
利用FastDNA Spin Kit For Soil(MP)试剂盒提取DNA,首先称取0.5 g土壤样品,在灭菌后的研钵中利用液氮研磨至粉状,装入Lysing Matrix Etube 中,之后步骤按照MP试剂盒说明书进行操作,利用核酸定量仪(NanoDrop 2000)测定DNA核酸浓度与纯度,DNA于-20 ℃保存备用。
1.4 16S rRNA基因PCR扩增及测序样品的准备
16S rRNA基因扩增使用带有不同barcode序列的通用引物V3-V4区534F(5'-CCAGCAGCCGCGGTA AT-3')和 783R(5'-ACCMGGGTATCTAATCCKG-3')[15]。PCR 体系(50 μL):2×TaqPCR Mix 25 μL,534 F与783 R引物各2 μL,DNA 30 ng,ddH2O补足至50 μL;PCR反应程序为:95 ℃预变性3 min;94 ℃变性45 s,53 ℃退火1 min,72 ℃延伸1 min,共35个循环;72 ℃延伸10 min。使用OMEGA纯化试剂盒进行胶回收。用Nanodrop检测已纯化产物,按照Marker 250 bp条带的净光密度值进行标准化后进行等质量DNA(150 ng)混样,采用双端Nova-PE 250模式进行测序,由北京诺禾致源生物信息科技有限公司完成。
1.5 扩增子数据处理和分析
将16S rRNA基因的下机测序数据(正向、反向序列文件)与barcode序列(txt文件)上传至Galaxy平台[16];有序去除barcode序列与正反向引物序列;利用Flash工具将正反向序列拼接;去除非细菌序列后,按照97%的相似度水平生成OTU(Operational Taxonomic Units)表,借助在线平台MicrobiomeAnalyst(https://www.microbiomeanalyst.ca/)[17]和 ImageGP(http://www.ehbio.com/ImageG/)[18]进行数据分析。选用Canonical Correspondence Analysis(CCA)对土壤养分和土壤细菌群落的相关性进行分析,明确驱动细菌群落发生变化的主要影响因子;利用Chao1、Shannon和Simpson指数来表征番茄根际土壤微生物群落α多样性;基于Bray-Curtis距离对微生物群落相似性进行主成分分析(Principle coordinate analysis PCoA);基于SparCC算法进行群落相关性网络分析,利用Gephi(v 0.9.2)软件可视化;利用软件SAS 9.4完成单因素方差分析。
1.6 根际细菌分离培养与16S rRNA基因测序分析
取5 g鲜土置于盛有玻璃珠和45 mL无菌水的三角瓶中,室温120 r/min、振荡30 min,取1 mL土壤悬液用无菌水进行梯度稀释,选取稀释度10-4~10-6的土壤悬液各0.1 mL分别涂布在R2A培养基[19],30 ℃恒温培养7 d,取单个菌落并进一步纯化。最后以20%甘油悬液保存在-80 ℃冰箱。采用细菌16S rRNA基因通用引物27F和1492R[20]扩增供试菌的16S rRNA基因片段。每个反应体系25 μL:DNA 3 μL,引物各1 μL,2×TaqPCR Mix 12.5 μL,ddH2O 补至 25 μL。PCR 反应程序为:94 ℃预变性 3 min;94 ℃变性 30 s,55 ℃退火30 s,72 ℃延伸2.5 min;35个循环后于4 ℃保存。PCR产物由生工生物公司(北京)进行测序。将获得的16S rRNA基因序列与EzBioCloud(https://www.ezbiocloud.net/identify)和GenBank (www.ncbi.nlm.nih.gov/GenBank/)中菌株序列数据库进行比对鉴定。
1.7 根际细菌拮抗功能筛选
将病原菌在NA固体培养基[21]上活化,再转接NA液体培养基,同时培养试验菌株,30 ℃、120 r/min振荡培养24 h后,离心去掉上清液,加入适量无菌水调至浓度为OD600≈1。取200 μL病原菌菌液涂布于NA平板上,晾干后,缓慢滴加10 μL试验菌株,每株菌株3个重复,30 ℃培养箱倒置培养,观察并测量抑菌圈。
2 结果与分析
2.1 不同采样地区的土壤理化性质分析
对顺义区、大兴区、通州区和昌平区4个区的6个采样地区的散土理化指标进行方差分析(表2),发现6个采样地区的土壤养分均存在显著性差异,其中兴寿镇(XS)地区样品pH最高,为8.63,全氮、全磷、有机质和碱解氮含量最低;大东各庄村(DDG)各个理化指标较高,但其土壤盐分含量显著高于其他采样地区,即土壤盐渍化程度较重。
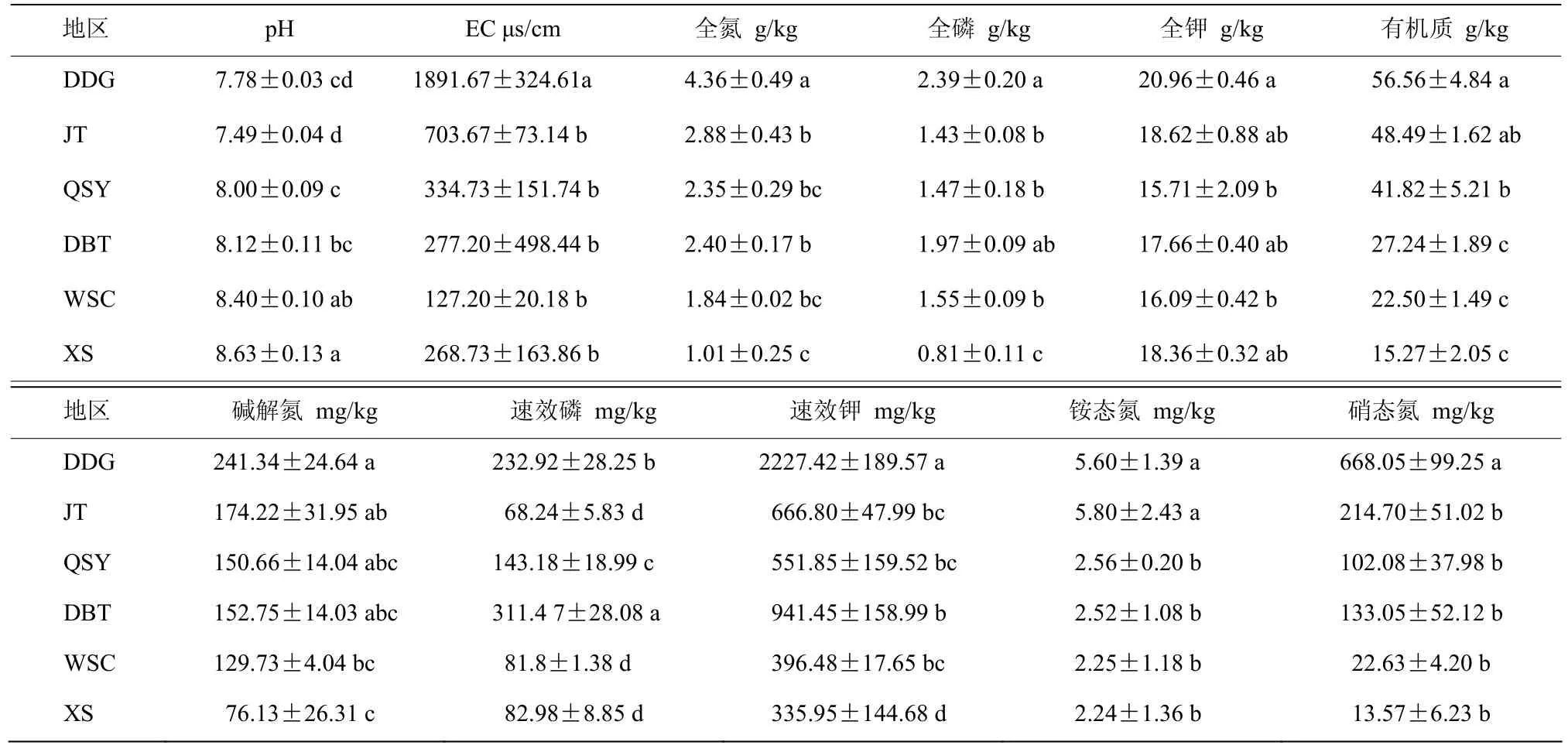
表2 不同采样地土壤样品的理化性质比较Table 2 Comparison on the physical and chemical properties of soil samples at different sampling sites
2.2 土壤养分对不同采样地细菌群落的影响
土壤养分与细菌群落相关分析结果表明(图1),pH和速效磷是影响土壤细菌群落变异最显著的环境因子,其次是速效钾、EC、硝态氮、全氮和有机质,而全钾、全磷、碱解氮和铵态氮对细菌群落差异化的贡献率较小。
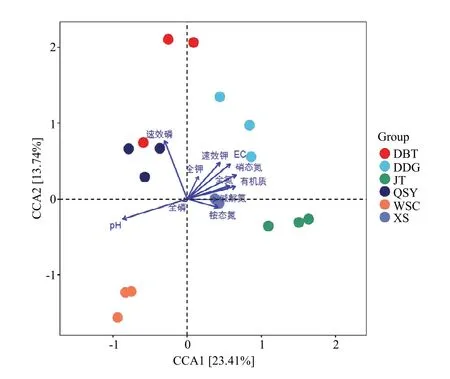
图1 土壤养分因子与散土细菌群落典型相关分析Fig. 1 Canonical correspondence analysis of relationship between soil nutrient factors with bulk soil bacterial community.Permutation test p value = 0.001
2.3 番茄根际土壤细菌群落的组成分析
北京6个地区36份番茄根际土壤样品共产生777540条16S rRNA基因序列。利用RDP数据库注释后,6个地区的样本共检测到16个细菌门(图2A,相对丰度大于1%的门类),各个地区番茄根际微生物群落以放线菌门(Actinobacteria 占比27%~46%)、变形菌门(Proteobacteria 占比21%~50%)和厚壁菌门(Firmicutes 占比7%~32%)为主。在属水平(图2B),大东各庄村(DDG)、姜屯(JT)和前桑园村(QSY)的番茄植株根际的优势菌群为芽胞杆菌属,占比42%~56%;兴寿镇(XS)和王上村(WSC)的优势菌群为节细菌属,占比16%~34%。所有采样点中,仅东北台村(DBTD和DBTH)和前桑园村(QSYD和QSYH)的健康和发病植株根际组成存在差异。整体来看,不同地区的番茄根际植株群落组成在属水平的优势菌群不同,而同一地区的健康和发病植株根际组成差异较小。
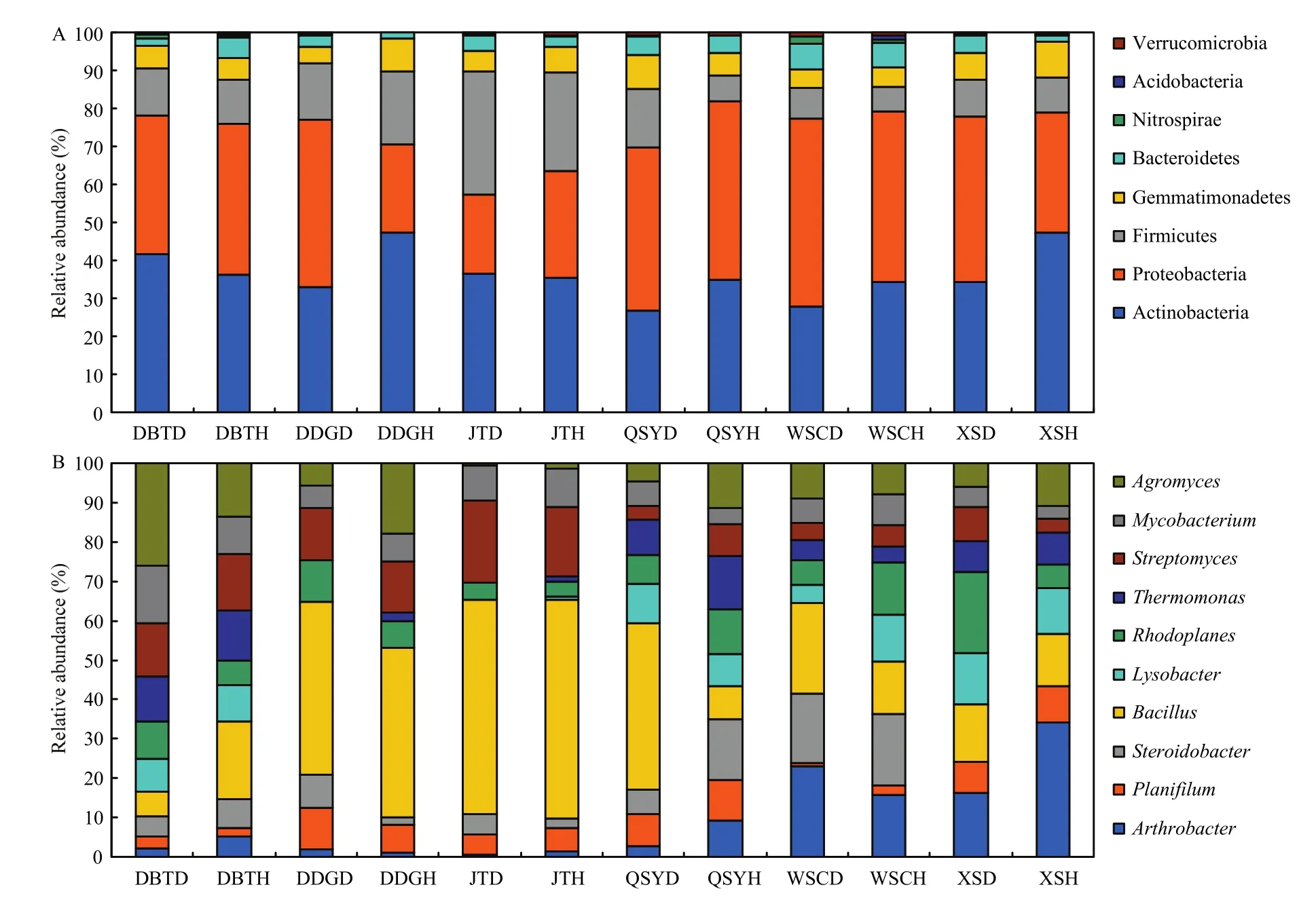
图2 各个地区健康和发病番茄根际土壤细菌群落组成比较(A:门水平,B:属水平,样品编号后的字母D和H分别是发病植株和健康植株)Fig. 2 Comparison of rhizosphere bacterial community composition of healthy and diseased tomato at different sampling sites (A: phylum level, B: genus level, the letters D and H after sample codes indicate the diseased and healthy plants respectively)
2.4 番茄根际土壤细菌群落多样性分析
多样性分析结果表明:健康植株根际群落Chao1、Shannon和Simpson指数均高于发病植株番茄根际土壤(图3)。主成分分析结果表明,6个地区的番茄根际细菌群落结构呈现显著的区域性分布(P<0.001,R2=0.46952,图4A),同时样品聚类分析也说明地域因素对于番茄根际细菌群体的影响大于病原微生物引起的群落结构变化(图4B)。
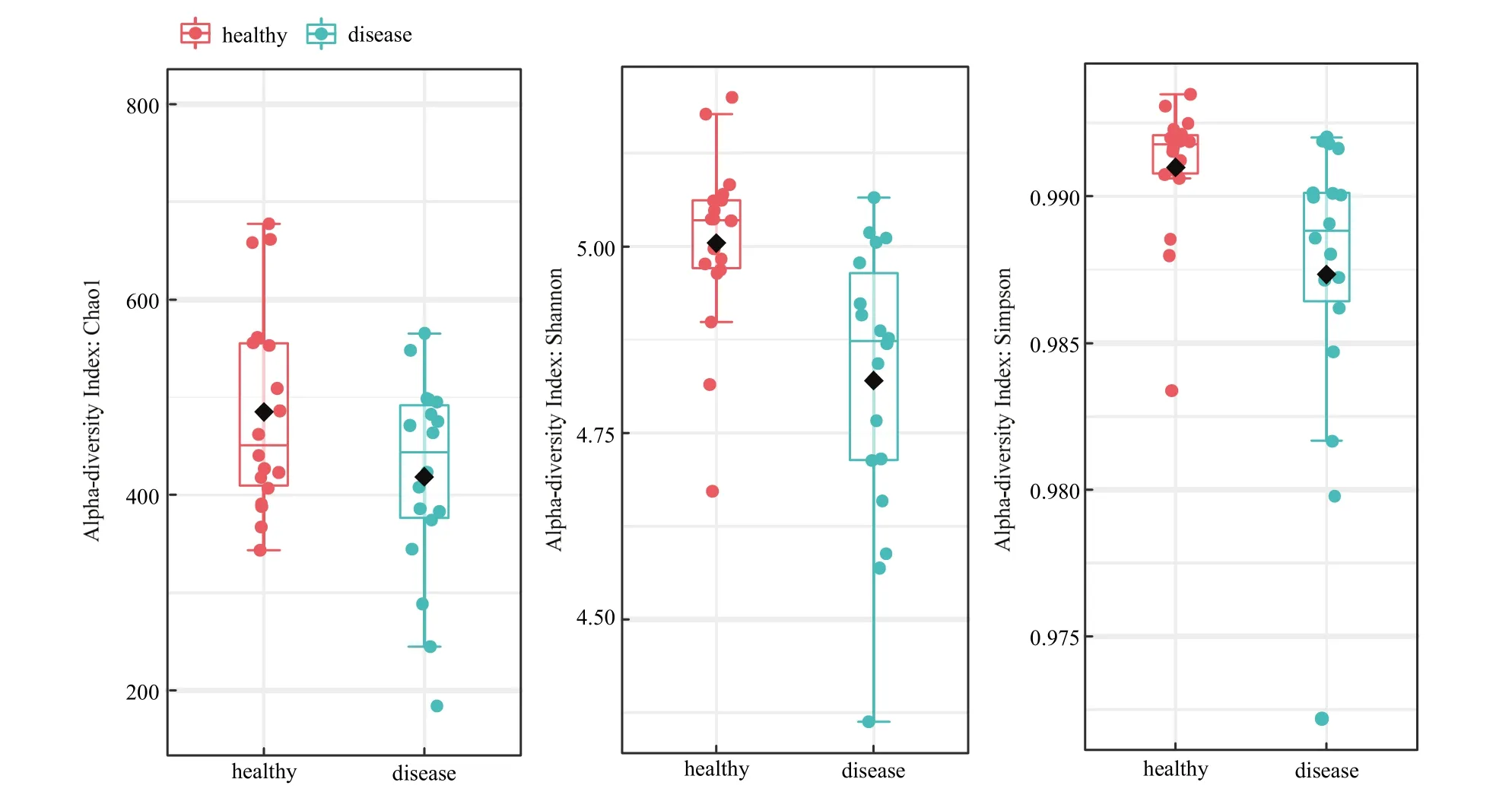
图3 各个地区健康和发病番茄根际土壤微生物群落α多样性分析Fig. 3 α diversity analysis of healthy and diseased tomato rhizosphere soil community at different sampling sites

图4 各个地区番茄根际土壤微生物群落β多样性分析和样品聚类分析(A:主成分分析,B:样品聚类分析)Fig. 4 β diversity and cluster analysis of tomato rhizosphere soil microbial community at different sampling sites
2.5 健康和发病植株根际细菌群落网络相关性分析
在健康植株根际土壤的群落相关性网络图中(图5A),点元素和边元素分别是93和113,正相互作用数量为71,负相互作用数量为42;在发病植株根际土壤的相关性网络图中(图5B),点元素和边元素分别是136和292,正相互作用数量为152,负相互作用数量为140。与健康番茄根际群落网络相比,发病番茄根际群落网络中的点元素和边元素的数量均明显升高,说明发病植株根际土壤的群落结构更为复杂且存在更多的相互作用,即微生物群落的平衡关系发生了变化。Bacillus在健康和发病植株根际细菌网络中都占据核心位置,且在发病植株根际细菌网络中与其他类群存在更多的互作关系(degree=14),而Stenotrophomonas,Lysinibacillus和Pseudoxanthomonas等类群仅在发病植株根际细菌网络中占据核心位置。

图5 健康和发病番茄根际群落相互作用的网络(A:健康植株根际土壤,B:发病植株根际土壤,不同的颜色代表不同的模块,黑色链接表示节点间的正交互作用,红色链接表示负交互作用)Fig. 5 Network of rhizosphere community interactions between healthy and diseased tomato (A: rhizosphere soil of healthy plants, B:rhizosphere soil of diseased plants, different colors represent different modules, black links represent positive interactions between nodes,and red links represent negative interactions)
2.6 Ralstonia相关类群分析
基于健康和发病土壤微生物群落相关性网络分析发现与Ralstonia直接相关的微生物有15个属(图6),其中与Ralstonia呈正相关的属有9个,分别是伯克霍尔德氏Burkholderia、索氏菌属Thauera、根瘤菌属Rhizobium、海洋芽胞杆菌属Oceanobacillus、肠杆菌属Enterobacter、侏儒囊菌属Nannocystis、泛菌属Pantoea、散生杆菌属Patulibacter和柯恩氏菌属Cohnella;与Ralstonia呈负相关的属有6个,分别是芽胞杆菌属Bacillus、寡养单胞菌属Stenotrophomonas、气微菌属Aeromicrobium、噬冷菌属Algoriphagus、珊瑚球菌属Corallococcus和假黄单胞菌属Pseudoxanthomonas。

图6 Ralstonia与番茄根际群落中其它细菌的网络相关分析Fig. 6 Network analysis of Ralstonia with other bacteria in tomato rhizosphere community
2.7 健康番茄根际土壤细菌的拮抗功能筛选
从健康番茄根际土壤中共分离得到 152株细菌,经过拮抗功能筛选得到 14株(表3)对青枯菌R.solanacearumGMI1000具有拮抗作用的菌株(图7),分别属于无色杆菌属Achromobacter、芽胞杆菌属Bacillus、剑菌属Ensifer、肠杆菌属Enterobacter,赖氨酸芽胞杆菌属Lysinibacillus、假单胞菌属Pseudomonas、红球菌属Rhodococcus寡养单胞菌属Stenotrophomonas和链霉菌属Streptomyces。
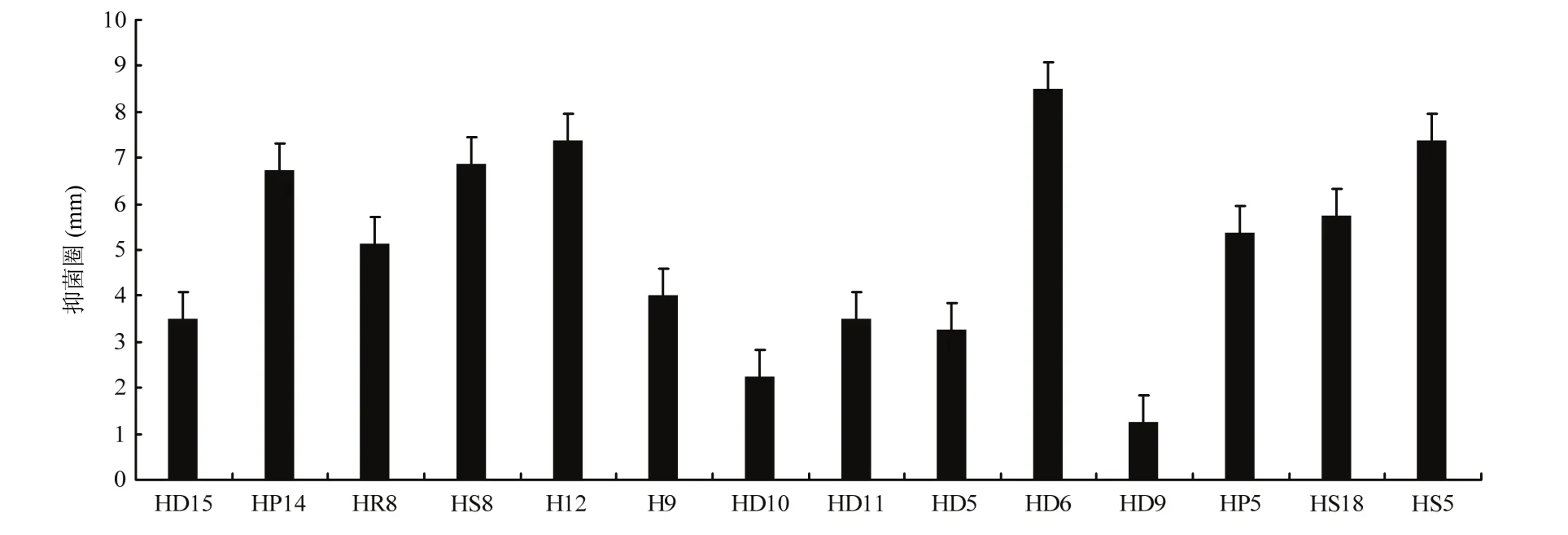
图7 14株根际细菌对青枯菌的拮抗活性测定Fig. 7 Antagonistic activity of 14 rhizosphere bacteria against Ralstonia solanacearum GMI1000

表3 青枯菌拮抗细菌鉴定结果Table 3 Isolation, culture and identification of bacteria from healthy tomato rhizosphere soil
3 讨论
尽管青枯病不是北方设施番茄的主要病害,但由于全球变暖、连作障碍等因素的影响下,番茄青枯病具有北移的趋势。本研究对顺义、大兴、通州、昌平4个区域6个采样点的健康与发病番茄植株进行研究,通过主成分分析和样品聚类分析发现地域因素对于番茄根际细菌群体的影响更大,即不同区域相比较,土壤养分因子对微生物组成影响更显著,但同一地区,青枯菌是根际细菌群落组装的主要影响因子。已有报道表明,pH、有效性养分磷和氮及有机质是调节根际群落组装的主要因素且影响病害的发生[22-26],Li等[27]首次证明土壤磷很可能通过微生物间的相互作用来促进青枯菌对土壤和植物系统的入侵。本研究对环境因子相关性分析表明,pH和速效磷是影响土壤细菌群落变异最显著的环境因子。有关病原菌侵染对微生物群落的影响,前人已有较多研究,Wei等[8]发现青枯菌的侵染会导致非致病细菌的多样性下降,与该研究结果相似,本研究发现健康植株根际的Chao1、Shannon,Simpson多样性指数均高于发病土壤,可能是由于病原菌的大量繁殖破坏了原有微生物群落的平衡。这些结果说明在抵抗青枯菌入侵的过程中不能忽略土壤养分对植株根际群落组装的重要性,也预示着不同区域的生物防治需要采取不同的生物制剂。
通过构建群落相关性网络发现拮抗病原菌的细菌已经成为研究微生物互作关系的有效方法,这些相关的类群可能是病原菌的靶点或者是助手[10],本研究通过构建群落相关性网络,发现15个与Ralstonia直接相关的菌属。Hu等[10]在研究根内生潜在致病性雷尔氏菌和其他微生物成员的相互作用网络时发现伯克氏菌属Burkholderia、根瘤菌属Rhizobium、肠杆菌属Enterobacter、散生杆菌属Patulibacter、芽胞杆菌属Bacillus和寡养单胞菌属Stenotrophomonas等与Ralstonia直接相关,除此之外,本研究还发现另外9个与Ralstonia直接相关的菌属,分别是索氏菌属Thauera、海洋芽胞杆菌属Oceanobacillus、侏儒囊菌属Nannocystis、泛菌属Pantoea、柯恩氏菌属Cohnella、气微菌属Aeromicrobium、噬冷菌属Algoriphagus、珊瑚球菌属Corallococcus和假黄单胞菌属Pseudoxanthomonas。以此为参考,筛选得到14株对青枯菌具有拮抗能力的菌株,分布在9个属,其中芽胞杆菌属、肠杆菌属、寡养单胞菌属、假单胞杆菌属和链霉菌属的细菌已被用于青枯病的防治[28]。此外,本研究还发现 3个可以抑制青枯菌生长但应用较少的类群:无色杆菌属Achromobacter、剑菌属Ensifer和赖氨酸芽胞杆菌属Lysinibacillus,这一发现增加了开发微生物制剂资源的多样性,后续研究将重点通过构建复合菌群用于青枯病的防治。
