小动物活体光学三维成像系统及其对乳腺癌的 定量分析
2022-09-14郑南南
郑南南,黄 钢
(1. 上海理工大学健康科学与工程学院,上海 200093; 2. 上海健康医学院,上海市分子影像学重点实验室,上海 201318)
乳腺癌位居全球女性常见癌症之首[1]。远处转移灶的扩散导致乳腺癌具有极高的死亡率,因此,对扩散性肿瘤细胞的生长过程不间断检测至关重要[2]。在临床前动物模型中进行体内分子成像是研究癌症发展中所涉及的病理生理机制和药物开发的方法。当前小动物无创活体成像技术主要有:使用X射线[3](X射线计算机断层扫描和传统的二维X射线成像)、核磁共振、核素成像(正电子发射断层扫描、单光子发射断层扫描)、超声成像和光学成像方法(生物发光和荧光)[4-5]。
生物发光断层成像(bioluminescence tomography,BLT)通过光传输模型确立生物体表面的光强测量值,并运用适当的三维重建算法将物体表面检测到的平面生物发光通量转化为物体内部重建的三维生物发光源[6-7]。许多有效的解决策略和先验信息[8-14]被用来解决BLT逆问题,主要包括基于压缩感知(compressive sensing,CS)理论的各种稀疏算法[15]、利用其他结构成像模式(如CT和MRI)分割肿瘤区域的先验信息、通过算法手动或自动分割肿瘤区域作为先验区域、对重建结果的结构分辨率进行改进。传统BLT重建通常存在前向问题不准确、逆向问题不适定、依赖先验知识、迭代计算代价高等问题。
随着深度学习的兴起,将深度学习模型应用于三维重建,为传统的基于视觉的三维重建方法带来了新的思路。深度学习使用神经网络从自然数据中提取不同尺度和空间的特征,然后以级联方式表达其特征,在高维数据上表现良好,因此,广泛用于多个领域,在医学成像中也发展了许多应用[16]。本文通过加入鲸鱼优化算法(whale optimization algorithm,WOA)的3D-En-Decoder的重建方法[17],直接建立了内部与另外边界区域荧光辐射信号数据分布之间的数据映射参数关系。它避免了重建的逆问题引起的误差,没有复杂的迭代计算,使重建效率提高。
本文工作流程如图1所示。

图1 本文工作流程Fig. 1 Workflow of this article
1 材料与方法
1.1 样品配置
慢病毒载体(pLenti-CBh-3FLAG-luc2-tCMV-td-Tomato-F2A-Puro)和人乳腺癌细胞(MDA-MB-231)均来自和元生物技术(上海)股份有限公司;重度联合免疫缺陷(server combined immune-deficiency,SCID)小鼠来源于上海吉辉动物饲养有限公司,饲养于上海健康医学院实验动物中心,符合实验动物福利伦理要求。
1.2 试验仪器
荧光显微镜(DMi8,Leica)、超净工作台(JBCJ-1500FX,佳宝)、CO2培养箱(311,Thermo)、低速离心机(TDZ4-WS,上海湘仪)、小动物气体麻醉机(Matrx, VIP 300)、小动物活体光学三维成像系统(IVIS Spectrum,PerkinElmer)。本研究使用的IVIS Spectrum小动物活体光学三维成像系统的结构图如图2所示,-90℃制冷的背照式电荷藕合器件(charge coupled device,CCD)相机,在可见光及近红外光谱范围内,可提供高于80%的高量子检测效率。其成像过程如图2所示,所有深度学习模型均在带有11 GB Nvidia GeForce RTX 2080 Ti GPU的服务器上训练,传统方法的重建工作都在CPU配置为Core i7-10700、主频为3.6 GHz的计算机上完成。训练3D-En-Decoder时,epochs设置为60,batch大小设置为32。
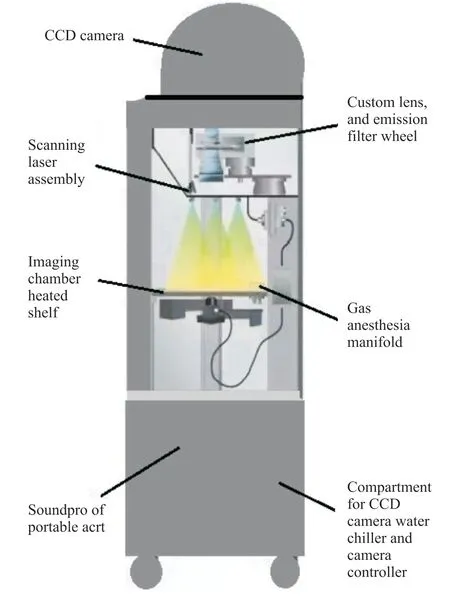
图2 IVIS Spectrum小动物活体光学三维成像系统结构图Fig. 2 Structure diagram of IVIS Spectrum small animal in vivo optical three-dimensional imaging system
1.3 细胞转染及稳定细胞系的筛选
将MDA-MB-231细胞用含10%的胎牛血清(fetal bovine serum,FBS)培养基培养,配成每毫升含有0.5×105个细胞的悬液,以每孔铺2 mL接种至6孔板,细胞密度为每孔1×105个细胞,放入37℃、5% CO2培养箱中生长。12~20 h后,每孔加4 μL 1 mg/mL聚凝胺(polybrene)转染试剂感染病毒。感染12~20 h后吸弃原培养基,磷酸盐缓冲液(phosphate buffer saline,PBS)洗涤,每孔加入5 mL新鲜的培养基稀释。培养72 h后,加入终质量浓度2μg/mL的筛选抗生素嘌呤霉素(puromycin)杀死未被有效感染的细胞。每隔2~3 d换一次液。药物筛选约2周后,使用荧光显微镜检测生物发光情况,并拍荧光照片,将稳定表达的细胞株MDA-MB-231-luc2细胞冻存。
1.4 SCID小鼠乳腺癌原位模型的构建
试验用6周龄SCID雌鼠建立原位乳腺癌模型,分别在右侧乳腺脂肪垫皮下原位注射5×106个生物发光标记的MDA-MB-231-luc2人乳腺癌细胞[18],并放于动物房无菌饲养。21 d后在SCID小鼠腹部注射发光底物,15 min后使用异氟烷-氧气混合气体(Matrx VIP 300小动物气体麻醉机)对小鼠进行麻醉。使用PerkinElmer公司的IVIS Spectrum小动物活体光学三维成像系统进行荧光图像的采集分析,活体荧光成像过程示意图如图3所示,动物试验流程遵守实验动物伦理委员会的管理规定。
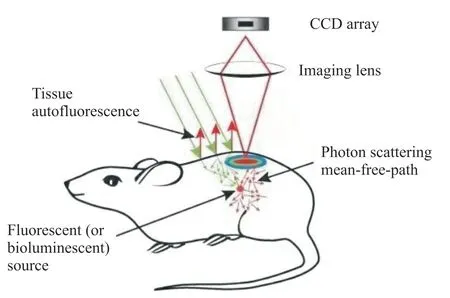
图3 小鼠活体荧光成像过程示意图Fig. 3 Schematic diagram of in vivo fluorescence imaging process
1.5 IVIS Spectrum小动物活体成像系统三维重建
IVIS Spectrum小动物活体光学三维成像系统具有生物发光和荧光三维成像能力,系统自带的生物发光成像断层扫描技术需要Living Image软件有结构照明或CT成像的图像,并能通过两个或两个以上的波长的滤光片获取生物发光图像。Living Image软件将被测体分解为体素的实体网格,表示实际光源近似的位置分布。每个体素的信号强度、光子密度或噪声传递函数效率之间存在线性关系,该线性关系可以通过格林函数来描述。通过一种非负最小平方算法寻找近似值解决方案,同时,仅选取信号高于阈值且有重大贡献的表面信号元素来减少变量数。
1.6 基于WOA优化的3D-En-Decoder的BLT重建算法
流程包括三个步骤:1)生成训练样本;2)构建深度编码器-解码器(encoder-decoder)模型;3)对模型进行训练。3D-Encoder和3D-Decoder包含了相同的模块,即卷积层、正则化、激活函数以及池化,卷积核大小都为3×3。3D-Encoder卷积核个数从256向32递减。3D-Decoder每次经过卷积之后,卷积通道数会依次递减一半,而特征通道图像会加倍。3D-Encoder主要用于提取出光学特征和Q特征的分布。Q特征主要为荧光团的位置、荧光团大小,光学特征主要为吸光系数和光散射系数大小。3DEncoder和3D-Decoder之间通过一个全连接层连接,进行不同形态数据特征时间的转换,即前面所提到的感知域和图像域。两种不同数据之间的低维的联合流形表示,可以进一步得到对噪声和其他干扰数据的具有鲁棒性的网络表示方式。
3D-En-Decoder基于三维卷积操作,使用编码器-解码器结构,其中编码器用于下采样提取特征,解码器用于上采样重构图像,并且参考了自编码器网络来学习新的表示。无论是二维还是三维卷积,都是对样本进行卷积池化采样等操作,虽然卷积神经网络的层数、池化方式、激活函数、分类器等结构参数可依据经验进行选择,不需要优化,但是卷积核的个数、大小、学习率等参数并没有明确的选取规则,显然随机选择容易使神经网络陷入局部最优。而WOA具有结构简单、参数少、搜索能力强且易于实现等特点,能对卷积神经网络(convolutional neural networks,CNN)卷积核的大小、个数、学习率等参数进行寻优,并利用所得进行全局最优参数重构。基于此,本文引进WOA优化算法对3D-En-Decoder编解码神经网络进行参数优化,避免陷入局部最优,从而获得更快的收敛速度以及更小的损失函数值。图4是WOA-3D-En-Decoder的网格模型设计流程图。
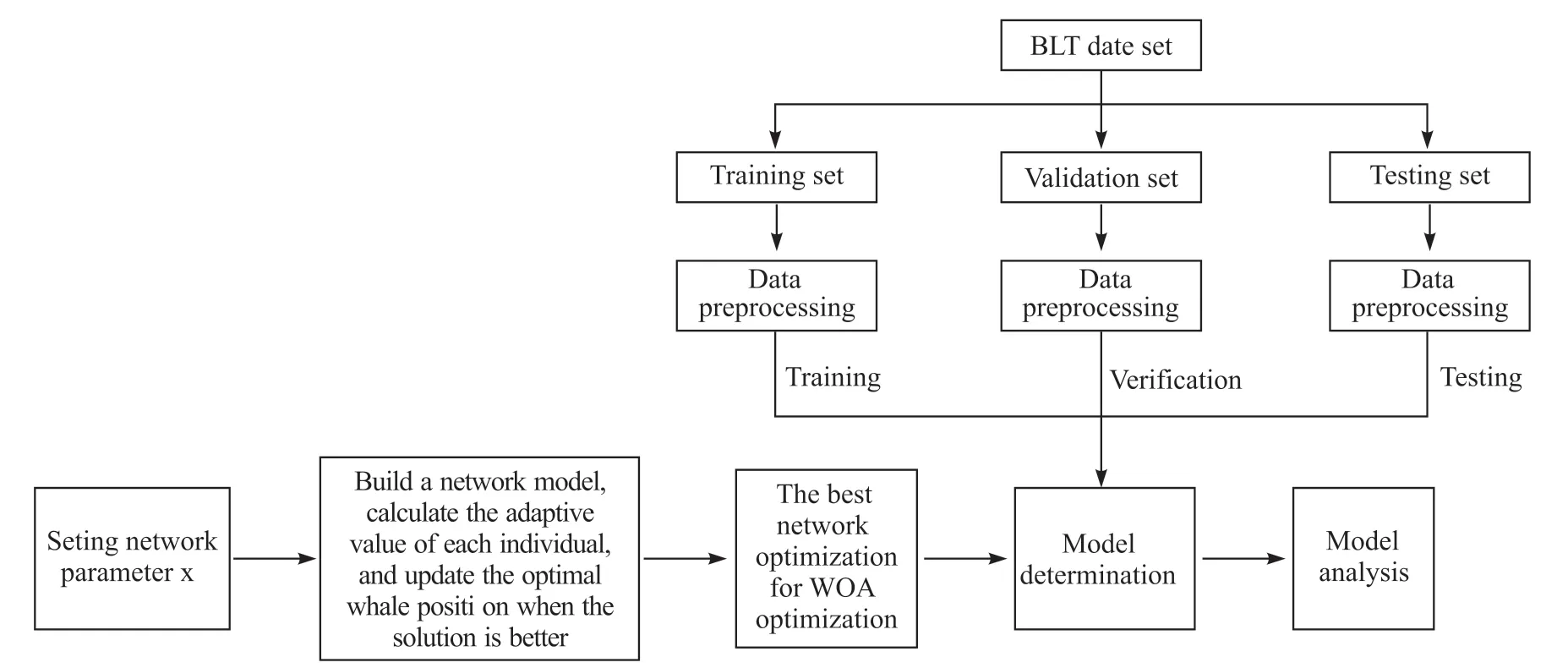
图4 WOA-3D-En-Decoder网络模型设计流程图Fig. 4 Design flow chart of WOA-3D-En-Decoder network model
2 结果与分析
2.1 MDA-MB-231-luc2转染的鉴定
加4 μL 1 mg/mL 的聚凝胺感染病毒后,经过2周筛选,荧光显微镜明场下观察到细胞生长良好,并加入荧光素酶底物进行生物发光观测luc2基因表达情况[19]。放大100倍的情况下,检测到信号较强,荧光分布较为均匀,能稳定表达荧光素酶,且随传代培养无明显减弱(图5)。这证明转染成功,同时表明慢病毒具有高效稳定的感染率。
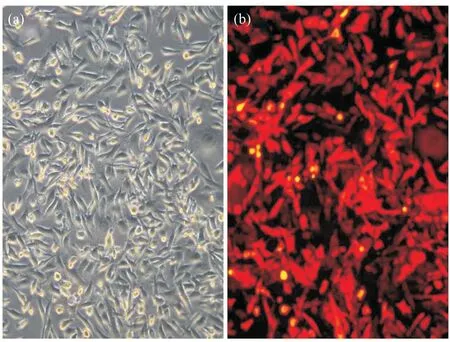
图5 稳定细胞系MDA-MB-231-luc2表达luc2 (100×)Fig. 5 Expression of luc2 in MDA-MB-231-luc2 cells (100×)
2.2 IVIS Spectrum小动物活体成像系统二维成像定量分析
在SCID小鼠乳腺脂肪垫皮下原位注射5×106个生物发光标记的MDA-MB-231-luc2乳腺癌细胞,并进行活体成像,IVIS Spectrum小动物活体成像系统长期监测肿瘤的生长情况,在注射后21 d观测到其他部位的转移信号。根据系统的成像向导选择生物发光程序、探针和滤光片。获取荧光图像后圈选感兴趣区域(region of interest,ROI),获得ROI区域的测量数据。根据二维图像可知,ROI 1标记的小鼠荧光值最大,手触摸其腹部,肿瘤体积最大。肿瘤的体积与其荧光值的变化一致,具有明显的相关性(图6)。

图6 二维成像对小鼠的定量分析Fig. 6 Quantitative analysis of 2D imaging

表1 不同ROI区域的肿瘤荧光值Tab. 1 Tumor fluorescence values in different ROI regions
2.3 IVIS Spectrum小动物活体光学三维成像系统的三维成像定量分析
通过多组滤光片得到生物发光的二维序列图像,结合表面拓扑结构重建得到生物发光光源的三维信号(图7,橘色区域),以及试验动物中信号光源的深度,并计算出光源的绝对强度。同时,得到了冠状面、矢量面和横断面的图像,体积的测定值Final voxel size (mm)为1.25,该体积是光通过3D散射形式产生的,并非物理体积。

图7 利用生物发光光源成像的荷瘤小鼠三维信号图Fig. 7 Three dimensional signal diagram of bioluminescent light source
利用异氟烷-氧气混合气体(Matrx VIP 300小动物气体麻醉机)对小鼠进行麻醉后,俯卧姿势下使用小动物PET/CT进行成像。图8中箭头所指的圆圈内,白色区域是肿瘤切片。通过结果比对,IVIS Spectrum小动物活体成像系统三维成像具有准确性,而CT需要一定的临床经验才能发现目标点,活体成像系统三维成像则更迅速直观。

图8 利用PET/CT进行成像的荷瘤小鼠断层扫描图Fig. 8 Tomography of tumor bearing mice imaged by PET/CT
2.4 WOA优化的3D-En-Decoder重建算法试验结果
本文中的WOA-3D-En-Decoder模型主要用于肿瘤样本重建问题以及BLT分辨率问题,因此,数据集是关于一个和两个荧光团的。本文通过翻转、平移等样本扩增手段,将数据集扩增为6 000组,按照7∶2∶1划分试验的训练集、验证集和测试集。训练集用于训练不同的模型,验证集用来挑选最优的模型以及验证模型的泛化能力,测试集用来评估模型的性能。
为了验证本文提出的WOA-3D-En-Decoder编解码网络的重构性能,本文将网络重建结果与基于L1正则化项约束的迭代收缩算法(L1-LS)和基于L2正则化项约束的吉洪诺夫算法(Tikhonov)这两种传统重建算法进行定量和定性的对比。图9为WOA参数优化前后的网络训练迭代图。
从图9可以看出,WOA-3D-En-Decoder比3DEn-Decoder收敛速度更快,代价函数值更小,证明WOA能有效地优化网络参数,使得重构误差更小。这主要是因为WOA优化算法可以对3D-En-Decoder卷积核的大小、个数、学习率等参数进行寻优,利用所得全局最优参数重构 WOA-3D-En-Decoder模型并进行肿瘤样本三维重建。

图9 3D-En-Decoder和WOA-3D-En-Decoder训练迭代图Fig. 9 3D-En-Decoder and WOA-3D-En-Decoder training iteration diagram
本文通过在二维投影图像中增加了10%的高斯噪声的方式来更好地模拟真实的情况。从上述训练过程可知,训练迭代次数从0到1 000时,训练误差快速降低,当训练迭代次数从1 000逐渐增加到8 000的时候,训练误差趋于稳定。最终均方根误差(root mean squared error,RMSE)值稳定在8左右。
图10是单肿瘤样本小鼠的切片图。从图11单肿瘤样本三维重建结果可以看到,通过深度学习方法可以得到一个质量较好的三维模型。通过建模,得到三维肿瘤模型。该肿瘤模型的三维尺寸为4.85、7.21、4.92 mm,与实际的4.90、7.10、5.00 mm的误差分别为1.03%、1.53%、1.63%。

图10 单肿瘤样本小鼠的切片图Fig. 10 Slice of mice with single tumor sample

图11 单肿瘤样本三维重建结果Fig. 11 3D reconstruction results of single tumor sample
深度学习模型训练有素,因此,重建深度学习模型消耗的3D图像的平均时间仅为0.58 s。三维重建结果证明了本文提出的深度学习网络模型用于生物发光断层成像重建的可行性、模型可扩展性以及性能的可提升性,对BLT领域的研究具有重大意义。
使用对比度噪声比(contrast-to-noise ratio,CNR)表示ROI信号与BCK信号的区分程度,以用于评估ROI的图像对比度信噪比。使用Dice系数来描述重建光源区域与实际光源区域的重叠程度。表2是不同深度荧光团边距为5 mm时的四种重建方法定量分析结果,根据本文使用的方法,三个结论指标为:CNR=6.21;Dice=0. 74;Time=1.25 s。从试验结果可知,在信噪比CNR值上Tikhonov方法最小,L1-LS方法和WOA-3D-En-Decoder网络都比Tikhonov要大一些,基于深度学习的重建结果优于其他三种算法。基于深度学习的重建结果重复度Dice值可以达到0.74,大于其他三种算法。这表明,基于深度学习的三维重建方案重建出的光源轮廓更清晰,具有较强的学习能力,能够更好地重建出三维模型。在试验时间方面,由于本文采取了先训练的方式,其测试时间明显优于Tikhonov方法和L1-LS方法。综上所述,基于深度学习的网络重建出的图像具有更好的质量、更高的效率。

表2 不同深度荧光团边距为5 mm时的四种重建方法定量 分析结果Tab. 2 Quantitative analysis results of four reconstruction methods when the edge distance of fluorophores in different depths of polybrene is 5 mm
3 讨论
随着科学技术的发展,基于大尺寸高量子效率CCD等硬件配置升级,活体光学成像仪检测灵敏度及成像速度都大大提高[20],还可以对完整的活体受试者进行断层成像,并将采集后图像的计算三维叠加、重建,同时将这些生物学指标进行量化,得到光学信号在体内的三维定量信息。
生物发光成像为完整细胞和小动物模型的生物过程提供了有价值的帮助,它有助于加快药物识别和开发,帮助评估新治疗方案的可行性[21]。生物发光成像面临的主要挑战是生成更稳定的红移荧光素酶和使用不同底物的新型荧光素酶,进一步增强成像效果。由于光的穿透深度有限以及荧光素的潜在毒性和荧光素酶的潜在免疫原性,目前无法将这一技术应用于临床。
本文通过luc2对人乳腺癌MDA-MB-231细胞进行标记,通过慢病毒感染法将目的基因导入细胞内。luc2能在体内外稳定表达后建立SCID小鼠原位乳腺癌模型,运用小动物活体光学三维成像系统对乳腺癌进行生物发光定量分析。同时,引入深度学习技术,运用WOA-3D-En-Decoder算法进行重建,通过小动物CT图像验证,试验结果表明,该网络模型与IVIS Spectrum小动物活体成像系统在重建的精度、效率和抗噪性方面都有了很大的提升。
