侵染白术的大豆花叶病毒的全基因序列测定及分析
2022-09-07马振男崔丽艳王舒婷王德富牛颜冰
马振男, 崔丽艳, 张 丽, 王舒婷, 王德富*, 牛颜冰*
(1)山西农业大学生命科学学院,山西 太谷 030801;2)山西农业大学草业学院,山西 太谷 030801)
白术(AtractylodesmacrocephalaKoidz)为菊科(Asteraceae)苍术属的多年生草本植物。早在2 000多年前的《神农本草经》中就已经将其列为上品药。现代研究进一步发现,白术富含挥发油、多糖、内酯类等化学成分,具有抗肿瘤、抗炎、调节消化系统等药理作用[1]。随着白术在现代临床应用和中成药生产等方面的不断需求,其种植面积也不断扩大,病虫害也随之增多,使得药材产量和质量均受影响。病害防治研究是保证药材质量及产量的关键之一,在影响白术生长的诸多病害中,病毒病害始终占据主导地位,因此,迫切需要对白术田间病害进行调查研究。
大豆花叶病毒(soybeanmosaicvirus, SMV),是马铃薯Y病毒属(potyvirus)最典型的成员之一,其基因组为被外壳蛋白(coat protein, CP)所包裹的长约10 kb的正义单链RNA;在基因组RNA的5′-末端为共价结合基因组连接蛋白(viral protein genome-linked, VPg),3′-末端则是多聚腺苷酸尾[2]。SMV基因组具有2个开放阅读框(open reading frame, ORF),其中一个ORF可翻译形成一个多聚蛋白,后经自身编码的具有蛋白酶功能的第一蛋白(first protein, P1)、辅助组分蛋白酶(helper component-proteinase, HC-Pro),以及核内含体蛋白a-蛋白酶(nuclear inclusion protein a proteinase, NIa-Pro)切割形成10个不同功能的成熟蛋白质,从N-端到C-端分别为P1、HC-Pro、第3蛋白(third protein, P3)、第1个6K蛋白(6-kilodalton protein 1, 6K1)、圆柱形内含体蛋白(cylindrical protein, CI)、第2个6K蛋白(6-kilodalton protein 2, 6K2)、核内含体蛋白a-基因组结合蛋白(nuclear inclusion protein a viral genome-linked protein, NIa-VPg)、NIa-Pro、核内含体蛋白b(nuclear inclusion protein b, NIb)和CP[3]蛋白。另一个ORF为移码翻译产生的PIPO(pretty interesting potyviridae ORF)蛋白(包含在P3蛋白中)[4]。
SMV的自然寄主范围极其有限,主要侵染以大豆为主的部分豆科植物[5-7],但是之后又有研究者发现SMV可侵染天南星科植物半夏[8-9]。SMV感染植株后会影响植株的正常生长发育,感病植株常出现顶枯和矮化现象,叶片则表现皱缩、黄化、花叶等症状[10]。本课题组在对白术进行田间调查时,发现一株疑似感病白术(见Fig.1),并对其进行非序列依赖性扩增(sequence-independent amplification, SIA)检测。结果表明,侵染白术的病毒病原为SMV,并将其命名为SMV-Am分离物。在此基础上,本文对侵染白术的SMV进行全基因组扩增,并对其序列相似性、进化关系、突变位点及重组事件进行分析,为防范SMV病害在白术上爆发流行及防治提供科学依据,并为明确SMV跨科侵染的分子机制研究奠定理论基础。
1 材料与方法
1.1 材料
自然发病具有典型花叶症状的白术叶片(见Fig.1(A)健康的植物。(B)感染的植物)采集于河北省白术种植基地,液氮速冻后于-80 ℃贮存备用,主要试剂见Table 1。

Fig.1 Healthy and infected field symptoms of Atractylodes macrocephala Koidz (A)Healthy plants.(B)Infected plants
1.2 dsRNA提取与SIA检测
取-80 ℃冰箱保存的疑似感病白术叶片10 g,按照已报道的方法[11-13]进行dsRNA提取,之后以所提dsRNA为材料参考牛颜冰等人[14]的方法进行SIA检测。
1.3 SMV-Am全基因组扩增
根据GenBank中已登录的SMV基因组保守序列,使用Primer Premier 5.0 软件设计用于SMV-Am基因组全长扩增的11对特异引物及RACE引物(Table 2),相邻片段之间无缝隙。再以所提白术dsRNA逆转录cDNA为模板,结合11对特异引物,使用Taq DNA polymerase进行PCR扩增,反应体系与反应条件参考牛颜冰等[15]的方法进行。cDNA末端快速扩增(rapid amplification of cDNA ends, RACE)采用巢式PCR的方法进行。其中,GSP和NGSP分别为一步和二步PCR反应所用引物,步骤按照SMARTer®RACE5′/3′Kit说明书进行。最终得到包含SMV-Am全基因组的11个特异片段和2个RACE扩增产物,将以上片段分别连接pMD18-T载体后导入大肠杆菌DH5α感受态细胞,阳性克隆通过菌落PCR鉴定后送至上海生工生物有限公司进行测序。

Table 1 Main reagents

Table 2 Primer pairs for SMV-Am whole genome amplification

Table 3 Comparison of nucleotide and amino acid sequence homology of SMV-Am and other 100 SMV isolates
1.4 SMV-Am生物信息学分析
对测序所得序列用DNAstar软件包中的SeqMan程序进行拼接,EditSeq程序进行修改和保存以获得SMV-Am全长基因组序列,并利用在线软件ORF finder用于预测核苷酸编码的氨基酸序列;DNAMAN软件对SMV-Am及其他已报道的100个SMV分离物进行核苷酸及氨基酸序列同源性分析;MEGA 7.0软件对用于系统进化分析;在线软件Network Protein Sequence(http://npsa-pbil.ibcp.fr)中的PROSCAN用于SMV-Am功能位点分析,I-TASSER和PyMOL用于分析SMV-Am的蛋白质结构变化;RDP4软件作为重组分析的工具。用于生物信息学分析的其他100条SMV分离物均来自于GenBank数据库。
2 结果
2.1 SIA检测
以感病白术叶片中提取出的dsRNA为模板,以M4T/M4为引物进行SIA检测。经凝胶电泳、测序和比对分析,得到大小为1 098 bp的非特异性片段(Fig.2)。将测序结果于NCBI数据库中进行Blast比对,最终发现该片段序列与SMV的相似性为84.5%-98.27%,并且与分离物SMV-pCB301-SC15(登录号:MH919386)的相似性最高,达到98.27%,因此,将该分离物命名为SMV-Am。
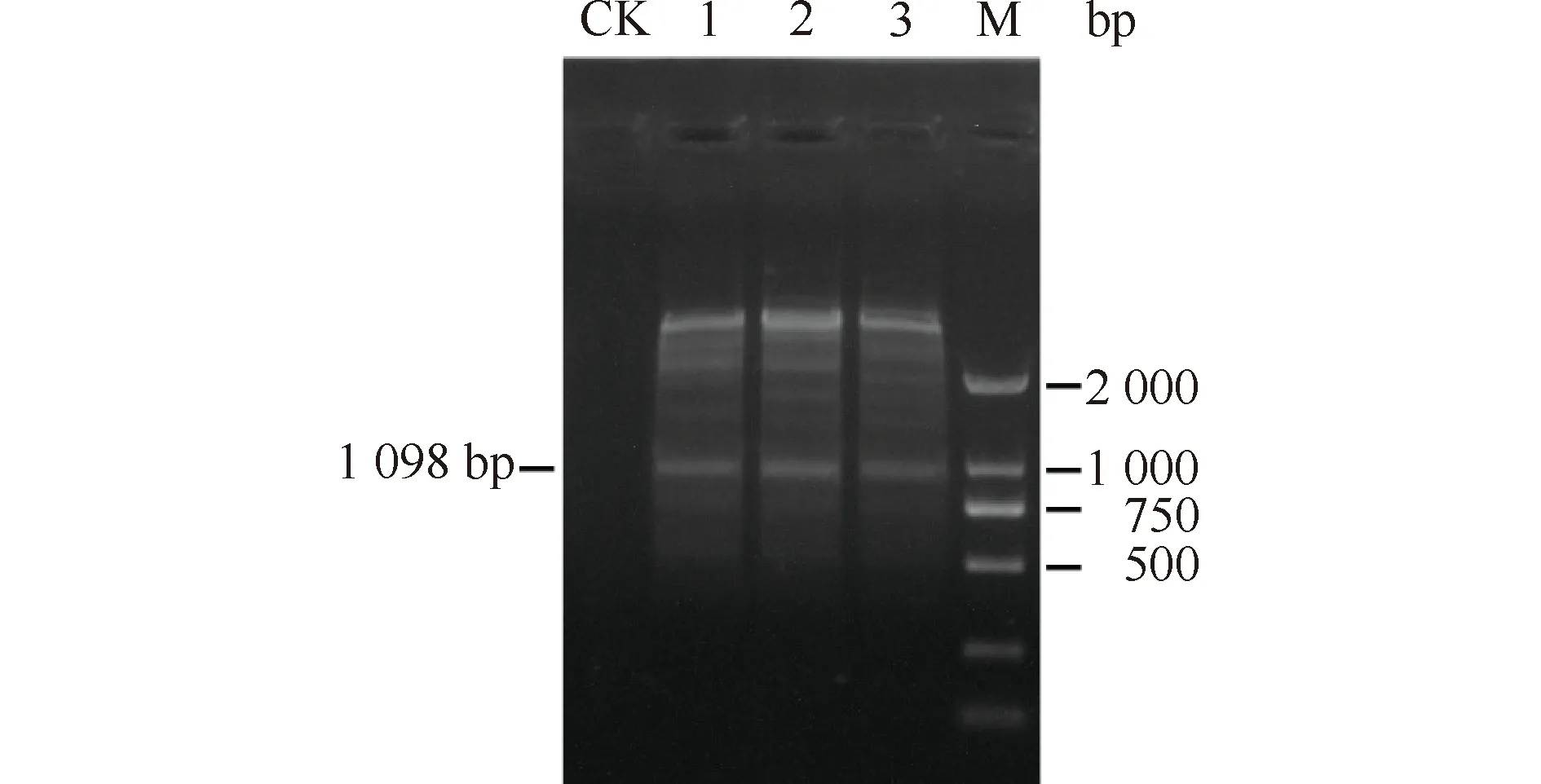
Fig.2 The dsRNAs were analyzed by the SIA method The degenerate primer M4,and the dsRNA,isolated from the susceptible Atractylodes macrocephala,were analyzed by the SIA method.M: DL 2 000TM DNA marker; Lanes 1-3: Infected plants; CK: Healthy plants
2.2 SMV-Am全基因组的扩增
以dsRNA逆转录所得cDNA为模板,首先用11对特异引物进行扩增,分别得到11个片段(Fig.3),再以GSP和NGSP为引物进行RACE扩增并获得SMV-Am基因组末端序列(Fig.4),所有相邻片段均无间隙,最终使用SeqMan软件对所得片段进行拼接,即获得全长为9 587 bp 的SMV-Am全基因组。
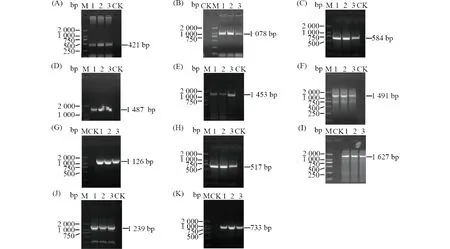
Fig.3 The specific fragments of SMV-Am were cloned by RT-PCR The specific primers,and the dsRNA,isolation from the susceptible Atractylodes macrocephala,were cloned by RT-PCR.(A)PCR product of SMV-F1422/SMV-R1422;(B)PCR product of SMV-F37-1 115/SMV-R37-1 115;(C)PCR product of SMV-F739-1 323/SMV-R739-1 323;(D)PCR product of SMV-F1298-2 785/SMV-R1298-2 785;(E)PCR product of SMV-F2538-3 991/SMV-R2538-3 991;(F)PCR product of SMV-F3802-5 293/SMV-R3802-5 293;(G)PCR product of SMV-F4524-5 650/SMV-R4524-5 650;(H)PCR product of SMV-F5628-6 145/SMV-R5628-6 145;(I)PCR product of SMV-F6080-7 707/SMV-R6080-7 707;(J)PCR product of SMV-F7404-8 643/SMV-R7404-8 643;(K)PCR product of SMV-F8540-9 273/SMV-R8540-9 273. M: DL 2 000TM DNA marker; Lanes 1-3: Infected plant; CK: Healthy plant

Fig.4 The RACE products of SMV-Am by the nested PCR method (A)5′-RACE amplification of SMV-Am genomic, PCR products using nest primers of SMV5′-GSP and SMV5′-NGSP;(B)3′-RACE amplification of SMV-Am genomic, PCR products using nest primers of SMV3′-GSP and SMV3′-NGSP.M: DL 2 000TM DNA marker; Lanes 1-3: Infected plant; CK: Healthy plant
对所得SMV-Am基因组进行分析,发现其具有典型的potyvirus基因组结构特征,编码一个大的多聚蛋白质,经自身编码的蛋白酶切割后,最终得到11个成熟的蛋白质。其中,P1、HC-Pro、P3、6K1、CI、6K2、NIa-VPg、NIa-Pro、NIb和CP蛋白之间的切割位点为Y/S、G/G、Q/A、Q/S、Q/S Q/G、E/S、Q/S、Q/S(Fig.5),PIPO蛋白则是由移码突变产生。
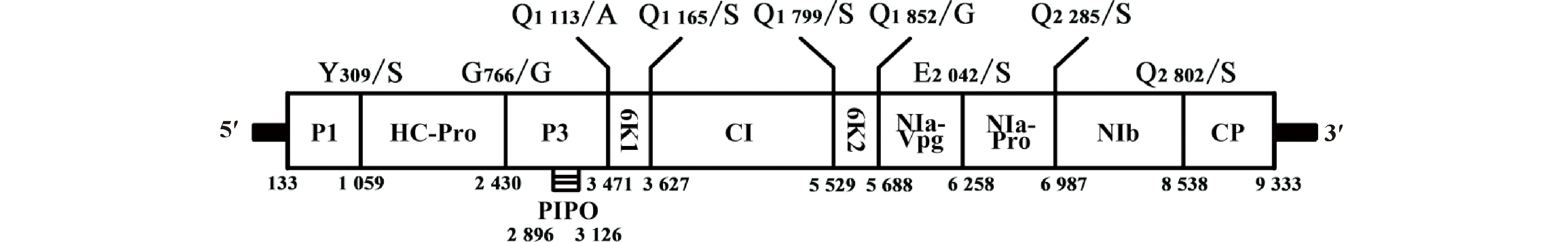
Fig.5 Genome organizations of SMV-Am Y309/S: Amino acid cleavage sites between P1 and HC-Pro; G766/G: Amino acid cleavage sites between HC-Pro and P3; Q1 113/A: Amino acid cleavage sites between P3 and 6K1; Q1 165/G: Amino acid cleavage sites between 6K1 and CI; Q1 799/S: Amino acid cleavage sites between CI and 6K2; Q1 852/G: Amino acid cleavage sites between 6K2 and NIa-Vpg; E2 042/S: Amino acid cleavage sites between NIa-Vpg and NIa-Pro; Q2 285/S: Amino acid cleavage sites between NIa-Pro and NIb; Q2 802/S: Amino acid cleavage sites between NIb and CP
2.3 序列相似性分析
利用DNAMAN软件对SMV-Am与GenBank中已登录的100个SMV分离物进行序列相似性分析,结果如Talbe 3所示。SMV-Am与其他100个SMV分离物的核苷酸相似性为89.51%~96.57%,氨基酸相似性为94.10%~98.86%,且在这100个分离物中,与SMV-Am核苷酸和氨基酸序列相似性最高的分离物为SMV-Liaoning(GenBank登录号:MK350280.1),分别为96.57%和98.86%,进一步证实感病白术为SMV侵染。
2.4 系统进化分析
利用MEGA 7.0软件完成SMV-Am与其他100条SMV序列的系统进化分析,采用邻接法(Neighbor-joining, NJ)以bootstrap = 1000构建系统进化树。结果正如Fig.6所示,SMV-Am与SMV-Liaoning分离株聚为一簇,亲缘关系最近。
2.5 功能位点突变及蛋白质结构分析
通过DNAMAN软件对SMV-Am和GenBank中100个SMV分离物的氨基酸序列比对后发现,P1、HC-Pro、P3、6K2、NIa-pro和NIb蛋白均发生了不同程度的氨基酸突变,经PROSCAN软件分析后发现,部分氨基酸突变发生在功能位点区域(Table 4)。例如:在P1蛋白中K35/T和V37/K 2个氨基酸的变化使蛋白激酶C磷酸化位点KQV变成TQK;HC-Pro蛋白中H65/N的变化使N-糖基化位点HLSW变化为NLSW;P3蛋白中L169/S的变化使蛋白激酶C磷酸化位点LEK变为SEK,等。其中,P1蛋白中在8个功能位点处检测到氨基酸突变,相较其他蛋白质最多。
根据PROSCAN分析结果,可得到SMV-Am中功能位点突变前后的氨基酸序列,之后利用I-TASSER软件对蛋白质进行三维结构模型预测。所得模型结果按照C值(C-score)评估,C值范围从-5到2,该分值越高,模型预测结果的置信度越高,因此选择各蛋白质C值最高的模型,利用三维结构分析软件PyMOL进行结构对比分析,结果正如Fig.7。
图中结果显示,6个蛋白质的整体空间构象均发生不同程度的变化。其中,P1蛋白存在7个氨基酸突变位点,三级结构变化最明显(Fig.7A),NIb也有较为明显的三级结构变化(Fig.7F),而HC-Pro、P3、6K2和NIa-Pro蛋白质的空间构象变化程度相对较小。并且在以上蛋白质中,P1蛋白的 K35/T、V37/K、A137/T、S140/T、K160/R、Y260/C突变位点,HC-Pro蛋白的H82/N突变位点,6K2蛋白的H3/Q突变位点,以及NIb蛋白中的S370/G和N460/S突变位点均引起了其所在功能区域二级结构的改变。
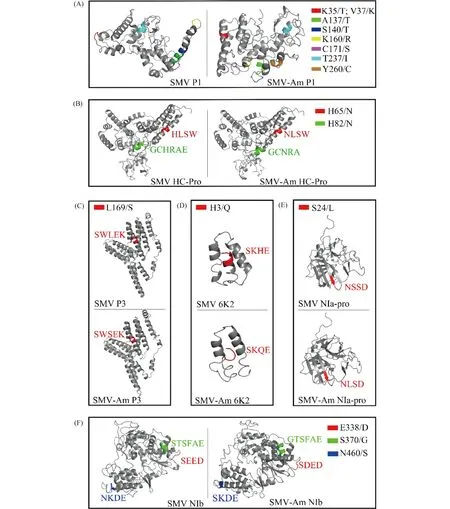
Fig.7 Effects of amino acid mutations at functional sites on protein structures (A)P1 protein; Red: Protein kinase C phosphorylation site; Green: N-myristoylation site including a Protein kinase C phosphorylation site; Blue: Protein kinase C phosphorylation site; Yellow: Protein kinase C phosphorylation site; Magenta: Protein kinase C phosphorylation site; Cyan: Casein kinase II phosphorylation site; Orange: Casein kinase II phosphorylation site.(B)Hc-pro protein; Red: N-glycosylation site; Green: N-myristoylation site.(C)P3 protein; Red: Protein kinase C phosphorylation site and Casein kinase II phosphorylation site.(D)6K2 protein; Red: Casein kinase II phosphorylation site.(E)NIa-pro protein; Red: N-glycosylation site.(F)NIb protein; Red: Casein kinase II phosphorylation site; Green: N-myristoylation site.Blue: Casein kinase II phosphorylation site
2.6 SMV-Am重组特征分析
利用RDP4软件对SMV-Am分离物以及NCBI中所得100个不同SMV分离物的全基因组进行重组分析。以RDP、GENENCONY、BootScan、MaxChi、Chimaera、SiScan和3SEQ 7种方法进行分析,当存在有至少3种方法检测到P≤ 1.0×10-6时即认为存在重组事件的发生。结果表明,RDP、GENENCONY、BootScan、MaxChi、Chimaera、SiScan、3SEQ 7种方法均在SMV-Am基因组6 560~8 950 nt位置处检测到重组事件的发生(Table 5和Fig.8),涉及到的蛋白质有NIa-Pro、NIb以及CP,并且RDP4的分析结果显示,SMV-Am的主要亲本为SMV-XFQ012分离物(GenBank登录号:KP710875.1),次要亲本为SMV-pCB301-SC15分离物(GenBank登录号:MH919386.1),两亲本均来自中国。

Table 4 Amino acid mutation analysis

Table 5 Recombination analysis of SMV-Am

Fig.8 Analysis of SMV-Am recombination based on RDP algorithm Blue line: identity of KP710875.1 and SMV-Am; Purple line: identity of MH919386.1 and SMV-Am; Yellow line: identity of KP710875.1 and MH919386.1; Red box: recombinant region
3 讨论
白术作为一种重要的药用植物,具有很高的经济价值,近年来日渐加重的白术病害严重影响着白术的产量与质量。本实验室之前在对山西省新绛县、河北省安国市的白术病毒病害调查中发现,白术的田间病株率可达90%以上,其中病毒病害植株皆表现花叶症状。通过对病样检测,发现侵染白术的病原为蚕豆萎蔫病毒2号和黄瓜花叶病毒,并分别命名为CMV-Am[15]和BBWV-2-Am[16]。除此之外,有关白术花叶病的报道十分有限。而本研究通过dsRNA提取以及SIA检测等方法,从河北省白术种植基地中的感病白术叶片中检测到SMV,并获得该分离物(SMV-Am)的全长基因组,经过序列比对分析以及系统进化分析发现,SMV-Am与SMV-Liaoning分离物亲缘关系最近。本研究是关于白术花叶症状病原的又一个报道,同时为白术病毒病的流行预防以及防控提供理论基础。
在长期的研究中,SMV的自然寄主范围很小,主要为大豆和豌豆等豆科植物,然而本研究报道了SMV的一种新寄主——白术。这可能是SMV在进化过程中发生了某种变异。基因组的突变和重组是病毒变异进化的重要因素。研究者们在对马铃薯Y病毒属病毒的长期研究中发现,该家族病毒基因组编码的HC-Pro[17-18]、P3[19]和CP[20]蛋白质与病毒寄主范围相关,而本文在对SMV-Am核苷酸与氨基酸序列分析后发现,该分离物除CP蛋白外,与寄主范围相关的蛋白质HC-Pro和P3均发生了不同程度的突变现象,而这一现象在另一个蛋白质——P1蛋白中表现的尤为明显;在以往的研究中发现,Potyvirus病毒的P1蛋白在不同病毒以及同一病毒不同株系之间均会出现较大的变异[21],其作用是在病毒基因组翻译形成多聚蛋白质前体后,P1会特异性的识别和切割P1与HC-Pro之间的酶切位点,将多聚蛋白质进一步加工,最为重要的是,P1蛋白对HC-Pro发挥促进作用,能够提高HC-Pro的致病能力和沉默抑制作用[22],那么SMV-Am可以侵染白术是否与P1和HC-Pro蛋白互作相关?还是P1蛋白本身的突变在一定程度上也直接影响到SMV-Am寄主范围的变化?Shan等[23]发现,A型P1蛋白的缺失突变会使李痘病毒(plum pox virus, PPV)的寄主发生变化。除了关键蛋白质的突变外,重组也是造成病毒能够适应新寄主的重要原因[24-27],Yang等[28,29]对SMV-SC7株系的研究发现,该株系由SMV和菜豆普通花叶病毒(bean common mosaic virus, BCMV)之间发生重组产生的,且属于强毒株,可以侵染非自然寄主烟草。本研究利用RDP4软件对SMV-Am基因组进行了重组分析,发现在其6 560~8 950 nt区域内存在重组事件,主要亲本和次要亲本分别为SMV-XFQ012和SMV-pCB301-SC15,重组的发生也可能是SMV能侵染白术的原因。然而,对于病毒侵染的具体机制,需要进一步构建侵染性克隆并进行相关试验才有望解答,而本研究为SMV的致病机制研究提供了新的切入点和科学依据。