扩增子测序法筛选结核分枝杆菌的耐药突变
2022-09-07杨鹏鹏罗娟秀宋玉竹李晓非夏雪山张阿梅
杨鹏鹏, 罗娟秀, 宋玉竹, 李晓非, 夏雪山*, 张阿梅*
(1)昆明理工大学 生命科学与技术学院 分子病毒实验室,昆明 650500;2)昆明市第三人民医院,临床检验科,昆明 650041)
结核病(tuberculosis,TB)是由结核分枝杆菌(Mycobacteriumtuberculosis,MTB)引起的传染性疾病,也是单一病原菌感染导致死亡的首位原因。2018年,全球约1/4人口感染了结核分枝杆菌,大约1 000万人(从900万到1 110万不等)发展为活动性结核病[1]。尽管多种抗结核药物已在临床上大量应用,但一线抗结核药物仍是主要治疗药物。然而抗结核药物的广泛使用带来了不可忽视的问题,例如耐药结核(drug-resistant TB,DR-TB)患者的增加。2018年,有484 000例结核病患者被诊断为利福平耐药结核病(rifampicin-resistant TB,RR-TB),78%的RR-TB患者属于耐多药结核病(multidrug-resistant TB,MDR-TB)患者。印度(27%)、中国(14%)和俄罗斯联邦(9%)的耐药结核患者约占全球耐药结核患者的一半[2]。据统计,2018年只有139 114例MDR-TB患者得到了有效的诊断和治疗[2]。而未被及时诊断和治疗的耐药患者中有43%存在于印度或中国,因此,对一线耐药结核患者进行快速准确的诊断是提高治愈率和降低死亡率的有效措施之一[2]。
及时准确的诊断是成功治疗的关键,但是临床应用的诊断技术和方法仍然存在一些问题[3-4]。药物敏感性试验(drug-susceptibility testing,DST)是判断耐药结核菌株的金标准,但该技术存在操作复杂、培养时间长等缺点[5]。分子诊断是目前常用的临床诊断方法之一,也是控制耐药结核病流行、传播和治疗的关键因素之一[6]。可以使用一代测序方法来鉴定耐药基因的突变,但它的缺点是通量较低且价格昂贵[7-8]。中等通量方法,例如基因芯片,但其在提高检测速度的同时却降低了准确性和灵敏度[9-10]。基于二代测序技术发展的扩增子测序是一种高通量、灵敏且准确的检测方法。目前,二代测序技术已被广泛用于诊断疾病、研究病因和致病机制等方面[11-13],但在耐药结核菌的突变检测中应用较少。
本文建立了一种快速、准确、灵敏且覆盖范围广的扩增子测序方法,用以筛选结核耐药菌株的17个一线耐药基因的突变。
1 材料与方法
1.1 结核分枝杆菌药物敏感性检测
由昆明市第三人民医院检验科的医生使用罗氏培养基培养26个临床结核分枝杆菌菌株,1个对药物敏感的结核分枝杆菌临床分离菌株作为阴性对照。使用一线抗结核药物和相对应的浓度对所有收集的结核菌株进行药物敏感性检测:异烟肼(0.2 mg/L);利福平(40 mg/L);链霉素(4.0 mg/L);乙胺丁醇(2.0 mg/L);吡嗪酰胺(100 mg/L)。该研究得到昆明理工大学医学伦理委员会的批准。
1.2 一线耐药基因的选择和PCR扩增
用G +细菌基因组DNA小量提取试剂盒(庄盟生物,中国)提取结核分枝杆菌菌株的基因组DNA。根据结核分枝杆菌菌株H37Rv的基因组序列设计引物(GenBank登录号:CP003248.2)。共设计了16对引物,用于扩增5种一线抗结核药物的17种耐药基因。抗异烟肼的基因包括katG、sigI、ahpC、oxyR、kasA、ndh、nat和mshA基因;抗利福平的基因包括rpoA、rpoB和rpoC基因;抗乙胺丁醇的基因包括embA、embB和embC基因;抗吡嗪酰胺的基因包括pncA和rpsA基因;抗链霉素的基因为rpsL基因。扩增引物见Table2。
PCR反应体系为50 μL,其中包含25 μL 2×Phanta®Turbo Master Mix(Vazyme,中国),30 ng 结核分枝杆菌基因组DNA和0.2 μL正向/反向引物。PCR条件如下:95 ℃预变性3 min,95 ℃变性15 s,65 ℃退火15 s,72 ℃延伸(延伸时间见Table 1),重复35个循环,72 ℃延伸5 min。通过使用琼脂糖凝胶DNA回收试剂盒(TIANGEN,中国)纯化扩增产物。
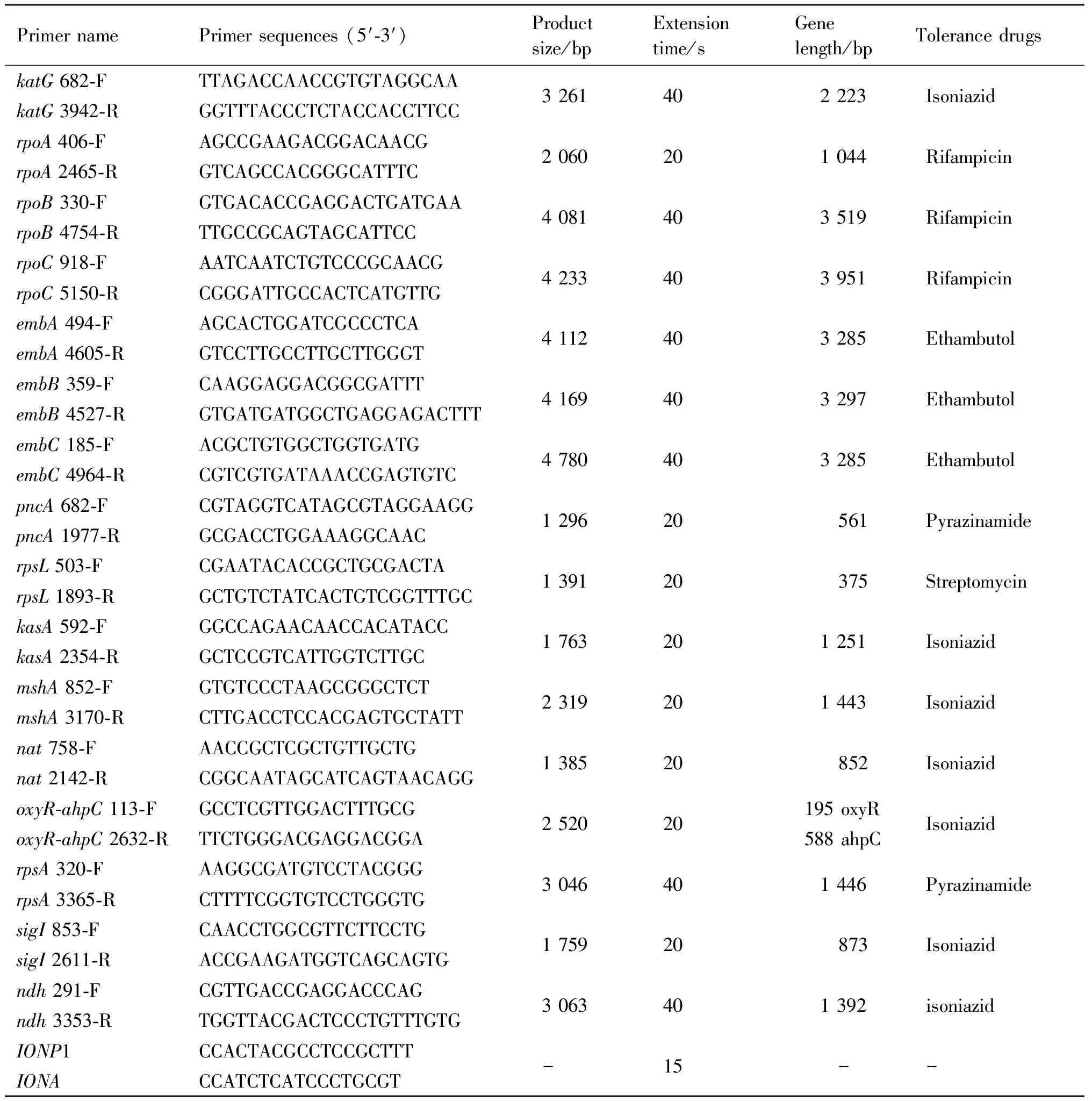
Table 1 The information of target fragment amplification primers and barcode evaluation
1.3 使用Ion TorrenTM PGM系统进行扩增子测序
1.3.1 构建扩增子文库 根据说明书,使用Ion XpressTMPlus Fragment Library Kit(Thermo Fisher Scientific,美国)将PCR扩增子构建到文库中。使用Qubit®dsDNA HS Assay Kit(Thermo Fisher Scientific,美国),将每个文库的浓度归一化至100 pmol/L,以进行下一步研究。
1.3.2 乳液PCR 根据Ion PGMTMTemplate OT2 Reactions 400 Kit(Thermo Fisher Scientific,美国)的说明书,在Ion OneTouchTM2 Instrument上初始化扩增子。再使用文库DNA模板进行乳液PCR。通过使用Ion OneTouchTMES(Life Technologies,美国)富集乳液PCR扩增子。
1.3.3 Ion TorrenTM PGM 测序 使用Ion 314TMChip Kit v2和Ion Personal Genome Machine®(PGMTM)System(Life Technology,美国)对富集的乳液PCR扩增子进行测序。
1.4 数据分析
用Ion Torrent Variant Caller软件分析测序结果,首先删除了低质量的(<50个reads)或潜在错误的数据。所有测定序列以H37Rv菌株作为参考序列进行分析。通过一代测序进一步验证了由二代测序鉴定出的突变,并在数据库(https://tbdreamdb.ki.se/Info/)中检索检测到的突变,初步分析这些突变是否已被报道。对检测到的新突变在9种分枝杆菌中进行保守性分析:结核分枝杆菌、非洲分枝杆菌、亚洲分枝杆菌、鸟分枝杆菌、牛分枝杆菌、卡氏分枝杆菌、胃分枝杆菌、堪萨斯分枝杆菌和猿分枝杆菌。从GenBank下载参考序列进行突变的保守性分析:WP_036362533.1、WP_003876960.1、WP_015294659.1、ORV60366.1、WP_023368850.1、KAA1250968.1、AMQ40394.1、KFW49971.1和NP_218311.1 用于分析embA基因;WP_051545718.1、WP_062908845.1、WP_015304020.1、WP_036411168.1、WP_152343010.1、KAA1250969.1、PRI02116.1、ALE45293.1和NP_218312.1用于分析embB基因;YP_009361183.1、WP_036362533.1、WP_010948786.1、WP_015304018.1、WP_036411191.1、WP_023368851.1、KAA1250966.1、NP_218310.1和KBF93337.1用于分析embC基因;NP_216424.1、CAA58266.1、WP_036359980.1、WP_019733604.1、WP_015290248.1、WP_036416255.1、WP_023364424.1、WP_044506092.1和KBI31267.1 用于分析katG基因;OBK93179.1、AYJ06713.1、ORV80661.1、EP43955.1、ORJ60792.1、NP_217974.1、AMQ40106.1、PHO98980.1和CCK65673.1用于分析rpoA基因;WP_051544984.1、WP_121035721.1、WP_015292360.1、WP_084293527.1、KEP43840.1、WP_084949953.1、NP_215181.1、AMQ37657.1和AKO23650.1用于分析rpoB基因;NP_215182.1、WP_036352589.1、WP_062899648.1、WP_014000349.1、ORV79512.1、ORB85546.1、ORJ60893.1、PRH95962.1和YP_009358020.1用于分析rpoC基因;WP_036355980.1、AYJ04498.1、WP_015303118.1、ORV74789.1、KZS77521.1、ORJ57881.1、NP_216146.1、PRH99054.1和KFW56032.1用于分析rpsA基因。用SWISS-MODEL(https://swissmodel.expasy.org/)预测蛋白质的局部结构是否发生变化。
2 结果
2.1 药物敏感检测分析
通过药物敏感性实验将26株临床菌株确认为耐药结核菌株,包括11株单耐药菌株和15株多耐药菌株。单耐药菌株分别为:5株吡嗪酰胺耐药株(45.5%),4株利福平耐药株(36.4%),乙胺丁醇和链霉素耐药株各1株,未发现针对异烟肼的单耐药菌株。26株临床耐药结核株中,耐异烟肼、利福平、链霉素、乙胺丁醇和吡嗪酰胺的比例分别为53.8%(14/26),61.5%(16/26),50.0%(13/26),26.9%(7/26)和 46.2%(12/26)(Table 2)。
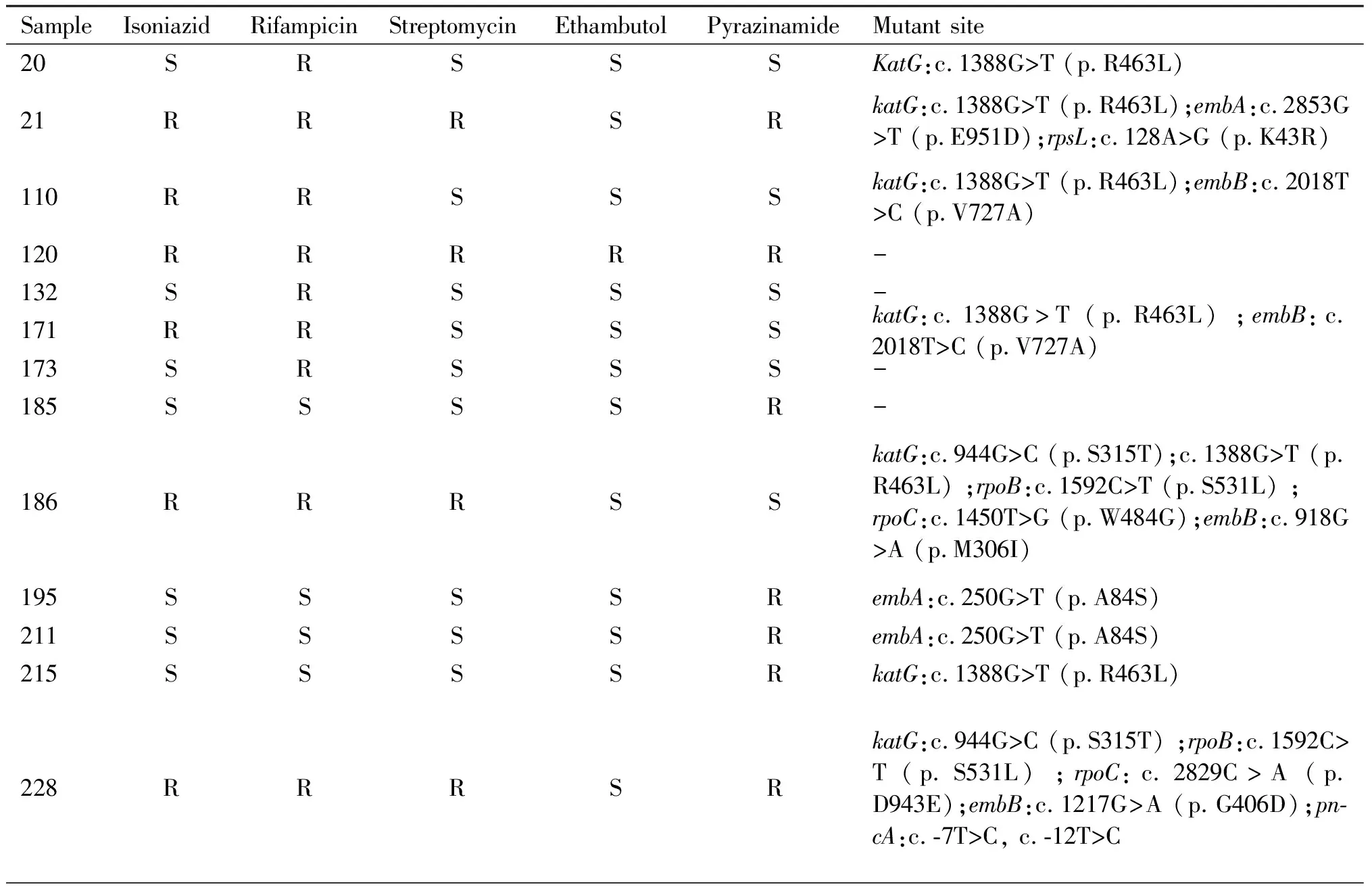
Table 2 Summary of DST results and mutations of each sample
2.2 Ion PGMTM 系统测序报告
每张Ion 314TM芯片可以检测10株结核分枝杆菌临床株。每张芯片最终获得71.1 M数据,覆盖率达到70%(Fig.1A),负荷率为49%(Fig.1B)。共获得622 597个reads。筛除无模板的reads(占总reads的1%),多克隆reads(占总reads的19%),低质量reads(占总reads的14%),测试片段(占总reads的9%)和衔接子二聚体reads(占总reads的1%),有效reads的数目为376 086。有效reads的扩增长度在100 bp至300 bp之间,平均长度为189 bp(Fig.1C)。67.6 M数据(约97%)成功匹配,平均覆盖深度为1 565.7X(Fig.1D)。当平均读取长度为1X时,精度达到99.2%(Fig.1E)。
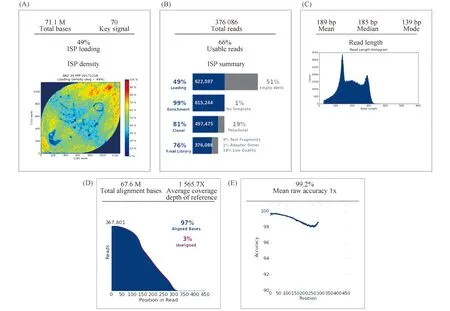
Fig.1 Report of the Ion PGMTM System (A)Total bases and ISP density;(B)Total reads and ISP summary;(C)Read length;(D)Total alignment bases and average coverage depth;(E)Mean raw accuracy
2.3 测序结果
仅在10个基因(包括katG、rpoA、rpoB、rpoC、embA、embB、embC、pncA、rpsA和rpsL基因)中检测到耐药突变的存在,共鉴定出65个突变位点,所有突变均经过一代测序验证,新发现的突变测序图见Fig.2,另外7个基因(kasA、mshA、nat、oxyR-ahpC、sigI和ndh基因)中未检测到突变。检测到的65个突变可分为38种类型,其中热点突变占总突变的50.8%(33/65),包括embB基因的c.916A>T/G和c.918G>A,katG的c.944G>C和c.1388G>T,rpoB基因的c.1592C>T,rpsL基因的c.128A>G和c.263A>G。9个稀有突变分别为embA基因的c.-12C>T,embB基因c.3070G>A,katG基因的c.1388G>A和c505G>A,rpoC基因的c.1448T>A、c.-12T>C、c.-7T> C和c.211C> T,pncA基因的c.233G> A。
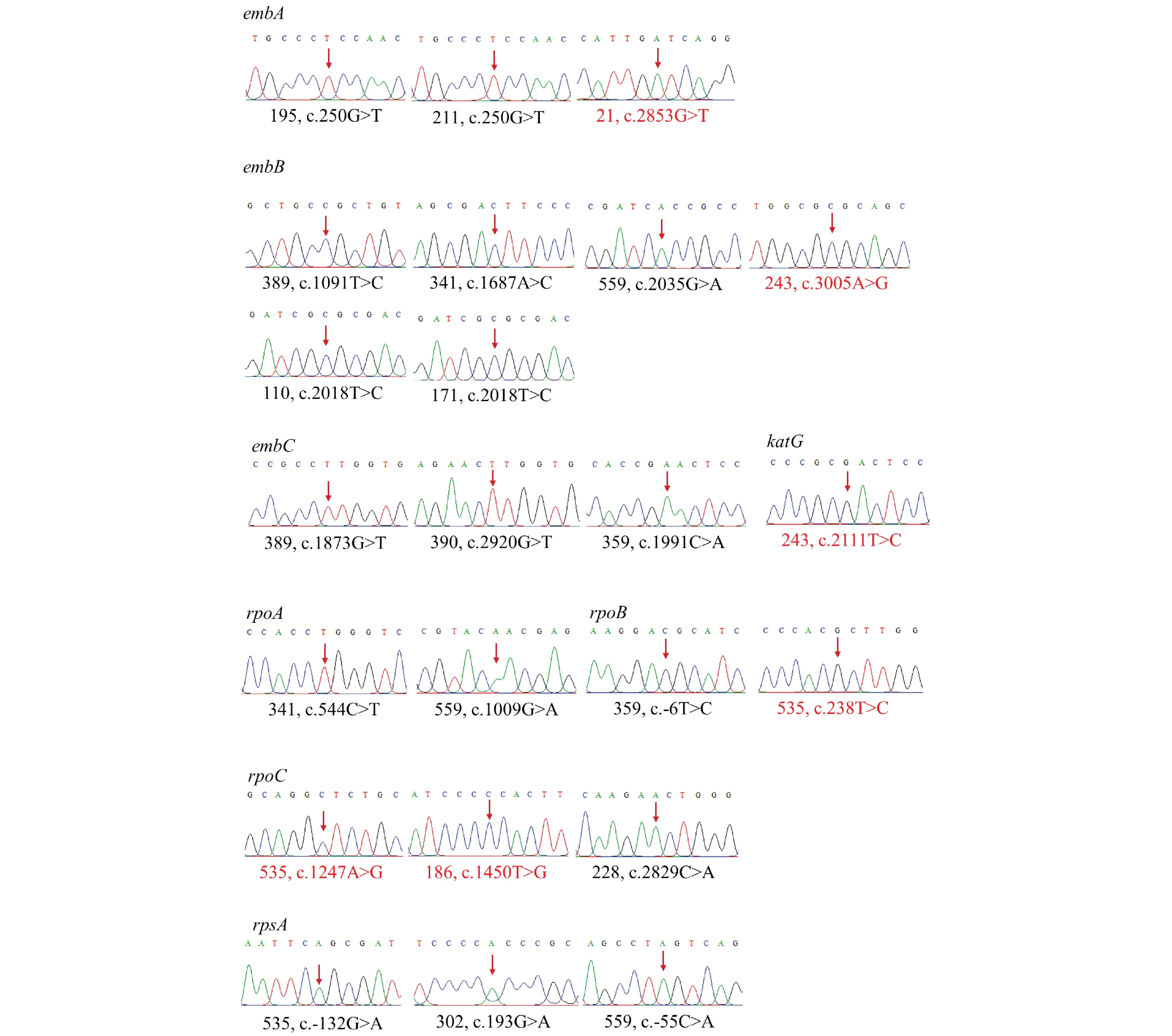
Fig.2 Sanger sequencing peak map of newly discovered mutation sites The mutation sites identified by the red characters are complementary to the reference sequence due to the use of reverse primers for sequencing
与结核病耐药抗性突变数据库检索对比后,发现23个检测到的突变未被报道,分属于21个突变类型。新突变分别为embA基因的c.2853G>T和c.250G>T,embB基因的c.1091T>C、c.1687A>C、c.2018T>C、c.2035G>A和c.3005A>G,embC基因的c.1873G>T、c.1991C>A和c.2920G>T,katG基因的c.2111T>C,rpoA基因的c.544C>T和c.1009G>A,ropB基因的c.-6T>C和c.238T>C,rpoC基因的c.1247A>G、c.1450T>G和c.2829C>A,rpsA基因的c.-55C>A,c.-132G> A和c.193G>A。
在5个临床耐药结核菌株(包括3个单耐药菌株和2个耐多药菌株)中未检测到任何突变,在9个结核菌株中发现了同一耐药基因的2个共存突变(Table 2),分别存在于katG(5个)、embB(2个)、rpoB(1个)和pncA基因(1个)。katG基因的所有5个共存突变中均包含突变c.1388G>T。突变数量最多的菌株(样品#535)中共检测到10个耐药突变。
2.4 新突变的基因序列保守性分析和蛋白质结构预测
保守性分析结果显示14个新突变具有高度保守性(Fig.3),分别是embA基因的p.A84S(c.250G>T)和p.E951D(c.2853G>T);embB基因的p.L364P(c.1091T>C)、p.I563L(c.1687A>C)、p.A679T(c.2035G>A)、p.V727A(c.2018T>C)和p.H1002R(c.3005A>G);katG基因的p.L704S(c.2111T>C);rpsA基因的p.A65T(c.193G>A);rpoA基因的p.R182W(c.544C>T)和p.D337N(c.1009G>A);rpoC基因的p.N416S(c.1247A>G)、p.W484G(c.1450T>G)和p.D943E(c.2829C>A)。用SWISS-MODEL对这些保守突变的结构进行预测(Fig.4)。结果表明,除rpoA基因的突变p.D337N(该突变未能准确预测),所有错义突变均导致氨基酸发生改变,进而改变蛋白质的空间结构,因此这些突变可能导致其蛋白质功能的改变。

Fig.3 Evolutionary conservation analysis of the amino acid changes with eighteen novel missense mutations Nine Mycobacterium species(Mycobacterium tuberculosis, Mycobacterium africanum, Mycobacterium asiaticum, Mycobacterium avium, Mycobacterium bovis, Mycobacterium canettii, Mycobacterium gastri, Mycobacterium kansasii, and Mycobacterium simiae)were used in this analysis
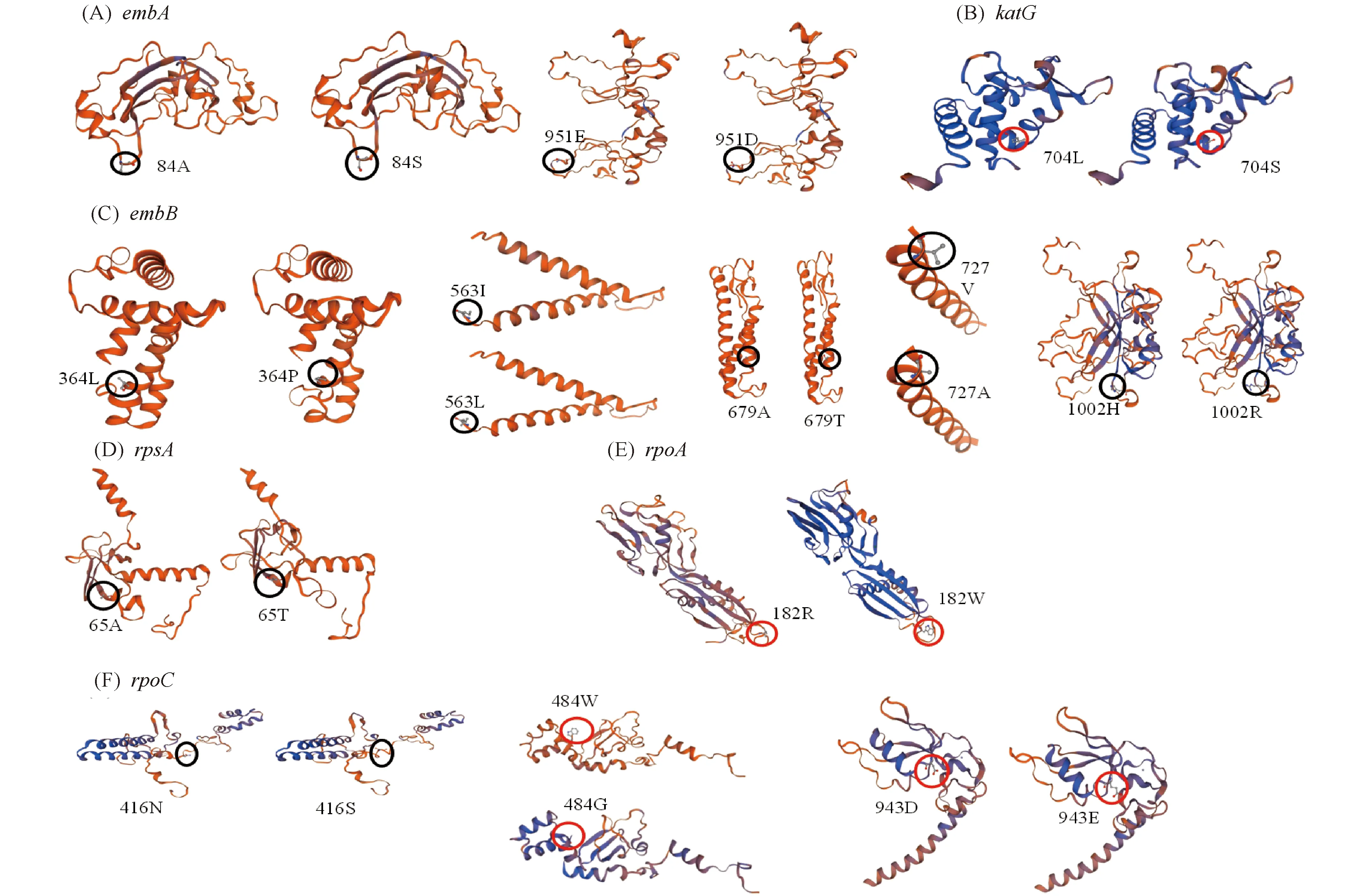
Fig.4 Partial structures prediction with missense mutation of 13 conserved mutations (A)Partial structure predictions with missense mutations in the embA gene;(B)Partial structure predictions with missense mutations in the katG gene;(C)Partial structure predictions with missense mutations in the embB gene;(D)Partial structure predictions with missense mutations in the rpsA gene;(E)Partial structure predictions with missense mutations in the rpoA gene;(F)Partial structure predictions with missense mutations in the rpoC gene
3 讨论
耐药结核病是亟待解决严重的社会和健康问题之一,对耐药结核病患者的快速准确诊断在标准化治疗中发挥重要作用[14]。目前检测耐药结核菌株的技术分为基于表型和基因型的检测方法[15-16]。尽管基于表型的药物敏感性实验是诊断耐药结核的“金标准”,但由于其实验条件严苛、耗费时间长、易污染等缺点,临床上应用效果不尽理想。分子诊断方法已广泛用于临床和实验室中检测结核菌的耐药突变,主要包括TaqMan探针法,Gene Xpert MTB/RIF 和基因芯片等[17-20]。但是,这些分子检测方法主要检测热点耐药基因的热点突变区域,导致一些稀有突变或新突变不能被发现,从而延误耐药结核患者的治疗。因此通过扩增一线抗结核药物相关所有基因进行测序分析,才能解决无法检测到稀有突变和新突变位点的问题。Sanger 测序法具有测定序列较长和准确性高等优点,曾在人类基因组计划中发挥了关键的作用,并应用于肿瘤学、遗传学和微生物学等多种学科的研究。但其检测成本高,检测通量低,无法满足大规模测序的需求。二代测序降低了检测成本,在保证准确性的同时缩短测序时间,因此已广泛的应用于全基因组分析、外显子组分析、转录组分析、宏基因组分析和基因分型等方面[21]。本文构建了扩增子测序的检测方法,可同时检测10株临床结核菌株的17个一线耐药基因。在26个耐药结核菌株中检测到21种新突变,这表明使用该方法可以有效地检测到耐药结核菌株的新突变,从而对临床患者是否耐药进行诊断。此外,在7个选定的基因中均未发现突变,这表明热点候选基因依然是结核菌的耐药突变的重点检测对象。
云南省耐药结核菌株中,单耐药结核和多耐药结核分别占42.3%和57.7%。Hu等人[22]的研究中多耐药结核病发生率为79.51%,明显高于本研究中的多耐药结核频率(P= 0.036)。可能是人群差异和样本量的不同导致多耐药比例存在差异。而耐药基因发生突变的频率在我国不同地区同样存在差异,这些差异可能是由于检测方法的差异所致,本研究中耐药基因突变频率高于文献报道的湖南省耐药突变频率[23]。这些结果提示,需要建立更全面的检测方法以便发现更多的耐药突变位点。
本研究中5个药敏试验确定的表型耐药菌株(包括3个单耐药菌株和2个耐多药菌株)未在17个耐药基因中检测到突变,而且一些表型耐药菌株的药敏试验结果和基因型不完全一致。该结果与一些报道具有相似性,Ko等人分析了36个表型耐药菌株的8个耐药相关基因,结果发现,基因型与表型的耐药分析不一致,不同耐药基因发生突变对耐药表型的预测值在44%~100%之间[24]。而辜吉秀等人的研究中耐药基因突变对耐药表型的预测值在54.55%~100%之间[25]。由此可见,耐药突变的基因型与表型之间存在一定的差异,且不同药物的差异度存在较大区别,可能是由以下原因引起这种差异:(1)耐药菌株中可能存在一些未被发现的耐药基因;(2)已知的针对某种药物的耐药基因突变也影响了其他药物的耐药性;(3)尽管在治疗药物相对应的耐药基因中未发现突变,但其他耐药基因突变的协同作用可能是多耐药的原因。本研究检测的120号菌株中,未在已知的17个耐药基因中检测到耐药突变,这一结果也提示以上推测的可能性。这些推测是否可以解释耐药菌株表型和基因型之间的矛盾结果,需要进一步研究。
总之,本研究构建了一种基于二代测序技术的扩增子测序方法,用以筛选已知一线抗结核药物耐药基因的所有潜在突变,该方法有望用于临床,为耐药结核患者的临床快速准确诊断和有效治疗提供技术依据。