tRNA衍生片段的鉴定及对靶分子的调节机制
2022-09-07黄炯杰陈乐意
黄炯杰, 陈乐意, 廖 奇
(宁波大学医学院预防医学系, 浙江省病理生理重点实验室, 浙江,宁波 315211)
tRNA是一类具有转运氨基酸功能的非编码RNA(non-coding RNA,ncRNA),成熟的tRNA具有三环四茎的结构:D环、反密码子环和TψC环,D茎、反密码子茎、TψC茎和接受氨基酸茎[1]。近年来,科学家们发现:在氧化、饥饿和应激等特殊条件下,细胞或组织中特定的核酸酶例如Dicer、Rnase A超家族中的血管生成素(angiopoietin,ANG)、核糖核酸酶5等会在前体或成熟tRNA的环上进行切割,产生特定的小片段RNA,这些RNA被称为tRNA衍生小分子(tRNA-derived small RNA,tsRNAs)[2-4]。
tsRNAs根据切割位点的不同可分为tiRNA(tRNA-derived stress-induced RNA,tiRNA)和tRF(tRNA-derived fragment,tRF)[5,6]。tiRNA是在应激条件下由血管生成素在tRNA反密码子环内或环附近切割而产生的一类RNA[7],可分为5′tiRNA(约30~35nt)和3′tiRNA(约40~50 nt)。前者来源于成熟tRNA的5′端至反密码子环,后者来源于反密码子环末端至成熟tRNA的3′端。tiRNA主要存在于细胞质中,但在人的血液循环系统中也可被检测到,因此,tiRNA可以作为疾病的生物标志物。
tRF是由特异性核酸酶例如Dicer、血管生成素等在D环、TψC环等或环附近切割产生,也可通过自身消化裂解产生,长度通常为14~32 nt。tRF根据切割位点的不同可分为1-tRF、3-tRF、5-tRF和i-tRF(尚未明确切割位点)。1-tRF来源于前体tRNA的3′末端序列,其3′端具有多聚尿苷酸(ploy-U)。3-tRF由Dicer、血管生成素等核酸酶在成熟tRNA的TψC环或环附近切割产生,长度约为13~22 nt,通常包括1个CCA的末端序列。3-tRF根据切割位点的不同又可分为tRF-3a(3′端至TψC环前)和tRF-3b(TψC环内裂解)[8]。5-tRF在D环或环附近裂解产生,长度约为14~30 nt,以腺嘌呤(A)作为尾巴。5-tRF根据切割位点的不同,同样也可以分为tRF-5a(5′端至D环前;14~16 nt)、tRF-5b(D环后剪切;22~24 nt)和tRF-5c(反密码子前剪切;28~30 nt)[8,9]。而i-tRF是近年来发现的一类新的tRF,来源于成熟tRNA内部区域,因此不能根据以上方法进行划分[10,11],其中2-tRF是一种只包含反密码子环的亚型,一般在缺氧条件下诱导产生。除2-tRF以外,还有一种源自前体tRNA的新tRF。据报道,它是由于tRNA剪接缺陷产生的,可能与神经元的丢失有关[12]。在以上4种tRF中,5-tRF存在于细胞核,1-tRF和3-tRF存在于细胞质,i-tRF尚不明确,然而这些tRF的定位仍然存在争议。Table 1列出了各类tsRNAs的特点。

Table 1 Classification and structural characteristics of tsRNA
目前,通过RNA高通量测序分析和定量实时逆转录聚合酶链反应(qRT-PCR)可对外周血的tRF进行检测和验证。随着高通量测序和芯片等技术的发展,相应的鉴定手段和工具也不断涌现,为tRF的深入研究提供了一定的技术支持,让越来越多的tRF被发现并深入研究。据报道,tRF具有多种生物学功能例如调节翻译水平、调控基因表达和抑制细胞凋亡等,并可作为肿瘤等疾病的生物标志物[13,14]。若对tRF的鉴定方法和相关数据库作进一步整理和归纳,将有利于日后开展对tRF的相关检测与研究,因此本文就关于tRF的鉴定方法、数据库、对靶分子的调节机制及其与人类疾病的关系作一综述。
1 tRNA衍生片段的鉴定
为了满足对tRF深入研究的要求,相关的鉴定方法和tRF数据库是必要的,他们也可以为开发鉴定tRF的软件提供一定的理论基础。目前,tRF的大规模鉴定主要基于已有的tRNA数据库和小RNA测序数据,即将所测的小RNA测序序列与已知tRF的序列进行比对,从而鉴定tRF的方法[15]。该方法准确性虽有待提高,但为tRF的研究提供了重要的线索。
由于tRF和miRNA的长度相当,研究人员早期主要采用类似鉴定miRNA的方法,即利用小RNA深度测序数据对tRNA进行表达检测,通过序列比对鉴定可能的tRF。早在2015年就出现2种基于测序序列和tRNA序列鉴定tRF的方法。1种是由Telonis等人提出的“完全匹配模式”,即将测序所得read序列(3′末端的3个核苷酸置换为CCA)与已知tRNA序列以完全匹配的方式进行比对,目的是为了避免由于同一反密码子tRNA的各个拷贝之间的序列相似性和不同密码子翻译相同氨基酸所造成的tRF来源的错误,以减少假阳性。他们以此确定了除5-tRF、3-tRF、1-tRF以外的第四类tRF:i-tRF[16]。另一种是由Selitsky和Sethupathy开发的tDRmapper,将筛选后的测序所得read序列(Phred+33质量分数在28以上;长度在14~40 nt;能在FASTQ文件中出现至少100次)与前体或成熟tRNA进行比对、鉴定[16,17]。此外,他们采用“错误型”层次比对方案来控制由化学修饰导致的错配[17]。每一步,tDRmapper都将上一步未匹配的序列作为输入,将其与前体或成熟tRNA进行比对(依次为1个碱基错配、1个碱基删除、2个碱基错配、2个碱基删除和3个碱基删除),然后每一步所发现的可以匹配到tRNA的序列将被收集、注释为候选的tRF。
这两者鉴定方法最大的区别在于:是否以“完全匹配”作为标准,将测序所得的read序列在参考基因组中进行比对。tDRmapper考虑到基因组中tRNA基因标注的不完整性和计算能力不足等问题,将完全匹配区域以外或非tRNA来源的序列也当作tRF或tiRNA,从而导致假阳性率较高;而Telonis等人由于采取完全匹配的模式,尽管假阳性率较低,但可能会漏掉一些潜在的tRF[16,17]。因此,tDRmapper是以增加假阳性为代价识别更多潜在的tRF;而Telonis等人的方法则以完全匹配的方式来确保tRF鉴定的准确性。
总结以上两种方法,目前出现1种基于“Telonis”和“tDRmapper”的鉴定方法——tRF2Cancer,一个基于小RNA测序数据鉴定tRF,并评估其在癌症中表达水平的网上服务[18]。tRF2Cancer由tRFfinder(基于小RNA深度测序数据的tRF鉴定工具)、tRFinCancer(提供tRF在不同癌症类型中的表达水平)和tRFBrowser(tRF的起源和化学修饰情况)组成。其鉴定tRF的流程如下:(1)提取前体tRNA基因中的tRNA基因和tRNA基因3′端上、下游100 bp的序列;(2)去除成熟tRNA序列的内含子,并在3′末端添加CCA序列;(3)利用bowtie比对工具将测序所得的read序列与人类基因组进行比对,去除未匹配的序列;(4)将上一步留下的序列与已知人类转录本序列例如mRNA、snoRNA、snRNA、rRNA、microRNA和重复序列进行比对,去除完全匹配的序列;(5)继续将上一步剩下未匹配的序列与(1)和(2)获得的前体tRNA和成熟tRNA序列进行比对,只有与前体tRNA转录本同源的序列才被认为是tRF;(6)最后,利用tRFfinder工具根据位点、长度和表达丰度进一步鉴定可能的tRF。tRFfinder能较为准确地识别tRF和区分降解片段,在完全匹配和完全否定之间取得了较好的平衡。但是对于某些发生了化学修饰的tRNA,tRFfinder也无法检测,这也是它的一个局限。
另外,SPORTS1.0是近年来开发的另一种注释和分析tRF的生物信息学工具[19]。主要包括4个步骤:预处理,序列比对,注释输出和注释汇总。即(1)预处理小RNA测序序列:去除接头序列,丢弃长度超过定义范围和那些不带ATUCG碱基的序列。(2)利用Bowtie比对工具将过滤所得的read序列与参考基因组和已知数据库(miRBase、rRNA数据库、GtRNAdb、piRNA数据库、Ensembl、Rfam等)的RNA序列进行比对。(3)根据比对结果获得与miRNA、rRNA、piRNA和tRF等相匹配的序列,并基于自编的程序识别tsRNAs的位置(5′末端、3′末端或3′CCA端)和计算表达水平。(4)对识别的tsRNAs进行统计,例如长度分布和错配情况等。SPORTS 1.0可以分析68个物种的sRNA-seq数据,提供了一个较全面的sRNA图谱,还可以显示tsRNA和sRNA的动态调节,为研究奠定了很好的基础。此外,还有Pagès[20]等人开发的SeRPeNT[20],尽管是为小非编码RNA而设计的鉴定流程,但也同样适用于tiRNA和tRF的鉴定。Table 2主要展示了不同tRF鉴定工具的异同点。

Table 2 The features of tDRmapper, tRF2Cancer, SeRPeNT, SPORTS1.0 and tRFdb
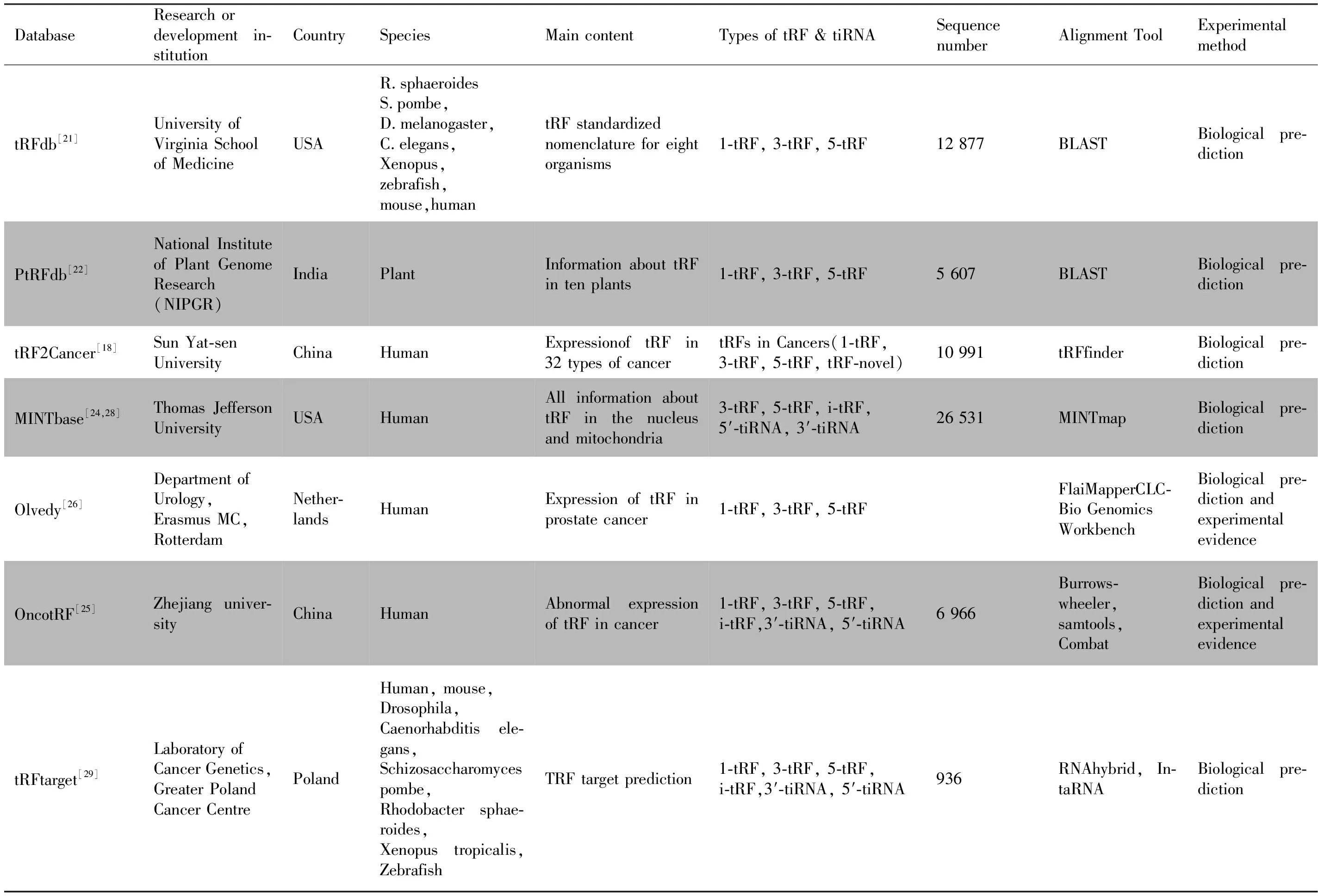
Table 3 The databases of tiRNA & tRF
目前的tRF鉴定工具主要是将测序序列与已知tRNA序列进行比对。此外,在全基因组范围内从头鉴定tRF的方法也已证明具有一定的潜力,未来可能需要结合序列、结构和功能等多方面特征,建立新的tRF鉴定策略和方法,以提高tRF鉴定的准确度。这些被鉴定的tRF将被整合到一个数据库中,用来分析其内在的调节机制和与人类疾病的关系。这些数据库的出现也方便了研究人员查询相关tRF。
2 tRNA衍生片段数据库
随着鉴定水平的提升和被鉴定的tRF量的增加,整合了tRF序列信息、表达水平和疾病关系的数据库越来越多。目前常用的tRF数据库有(1)由美国弗吉尼亚大学医学院研发的tRFdb[21]:包含8个物种的tRF信息;(2)由印度国家植物基因组研究所开发的PtRFdb[22]:收录植物中tRF的相关信息;(3)由意大利卡塔尼亚大学建立的tRFexplorer[23]:收录NCI-60数据库中每种细胞系以及TCGA数据库中每种癌症类型鉴定的tRF及其表达谱;(4)由美国杰斐逊大学维护的MINTbase[24]:收录细胞核、线粒体中的tRF及TCGA数据库中每种癌症类型的tRF信息,;(5)由中国中山大学研发的tRF2Cancer[18]:包括TCGA数据库中已识别的tRF及其在癌症的表达信息。另外还有一些tRF数据库也较为常用,例如tRF在癌症中异常表达的数据库OncotRF[25];前列腺癌tRF数据库Olvedy[26];tRF靶标预测数据库tRFtarget[27]等。下面主要介绍tRFdb、MINTbase、tRF2Cancer、OncotRF和tRFtarget这五种数据库。
2.1 tRFdb
tRFdb是第一个总结有关tRF和其他相关tRNA片段的数据库(http://genome.bioch.virginia.edu/trfdb/)[21]。该数据库记录了从细菌到人类8个物种的tRF信息,并且是第一个对tRF进行统一命名和赋予ID的数据库,展示了每个tRNA序列上所测得的read分布情况,方便研究人员对tRF进行鉴定。这些物种分别为浑球红细菌(R.sphaeroides),裂殖酵母(S.pombe),果蝇(D.melanogaster),秀丽线虫(C.elegans),非洲爪蟾(Xenopus),斑马鱼(zebrafish),鼠(mouse)和人类(human)。但是该数据库只包含了5-tRF、3-tRF和1-tRF这3种tRF类型,不包含最近发现的i-tRF或其他tRNA衍生片段信息,并且由于该数据库的数据都是基于生物信息学预测,因此有待进一步实验验证。
2.2 tRF2Cancer
tRF2Cancer由tRFfinder、tRFinCancer和tRFBrowser三个部分组成(http://rna.sysu.edu.cn/tRFfinder/)[18]。tRFfinder是基于癌症小RNA测序数据对tRF进行鉴定和表达分析的网上服务;tRFinCancer主要记录癌症tRF相关信息的数据库;而tRFBrowser则是用于显示tRF来源和化学修饰位点的工具。
tRF2Cancer数据库收录了基于32种人类癌症的小RNA深度测序数据而鉴定的tRF信息,包括5-tRF、3-tRF、1-tRF和tRF-novel。研究人员可以使用tRFfinder来识别tRF,检测其在癌症中的表达水平,借此来鉴定癌症相关的tRF,以发现癌症新的生物标志物和潜在的治疗靶点。
2.3 MINTbase
MINTbase是一个收录了来源于人体组织细胞中线粒体和细胞核来源的tRF数据库(http://highcharts.com)[24,28]。该数据库包含3种类型的tRF(5-tRF、3-tRF、i-tRF)和2种类型的tiRNA(5′-tiRNA、3′-tiRNA),相比于tRFdb和tRF2Cancer,MINTbase数据量更多,在算法方面有更好的敏感性和特异性,目前已更新到2.0版本。
在MINTbase数据库中,用户可以采用多种方法检索tRF,例如输入tRF类型、名称、序列等,较为灵活、方便。MINTbase数据库关于tRF信息主要有以下几方面:(1)基本信息:tRF序列、tRF可能的基因组来源及其来源tRNA的相关特征,例如tRNA转运密码子等;(2)RNA二级结构及比对情况:tRF在tRNA结构上的分布情况、与成熟tRNA的比对信息;(3)tRF在不同组织和疾病等的表达情况。同时MINTbase还可以接受研究人员上传tRF信息,实时更新,是一个“活”的数据库。但与tRFdb一样,所有的记录都来源于生物信息学预测,数据可信度有待进一步实验验证。
2.4 OncotRF
OncotRF是近年来构建的关于tRF的综合数据库(http://bioinformatics.zju.edu.cn/OncotRF)[25]。该数据库包含4种tRF类型(3-tRF、5-tRF、i-tRF、3′U-tRFs)和2种tiRNA(5′-tRNA、3′-tiRNA)。以往的数据库不是侧重于从小RNA测序数据中识别tRF,就是只包含tRF在不同癌症类型中的表达情况,而OncotRF数据库包含了tRF的异常表达、潜在功能、与基因的共表达关系网络、与癌症临床特征的关联性等信息,是较为实用的数据库。OncotRF数据库分析了11 211个小RNA测序样本,8 776个RNA测序样本,以及TCGA的临床病理注释数据,得到6 966个来自核和线粒体tRNAs的tRF,包括992个5-tRF、799个3-tRF、271个3′U-tRF和4 933个i-tRF。在OncotRF数据库中,用户可以输入想要查询的tRF ID、类型、序列等进行检索,从而获得tRF的相关信息,例如ID、tRF类型、来源tRNAs、基因组位点、tRF长度、序列及在癌症中的表达情况等。
2.5 tRFtarget
tRFtarget是第一个关于tRF靶标预测的数据库(http://trftarget.net)[29]。该数据库记录了8个物种的tRF,这些物种包括人类(human),鼠(mouse),果蝇(Drosophila),秀丽隐杆线虫(Caenorhabditiselegans),粟酒裂殖酵母(Schizosaccharomycespombe),类球红细菌(Rhodobactersphaeroides),非洲爪蟾(Xenopustropicalis)和斑马鱼(Zebrafish),并用2个最新的预测工具RNAhybrid和IntaRNA来预测tRF与mRNA转录本之间的相互作用,以及tRF在靶RNA上的结合位点、结合区域和结合稳定性等信息。该数据库为研究tRF的分子功能和调节机制进一步提供了有力的线索。
2.6 各数据库的优缺点
对比以上几种数据库,tRFdb和MINTbase数据库几乎包含了所有已知的tRF记录,而MINTbase在tRF数据方面会更加全面具体,并且是一个“活”的数据库,但这2种数据库在肿瘤tRF方面的信息相对较少。在疾病谱方面,tRF2Cancer提供了不同肿瘤类型中tRF的表达水平,而OncotRF则包含tRF在癌症中的异常表达情况,更加偏向于临床应用。另外,tRFtarget是一个偏向于基础学术研究的数据库,主要为研究人员对tRF的生物学功能和调节作用机制研究提供可能的线索或依据,因此这几种数据库都各有优缺点。基于计算机预测算法在全基因组范围内鉴定tRF需要更加完美的数据库支持,而数据库中数据量大小及准确性将会影响tRF鉴定的速度及精度。因此,我们迫切需要对tRF数据库作进一步开发和完善,以更好地研究tRF。Table 3展示了目前 tiRNA&tRF主要数据库的重要特征。
3 tRNA衍生片段和tRNA应激诱导RNA对靶分子的调节机制
有了上述tRF鉴定方法和数据库的基础之后,研究人员系统地开展了对tRF的研究,对tRF与RNA、DNA及蛋白质等相关靶分子的调节机制进行了深入挖掘,意在揭示其内在的生物学功能。根据目前的研究显示,tRF具有调节RNA稳定性和基因表达,参与众多生物过程,例如细胞周期与凋亡等功能,但具体的调节机制尚未完全阐明。下面主要介绍tRF对相关靶分子的调节机制。
3.1 与mRNA的相互作用
有研究提到tRF与Ago蛋白可形成RNA诱导沉默复合体(RNA-induced silencing complex,RISC),结合到mRNA上,调节mRNA的稳定性从而影响翻译水平[30]。事实上,tiRNA与tRF似乎可以不依赖Ago蛋白,直接或间接与mRNA相互作用来发挥生物学功能。有研究表明,tRNA-Ala和tRNA-Cys来源的5′-tiRNA可以组装成分子间RNAG四联体(RG4),取代mRNA帽上的翻译起始复合物eIF4G/eIF4A,从而抑制mRNA的翻译(Fig.1A)[31,32]。但是这类特定的5′-tiRNA都有末端寡G序列(TOG),对于无该序列的5-tiRNA是否具有这样的功能仍有待研究。而特异性的5-tRF也会和5′-tiRNA一样,组装成RG4结构,竞争性结合真核起始因子4 G(eIF4G)和4A(eIF4A),直接影响蛋白质的翻译过程[33,34]。
与上述调节方式不同,tRF可以与靶mRNA的编码区和非编码3′UTR区结合,通过改变tRF的二级结构来增强翻译速率(Fig.1B)。根据Kim等人的报道,来源于Leu-CAGtRNA的3-tRF可以与核糖体蛋白PRS28和RPS15对应的mRNA结合[35],通过改变RPS28的二级结构来增强其翻译速率[36]。但是,tRF是否可以和一般的mRNA结合以增强翻译速率仍有待进一步研究。此外,一种名为5′-tiRNA-Glu的tiRNA可以作为向导RNA,诱导tRNaseZL来裂解靶mRNA,例如PPM1F mRNA[37,38],从而抑制相关细胞的凋亡。
在卵巢癌和乳腺癌中,Zhou等人发现,tRF-5-Glu可以直接靶向影响乳腺癌抗雌激素3(BCAR3)的mRNA,干扰其在卵巢癌中的表达水平,从而抑制卵巢癌细胞的增殖[39]。而Mo等人发现,5′-tiRNA-Val过表达可以靶向影响FZD3基因,从而抑制FZD3/Wnt/β-Catenin信号通路,进而抑制乳腺癌细胞的增殖和迁徙[40]。
3.2 与ncRNA的相互作用
研究表明,在特定压力条件下产生的tRF可以和siRNA、前体dsRNA竞争结合Dicer和Ago2蛋白,但不形成新的RISC,从而减弱siRNA对各类mRNA的抑制作用(Fig.1C)[41,42]。此外,Rando等人发现来自tRNA-Gly-GCC的tRF-GG可以通过调控ncRNA——U7snRNA水平来影响MERVL靶基因的转录[43],这也间接说明了tRF可以与U7 snRNA相互作用[44]。

Fig.1 Regulation mechanisms of tRF on mRNA, ncRNA and DNA (A)TRF inhibits mRNA translation by forming RG4 to replace eIF4G/eIF4a on the mRNA cap.(B)TRF binds to the 3′ UTR of the target mRNA to enhance the translation rate by altering the secondary structure.(C)TRF competes with siRNA or precursor dsRNA for Ago and Dicer to weaken the inhibitory effect of siRNA on mRNA.(D)TRF and HIV RNA form double-stranded RNA, which is transcribed into cDNA by reverse transcriptase to form complementary DNA primers, thus promoting the proliferation of the virus
由于tRF一开始被认为是miRNA的一种[45],因此后续研究的开展也主要着重于检验其是否以miRNA的方式发挥作用,而对tRF与其他ncRNA相互作用的研究相对较少。以上例子说明,tRF能直接或间接与少数特定的ncRNA相互作用,但tRF是否能和更多的ncRNA相互作用发挥调控作用,以及具体的作用途径仍有待深入研究。
3.3 与DNA的相互作用
tRF可以和DNA结合发挥调控作用。有文献报道,被HIV-1病毒感染的细胞富集了来自tRNALys的tRF-3006,而HIV RNA可以和tRNALys的3′端杂交形成双链RNA,再通过逆转录酶转录为cDNA,合成互补DNA的引物(Fig.1D)。这说明tRF-3006和tRNALys的56~67 nt能形成引物结合位点(primer binding site,PBS),促进病毒增殖[46]。类似的机制在人T细胞白血病病毒I型(HtLV-1)细胞中也有发现。即宿主细胞中的3-tRF(tRF-3019)可以作为引物与HtLV-1的PBS结合,促进病毒的翻译水平并加快病毒的增殖[47]。
3.4 与特异性蛋白质相互作用
3.4.1 与Ago蛋白家族的结合 由于tRF的大小与miRNA和siRNA相似,因此大量研究人员参考miRNA的分析方法来预测tRF的功能,认为tRF可以与Argonautes(Ago)蛋白家族结合,形成RISC,并作用于mRNA,从而影响翻译水平[48](Fig.2A)。研究表明,1-tRF、3-tRF和5-tRF确实可以与Ago1、Ago3、Ago4蛋白质相互作用发挥基因沉默的功能。例如Haussecker等人发现,来自前体tRNA-Ser-TGA的1-tRF-Ser-TGA可以和Ago3、Ago4蛋白质结合,发挥基因沉默的作用[49];Kumar等人也通过测序技术发现,3-tRF和5-tRF可以与Ago1、Ago3、Ago4相互作用,影响靶mRNA的翻译水平[8]。其具体的作用机制如下:首先,tRF可以像miRNA一样与Ago蛋白结合形成tRF-Ago蛋白复合体。该复合体tRF的5′端上有7~8个碱基大小的种子序列,可以与靶mRNA结合。由于在该复合体上还存在GW182蛋白(翻译抑制和mRNA降解的效应蛋白[50]),可以通过RNA诱导的RISC来降解mRNA或通过序列互补在转录后水平抑制mRNA的表达,从而发挥基因沉默功能。此外,Green等人发现,由tRNA-CysGCA裂解产生的3-tRF(tRF-3003a)可以与Ago2蛋白形成tRF-Ago复合物,抑制骨性关节炎(OA)软骨细胞中JAK3mRNA的表达,这种抑制效果可能与Ago2或GW182蛋白有关,如果复合物中Ago2或GW182蛋白缺失,这种抑制效果也随之消失[51]。以上结果表明,Ago蛋白与GW182蛋白对tRF发挥基因沉默功能都缺一不可。tRF-Ago蛋白复合体除了通过GW182效应蛋白质发挥基因沉默功能外,还可以让大量的mRNA降解酶进入细胞质加工体,造成靶mRNA的降解[5]。这点在结核分枝杆菌(Mtb)的相关研究中得到了验证,即来自线粒体的tRF可以与Ago蛋白结合并降解靶mRNA[52]。
tRF与Ago蛋白结合的破坏将导致一系列生物过程发生紊乱。例如在Burkitt淋巴瘤细胞系中,tRF-3027b的下调将导致RPA1蛋白的上调,从而导致细胞异常增殖[2]。通常情况下,tRF-1001与Ago3、Ago4蛋白质发生相互作用[49],但敲除tRF-1001后,细胞将被阻滞在G2期并抑制DNA的生物合成,从而干扰细胞的正常增殖[53]。从以上结果可知,部分特定的tRF可以与Ago蛋白家族结合,通过类miRNA-RISC途径或召集mRNA降解酶,直接或间接调控mRNA的翻译水平,但其具体的调节机制与作用靶点仍然有待进一步研究。此外,有些tRF被认为和Ago蛋白无关系,例如Kumar等人就认为,tRF-1不与任何Ago蛋白结合[8]。
3.4.2 与RNA结合蛋白的相互作用 RNA结合蛋白(RNA binding proteins, RBPs)是一类同RNA结合并发挥调控作用的蛋白质。RBPs可以结合和调节ncRNA的表达及功能,例如ncRNA的合成和降解[54,55]等,同时RBPs也是许多疾病的关键蛋白质,例如癌症、心血管疾病、遗传疾病、发育障碍和神经退行性疾病等[56]。研究表明,tRF也可以和RBPs结合发挥生物学功能,例如抑制细胞增殖、抑制翻译水平等。因此,tRF-RBP相互作用是影响细胞稳态的关键,是导致细胞功能障碍和疾病的重要原因之一。
YBX-1(Y-box binding protein,YB-1)是一种具有多个相互作用配体的RBP,在多条信号通路中发挥作用,并在多种癌症类型中高度过表达。研究发现,在特定的条件下,2-tRF可以与肿瘤促癌基因的mRNA 3′UTR竞争结合YBX-1蛋白,从而阻断YBX-1蛋白与这些mRNA的相互作用(Fig.2B),导致其不稳定,抑制癌细胞的浸润。类似的机制在乳腺癌细胞的相关研究中也得到了证实[4,10,57,58]。此外,tRF还会诱导应激颗粒(SGs)的形成。SGs在细胞质中发生组装,导致细胞中的mRNA暂时沉默,使细胞暂时进入一种“冬眠状态”,从而提高细胞在不利条件下的生存能力。
最近Falconi等人通过研究发现,来自tRNAGlu的tRF3E以一种不同于上述2-tRF调节机制的方式,特异性地与RBP相互作用[59]。NCL是一种在乳腺癌过表达的RBP,能够抑制p53 mRNA的翻译水平,NCL和tRF3E可以形成NCL-tRF3E复合物,使得p53 mRNA的表达水平提高(Fig.2C),从而抑制癌细胞的生长。可见,tRF在抑制肿瘤生长机制中也发挥重要的作用。
3.4.3 与其他蛋白的相互作用 除了对蛋白家族进行系统的研究外,蛋白家族的分支蛋白质和一些散在的蛋白质也有被研究。细胞色素C是在一种在氧化还原过程中发挥重要作用的电子传递蛋白质。研究发现。一些tRF和tiRNA可以与细胞色素C结合,抑制其与Apaf-1的结合,阻止胱天蛋白酶9(caspase-9)的活化(Fig.2D),从而抑制凋亡小体的生成,提高细胞的生存率[60,61]。
Piwi蛋白是Ago蛋白家族的一个亚分支,可以与3-tRF和5-tRF相互作用来调节基因的表达[62]。研究发现,在单核细胞发展为树突状细胞的过程中,来源于tRNAGlu的5-tRF可以和Piwil1形成复合物,募集SETDB1、SUV39H1和HP1β等蛋白质,使得启动子区域发生组蛋白H3K9甲基化修饰,进而抑制CD1A的表达(Fig.2E)[63]。Pekarsky等人发现ts-3676和ts-4521可以与PIWIL2蛋白结合,对造血系统的恶性肿瘤和实体瘤发挥抑制作用[64]。此外,嗜热四膜虫中的3-tRF可以特异性地结合Twi12蛋白,募集Tan1蛋白和核酸外切酶Xrn2,形成前体rRNA剪切复合体TXT(Twi12/Xrn2/Tan1 complex),并通过剪切、加工前体rRNA来增强rRNA的合成(Fig.2F)[65],调节表观遗传修饰。
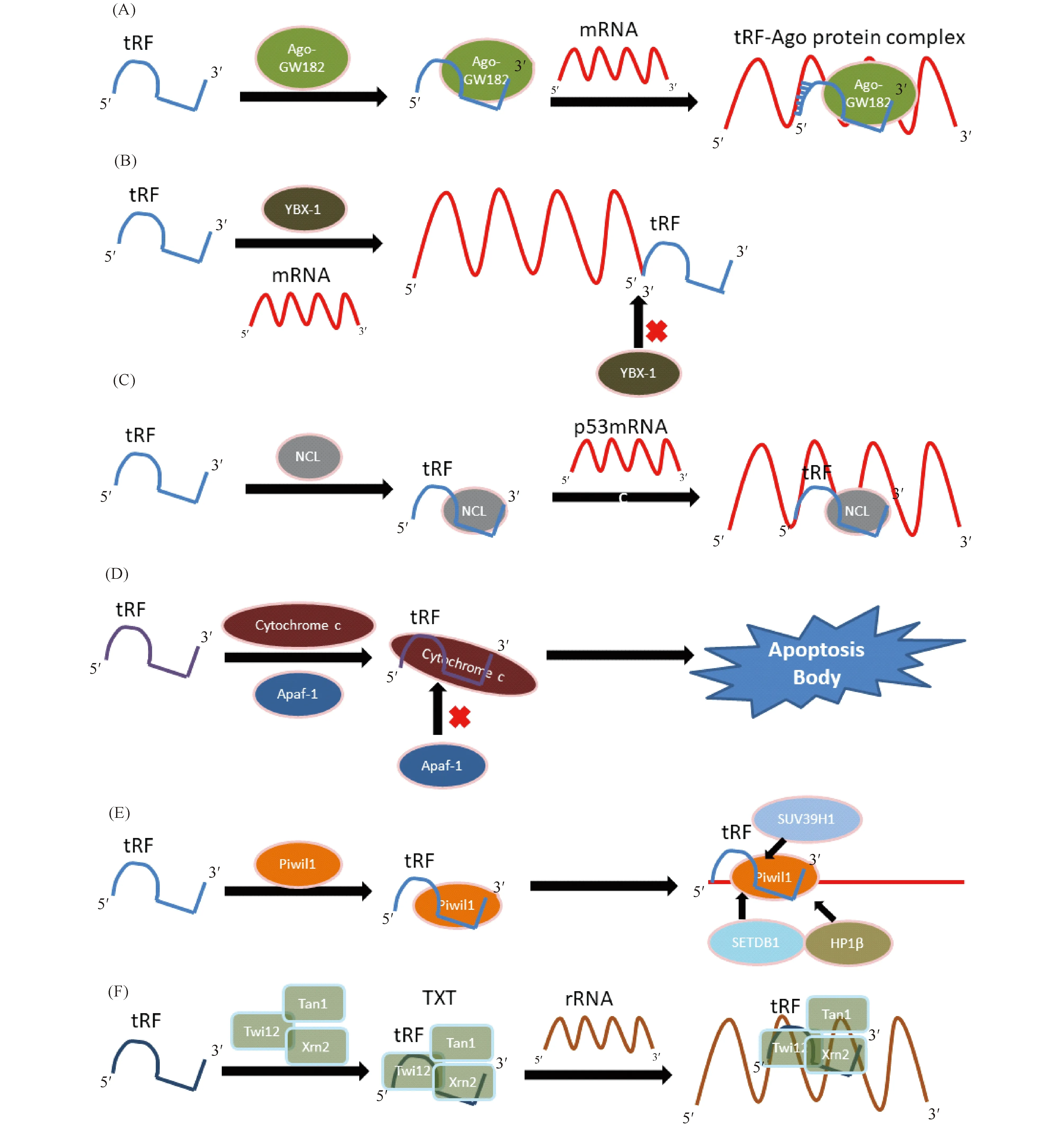
Fig.2 The regulatory mechanism of tRF on proteins (A)TRF forms a complex with the AGO-gw182 protein, which binds to the target mRNA to degrade mRNA or inhibit its expression.(B)TRF and YBX-1 competed for the 3′UTR of target mRNA and blocked the binding of YBX-1 to mRNA, thereby inhibiting cell proliferation.(C)TRF and NCL form a complex to bind to P53 mRNA and inhibit the expression level of this mRNA.(D)TRF binds to cytochrome C, inhibits the binding of cytochrome C to Apaf-1, and inhibits the formation of apoptotic bodies.(E)TRF and PIWIL1 form a complex that recruits proteins such as SETDB1, SUV39H1 and HP1β, resulting in histone H3K9 methylation in the promoter region, thereby inhibiting the expression of CD1A.(F)TRF binds to the TBI12 protein to recruit TAN1 and XRN2 to form TXT, which can enhance rRNA synthesis
tRF还可以与一些特殊酶相互作用而发挥特定的生物学功能。Gebetsberger等人发现在特定的应激条件下,沃氏嗜盐富饶菌可以从tRNA-Val的5′端产生5-tRF。而5-tRF可以抑制肽基转移酶的活性,从而抑制翻译水平[44]。Guzzi等人在胚胎干细胞的研究中发现,假尿苷酸合酶7(pseudouridylate synthase 7,PUS7)可以介导tRF-5的假尿苷化,抑制干细胞中部分RNA的翻译,如果PUS7失活,抑制作用则减弱,从而导致蛋白质合成的增加和一些胚胎特异性缺陷的出现[66]。Elbarbary等研究人员发现,负责tRNA 3′端成熟的内切酶tRNaseZ(L)可以与tRF相互作用[37,67],直接裂解匹配到RNA的序列,例如5′-tiRNA(来自tRNAGlu)可以与PPM1F mRNA在体内和体外相互作用,促进mRNA靶蛋白的降解。
在病毒方面,Wang等人对感染了呼吸道合胞病毒(RSV)的细胞进行深度测序后,发现有大量的5′-tiRNA存在,这种源自tRNAGlu-CTC的tiRNA可以与载脂蛋白E受体2(APOER2)结合并抑制其表达。而APOER2是一种抗RSV蛋白,该蛋白质的表达被抑制后会促进RSV病毒的增殖[68,69]。
以上关于tRF作用机制的总结主要从RNA、DNA和蛋白质三个方面进行阐述,其中具体的调节机制错综复杂,有些调节关系仍然不是很明确。例如,5-tRF可以抑制靶基因的表达,但却不需要与靶mRNA的互补位点结合,其具体机制不清楚[70]等。此外,目前文献报道的tRF调节机制仅是冰山一角,tRF是否存在其它的调节机制仍有待进一步研究和发现。但是基于目前已有的研究,tRF可以对相关靶分子的表达水平进行影响,调节人体内某一疾病的危害程度,例如癌细胞的分裂和浸润程度。整合这些tRF与疾病的关系,将加深我们对tRF的理解。
4 tRNA衍生片段与疾病的关系
在大量tRF对靶分子调节机制的研究中提示,tRF是众多癌症包括乳腺癌、肺癌、前列腺癌、结直肠癌等的理想生物标志物。在乳腺癌中,2-tRF可以与YBX-1相互作用,对乳腺癌细胞的增殖产生抑制作用[4,10,57,58];5′-tiRNA也可以通过FZD3/Wnt/β-Catenin信号通路抑制乳腺癌细胞的增殖和迁徙[40]。在前列腺癌中,Lee等人发现tRF-1001表达水平升高,促进癌细胞从细胞周期的G2期过渡到M期,影响癌细胞的增殖速度[13];在结直肠癌中,tRF/miR-1280可以通过3′UTR的结合位点直接调控JAG2蛋白的表达水平,抑制Notch信号转导通路,从而阻止结直肠癌细胞的扩增[71,72]。
除癌症外,tRF与其他疾病也存在关联。在神经系统疾病中,由ANG生成的tRF可以形成RG4,促进运动神经元的恢复,从而揭示了肌萎缩性侧索硬化症(amyotrophic lateral sclerosis,ALS)的潜在机制[73];在遗传病中,精子的tRF水平与雄性后代抗焦虑行为表现有关[74];在病毒方面,Wang等人对感染了呼吸道合胞病毒(respiratory syncytial virus,RSV)的细胞进行深度测序后发现,有大量源自tRNAGlu-CTC的5′-tiRNA产生,且可以与载脂蛋白E受体2(apolipoprotein E receptor 2,ApoER2)结合并抑制其表达。而APOER2是一种抗RSV蛋白质,该蛋白质表达水平的抑制会促进RSV病毒的增殖[68,69];此外,在病毒感染时,3-tRF tRF-3006和tRF-3019可以作为PBS,促进HIV-1病毒和HtLV-1病毒的增殖[46,47]。
上述例子都说明,tRF在多种疾病中具有作为生物标志物和治疗靶点的潜力,但实现上述想法仍有待于进一步的实验和论证,而实验的开展又需要更加完善的鉴定方法和工具,因此这些方面都有待提升。Table 4列出了部分目前已发现的在疾病中发生的tRF调节关系。

Table 4 tRF and related diseases
5 问题与展望
tRF是近年来研究较热的调节性非编码RNA。本文依次从tRF鉴定方法,相关数据库及对靶分子的调节机制这几个方面展开说明。首先介绍了鉴定tRF的两大基本策略,他们相互补充,为tRF鉴定方法的改进打下了基础和提供了思路。而日后发展的几个tRF鉴定方法主要是进一步完善了tRF的序列比对方法,例如tRF2Cancer,SeRPeNT和SPORTS1.0。其次介绍了有关tRF的5种数据库,包括tRFdb,tRF2Cancer,MINTbase,OncotRF和tRFtarget,他们各有特点及作用,为后续计算机大规模识别鉴定tRF提供了数据资源。这两者相辅相成,为tRF对相关靶分子调节机制的研究提供了更为有效的工具。然后从tRF与RNA,DNA及蛋白质的调节机制角度进行总结,列举了几种目前研究较为热门的靶分子对象。从这些研究中也提示了tRF与疾病的关系,通过检测tRF表达水平的高低,可以探索其作为疾病生物标志物和治疗靶点的潜在价值。目前在癌症等多种疾病中已发现一些差异表达的tRF,但其在临床上的应用和转化仍有待进一步探讨。
目前有关tRF和tiRNA的研究层出不穷,但我们只看到了冰山一角,仍有更多的未知等待探索。其次,更多关于tRF和tiRNA的产生机制有待进一步发掘,其命名也有待进一步统一。同时,tRF的鉴定方法和工具也稂莠不齐,但都主要以序列比对为主要依据,对其预测的可信度仍无进一步把控。未来可从空间结构,表达情况,功能分析,调节关系等更多角度综合考虑,以更加准确地鉴定tRF,减少假阳性。此外,目前已有的数据库缺乏相互整合,也迫切需要进一步完善和综合,有必要建立一个集tRFdb,tRF2Cancer,MINTbase等各种数据库内容在内的综合性tRF数据库,以方便研究人员查询和分析、实时上传数据,分享潜在的tRF类型等。至于有关tRF对靶分子调节机制的研究,目前主要关注蛋白质方面,对于tRF与RNA和DNA调节机制的报道仍然较少,并且这些都来自于少数特定的tRF或tiRNA,缺乏更多组织样本的数据结果,未来可进一步结合生物信息学技术与生物实验,预测和验证tRF更多可能的靶基因和功能,使我们进一步了解tRF。