四个竹秆变异毛竹变型的全基因组序列分析
2022-09-07牟少华李雪平
牟少华, 李 娟, 李雪平, 高 健
( 国际竹藤中心 竹藤科学与技术重点实验室, 北京 100102 )
毛竹 () 是我国独有的传统经济竹种(江泽慧,2002),分布范围较广,现有毛竹变型20多种(马乃训等,2014)。毛竹变型在形态上表现出丰富的多态性,尤其是秆色性状方面差异表现显著,例如花毛竹秆为黄色,有宽窄不等的绿色纵条纹;而绿皮花毛竹秆为绿色,但节间有淡黄色细纵条纹。秆色的变异大大丰富了园林观赏种类,提高了园林观赏价值。毛竹竹秆性状的遗传变异是遗传育种工作关注的重点。
目前,高通量测序技术可以分析一个物种的基因组的全貌,已经在谷子 (Bai et al., 2013; 贾小平等,2019)、水稻 (Takagi et al., 2013)、大豆 (Qi et al., 2014; Zhou et al., 2015; 张彦威等,2016)、黄秋葵(张少平等,2017)、厚朴(尹彦棚等,2020)、菜豆 (Jeremy et al., 2010)和番茄 (Lin et al., 2014)等植物上得到了广泛应用。
近年来,随着分子生物学和组学技术的发展,毛竹全基因组序列获得公布 (Peng et al., 2013),一些与毛竹性状相关的基因家族,如2(Wu et al., 2015)、( Bai et al., 2016)、(Sun et al., 2016)、-like(Pan et al., 2016)、-(Chen et al., 2017)、(Xie et al., 2019)、-(Liu et al., 2016)等已进行鉴定和功能验证,但是在基因组层面上的研究较少,特别是毛竹变型秆色相关的研究薄弱,仅对黄槽毛竹和黄皮花毛竹两个毛竹变型基因组序列变异进行初步探索 (牟少华等,2020),这在一定程度上限制了毛竹遗传育种的应用发展。从基因水平上揭示毛竹的变异程度,是分析毛竹变型形态差异产生原因的重要手段之一。因此,开展毛竹变型基因组研究,揭示毛竹变型全基因组突变类型,探究黄酮类和硝酸还原酶等代谢途径相关基因,对解析毛竹丰富的遗传多样性以及性状相关的遗传变异具有重要意义。
本研究以黄皮毛竹等4个有代表性的竹秆变异毛竹变型为研究对象,毛竹全基因组作为参考基因组,采用高通量测序技术,构建全基因组数据库,并利用生物信息学的方法对获得的核酸序列组装,检测并注释其单核苷酸多态性(single nuclotide polymorphisms,SNP)、结构变异(structural variation,SV)和小片段插入缺失(insertion and deletion,InDel)等,注释变异基因功能,积累基因组序列数据,以便为从全基因组水平上深入地分析毛竹的遗传变异,为遗传育种提供遗传基础。
1 材料与方法
1.1 材料
供试材料选自国际竹藤中心安徽太平试验中心种质资源圃。选取4个毛竹变型(表1)适量新鲜幼嫩的叶片,经液氮罐中速冻后,放入-80 ℃冰箱冻存备用。
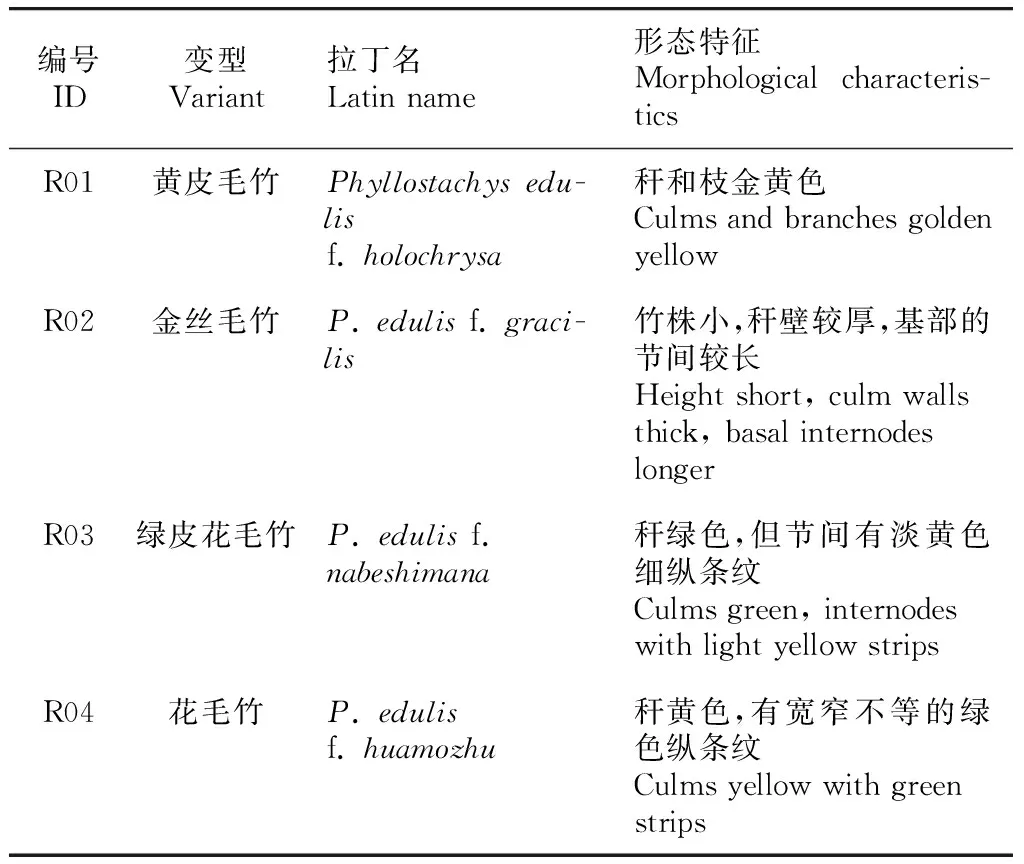
表 1 四个毛竹变型样品简表Table 1 Brief introduction of four variant samples of Moso bamboo
1.2 方法
1.2.1 基因组测序 毛竹变型叶片DNA提取(Zidani et al., 2005)后,经过打断、损伤修复及连接接头、PCR富集、文库质量检测,建成测序文库,在Illunima Hiseq 2500测序平台上运行获得原始数据,将数据过滤后得到高质量数据。
1.2.2 比对统计 使用BWA软件 (Li & Durbin, 2009) 将测序数据比对定位到已测序的毛竹基因组的位置,统计测序深度和基因组覆盖度等信息。
1.2.3 检测SNP、InDel和SV 使用Picard软件 (Gordon et al., 2012) 去重复和GATK软件 (Mckenna et al., 2010) 预处理后,检测SNP和InDel变异。使用BreakDancer软件 (Chen et al., 2009) 检测SV变异,具体方法参照牟少华等(2020)。
1.2.4 注释SNP、InDel和SV 运用SnpEff软件 (Cingolani et al., 2012)注释SNP、InDel和SV,具体方法参照牟少华等(2020)。
1.2.5 注释功能基因 运用BLAST软件,对筛选得到的功能可能变异基因的基因序列与GO (Ashburner et al., 2000)、COG (Tatusov et al., 2000)和KEGG (Minoru et al., 2004)三大功能数据库,进行BLAST比对,得到基因注释。
2 结果与分析
2.1 与毛竹基因组比对
4个竹种通过高通量测序得到测序数据。金丝毛竹样品(R02)过滤后的Clean Reads最少,为82 276 884 bp;花毛竹样品 (R04) 的Clean Reads最多,为112 054 728 bp。定位到毛竹参考基因组的占所有Clean Reads数的百分比在99.45%以上,双端均定位到毛竹参考基因组上并且距离符合测序片段的长度分布的占所有Clean Reads数的百分比在88%左右,说明参考基因组选择合适,且相关实验过程不存在污染,测序Reads的比对率会高于70%。另外,比对率的4个毛竹变型与毛竹参考基因组亲缘关系较近、基因组组装质量高,而且Reads测序质量高。4个样品平均覆盖深度均在10×左右(表2)。
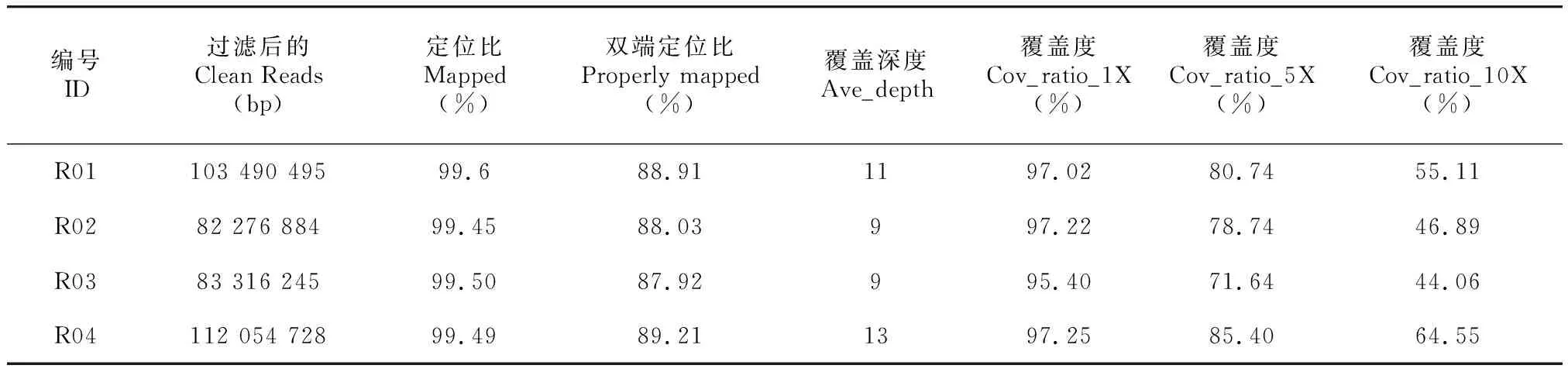
表 2 四个样品数据产出统计表Table 2 Output statistics among four samples
2.2 SNP的检测与注释
2.2.1 SNP检测 4个毛竹样品检测后获得SNP位点统计表(表3)。其中,花毛竹样品(R04)的SNP数量最多,为1 691 715;绿皮花毛竹样品(R03)的SNP数量最少,为1 534 648。4个样品中,转换类型(transition, Ti) SNP数量与颠换类型(transversion, Tv) SNP数量的比值Ti/Tv在3.05~3.10之间,说明这些毛竹变型转换比颠换更容易发生。杂合类型 (heterozygosity, Het) SNP数量为纯合类型 (homozygosity, Homo) SNP数量的10倍左右,杂合比率为88.53%~92.01%。其中,花毛竹样品(R04)杂合比率最高,为92.01%,说明其杂合程度最高。绿皮花毛竹样品(R03)杂合比率最低,为88.53%。
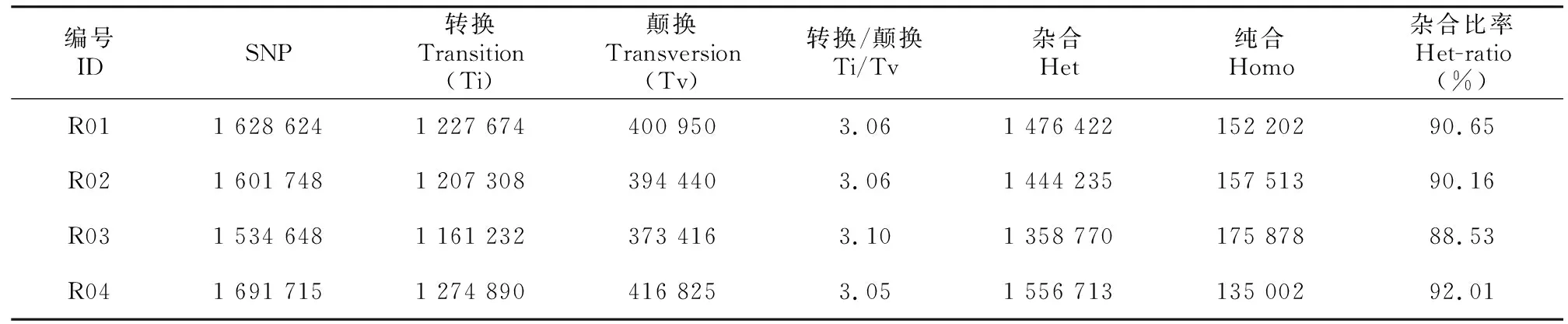
表 3 四个样品SNP位点统计表Table 3 SNP loci statistics in four samples
根据4个毛竹样品与参考基因组的比对结果,汇总样品间SNP的统计结果见表4,表中各数值为对应的横纵两样品之间的SNP数。从表中可以看出,金丝毛竹(R02)与绿皮花毛竹样品(R03)间的SNP数最多。
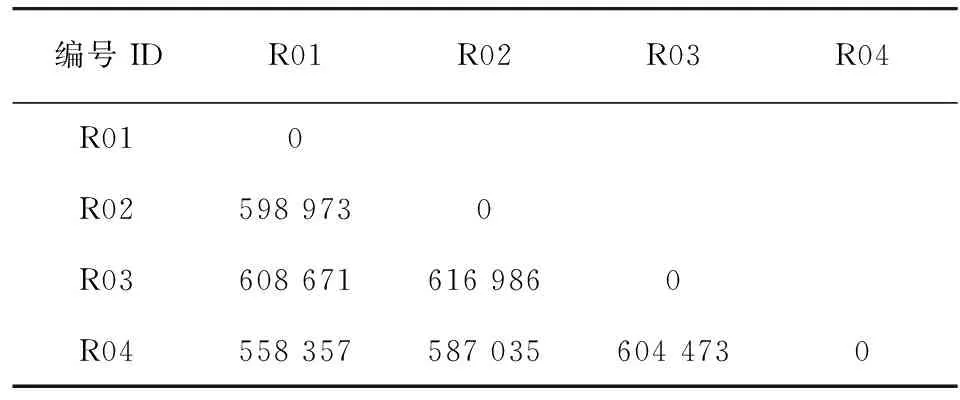
表 4 四个样品间的SNP统计表Table 4 Summary of SNPs detected between four samples
2.2.2 SNP注释 对4个样品SNP进行注释,获得其变异位点发生的区域或类型(图1)。4个毛竹变型发生在编码区(coding sequence,CDS)区域内的SNP数量占比均为2%左右,其中同义突变占48%左右,非同义突变占51%左右。非同义突变率与同义突变率的比值大于1,预示着有正向选择效应。
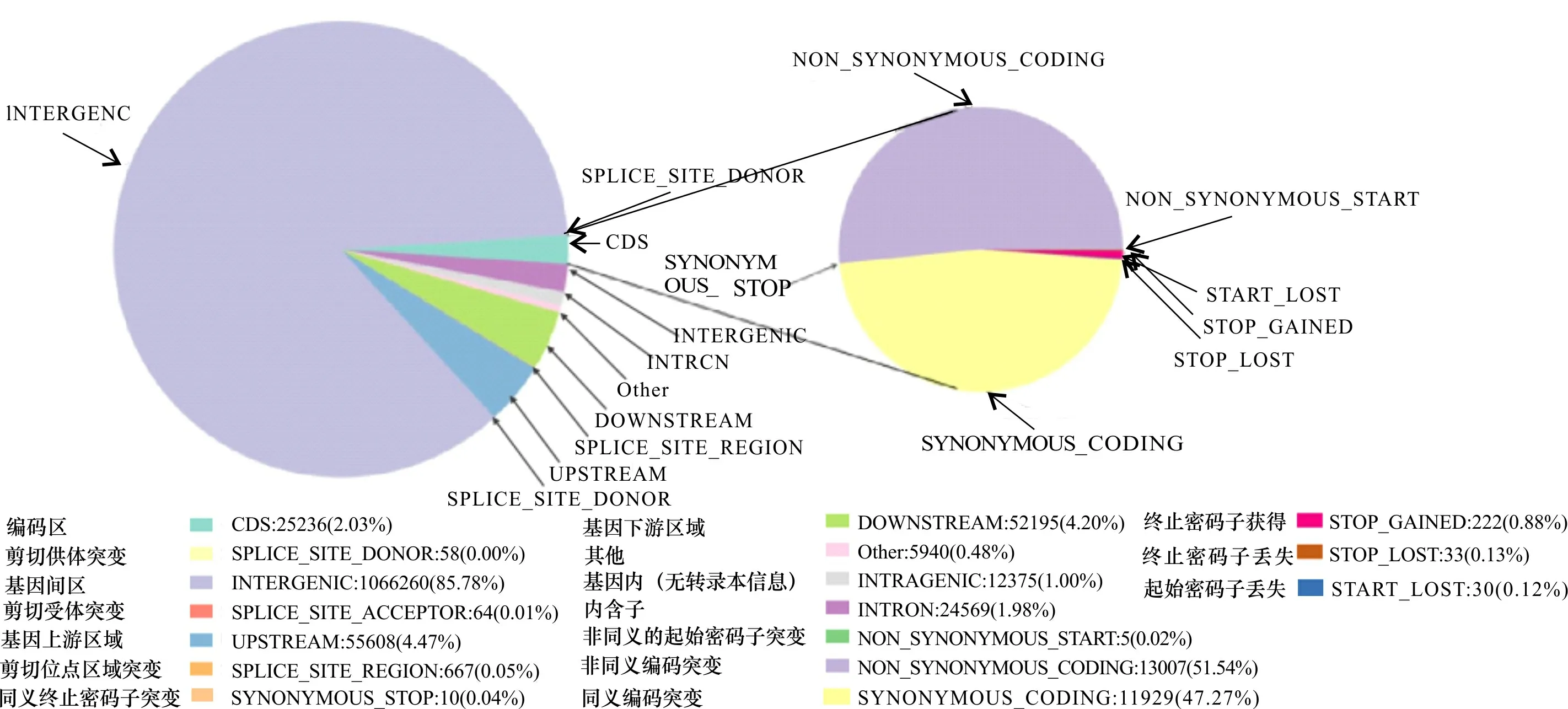
图 1 黄皮毛竹(R01)的SNP注释图Fig. 1 SNP annotations pie of Phyllostachys edulis f. holochrysa (R01)
2.3 InDel检测与注释
2.3.1 InDel检测 对4个毛竹变型InDel进行统计(表5),可以发现4个样品全基因组范围检测出的InDel总数范围为271 648~292 253,其中插入类型的突变总数略低于缺失突变总数;编码区检测出的InDel总数为4 711~4 877,其中插入突变总数为缺失突变的67%左右。各样品中,全基因组范围内纯合突变数约为杂合突变数的2倍,编码区纯合突变数略低于杂合突变数。

表 5 四个样品InDel统计表Table 5 Summary of InDels detected in four samples
对4个样品各区域的InDel长度进行统计发现,编码区存在较多的+1、-1、+ 3、-3 类型突变,而基因组范围存在较多的+1、-1、+ 2、-2 类型突变。其中,数值代表InDel的长度(10 bp以内);大于0为插入;小于0为缺失。
将4个样品的InDel进行两两比较,统计结果见表6。表中各数值为对应的横纵两样品之间的InDel数。
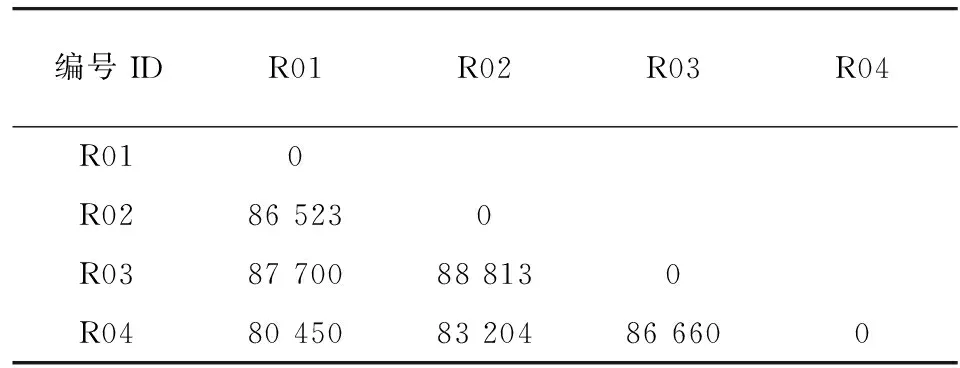
表 6 毛竹变型间的InDel统计表Table 6 Summary of InDels detected between variants of Moso bamboo
2.3.2 InDel注释 对比毛竹参考基因组的基因、CDS位置等信息,注释各样品InDel位点的发生位置以及是否为移码突变等,具体注释结果如图2所示。4个毛竹变型发生在编码区的InDel数量均在1.7%左右。移码突变的InDel有可能会引起基因功能的改变。
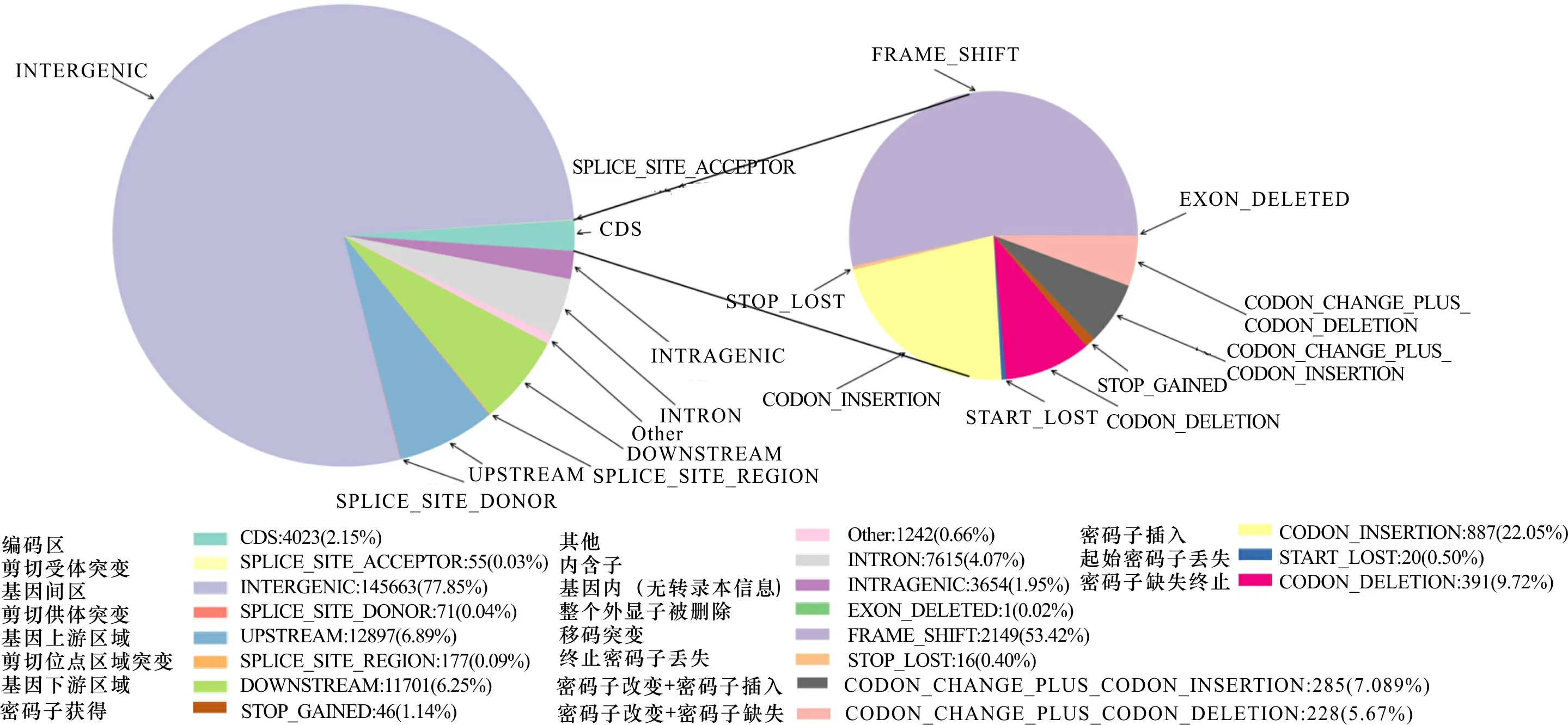
图 2 黄皮毛竹(R01) InDel注释图Fig. 2 InDel annotations pie of Phyllostachys edulis f. holochrysa (R01)
2.4 SV检测与注释
2.4.1 SV检测 检测4个样品与参考基因组间的插入(insertion,INS)、缺失(delection,DEL)、反转(inversion,INV)、染色体内部易位(intra-chromosome translocation,ITX)、染色体间易位(inter-chromosomal translocation,CTX),得到的各类型SV数量统计见表7。其中,4个毛竹变型都表现为缺失类型的SV数量最多,其次为染色体内易位类型。
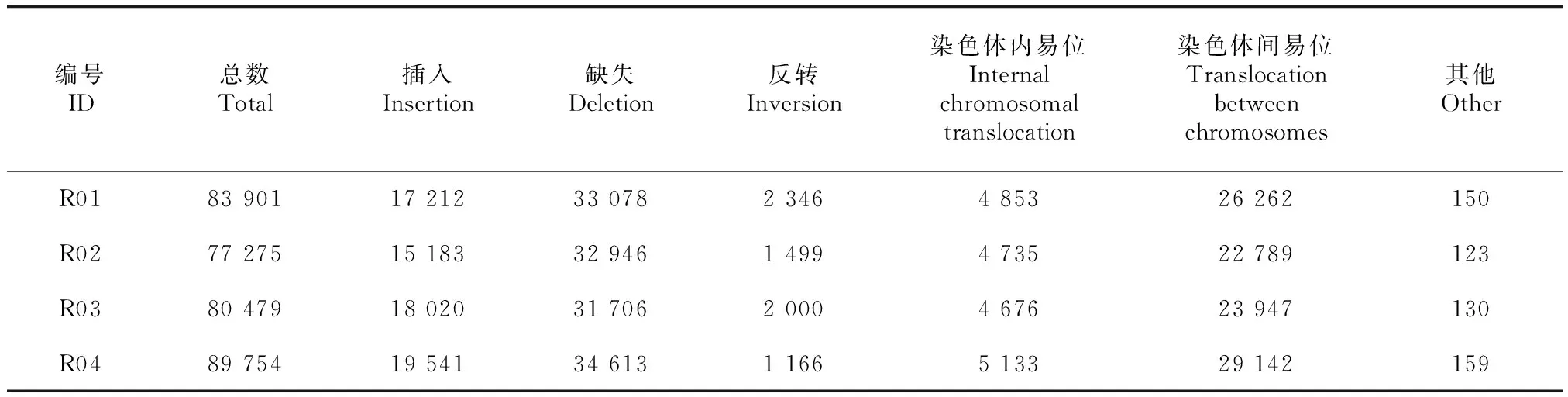
表 7 四个样品SV数量统计Table 7 Quantity statistics of SVs detected in four samples
2.4.2 SV注释 检测4个样品SV发生位置信息,并对缺失、插入和反转3种类型的结构变异进行注释。结果表明,4个毛竹变型在各区域分布的SV总体情况一致,注释到的变异基因数目以基因间区的缺失类型最多,其次为基因间区的插入类型(表8)。
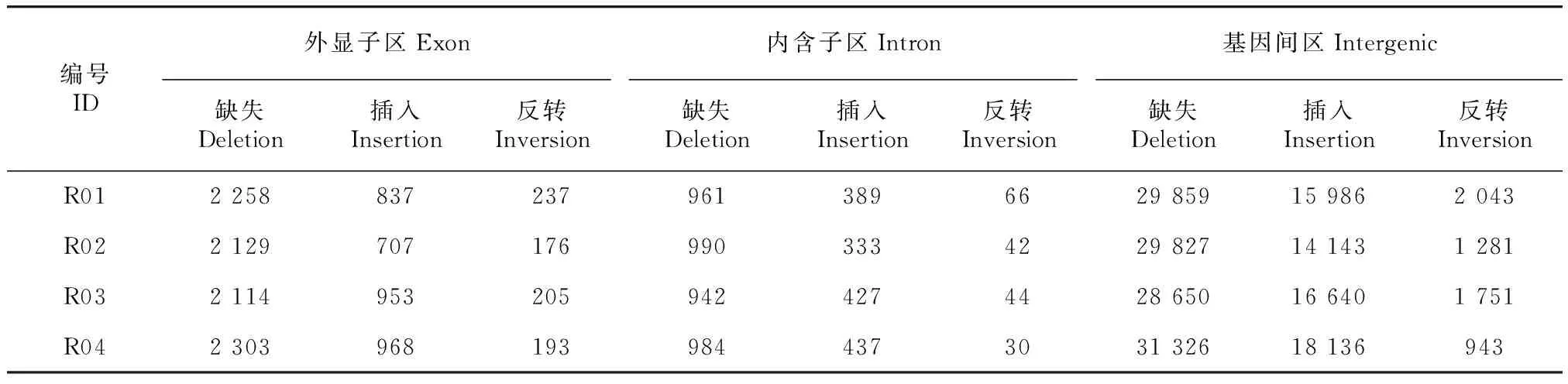
表 8 四个毛竹变型SV注释结果统计表Table 8 SV annotations in four variants of Moso bamboo
2.5 变异基因功能注释与分析
2.5.1变异基因挖掘 分别统计4个样品的非同义突变的SNP以及CDS区发生InDel和SV的基因(表9),寻找可能存在功能变异的基因。在4个毛竹样品中,花毛竹(R04)基因组存在12 555个基因变异,其中,非同义突变SNP基因为5 563个,InDel基因为4 006个,SV突变的基因为2 986个,差异基因总数和SV突变基因数最多。在3类变异基因中,非同义突变SNP基因数最多,InDel基因数量次之,SV突变的基因最少。
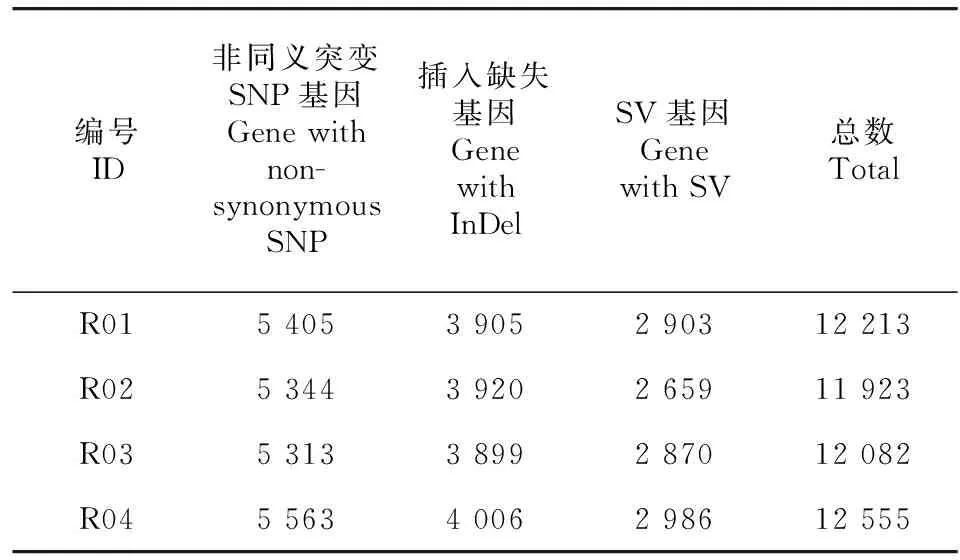
表 9 四个毛竹变型的变异基因统计表Table 9 Summary of mutant genes in four variants of Moso bamboo
2.5.2 变异基因的功能注释 黄皮毛竹、金丝毛竹、绿皮花毛竹和花毛竹注释到数据库中的变异基因数分别为7 575、7 538、7 476和7 728。变异基因GO分类统计结果图(图3)中,显示出在3大基因功能分类体系(分子功能、细胞组件和生物过程)的56个分类内容中所对应的基因数和基因占比。其中,在细胞组件分类中,与叶绿素合成相关的基因有2 431个;在生物过程分类中,参与类胡萝卜素合成过程的基因有75个;在分子功能分类中,与花青素合成调控以及紫外光下组织中花青素积累的相关基因有80个。4个毛竹变型在相应的基因功能中的变异基因数目有差异,例如:花毛竹有21个与类胡萝卜素合成相关的基因;绿皮花毛竹有17个相关基因;而黄皮毛竹有18个相关基因,基因数量和种类的差异,可能引起相应的功能变化。深入研究叶绿素、类胡萝卜素和花青素合成相关基因以及这些差异基因的调控途径,有利于从DNA水平上解释秆色的变异。
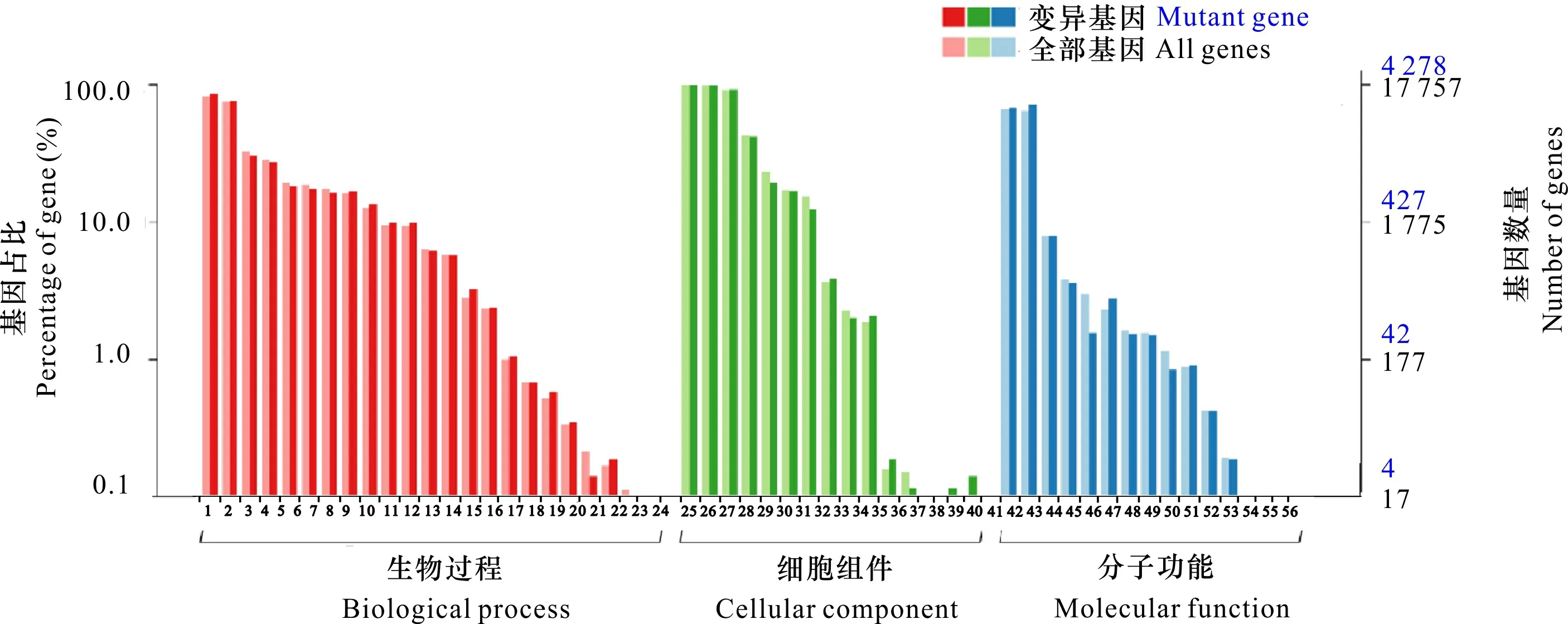
横坐标为GO的分类内容,纵坐标的左侧为基因数占比,右侧为基因数量。 1. 代谢过程; 2. 细胞过程; 3. 对刺激的反应; 4. 生物调节; 5. 定位; 6. 定位确立; 7. 细胞组成或生物形成; 8. 分化过程; 9. 多细胞生物过程; 10. 繁殖; 11. 生殖过程; 12. 信号; 13. 多组织过程; 14. 生长; 15. 免疫系统过程; 16. 死亡; 17. 细胞增殖; 18. 生物粘附; 19. 节律过程; 20. 病毒繁殖; 21. 色素沉着; 22. 运动; 23. 细胞死亡; 24. 碳利用; 25. 细胞部分; 26. 细胞; 27. 细胞器; 28. 膜; 29. 细胞器部分; 30. 膜部分; 31. 高分子复合物; 32. 细胞外区域; 33. 膜内腔; 34. 细胞连接; 35. 细胞外基质; 36. 类核; 37. 病毒粒子; 38. 细胞外基质部分; 39. 细胞外区部分; 40. 病毒粒子部分; 41. 绑定; 42. 催化活性; 43. 运输活动; 44. 核酸结合转录因子活性; 45. 结构分子活性; 46. 电子载体活性; 47. 酶调节活性; 48. 活动分子传感器; 49. 抗氧化活性; 50. 受体活性; 51. 蛋白结合转录因子活性; 52. 营养库活性; 53. 翻译调节活性; 54. 金属伴侣活性; 55. 蛋白质标记; 56. 通道调节活性。Abscissa is GO classification, the left side of the ordinate is the percentage of genes, and the right side is the number of genes. 1. Metabolic process; 2. Celluar process; 3. Response to stimulus; 4. Biological regulation; 5. Localization; 6. Establishment of localization; 7. Cellular component organization or biogenesis; 8. Developmental process; 9. Multicellular organismal process; 10. Reproduction; 11. Reproductive process; 12. Signaling; 13. Multi-organism process; 14. Growth; 15. Immune system process; 16. Death; 17. Cell proliferation; 18. Biological adhesion; 19. Rhythmic process; 20. Viral reproduction; 21. Pigmentation; 22. Locomotion; 23. Cell killing; 24. Carbon utilization; 25. Cell part; 26. Cell; 27. Organelle; 28. Membrane; 29. Organelle part; 30. Membrane part; 31. Macromolecular complex; 32. Extracellular region; 33. Membrane-enclosed lumen; 34. Cell junction; 35. Extracellular matrix; 36. Nucleoid; 37. Virion; 38. Extracellular matrix part; 39. Extracellular region part; 40. Virion part; 41. Binding; 42. Catalytic activity; 43. Transporter activity; 44. Nucleic acid binding transcription factor activity; 45. Structural molecule activity; 46. Electron carrier activity; 47. Enzyme regulator activity; 48. Molecular transducer activity; 49. Antioxidant activity; 50. Receptor activity; 51. Protein binding transcription factor activity; 52. Nutrient reservoir activity; 53. Translation regulator activity; 54. Metallochaperone activity; 55. protein tag; 56. Channel regulator activity.图 3 黄皮毛竹(R01)变异基因的GO注释分类图Fig. 3 Classification of Phyllostachys edulis f. holochrysa (R01) mutant genes compared with GO database by BLAST
变异基因COG注释分类图(图4)直观显示出COG功能分类条目上分别对应的频率,其中涉及到功能注释、转录、复制重组修复和信号转导机制的对应数值高。获得的功能注释基因为1 630个,参与复制、重组和修复的基因数为369个,信号转导机制的基因数为291个,转录的相关基因为222个。

图 4 黄皮毛竹(R01)变异基因的COG注释分类图Fig. 4 Classification of Phyllostachys edulis f. holochrysa (R01) mutant genes compared with COG database
KEGG数据库系统地分析4个毛竹变型的基因产物在生物学过程中的功能。以黄皮毛竹(R01)的缬氨酸、亮氨酸和异亮氨酸生物合成通路为例(图5),注释到57个基因参与该通路,其中23个变异基因。整个通路涉及不同的酶连接一系列生化反应形成,其中,框内的数字代表enzyme的号码,红色的框代表通路相关变异基因。
3 讨论
全基因组重测序可以在已知植物的基因组序列基础上,对其不同品种的基因组序列进行测序,从而找出个体与该物种间的差异性(Ley et al., 2008)。随着毛竹全基因组序列的公开发表(Peng et al., 2013),研究毛竹不同变种或变型基因组序列差异成为可能。全基因组重测序可以检测个体的全部基因组序列,扫描出一些与该个体生长性状密切相关的变异位点(宋志芳等,2017)。毛竹的地下茎中单轴散生,其不同变异类型也都是散生竹。在毛竹自身的遗传基因漂移,以及长期的栽培措施和自然环境变迁等因素的影响下,毛竹种内产生了很多遗传变异,产生各种独特的结构形态,表现出丰富的园林观赏性状。其中,黄皮毛竹、花毛竹、绿皮花毛竹在竹秆颜色方面表现出不同程度的变异,使其具有更高的园林观赏价值。
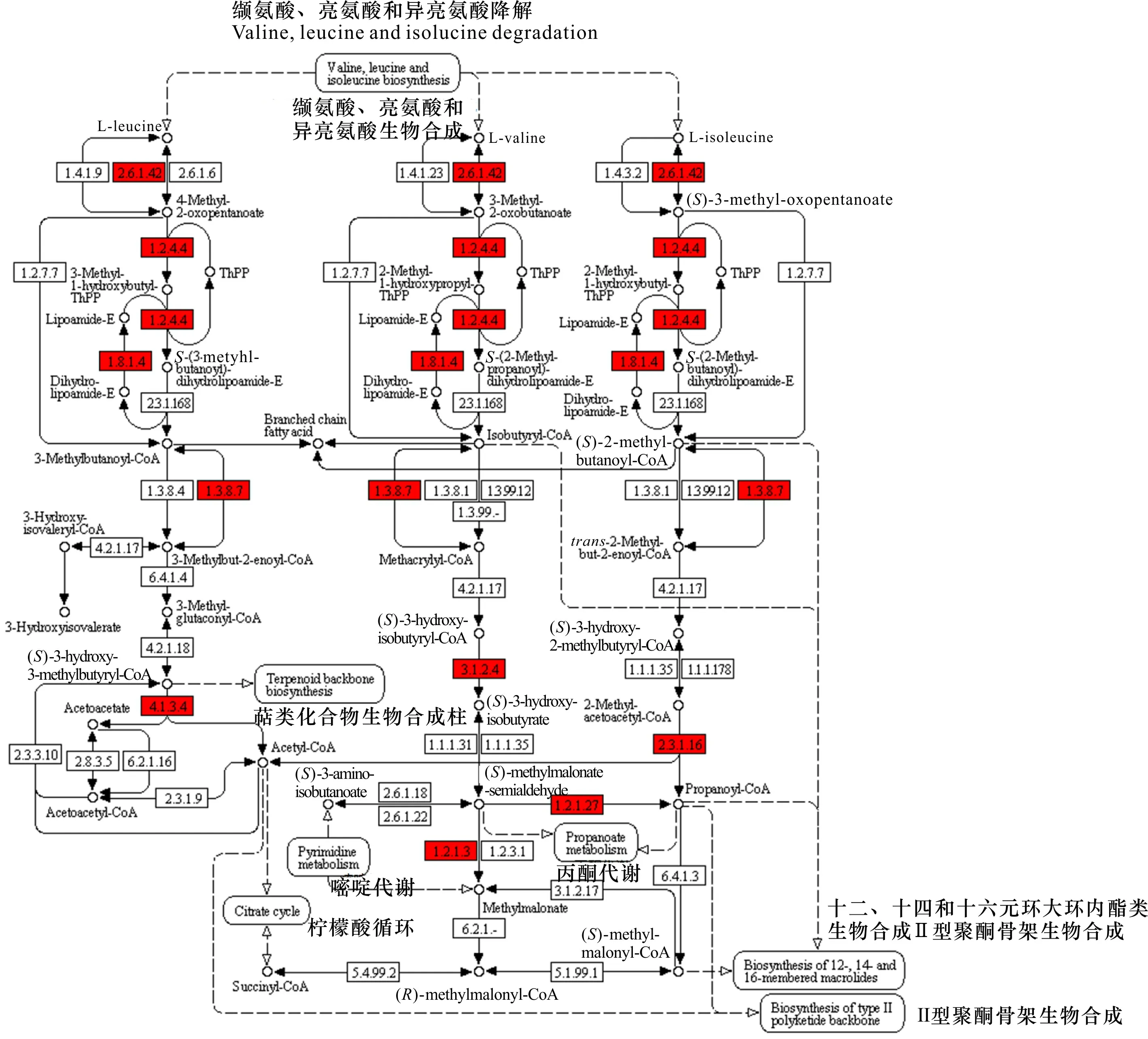
图 5 黄皮毛竹(R01)变异基因的KEGG通路代谢图Fig. 5 Pathway of Phyllostachys edulis f. holochrysa (R01) mutant genes compared with KEGG database by BLAST
对4个毛竹变异类型全基因组重测序,初步统计分析了其基因组数据,与毛竹参考基因组进行比对,检测其SNP、InDel和SV。SNP类型的变异分为转换和颠换两种,4个毛竹样品的转换/颠换(Ti/Tv)的比值均约等于3,说明转换类型比颠换类型更容易发生。SNP杂合比例约为90%,说明样品有很高的杂合度,即同源染色体上SNP位点含不同类型的碱基比例高。InDel位点数同样能反映不同样品与毛竹基因组之间的差异,并且编码区的InDel会引起移码突变,影响基因功能。SV中缺失、插入、反转、易位4种类型的数量,反映出基因组水平上大片段的缺失、插入、倒置、易位等序列差异。通过生物信息学分析,比较不同秆色的变异类型在全基因组水平上的结构差异,并进行差异注释,从而为毛竹选育提供遗传基础,也为重要基因的功能研究提供有利依据。
颜色变异是植物中较常见的表型变异,其中关于水稻、拟南芥、菊花等多种植物均有叶色变异的报道。据不完全统计,水稻叶绿体含量基因超过140个(赵绍路等,2018)。竹子中的色素分为3大类:叶绿素、花青素和类胡萝卜素。通过功能数据库比对,对4个毛竹变型的变异基因,进行基因功能注释和分析。GO数据库注释聚类反映了毛竹变型在不同功能组分类中基因数目和基因产物的属性,其中与秆色变异有关的叶绿素、类胡萝卜素和花青素等色素合成相关基因作为重点关注对象进行分析。COG数据库注释了基因产物的直系同源分类,不同分类对应的基因数目差别很大,反映了不同条件下的生理或者代谢偏好等。 KEGG数据库将基因和多种酶形成通路,有氨基酸生物合成、类胡萝卜素生物合成、类黄酮生物合成、 萜类化合物的生物合成、植物激素信号转导、参与卟啉和叶绿素代谢等显著富集。其中,叶绿体、类胡萝卜素和花青素等色素合成相关通路,是与秆颜色相关的主要代谢通路。
结合不同秆色毛竹变型的生物学和生理学特性,研究全基因组序列色素合成相关基因,有助于从基因水平上解析其秆色变异原因。有研究表明,毛竹不同变异类型在叶绿素含量、β胡萝卜素含量等生理指标中存在显著性差异(陈建华等,2011),株型较大的花毛竹生理指标值比较小型的龟甲竹、绿槽毛竹大(晏育存,2011)。毛竹变型ISSR和AFLP分子标记分析表明,变型间的遗传变异程度较小(阮晓赛,2008)。在参考黄槽毛竹和黄皮花毛竹两个秆色变异毛竹变型的研究结果(牟少华等,2020)基础上,通过对黄皮毛竹等4个毛竹变异类型重测序, 进行DNA水平的变异基因功能注释,可以分析基因产物在细胞中的代谢途径及功能,尤其是对黄酮类、类胡萝卜素、硝酸还原酶等合成通路的深入分析,为揭示相关代谢通路有关基因提供重要理论依据,对于探究毛竹变型秆色变异有重要意义。另外,颜色变异通常是一个不稳定的性状。例如,花毛竹在不同的生境条件下有可能变回全部绿色或者变成绿皮花毛竹,这表明竹类植物的颜色变异在遗传上不是一个稳定的性状。因此,从分子机制上探索颜色变异有其复杂性,其代谢调控还需要进一步研究。
4 结论
采用第二代高通量重测序技术,对4个毛竹变型材料进行全基因组重测序研究,对其单核苷多态性、小片段插入缺失和结构变异进行分析和注释,筛选可能发生功能变异的基因。将变异基因与GO、COG、KEGG等功能数据库进行比对,每样品都有7 000多个变异基因得到功能注释。GO注释分类包括细胞组件、分子功能和生物过程3个基因功能分类体系的56个功能组,在细胞组件分类中,叶绿素合成相关基因有2 431个;在生物过程分类中,参与类胡萝卜素合成过程的基因有75个;在分子功能分类中,参与花青素合成调控以及紫外光下组织中花青素积累的相关基因有80个。COG分类表明参与复制、重组和修复的基因数为369个,信号转导机制的基因数为291个,转录的相关基因为222个。通过KEGG数据库系统地分析变异基因参与的黄酮类、类胡萝卜素等物质代谢合成途径。后续数据的深入分析将解析不同变异类型的基因家族和基因功能,初步阐析不同竹秆变异毛竹变型的分子遗传基础。
