基于序列比对分析木聚糖1,4-β-木糖苷酶结构差异和分子进化规律
2022-09-02林燕玲蔡雨晨李利君倪辉
林燕玲,蔡雨晨,李利君,2*,倪辉,2
(1.集美大学海洋食品与生物工程学院,福建厦门 361021)
(2.福建省食品微生物与酶工程重点实验室,厦门市食品生物工程技术研究中心,福建厦门 361021)
木聚糖1,4-β-木糖苷酶(xylan 1,4-β-xylosidae,EC 3.2.1.37)属于木聚糖酶系[1],是一种糖苷水解酶[2],具有外切性质[3]。大多数的木聚糖1,4-β-木糖苷酶存在于真菌中,也有部分存在于细菌中[4],极少存在于酵母菌中[5]。因其可以作用于低聚木糖的非还原端释放木糖[6],不仅在造纸工业、医药领域应用广泛[7],在食品技术中有很大的应用潜能,如利用木聚糖1,4-β-木糖苷酶水解低聚木糖的性质制备天然食品甜味剂木寡糖;在啤酒酿造中代替乳化剂、氧化剂,降低啤酒的黏度与浊度;水解小麦、玉米等谷物中的半纤维素,促进营养的消化吸收等[4]。
目前木聚糖1,4-β-木糖苷酶的研究集中在筛选、纯化、表征和克隆表达上,缺乏对结构的系统研究。对木聚糖1,4-β-木糖苷酶的结构进行系统的分类研究,有助于今后研究其结构与功能之间的关系,进一步拓展其在食品等领域的应用。多序列比对(Multiple sequence alignment)[8]是将三个及三个以上的生物学序列进行比对,比对结果作为探究序列同源性的依据,揭示序列保守性,与此同时多重序列比对在构建系统进化树、预测蛋白质二级结构以及三级结构、确定未知序列家族等方面也有广泛应用。系统进化树[9]是研究生物进化过程的基本框架,三维结构叠合可以反映蛋白质立体结构差异。分子对接可以预测两个或两个以上已知结构结合方式的计算机算法,广泛应用与分子识别研究[10]。本研究利用生物信息学方法,分析木聚糖1,4-β-木糖苷酶的基因序列,对基因序列进行多重序列比对,构建系统进化树,同时预测其理化性质,并对该酶的三维结构进行建模、结构叠合与分子对接,进而分析木聚糖1,4-β-木糖苷酶分子进化规律以及结构差异,为木聚糖1,4-β-木糖苷酶功能的进一步研究提供生物信息学参考。
1 材料与方法
1.1 数据获取
国际生物技术信息中心NCBI(http://www.ncbi. nlm. nih.gov/)。
1.2 实验方法
登录NCBI网站,在搜索栏中输入关键词“xylan 1,4-β-xylosidase”,收集所有木聚糖1,4-β-木糖苷酶的基因序列、氨基酸序列以及相关信息,以fasta格式保存。利用MEGA X 10.1.8[11]软件构建系统进化树进入ClustalW 2.0[12]软件,设置Gap opening penalty为10.00,Gap extension penalty为0.20,Delay divergent sequence为30%,其他参数默认,对蛋白质序列进行多重序列比对,序列比对结果用 Espript 3[13](http://espript. ibcp.fr/ESPript/ESPript/)与WebLogo[14](http://weblogo.berkeley.edu/logo.cgi)在线工具进行显示。通过ExPASy-ProtParam(https://web.expasy.org/ protparam)、ExPASy-ProtScale[15](https://web.expasy. org/protscale/)在线分析工具分析氨基酸序列的理化性质、疏水性,采用TMHMM 2.0(http://www.cbs.dtu.dk/ services/TMHMM/)、PSIPRED[16](http://bioinf.cs.ucl.ac. uk/psipred/)、Signal P 5.0[17](http://www.cbs.dtu.dk/ services/SignalP-5.0/)在线分析工具寻找蛋白质跨膜区域以及跨膜区域位置、分析信号肽。蛋白质的三维结构建模采用了两种方法,第一种为同源建模法,Modeller 9.24[18]软件,穿针引线法选用了Phyre2.0[19](http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)在线服务器进行建模。建模结构用SAVES 5.0(https://servicesn.mbi.ucla.edu/SAVES/)进行模型质量评估。使用UCSF Chimera[20]1.14软件进行结构叠合,分析结构差异。利用Autodock Vina[21]工具进行分子对接,结合Proteins Plus(https://proteins.plus/)计算结合口袋大小,探讨结构进化规律。
2 结果与分析
2.1 木聚糖1,4-β-木糖苷酶基因序列系统进化树构建以及代表序列筛选
利用NCBI网站收集木聚糖1,4-β-木糖苷酶的基因序列,除去不完整序列,共获得来自GH3、GH39、GH43家族的72条完整序列。72条序列来源广泛,包括原核生物链霉菌、单胞菌,真核生物曲霉、酵母菌、镰刀菌,以及真核动、植物。将获得的72条序列进行多序列比对构建系统进化树(图1)。从进化树的结果来看,木聚糖1,4-β-木糖苷酶可分成两大类。第一类含有38条序列,进一步分为三小类,编号为Ⅰ、Ⅱ、Ⅲ,第一小类包括8条真菌来源序列,第二小类包括10条细菌与真菌来源序列,第三小类包括20条真菌来源与真核动物来源序列。剩余序列构成第二类,同样分为三小类,编号为Ⅳ、Ⅴ、Ⅵ。第四小类的12条序列都源于细菌,第五小类仅有1条细菌来源与3条真核动物来源的序列,第六类则全为细菌来源。
图1的进化分类结果表明木聚糖1,4-β-木糖苷酶的进化进程不仅存在细菌与细菌、真菌与真菌之间的基因转移,同时也存在细菌与真菌之间、细菌与真核动物、真菌与真核动物之间的基因转移。分支代表进化谱系随时间的变化,分支长度越长,代表序列的变化越大,进化树中细菌来源的序列分支长度较短,而真菌以及真核生物来源的序列分支长度相对长,说明在木聚糖1,4-β-木糖苷酶进化过程中,真菌来源与真核动物来源更易发生变化,这可能与原核生物与真核生物细胞以及基因组之间的差异有关。以进化树的聚类为样本单位,按照分层抽样的原则,从进化树中挑选12条木聚糖1,4-β-木糖苷酶代表性序列(表1),进行进一步的结构分类研究。
表1 12条代表性序列对应的蛋白登录号 Table 1 Protein accession numbers of the 12 representative sequences
2.2 木聚糖1,4-β-木糖苷酶氨基酸序列分析
2.2.1 序列保守位点分析
利用ClustalW 2.0软件对12条代表性氨基酸序列进行多重序列比对,构建保守序列谱[22],用Espript 3在线工具显示12条序列的保守位点(图2)。木聚糖1,4-β-木糖苷酶存在15个较保守位点,位于以BV401_RS11760为基准的25、66、81、87、102、125、174、294、310、311、355、387、400、433、452号位点。虽然保守位点分布分散,但在进化过程中的保守性相对较好,推测可能是与酶的结构或功能密切相关的氨基酸位点。没有发现超保守位点,说明已选择的木聚糖1,4-β-木糖苷酶代表性序列进化过程相对独立,可以从中总结其进化及结构规律。
图2 12条木聚糖1,4-β-木糖苷酶代表性序列保守位点 Fig.2 12 Representative sequence conserved sites of xylan 1,4-β-xylosidase
2.2.2 氨基酸序列系统进化树分析
利用MEGA X软件,对12条代表性序列的氨基酸序列进行系统进化树的构建。氨基酸序列系统进化树的分析结果(图3)将12条代表性序列分成三个大类,GTNG_RS09260、BXA11_RS32660、BV401_RS11760、LK06_RS05470为第一类,属于GH39家族,GH3家族的LOC112950837、MgXBX3、LY89DRAFT_679094为第二类,FOXG_09848、FOXG_02672、SPSK_01007、MgXBX4、A1O5_02121为第三类,属于GH43家族。系统进化树的分类与其家族相对应,且与基因序列进化的分类具有一致性,表示木聚糖1,4-β-木糖苷酶的序列的家族划分是以序列相似性为基础。利用Signal P 5.0预测氨基酸序列的信号肽(表2),发现只有LY89DRAFT_679094、MgXBX3、FOXG_09848、FOXG_02672四条序列存在信号肽,均匀的分布在GH3家族与GH43家族的聚类上,说明木聚糖1,4-β-木糖苷酶在进化过程中分出了胞外酶与胞内酶,但信号肽的有无并不能体现其结构进化规律。
图3 12条氨基酸序列系统进化树 Fig.3 Phylogenetic tree of 12 amino acid sequences
表2 信号肽预测结果 Table 2 Signal peptide prediction results
2.2.3 氨基酸序列理化性质分析
用ExPASy-ProtParam在线工具对12条代表性氨基酸序列进行理化性质的预测[23],结果如表3所示。代表序列的氨基酸数在324~829之间,分子质量(Mr)最大为89769.18,最小为36301.39,相差较大。除了LOC112950837、BV401_RS11760的预测等电点分别为7.95、8.81,大于7.40为偏碱性蛋白质外,其余的都为酸性蛋白质。LOC112950837源于无尾线虫,而BV401_RS11760源于自溶链霉菌,对应物种都能较好的适应偏碱性的环境,符合酶的特征与环境相互适应的关系。比较负电荷残基总数(Asp+Glu)与正电荷残基总数(Arg+Lys)的数量,从整体上来看,几乎所有的序列都带负电荷较多,说明其大多为酸性蛋白,这一预测结果与等电点预测结果相符。序列的总原子数相差较大,极差为7514,脂肪系数的范围为65.68~84.59,序列的总平均亲水性为-0.04~-0.64,皆为负数,表现为亲水性蛋白,易溶于水,这为木聚糖1,4-β-木糖苷酶表现出水解性质提供基础条件。
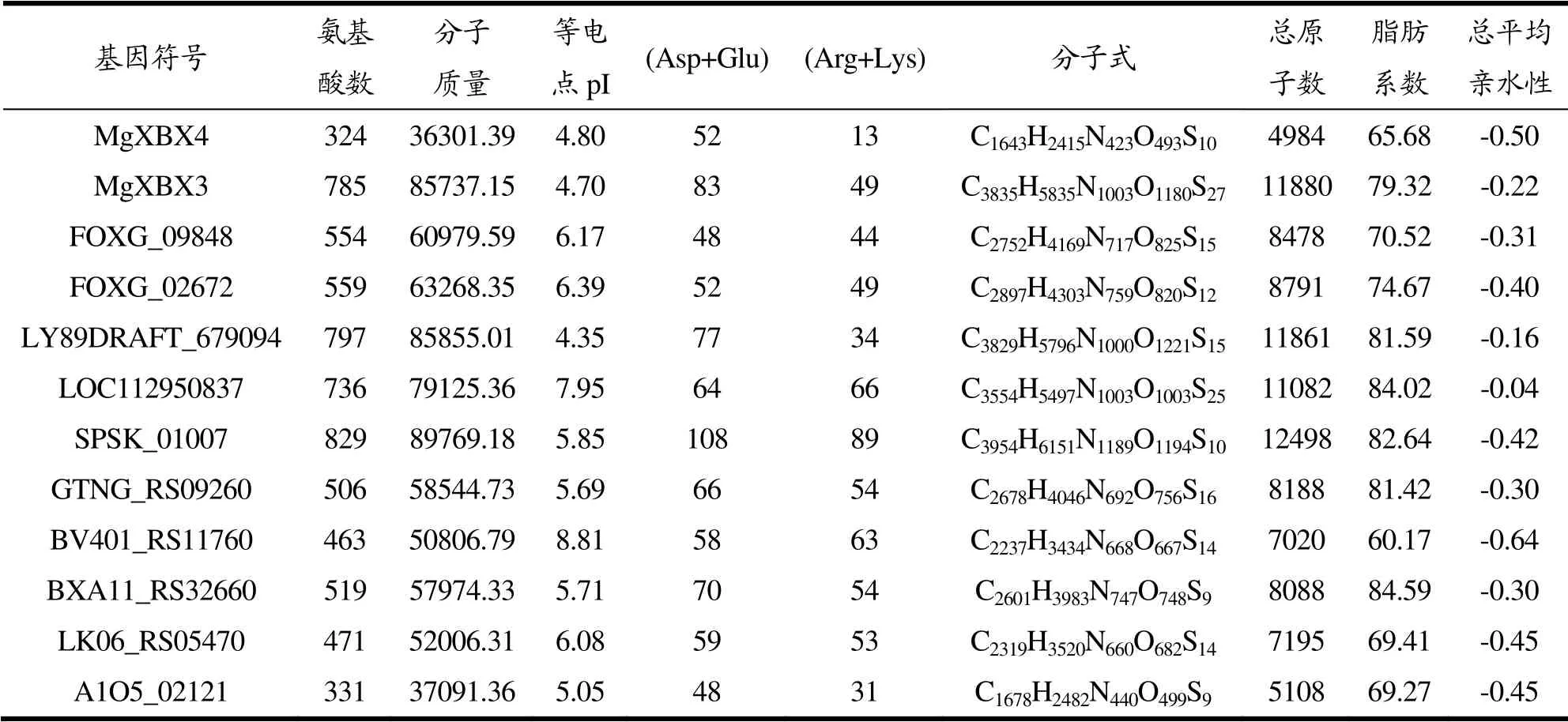
表3 12条代表性木聚糖1,4-β-木糖苷酶理化性质 Table 3 Physicochemical properties of 12 representative xylan 1,4-β-xylosidase
2.2.4 氨基酸序列疏水性分析
使用ExPASy-ProtScale在线工具对12条代表性氨基酸序列进行疏水性分析(图4)。分析结果以0位基准,在0以下的峰为亲水峰,以上的峰则为疏水峰,根据亲水峰和疏水峰的数量比较来判断蛋白质的疏水性。木聚糖1,4-β-木糖苷酶12氨基酸序列疏水性表明,所有的氨基酸序列亲水峰的数量都明显多于疏水峰的数量,表现为亲水性,与ExPASy-ProtParam对氨基酸序列疏水性的一致,进一步证明木聚糖1,4-β-木糖苷酶为亲水性蛋白,说明在蛋白肽链折叠的过程中,亲水性氨基酸更倾向于在蛋白外部,使蛋白具有可溶性。
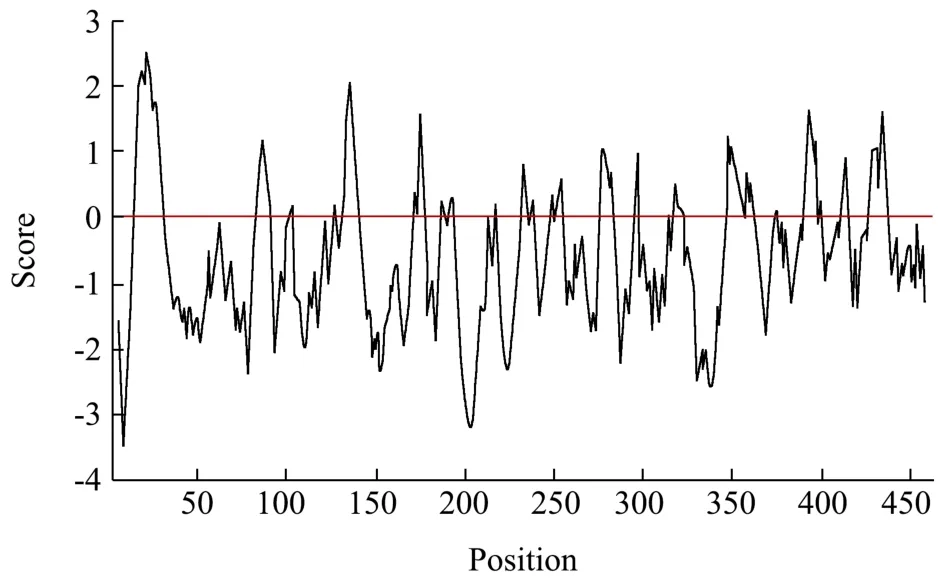
图4 BV401_RS11760疏水性预测图 Fig.4 Prediction chart of BV_401RS 11760 hydrophobicity

表5 结构分类表 Table 5 Structural classification
2.3 木聚糖1,4-β-木糖苷酶结构分析
利用Modeller 9.24软件以及Phyre2在线服务器对12条代表性氨基酸序列进行三维结构建模,利用SAVES 5.0对建模结果进行Verify 3D得分计算,评估模型质量。根据SAVES 5.0提供的Verify 3D得分,木聚糖1,4-β-木糖苷酶皆在80%以上(表4),说明模型质量良好,可进行进一步的结构分析。
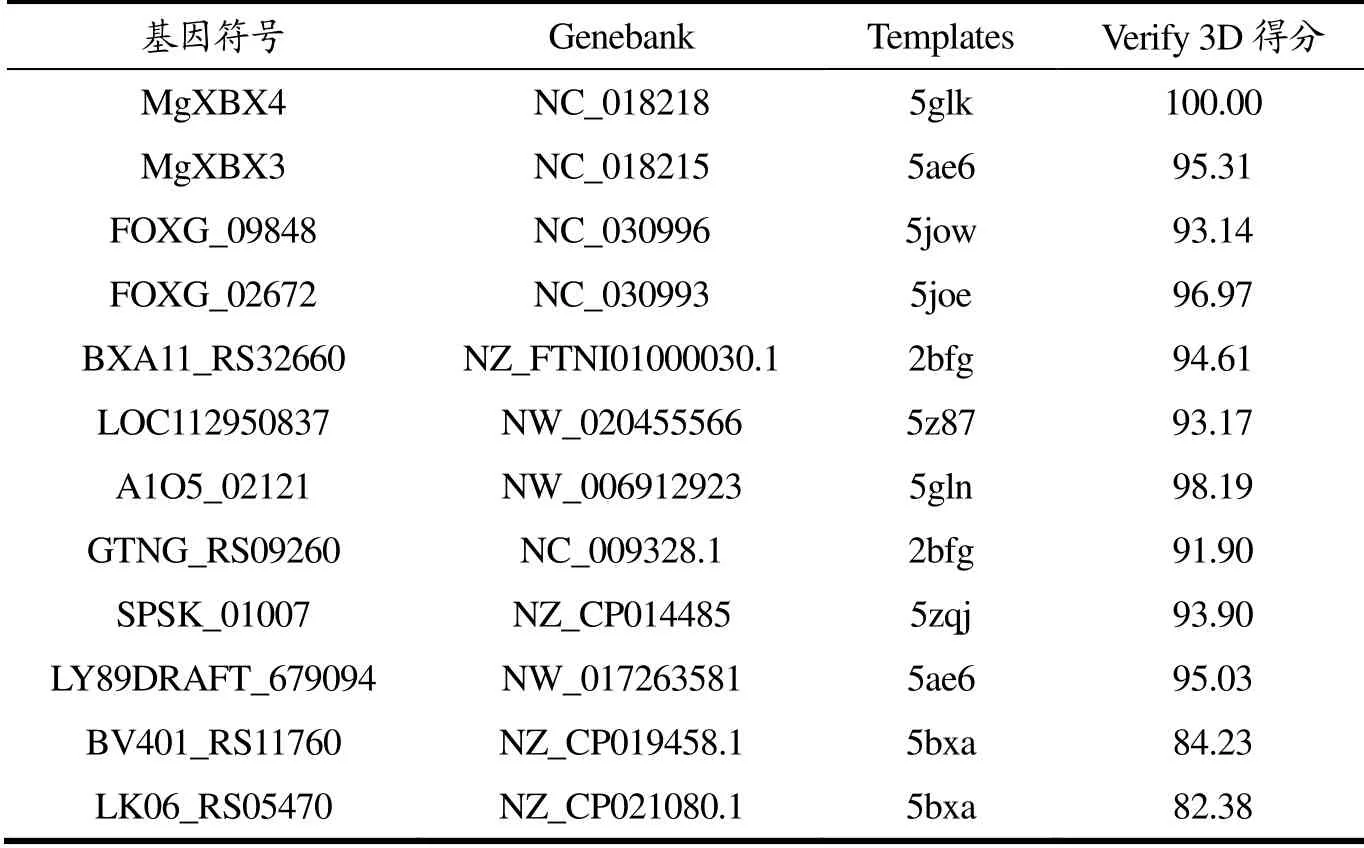
表4 12条代表性氨基酸序列建模结果 Table 4 Results of modeling 12 representative amino acid sequences
借助Chimera 1.14软件进行可视化以及结构叠合,叠合结果显示,依据木聚糖1,4-β-木糖苷酶的特征结构,可分成三个大类。糖苷水解酶GH39家族的GTNG_RS09260、BXA11_RS32660、BV401_RS11760、LK06_RS05470四条序列组成第一类(图5),此类结构都拥有一个(α/β)8TIM折叠桶结构;第二类(图6)包含LOC112950837、MgXBX3、LY89DRAFT_679094三条序列,属于GH3家族,由(α/β)8TIM折叠桶、(α/β)6三明治结构、FnⅢ型β折叠片层三个结构域共同组成,(α/β)8TIM折叠桶、(α/β)6三明治结构形成了一个上方开口大,下方开口小的碗状结构;A1O5_02121、MgXBX4、FOXG_09848、FOXG_02672 和SPSK_01007五条GH43家族序列共同组成了第三类(图7),这一类的木聚糖1,4-β-木糖苷酶都存在一个五叶的β折叠桶。此外,通过结构叠合,发现C末端延伸的β折叠片层结构可将第三类更为细致的划分成两个小类,A1O5_02121、MgXBX4为第一小类(图8a),只包含五叶β折叠桶这一基本结构,第二小类(图8b)的FOXG_09848、FOXG_02672和SPSK_01007则在C末端连接一个β折叠片层,与Rohman等[24]对木糖苷酶的结构分类结果一致,有文献报道这种C端的β折叠片层对木聚糖1,4-β-木糖苷酶的催化活性至关重要[25]。这一分类与GH43家族氨基酸序列进化树聚类相符,暗示C端延伸的β-折叠片层可以反映GH43家族木聚糖1,4-β-木糖苷酶的结构规律。
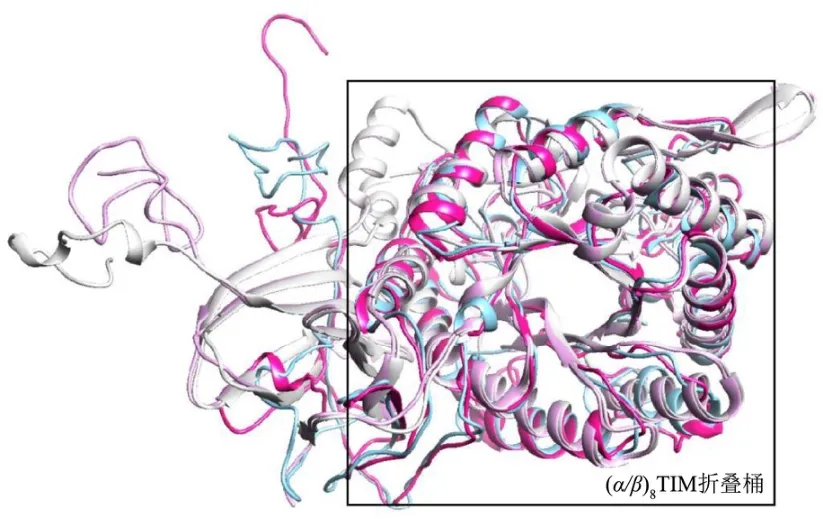
图5 木聚糖1,4-β-木糖苷酶第一大类叠合图 Fig.5 Overlay of the first major class of xylan 1,4-β-xylosidase
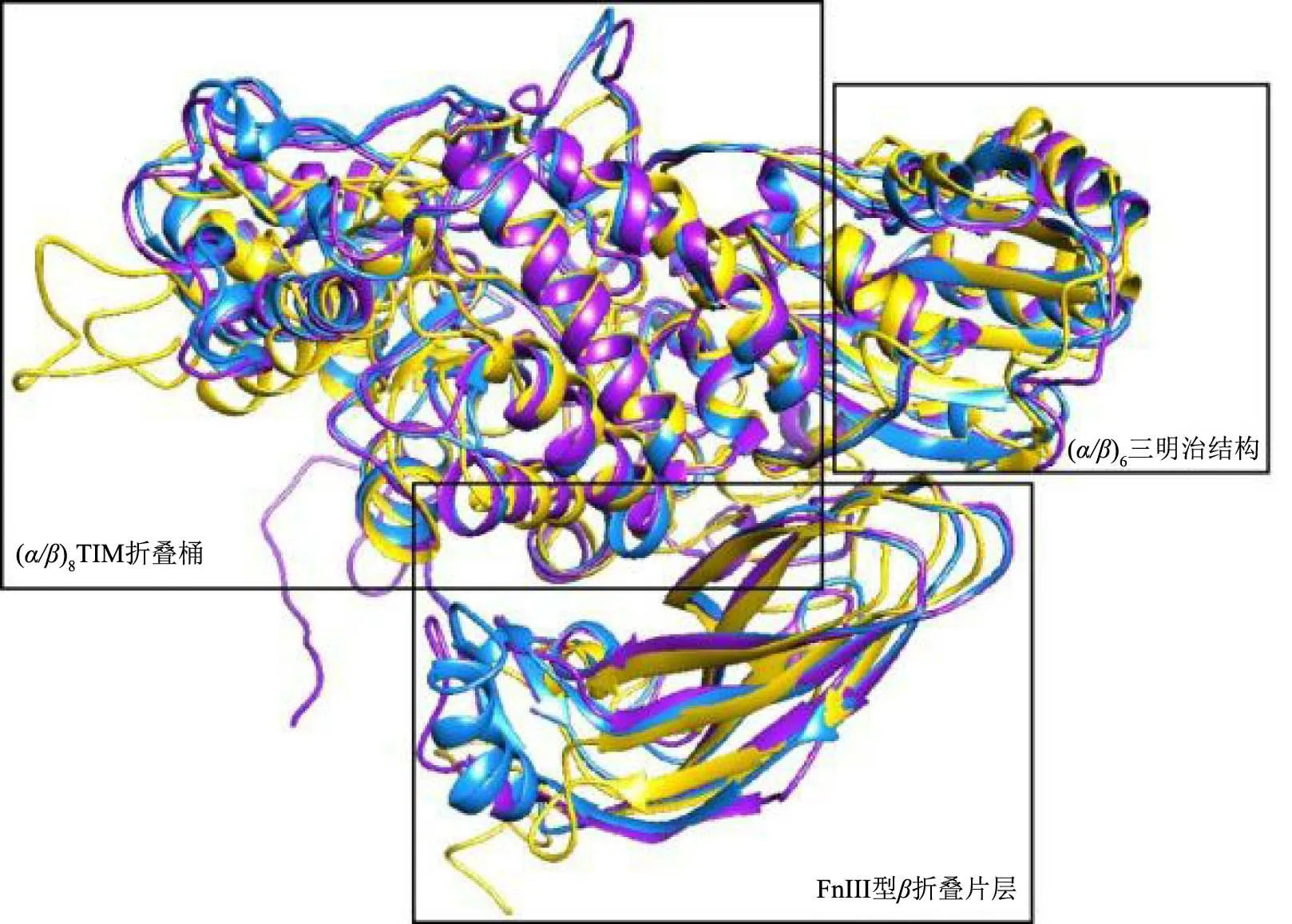
图6 木聚糖1,4-β-木糖苷酶第二大类叠合图 Fig.6 Overlay of the second major class of xylan 1,4-β-xylosidase
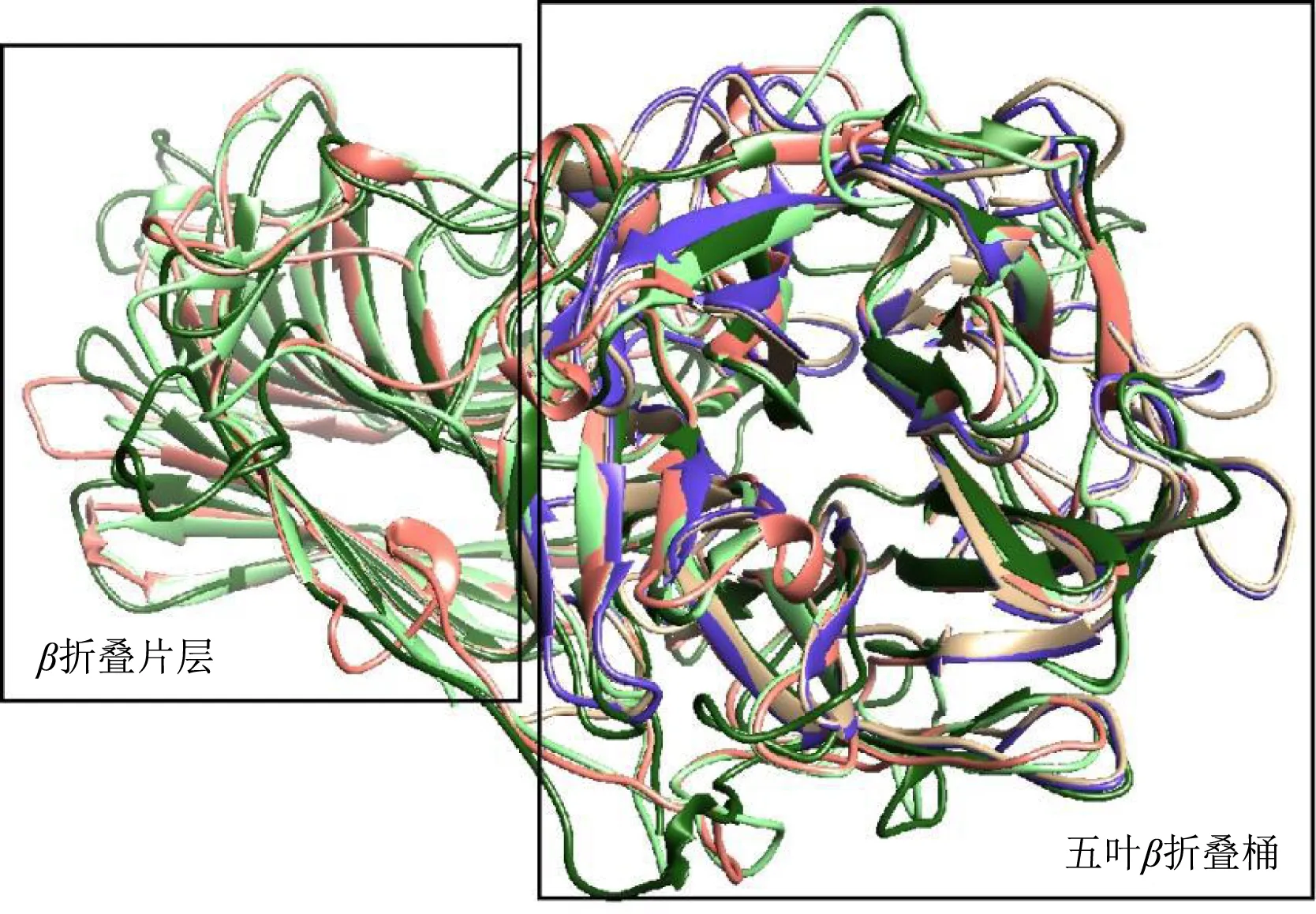
图7 木聚糖1,4-β-木糖苷酶第三大类叠合图 Fig.7 Overlay of the third major class of xylan 1,4-β-xylosidase
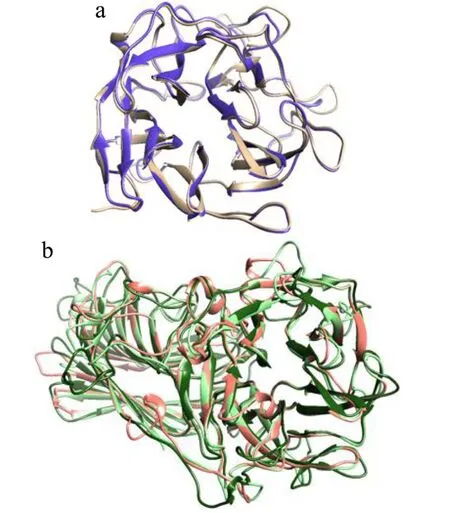
图8 木聚糖1,4-β-木糖苷酶第三大类的两个小类叠合图 Fig.8 Overlay of two subclasses of the third major class of xylan 1,4-β-xylosidases
目前已有的研究报道中,木聚糖1,4-β-木糖苷酶的底物谱较广,不同家族的酶对应水解不同的底物。表6列举了不同家族木聚糖1,4-β-木糖苷酶的酶学性质,GH3家族的木聚糖1,4-β-木糖苷酶无法水解低聚木糖,但可以作用于天然糖苷类底物以及多种人工底物,如pNPX(对硝基苯基-β-D-吡喃木糖苷)、pNPA(对硝基苯基-α-L-阿拉伯呋喃苷)、pNPG(对硝基苯基-β-D-吡喃葡萄糖苷);GH39家族木聚糖1,4-β-木糖苷酶表现出广泛的底物特异性,不仅能水解天然糖苷类底物,对低聚木糖和多聚木糖同样也有水解作用;GH43家族木聚糖1,4-β-木糖苷酶仅能水解人工底物pNPX、pNPG以及低聚木糖,需要内切木糖苷酶的协同作用下才能水解多聚木糖。
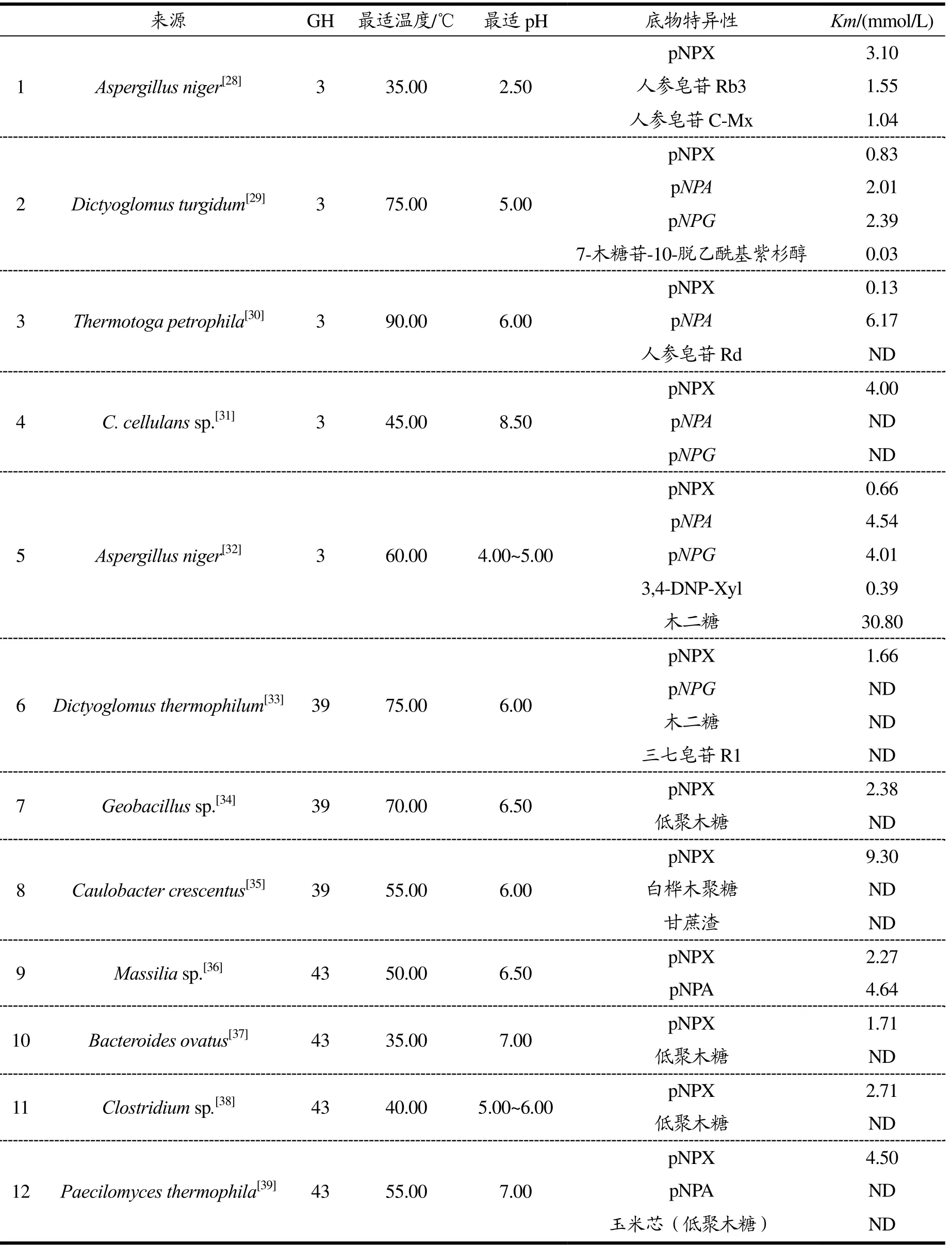
表6 部分来源木聚糖1,4-β-木糖苷酶学性质表 Table 6 List of enzymatic properties of xylan 1,4-β-xylosidase from some sources
以所有木聚糖1,4-β-木糖苷酶的共有底物pNPX为配体,与属于不同分类的木聚糖1,4-β-木糖苷酶LOC112950837、BXA11_RS32660、A1O5_02121进行分子对接确定结合口袋位置,计算结合口袋大小(图9,表7)。计算结果显示GH3家族的结合口袋宽且浅(图9a),适合分子量大的天然糖苷类底物如人参皂苷等与之结合;此外GH3家族木聚糖1,4-β-木糖苷酶蛋白质三维结构与α-阿拉伯呋喃糖苷酶以及β-葡萄糖苷酶结构相似度极高,可以水解pNPX、pNPA、pNPG,表现出三酶性质[26]。GH39家族木聚糖1,4-β-木糖苷酶结合口袋大、开口开放(图9b),这种大且开的口袋赋予了酶容纳大分子量底物与木糖分支链的能力,水解底物囊括人工底物pNPX、天然糖苷、低聚木糖与多聚木糖,拥有宽广的底物谱[27]。GH43家族木聚糖1,4-β-木糖苷酶的结合口袋相较于GH39家族木聚糖1,4-β-木糖苷酶显得窄、深,仅能支持与只有2~7个木糖分子的低聚木糖结合(图9c)。
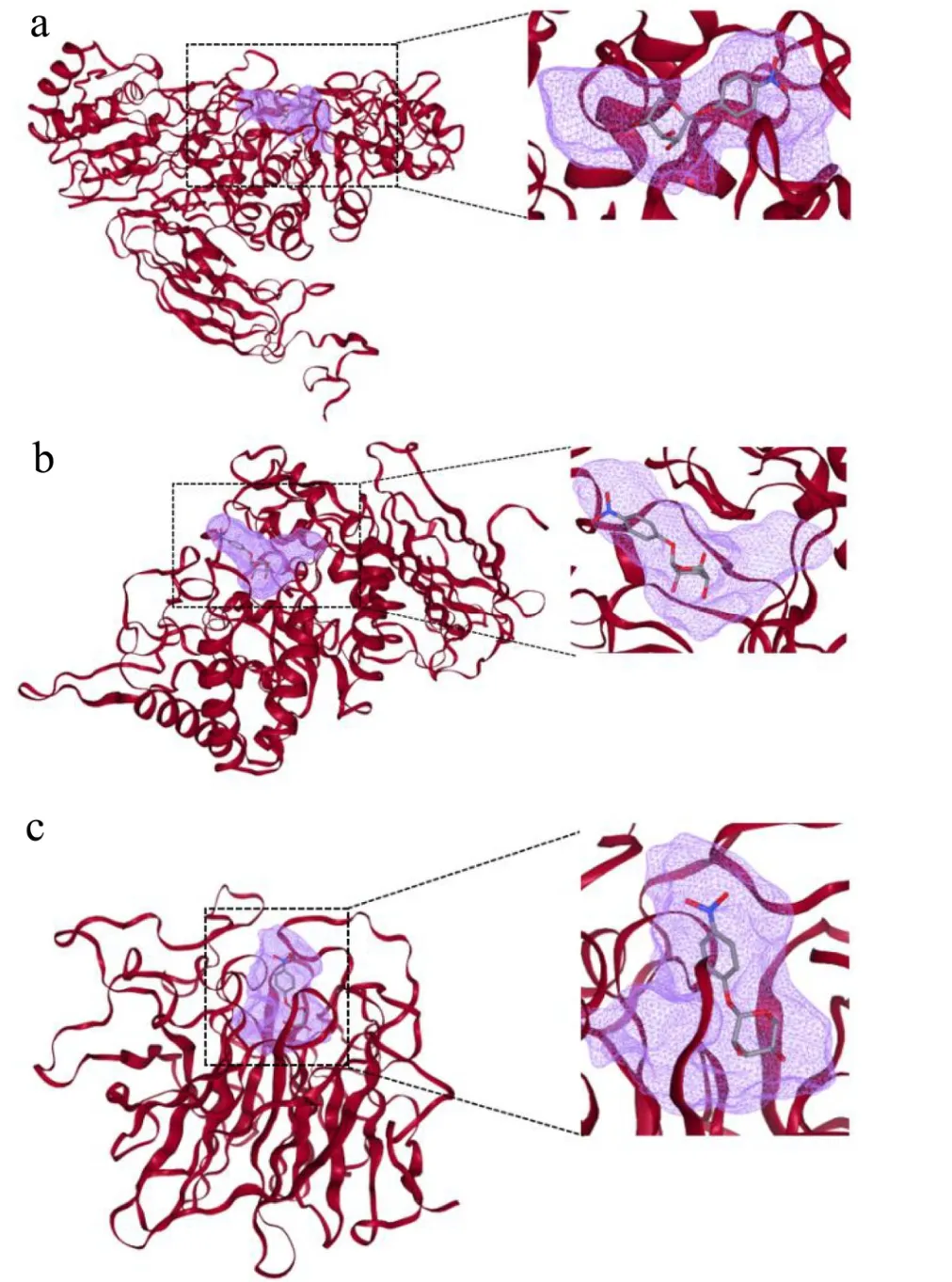
图9 不同分类木聚糖1,4-β-木糖苷酶结合口袋 Fig.9 Binding pockets of xylan 1,4-β-xylosidases from different classifications
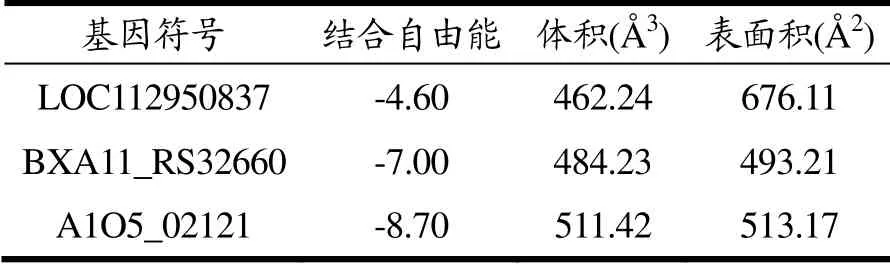
表7 不同分类木聚糖1,4-β-木糖苷酶结合口袋 Table 7 Binding pockets of xylan 1,4-β-xylosidases from different classifications
综上所述,木聚糖1,4-β-木糖苷酶的底物特异性与结构中结合口袋的大小与位置呈现了一定的相关性,说明酶在进化过程中为了适应底物,可能会对结构进行调整以获得不同的底物结合口袋。木聚糖1,4-β-木糖苷酶的进化规律不仅体现在三级结构上,水解底物的大小对其结构进化也产生了一定程度的影响。
3 结语
本文利用NCBI数据库,收集了木聚糖1,4-β-木糖苷酶的基因序列,成功构建系统进化树后,从中筛选了12条代表性序列,对其进行多重序列比对以及一级结构、二级结构、三级结构建模等分析。多序列比对结果显示木聚糖1,4-β-木糖苷酶的保守位点较少,进化过程并不保守,在长期进化的过程中这些位点能始终存在,说明其可能对于结构或功能的意义很大。蛋白质理化性质以及疏水性预测性都表明了这12条代表性序列都是亲水蛋白质,信号肽预测结果提示木聚糖1,4-β-木糖苷酶分泌方式可分为胞内分泌与胞外分泌;根据蛋白质结构叠合的结果,可以将木聚糖1,4-β-木糖苷酶分成三个大类,第一大类共有一个(α/β)8折叠桶;第二大类的结构较为复杂,由三个结构域共同组成,从N端到C端依次是(α/β)8TIM折叠桶、(α/β)6三明治结构、FnⅢ型β折叠片层;第三大类是一个五叶β折叠桶状结构,C端β折叠片层的有无将第三大类划分成两个小类,这三种结构共同构成了木聚糖1,4-β-木糖苷酶的代表性结构。结构分类结果与氨基酸序列进化树分类结果一致,说明木聚糖1,4-β-木糖苷酶的进化规律可以体现在三级结构上,每种类型的酶对应底物不同,说明底物大小能在一定程度上影响木聚糖1,4-β-木糖苷酶的进化。本文通过分析木聚糖1,4-β-木糖苷酶的12条代表性氨基酸序列,说明了木聚糖1,4-β-木糖苷酶的进化规律,并总结归纳其结构特征,为木聚糖1,4-β-木糖苷酶的定向改造、酶结构与功能关系研究奠定生物信息学基础。
