基于转录组测序技术研究三角鲂和团头鲂抗嗜水气单胞菌感染基因表达差异
2022-08-27戴瑜来阮松林冯晓宇
刘 凯,谢 楠,戴瑜来,阮松林,冯晓宇
(杭州市农业科学研究院,浙江 杭州 310024)
鲂属(Magalobrame)鱼类,在分类地位上属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)和鲌亚科(Cultrinae),该属主要包括4种鱼类:三角鲂(Megalobramaterminalis)、团头鲂(M.amblycephala)、广东鲂(M.hoffmanni)和厚颌鲂(M.pellegrini)等。团头鲂,又名“武昌鱼”,是我国特有的优良草食性鱼类,因食性广、生长快、养殖成本低等优点,其还是我国主要的大宗淡水水产养殖品种之一[1]。三角鲂,在钱塘江附近地区被称为“三角鳊”“塔鳊”,在黑龙江附近地区被称为“法罗”。相对于团头鲂,三角鲂的养殖区域较小,但因具有快速生长期长、商品个体大等特点,三角鲂在浙江、湖南、山东、江苏等地也具有一定的养殖规模,具有良好的养殖前景[2-3]。
近年来,团头鲂病害频发,由嗜水气单胞菌(Aeromonashydrophila)引起的细菌性败血症是团头鲂养殖过程中危害较大的疾病之一,严重影响养殖效益[4]。而养殖实践发现,与同属的团头鲂相比,三角鲂的细菌性败血症发生率较低,即使发生了该病,使用常规药物就可较好地控制,未有大面积发病的情况,且病死率低。三角鲂和团头鲂之间差异小,亲缘关系较近[5],但2种鱼类在感染嗜水气单胞菌后所表现出的差异,除去环境因素外,遗传因素是其中一个重要的原因。此前,方献平等[6]采用非标记定量蛋白质组学技术分析了在嗜水气单胞菌侵染胁迫后3 h、10 h和24 h肝组织蛋白质组的变化,结果表明三角鲂在戊糖磷酸途径、脂肪酸代谢、亮氨酸降解等通路与团头鲂存在显著差异。为在基因组注释的丰富度以及期望于基因水平上了解三角鲂和团头鲂在嗜水气单胞菌侵染过程中的表达差异,本文基于转录组测序技术研究了三角鲂和团头鲂在感染嗜水气单胞菌后转录组的变化,以期了解三角鲂和团头鲂在嗜水气单胞菌感染上的差异性,为阐明嗜水气单胞菌致病机制乃至抗病育种提供研究基础。
1 材料与方法
1.1 实验材料
实验用三角鲂和团头鲂由杭州市农业科学研究院水产研究所提供,嗜水气单胞菌株分离自江苏武进某养殖场。致病实验与方献平等[6]报道实验相同,选取了3 h和24 h两个时间点,每个时间点分别取三角鲂和团头鲂各5尾,体质量为130.5~150.5 g。每5尾鱼分别取肌肉、肝脏、血液3个组织并分别浸泡于3份RNAfixer无液氮RNA样品储存液中,于-80℃冰箱中保存备用。测序时,每3个组织等质量混合后作为1份实验材料,进行建库和测序。各实验样品被分别标记为SJF3、SJF20、TTF3、TTF20,表示3 h和24 h两个时间点采集的三角鲂和团头鲂样品。
1.2 RNA提取及建库测序
RNA提取及建库测序委托杭州英睿生物科技有限公司完成。取总RNA样品,利用试剂盒合成cDNA,通过末端修复、连接接头和纯化,获得三角鲂和团头鲂的cDNA文库,建好的文库通过Illumina HiseqTM 2000平台进行测序。测序样本的原始数据(Raw Reads)过滤与组装等步骤参照刘凯等[7]的方法进行,过滤数据时,采用Cutadapt去除3′端带接头的序列以及去除平均质量分数低于Q20的Reads,使用Trinity软件对所有样本的Clean Reads进行拼接,最终组装获得Unigenes,作为参考序列。
1.3 功能注释、分类及代谢途径分析
通过blastx、Blast2GO和KOBAS等程序将Unigenes比对到数据库NR(NCBI non-redundant protein sequences)、COG(Cluster of orthologous groups of proteins)、GO(Gene ontology)和KEGG(Kyoto encyclopedia of genes and genomes)等,进而得到Unigenes的功能注释信息。
1.4 差异基因分析
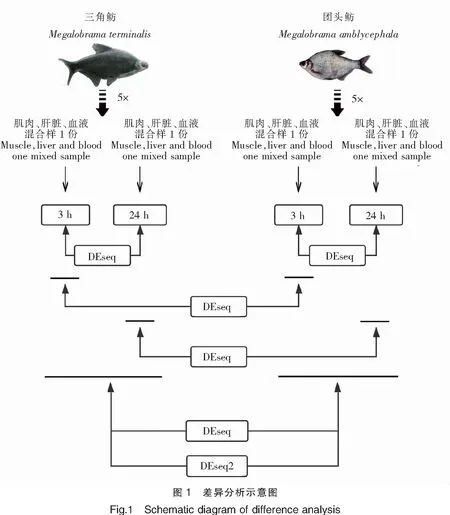
使用转录组表达定量软件RSEM,以组装获得的Unigenes作为参考序列,分别将每个样品的Clean Reads比对到Unigenes序列上,统计每个样品比对到每一个基因上的Reads数,并计算每个基因的FPKM值,最终获得基因表达谱。分别使用DESeq[8]和DESeq2[9]的R包对比分析三角鲂和团头鲂在感染嗜水气单胞菌后的差异基因表达,将P<0.05并且log2|(Fold change)|>1的基因定义为差异基因,分析示意图见图1。使用clusterProfiler[10]的R包通过GO和KEGG数据库对主要的差异表达基因进行富集分析。
2 结果与分析
2.1 转录组数据与组装
三角鲂和团头鲂的样本经建库、测序后,每个样本获得了超过4 G的数据量,共获得Raw Reads 114 827 718条。去除接头序列、低质量序列后,共获得Clean Reads 114 824 154条,占总Reads的99.996 8%(表1)。序列经拼接和组装后,共获得62 780个Unigenes,平均长度达到531.883 bp,Unigenes的N50为652 bp,组装完整性较高(表2)。

表1 测序数据质量评估

表2 测序数据的统计汇总
2.2 转录组功能注释
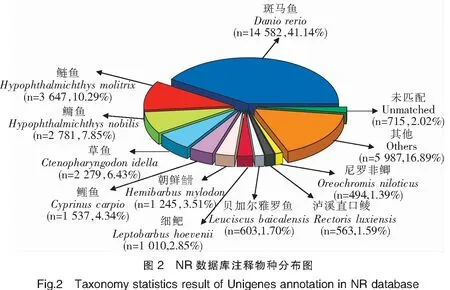
在拼接获得Unigenes后,与公共数据库进行比对注释,62 780个Unigenes中有35 443个可注释到NR数据库,比例最高,为56.46%;仅有8 632个可注释到COG数据库,比例最低,为13.75%(表3)。在NR数据库注释中,注释来源最多物种为斑马鱼(Daniorerio),其次为鲢鱼(Hypophthalmichthysmolitrix)、鳙鱼(Hypophthalmichthysnobilis)等(图2)。在GO数据库注释中,11 622个Unigenes被注释到38个GO Term分类上,其中生物学过程GO Term分类的相关基因较多,如GO:0009987 细胞过程GO Term分类上注释了4 599个Unigenes;分子功能GO Term分类中,GO:0005488 细胞结合GO Term分类相关基因最多,注释了7 413个Unigenes。在COG数据库注释中,一般功能预测分类注释的Unigenes最多,为1 979个;其次为翻译后修饰、蛋白质周转、伴侣的分类,注释Unigenes 1 353个;之后为转录分类,注释Unigenes 684个。在KEGG数据库注释中,注释Unigenes 最多的前3个通路分别是补体和凝血级联、金黄色葡萄球菌感染、吞噬体,注释Unigenes 达到9 898、3 721和1 653个。
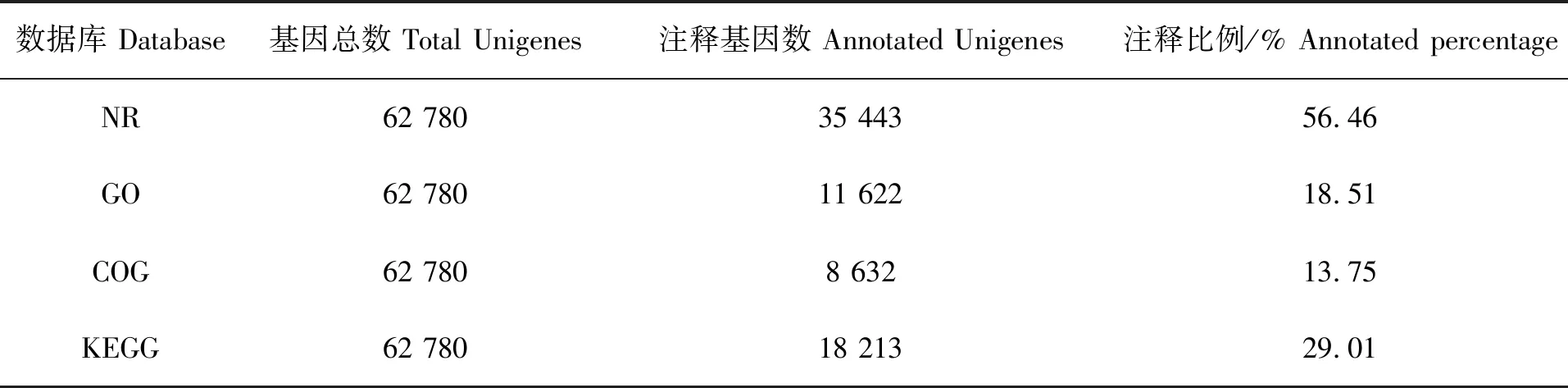
表3 基因注释结果统计
2.3 基因表达的差异分析
62 780个Unigenes中,有33 693个Unigenes在三角鲂中表达,有32 936个Unigenes在团头鲂中表达,其中有24 834个Unigenes在三角鲂和团头鲂中共表达。基于DESeq,分别比较了三角鲂、团头鲂在3 h和24 h时间点上基因表达的差异变化(图3)。对TTF20-TTF3的差异分析表明,团头鲂感染24 h后与感染3 h后相比,显著上调表达的基因有4个,分别为FN1b、PAH、CBLN14和INIH3b.2,显著下调表达基因的前10个主要有ApolipoproteinA-I、C-typelectin14以及UBA、MHCclassⅠantigin和MHCclassⅡalphaantigen等。对SJF20-SJF3的差异分析表明,三角鲂感染24 h后与感染3 h后相比,显著上调表达的基因的前10个主要有Ovary-specificC1q-likefactor、ComplementC3以及bain-DAA*0101、Hymo-UA和Meam-UBA等主要组织相容性复合物(Major histocompatibility complexes,MHC)相关基因,显著下调表达基因的前10个主要有CBLN6LOC793037、Phosphotransferase、TBL2和Cyca-UA1*01等。对SJF3-TTF3的差异分析表明,在感染3 h后,三角鲂相对于团头鲂,显著上调表达的基因的前10个主要有Preprohepcidin、FN1b、TBL2、Phosphotransferase和Cyca-UA1*01等,显著下调表达基因的前10个主要有MHCclassⅡalphaantigen、HBB、HBBLOC113082649和Ba1globin等。对SJF20-TTF20的差异分析表明,在感染24 h后,三角鲂相对于团头鲂,显著上调表达的基因的前10个主要有ApolipoproteinA-I、Ovary-specificC1q-likefactor、Serotransferrin、Hymo-UA和bain-DAA*0101等,显著下调表达基因的前10个主要有CFHL4、BHMTwu:fb53h01、G6PC1a.2和CBLN10RP71-1G18.9等。将以上显著上调或下调表达的基因进行表达趋势聚类分析(图4),三角鲂、团头鲂在3 h和24 h时间点上基因表达趋势可划分为6类,其中聚类模式4所包含的差异表达基因最多,为16个;聚类模式1所包含的差异表达基因数量为15个;聚类模式6包含基因12个;聚类模式3包含基因10个;聚类模式2、5分别包含基因5个、4个。由此可见,三角鲂、团头鲂基因表达趋势聚类主要模式为4、1、6和3。从以上主要聚类模式可见,三角鲂、团头鲂之间差异表达的基因主要以三角鲂或团头鲂下调表达的较多,而上调表达的基因较少。
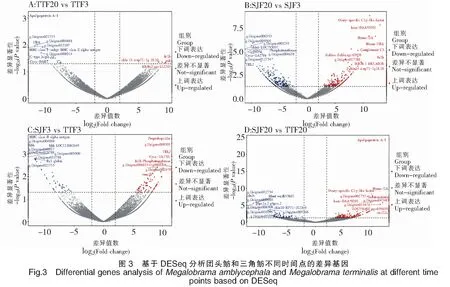
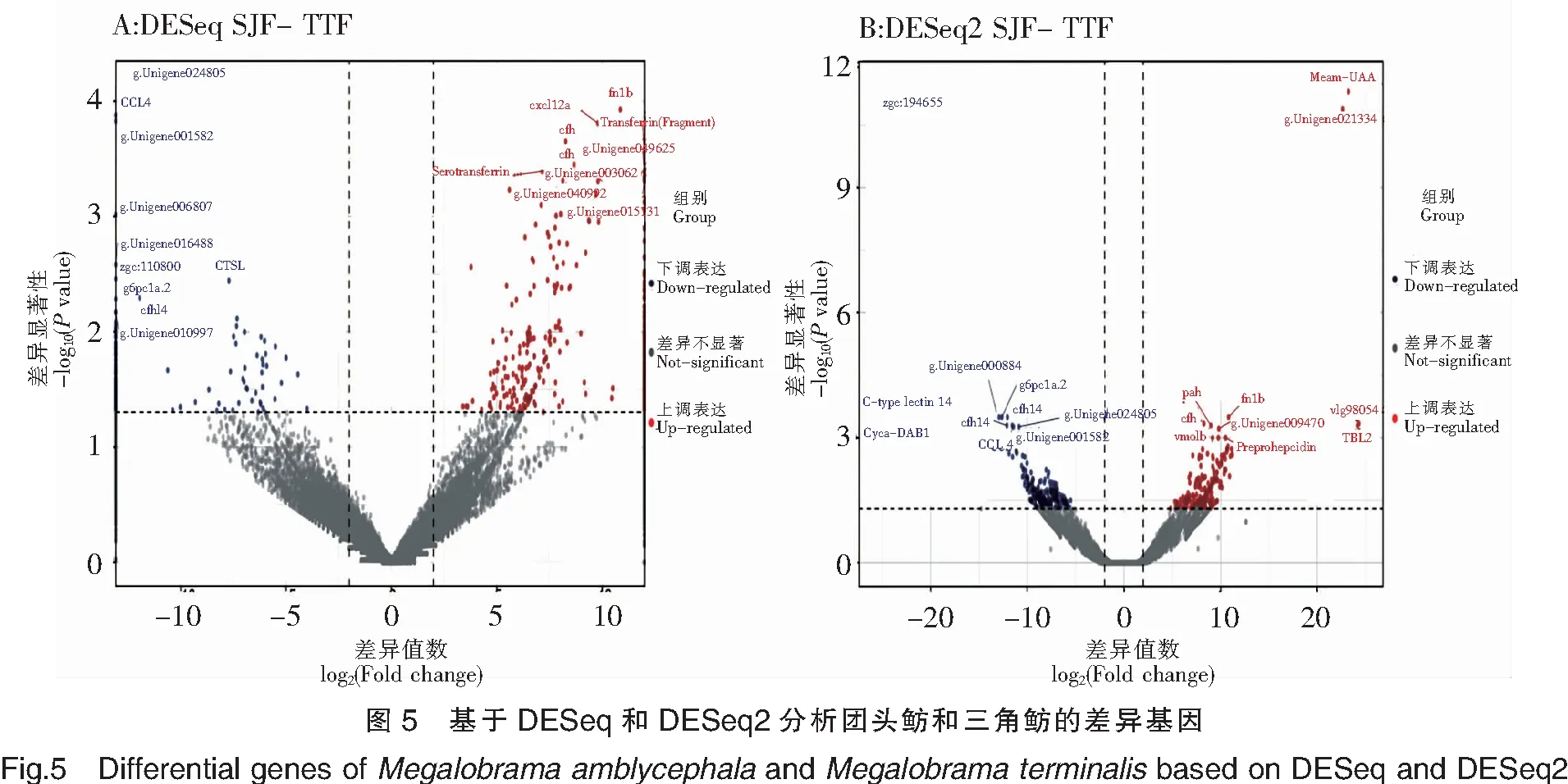
注:红色点表示显著上调的基因,其中显著上调的前10个基因标注了基因名。蓝色点表示显著下调的基因,其中显著下调的前10个基因标注了基因名。图5同此。
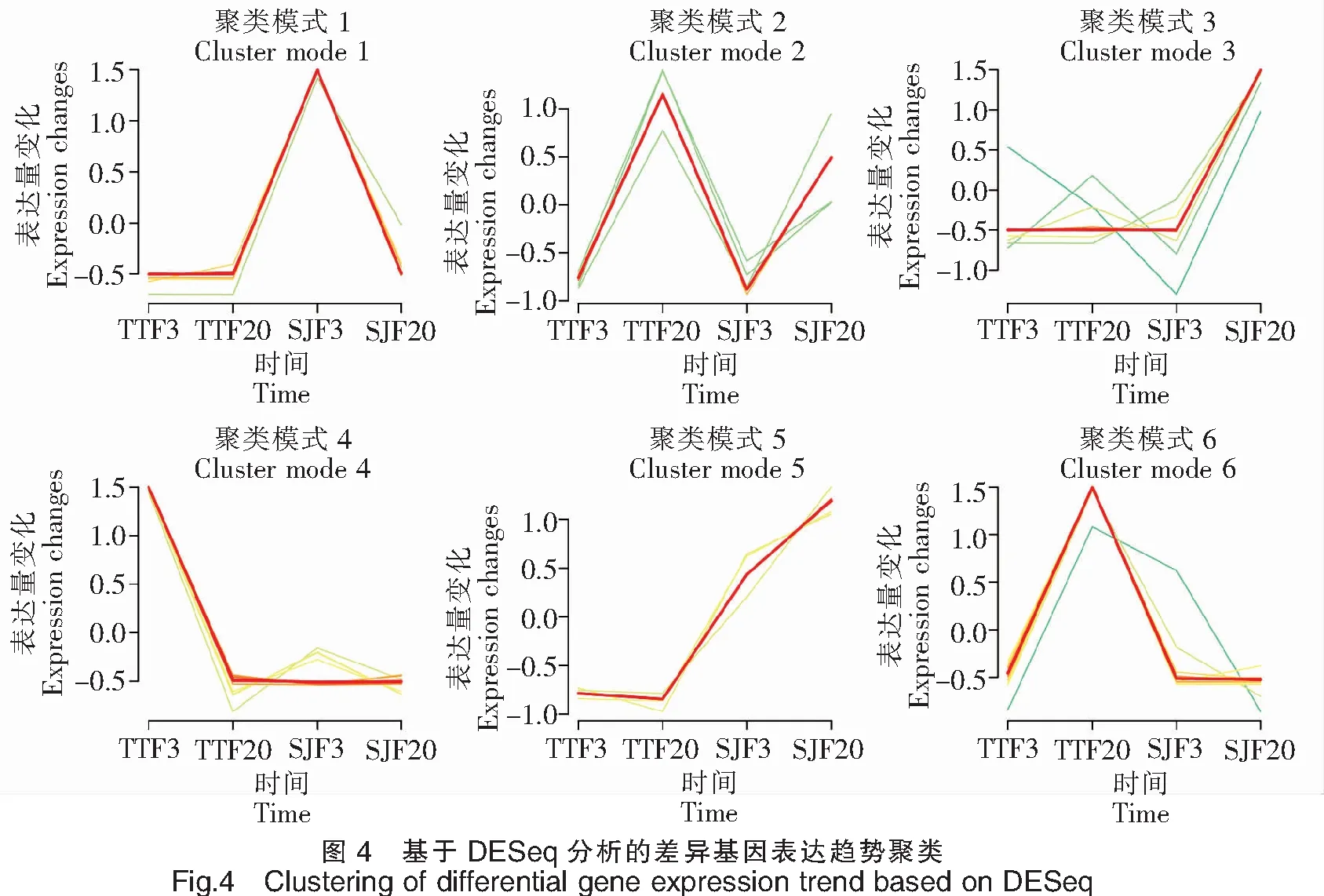
注:不同颜色线表示基于DESeq分析的差异基因在不同时间点和样本之间的表达变化。
基于DESeq和DESeq2,从总体上比较了三角鲂相对于团头鲂基因表达的差异变化(图5)。基于DESeq对SJF-TTF的差异分析表明,显著上调表达的基因的前10个主要有FN1b、CXCL12a、CFH、Transferrin(Fragment)和Serotransferrin等,显著下调表达基因的前10个主要有CCL4、CFHL4、G6PC1a.2和CTSL等。基于DESeq2对SJF-TTF的差异分析表明,显著上调表达基因的前10个主要有Meam-UAA、TBL2、CFH、PAH、FN1b和Preprohepcidin等,显著下调表达基因的前10个主要有Cyca-DAB1、CFHL4、C-typelectin14、G6PC1a.2和CCL4等。比较DESeq和DESeq2分析结果发现,有7个差异表达的基因在DESeq和DESeq2中均被鉴别出来,分别是上调表达基因FN1b和CFH,下调表达基因CCL4、CFHL4、G6PC1a.2,以及2个未注释的基因g.Unigene024805和g.Unigene001582。
2.4 差异表达基因的富集分析
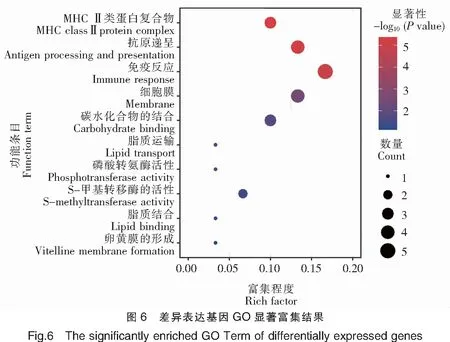
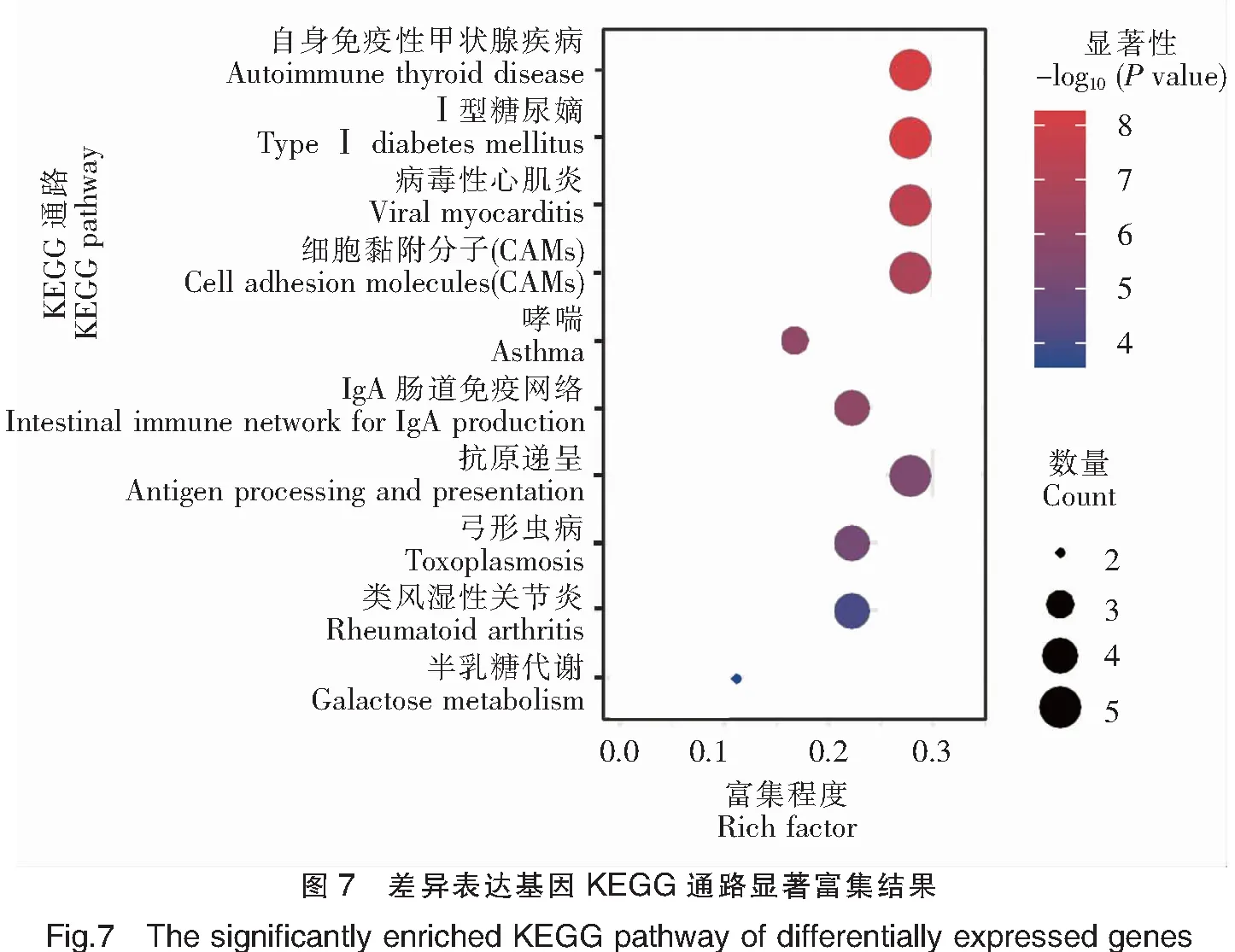
对基于DESeq和DESeq2方法分别获得的前20个表达差异最显著的基因进行GO富集分析,通过Rich factor、Pvalue值和富集到GO Term上的基因个数来衡量富集的程度(图6)。其中,免疫反应(GO:0006955)条目下富集的差异表达基因最多,30个GO注释的差异基因中有5个差异基因在此条目下;其次为抗原递呈(GO:0019882)和细胞膜(GO:0016020)2个条目,以及MHCⅡ类蛋白复合物(GO:0042613)和碳水化合物结合(GO:0030246)等条目。以上结果表明,三角鲂和团头鲂之间的抗感染差异与抗原递呈相关基因关系密切。进一步对以上获得的差异表达基因进行KEGG富集分析,富集程度的表征与GO富集分析类似(图7)。其中,自身免疫性甲状腺疾病(ko05320)、Ⅰ型糖尿病(ko04940)、病毒性心肌炎(ko05416)、细胞黏附分子(ko04514)和抗原递呈(ko04612)5个KEGG通路是富集程度最高的,18个KEGG注释的差异基因中,均有5个基因富集在这些通路下。剩下的通路还包括IgA肠道免疫网络(ko04672)、弓形虫病(ko05145)和类风湿性关节炎(ko05323)等。综上,依然可见抗原递呈相关基因与三角鲂和团头鲂之间的抗感染差异有关,此外细胞黏附通路(ko04514)也与细菌感染密切相关。
3 讨论
由于转录组测序无需事先以已知序列为目标,因此可以对任意物种进行测序而无需知道物种的基因组背景,这为研究物种间基因表达差异提供了方便[11-12]。因此,本研究基于转录组测序分析三角鲂和团头鲂抗嗜水气单胞菌感染的基因表达的差异。将三角鲂和团头鲂样本的测序数据经过滤后组装Unigenes作为参考序列,使用RSEM软件比对Unigenes序列确定表达量,以团头鲂为对照,分析三角鲂Unigenes表达量的相对变化。此前,方献平等[6]采用非标记定量蛋白质组学技术,分析在嗜水气单胞菌侵染胁迫后3 h、10 h和24 h时三角鲂和团头鲂肝组织蛋白质组的变化。基于方献平等[6]的蛋白应答聚类分析发现,10 h和24 h的应答蛋白质优选聚类后再与3 h的应答蛋白质聚类,表明10 h和24 h蛋白质应答具有相似性。基于此,本研究选择3 h和24 h作为时间节点开展转录组分析。由于方献平等[6]的分析仅基于肝脏组织,不能完全反映整个机体对嗜水气单胞菌侵染的应答情况。因此,本研究进一步分析了肌肉、肝脏和血液的混合组织样,以更全面了解三角鲂和团头鲂在抗嗜水气单胞菌侵染过程中基因表达的差异性。此外,由于DESeq2可以消除DESeq的边界效应,因此在进行整体分析中采用了DESeq2方法与DESeq方法共同进行了分析,减少单方法分析的误差。
基于DESeq的基因表达趋势聚类分析表明,三角鲂和团头鲂在3 h和24 h时间节点上的基因表达均表现为表达量无变化、表达上调和表达下调等3种情况,但其中主要以三角鲂表达量无变化、团头鲂表达下调(Cluster 4),团头鲂表达量无变化、三角鲂表达下调(Cluster 1)为主。可见,三角鲂、团头鲂之间差异表达的基因主要以三角鲂或团头鲂下调表达为主,这与罗非鱼(Oreochromisniloticus)的情况类似[13],但与蛋白质组学分析结果存在差异[6]。此外,为从总体上评估三角鲂和团头鲂感染后基因表达差异,进一步基于DESeq和DESeq2方法进行了分析,并进一步鉴定出上调表达基因FN1b和CFH、下调表达基因CFHL4和CCL4等,提示以上基因表达与三角鲂和团头鲂的感染差异相关。FN1b是编码纤维连接蛋白(Fibronectin,FN)的两种基因亚型之一[14],纤维连接蛋白则是细胞外基质的重要成分之一,而细胞外基质被认为是影响器官形成与修复的关键因子[15]。CFH(Complement factor H)即补体因子H ,调节血浆中补体的替代途径,抑制补体活性并介导细胞表面对替代途径激活剂和非激活剂的区分[16]。CFH蛋白产生缺陷后可致替代途径异常激活,导致细胞损伤[17]。CCL4[Chemokine(C-C motif)ligands 4 ]即CC型趋化因子配体4,是CC型趋化因子家族的一员,因其从脂多糖激活的巨噬细胞中被分离出来,亦称MIP-1β[18],它在免疫细胞迁移、活化、分化和增殖等方面具有重要作用[19]。三角鲂相对于团头鲂,FN1b和CFH基因表达显著上调,表明三角鲂在受到细菌攻击时更倾向于自我修复,而CCL4基因的下调,则表明团头鲂调动了更多的细胞因子用于消除细菌。CFHL4则是补体因子H相关蛋白(Complement factor H-related protein,FHRs)之一[16],目前关于其具体机制并未明确,但越来越多的数据表明,FHRs与CFH具有相反的作用,即它们直接增强补体活性[20]。因此,推测三角鲂相对于团头鲂CFHL4基因表达的下调也与三角鲂细胞自我修复基因有关。
基于GO和KEGG富集分析,对DESeq和DESeq2方法鉴定出的主要差异基因进行了富集分析,均表明三角鲂和团头鲂的感染差异与抗原递呈相关基因密切相关。基于转录组学对罗非鱼感染后的基因表达分析表明,吞噬作用在罗非鱼的抗感染免疫中起到重要作用,其中主要组织相容性复合物基因就是主要的吞噬相关基因[13]。本文结果与此类似,也确定了MHC基因在感染后的重要作用,并表明其与三角鲂和团头鲂抗感染差异相关。基于团头鲂MHCⅡα基因的研究也表明,MHC基因与团头鲂抗病性密切相关[21]。在DESeq和DESeq2共同鉴定出的差异基因中,仅FN1b被KEGG注释,其富集在ECM-受体相互作用(ko04512)通路(图7中未显示),表明ECM即细胞外基质与三角鲂和团头鲂抗感染差异密切相关。此外,在GO和KEGG富集分析中均富集到CXCL12a基因,分别富集于免疫反应(GO:0006955)条目和IgA肠道免疫网络(ko04672)通路下。CXCL12[Chemokine(C-X-C motif)ligand 12]即CXC型趋化因子配体12,是一种小分子的细胞因子,与CCL4同属于趋化因子家族,对淋巴细胞有强趋化作用[22]。而CXCL12基因在DESeq分析中,三角鲂相对于团头鲂显著上调,与团头鲂CCL4基因显著上调不同,表明三角鲂和团头鲂抗感染差异可能与招募淋巴细胞、促进细胞吞噬的机制不同相关。方献平等[6]基于蛋白质组学技术发现,相对于团头鲂,三角鲂β-免疫球蛋白感染后显著上调表达。但在本研究中,尽管检测到了β-免疫球蛋白基因表达,但在三角鲂和团头鲂间差异不显著,这可能是由于蛋白应答与基因表达的不同步性[23],或存在其他原因,都有待进一步研究。
