宁夏枸杞根际土壤链霉菌V-1-3次级代谢产物分析
2022-08-18胡运琪马晓莉贾荣亮林文星郭欢欢李彬李舂龙南泽东吴秀丽江志波
胡运琪 马晓莉 贾荣亮 林文星 郭欢欢 李彬 李舂龙 南泽东 吴秀丽 江志波,*
(1 北方民族大学化学与化学工程学院 化工技术基础国家民委重点实验室,银川 750021;2 中国科学院西北生态环境资源研究院甘肃省寒区旱区逆境生理与生态重点实验室,兰州 730000;3 宁夏医科大学药学院,银川 750004;4 北方民族大学实验室建设与管理处,银川 750021)
微生物次级代谢产物是新药研发的一个重要来源。20世纪青霉素和链霉素研发成功后,从微生物次级代谢产物中发现新抗生素曾经历过黄金时代[1-2],从放线菌,尤其是链霉菌次级代谢产物中研发出了具有抗细菌、抗病毒、抗肿瘤和免疫调节等作用的药物[3-5]。然而,近几十年来,以新抗生素为代表的微生物药物开发速度明显变缓[6],进入21世纪以来,36个成功上市的抗生素中,直接来源于微生物次级代谢产物的仅有达托霉素和非达霉素[7]。形成强烈对比的是,抗生素多重耐药及泛耐药菌的出现和快速流行,导致全球每年有数百万人死于耐药细菌感染[8-9]。2016年5月19日,英国吉姆·奥尼尔爵士(Jim O'Neill)在《全球抗生素耐药回顾:报告及建议》预测,如果目前的情况得不到改善,到2050年全球每年将有1000万人死于耐药菌感染[10]。可见,人类正逐渐步入“后抗生素”时代,因此,发现新结构、新作用机制的抗耐药抗生素迫在眉睫。对陆地微生物长达半个多世纪的研究中,普通生态环境放线菌次级代谢产物的重复筛选已成为新抗生素发现的瓶颈之一,从中寻找新结构抗生素已极具挑战性[11]。而深海[12]、冰川[13]、火山口[14]、红树林[15]等极端或特殊生态环境(简称“特境”)微生物的研究,拓宽了药用微生物来源,为基于物种多样性,发掘新次级代谢产物生物合成基因,进而开展新结构抗生素发现奠定了基础。
据不完全统计,宁夏回族自治区共有盐碱地约263.3万亩,其中银川北部(简称宁北地区)占58%,该地区常年干旱少雨,年蒸发量远大于降水量,土壤盐碱化十分严重。盐碱土具有与普通土壤明显不同的特点,如无机盐组分多、pH偏碱性,土壤腐殖质淋失,通气和透水性不良,严重时导致植被难以存活[16]。盐碱环境微生物在诸多逆境因子作用下,经过长期演化,能获得多种特殊的代谢能力,是发掘潜在新抗生素的药用资源。放线菌,尤其是链霉菌,蕴藏着丰富的次级代谢产物生物合成基因,采用新一代测序技术、对其次级代谢产物的生物合成基因簇进行预测,已成为常用的研究方法[17-18]。前期,本课题组对宁北地区宁夏枸杞根际盐碱土进行了微生物多样性的分离工作,获得了56株放线菌,根据菌株形态和16S rRNA鉴定,获得了一株与Streptomyces alfalfae相似性较高的链霉菌,命名为V-1-3。本研究以发掘宁北地区盐碱地链霉菌的次级代谢产物为目标,结合全基因组测序技术与HPLCQ-TOF MS分析,开展了链霉菌V-1-3次级代谢产物的预测工作。
1 材料与方法
1.1 仪器、材料及试剂
NanoDrop2000超微量分光光度计购自赛默飞世尔科技公司;SangNond SN-300显微镜购自深圳市叁诺西努科技有限公司;Quantus荧光计购自普洛麦格(北京)生物技术有限公司;DYY-6D电泳仪购自北京六一仪器厂;YT-CJ-1ND超净工作台购于北京亚泰科隆仪器有限公司;SQ810C高压蒸汽灭菌锅购自雅马拓科技贸易(上海)有限公司;ZHWY-211C摇床购自上海智城分析仪器制造公司;6530 Q-TOF LC/MS液相-质谱联用仪购自安捷伦科技(中国)有限公司;细菌基因组DNA提取试剂盒购自上海美吉逾华生物医药科技有限公司;SPE-C18微型固相萃取柱购自沃特世科技(上海)有限公司;Zorbax XDB-C18(4.6 mm× 250 mm,5.0 μm) 购自安捷伦科技(中国)有限公司。可溶性淀粉由天津市北辰试剂厂生产;琼脂粉为青岛高科技园海博生物技术有限公司生产;酵母浸膏和牛肉膏为上海博微生物科技有限公司生产;蔗糖为福晨(天津)化学试剂有限公司生产;BR级水解酪蛋白为上海瑶永生物科技有限公司生产;其他化学试剂无特殊说明均为分析纯,由天津市大茂化学试剂厂提供。
1.2 菌种来源
菌株V-1-3分离自宁夏枸杞植物根际盐碱性土壤(东经106°08′,北纬38°38′),菌株保存于北方民族大学化学与化学工程学院,化工技术基础国家民委重点实验室,同时保存于中国微生物菌种保藏管理委员会普通微生物中心(China General Microbiological Culture Collection Center,CGMCC),编号为CGMCC 23478。
1.3 培养基
高氏一号培养基:可溶性淀粉20.0 g,KNO31.0 g,K2HPO40.5 g,MgSO4·7H2O 0.5 g,NaCl 0.5 g和FeSO4·7H2O 0.01 g,琼脂20 g,蒸馏水1 L,pH 7.1~7.5。
本尼特(Bennett)液体培养基:酵母膏1.0 g,牛肉膏1.0 g,水解酪蛋白2.0 g,葡萄糖10.0 g,蒸馏水1 L,pH 7.3。
无铁察氏培养基:NaNO33.0 g,K2HPO41.0 g,MgSO4·7H2O 0.5 g,KCl 0.5 g,蔗糖30.0 g,pH自然。
PBS缓冲液:NaCl 8.0 g,KCl 0.2 g,Na2HPO41.44 g,KH2PO40.24 g,蒸馏水1 L,pH 7.4。
1.4 方法
1.4.1 菌种鉴
菌株V-1-3接种至高氏一号培养基观察培养特征,革兰染色观察菌丝特征和染色特性。菌株DNA的提取按照DNA提取试剂盒的操作流程进行。16S rRNA基因PCR扩增的引物、扩增体系和反应条件参照文献方法[19]。PCR阳性结果由生工生物工程(上海)股份有限公司完成测序。测序结果利用NCBI数据库(https://www.ncbi.nlm.nih.gov/)进行16S rRNA基因相似性的比对,构建菌株V-1-3基于16S rRNA基因的系统发育树,确定菌株的分类地位。
1.4.2 菌株全基因组测序和生物合成基因簇分析
将链霉菌V-1-3接种到高氏一号固体培养基上,28℃培养5 d,灭菌竹签挑取新鲜菌体接种于盛有50 mL Bennett培养基的150 mL三角瓶中,28℃、220 r/min培养48 h后离心收集菌丝体,PBS缓冲液冲洗2~3 次,液氮速冻,于-80℃中保存备用。冻存的菌体按照细菌DNA提取试剂盒的操作流程进行基因组DNA的提取,委托美吉生物科技有限公司进行第二代+第三代(即Illumina Hiseq+PacBio)测序以及相关组装工作,获得菌株基因组完成图。利用antiSMASH v6.0.1 (https://antismash.secondarymetabolites.org)在线工具对菌株V-1-3中的次级代谢产物合成基因簇进行预测[20]。
1.4.3 液-质联用分析
①样品制备:菌株V-1-3于液体无铁察氏培养基中28℃、180 r/min发酵7 d后,5000 r/min离心20 min,取上层清液(10 mL)至微型固相萃取柱,依次使用纯水、50%和95%甲醇水溶液洗脱,得到洗脱液浓缩后,用于液-质联用分析。②色谱条件:乙腈、水为流动相,其中乙腈在30 min内由5%递增至95%;色谱柱:Zorbax XDB-C18;流速为0.8 mL/min,柱温设定为25℃。③质谱条件:高纯氮(液氮)用作雾化和干燥气体;高纯氦作为碰撞气体;采集模式为正和负离子两种模式;毛细管喷雾电压设定为3.5 kV;雾化室温度设定为320℃,雾化气体流速为5.0 L/min;一级和二级质谱扫描范围分别为m/z300~2000和m/z20~2000。Autoscan模式下选择丰度大于105的离子进行二级质谱分析。
2 结果
2.1 菌株形态与菌属鉴定
菌株V-1-3在高氏一号培养基上形成黄色、圆形、较小菌落。菌落表面褶皱,中心呈不平整凸起,四周呈绒毛状,菌落较致密不易挑取,可产生不溶性黄色色素。显微镜下观察,可见细长菌丝,革兰染色阳性。16S rRNA基因比对结果及系统发育树(图1)显示菌株V-1-3隶属于链霉菌,并与Streptomyces alfalfaeXY25T具有100%同源性。
2.2 菌株基因组特征
菌株V-1-3基因组大小为8243417 bp (GenBank号:SAMN20858213),平均(G+C)含量为72.14%,含有7578个基因,编码基因序列总长度7285665 bp,占DNA全长88.38%。在7578个基因中,长度在101~1000 bp的基因有4776个;长度大于1000 bp的基因有2802个。此外,基因组上还检测到了67个tRNA、18个rRNA、以及一些其他特征信息。链霉菌V-1-3的基因功能注释显示有174个为合成酶基因,主要参与聚酮类(polyketide synthases,PKSs)以及非核糖体肽类(nonribosomal peptide synthases,NRPSs)等酶的合成。
2.4 次级代谢产物合成相关基因簇分析
利用antiSMASH(v6.0.1)对菌株V-1-3基因组的次级代谢产物合成基因簇进行预测及分析,检测到33个与次级代谢产物合成相关的基因簇(表1),其中:①6个为萜类合成相关,即clusters 1、8、10、12、16和25,其中clusters 10和25分别与文献报道的Geosmin和Isorenieratene的生物合成基因簇在全长范围内完全相同;②16个为非核糖体肽类合成相关,clusters 2-7、13、15、18、21、23、26、27、28、31和33,其中clusters 4、13、15和23不与其它合成基因邻近,而clusters 2、21、26和28与T1PKS处于上下游,可能编码杂合型化合物;此外,clusters 3和6与trans AT-PKS交合,clusters 5和7与T3PKS交合,clusters 18和27分别与Ladderane和Terpene交合,clusters 31和33与RiPP交合;③4个为单纯聚酮类合成相关,即clusters 9、17、24和30;④1个为非NRPS铁载体类合成相关,即cluster 14;⑤1个为黑色素类合成相关,即cluster 19;⑥1个为吩嗪类合成相关,即cluster 20;⑦1个tRNA依赖性环二肽类合成相关,即cluster 29;⑧2个为翻译后修饰肽类合成相关(RiPP) 即clusters 11和32;⑨1个为嘧啶羧酸类合成相关,即cluster 22。
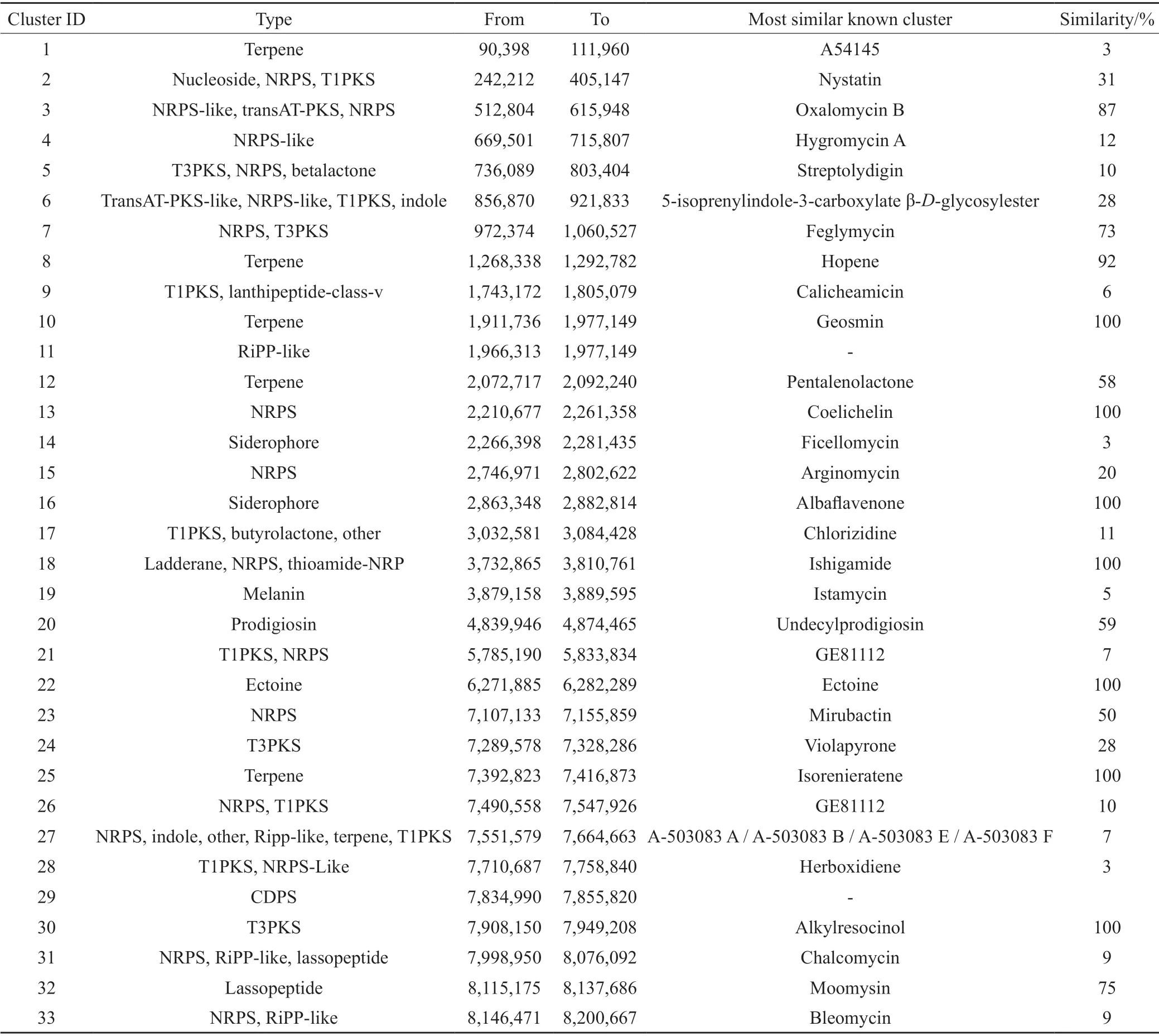
表1 菌株V-1-3次级代谢产物生物合成基因簇Tab.1 Biosynthesis gene clusters in strain V-1-3
相似度筛查发现,有10个和已知生物合成基因簇相似度超过50%,其中clusters 3、7、8、12和20分别与oxalomycin B(87%)、feglymycin(73%)、hopene(92%)、pentalenolactone(58%)以及undecylprodigiosin(59%)具有较高的相似度,而clusters 10,13,18,22和25分别与geosmin、coelichelin、ishigamide、ectoine和isorenieratene的生物合成基因簇相似度高达100%。
2.5 液-质联用分析
根据生物合成基因簇分析结果,本研究进一步采用高效液相色谱-高分辨质谱联用 (HPLC-Q-TOF/MS)方法对链霉菌V-1-3的发酵液进行分析。一级分子离子峰扫描检测出发酵液中的主要化学成分后,利用自动化采集技术,在ESI离子源模式下,对其中丰度高于105离子采集二级质谱,得到样品总离子图。对基因簇中相似度高于50%的潜在化合物的分子离子进行手动提取,结果发现cluster 3可能编码并表达了化合物oxalomycin B[21]。在正负离子模式高分辨质谱中,oxalomycin B(1)的分子离子峰被提取得到,分别为m/z656.3413 [M+H]+和654.3336 [M-H]-,与计算值之间的误差小于5 ppm;cluster 10可能编码了geosmin(2)[22],该分子为易挥发单萜,在水洗样品中有残留,保留时间为8~9 min,准分子离子峰为m/z181.0694 [M-H]-;化合物coelichelin(3)[23]和ishigamide(4)[24]的化学结构在总离子流图中被提取到,且丰度高于105,提示这些结构可能存在于发酵液中,化合物1~4的化学结构和相关信息见图2,二级质谱裂解如图3所示。
3 结论与讨论
宁夏盐碱地生物多样性的研究报道不多,而对其它地区的盐碱地的报道表明盐碱环境下蕴含丰富的微生物资源。贾晓宇等[25]研究新疆湿地盐碱土发现,0~50 cm土层中放线菌总数最多,大部分属于放线菌纲中的3个亚纲:放线菌亚纲(Actinobacteridae)、酸微菌亚纲(Acidimicrobidae)和红色杆菌亚纲(Rubrobacteridae)。阎薇如[26]自河西走廊石羊河流域5种盐碱土壤中共分离出298株线菌,80%为链霉菌属,初步筛选得出有105株放线菌对动物病原菌具有不同程度的抑菌效果。张晋龙[27]以内蒙古赤峰宁城县青山林场土样为材料,分离得到63株盐碱地放线菌,并筛选出7株高活性的可以同时拮抗真菌和细菌的菌株。于洋[28]对杜尔伯特蒙古自治县地区的盐碱化土壤中的放线菌进行分离得到43株链霉菌属放线菌,抗真菌活性初筛得到7株具有广谱抗菌活性的菌株。本研究采用第二代和第三代测序技术,获得了宁夏枸杞根际盐碱土壤来源链霉菌V-1-3的全基因组序列,并利用anti SMASH(v6.0.1)在线工具预测了该菌株的潜在生物合成基因簇,发现其具有33条生物合成基因簇,至少可编码33种化学结构。
HPLC-Q-TOF MS具有快速、高通量、高灵敏度和特异性强的特点,适用于发酵产物中已知化合物的早期识别和去重复[29]。本研究对链霉菌V-1-3的发酵液进行了HPLC-Q-TOF MS分析,并对生物合成基因簇与其相似度高于50%的化学组分进行提取解析,发现链霉菌V-1-3可能编码了化合物oxalomycin B(1)、geosmin(2)、coelichelin(3)和ishigamide(4),对后续菌株发酵产物的分离具有指导意义。本文预测出33条生物合成基因簇,除编码化合物1~4之外,还存在和已知化合物生物合成基因簇相似度不高的基因簇,它们可能编码了新结构类型的次级代谢产物,说明菌株V-1-3具有发现新结构天然产物的潜力。HPLC-Q-TOF MS技术与antiSMASH(v6.0.1)预测相结合解析菌株次级代谢产物的方法为新结构抗生素的发掘提供了新思路。
致谢:感谢中国科学院西北生态环境资源研究院房嬛研究员和陈永欣副研究员在液-质联用测试方面提供的帮助;致谢宁夏石嘴山市委组织部、市科技局、平罗县政府提供的挂职高层次人才科研课题的经费支持,课题名称:平罗县盐碱地生物结皮中微生物、化学多样性研究及其在盐碱地治理中的应用;感谢河北北方学院李小俊副教授、广西医科大学卢覃培博士和广东省科学院动物研究所动物生态与进化研究中心郭敏博士对本文的修改和帮助。
