土壤噬菌体的研究进展及生态功能解析
2022-08-02高瑞川李芳柏陈冠虹方利平
高瑞川,胡 敏,李芳柏,陈冠虹,方利平
(1 中国科学院 广州地球化学研究所, 广东 广州 510640; 2 广东省科学院 生态环境与土壤研究所,广东 广州 510650; 3 中国科学院大学, 北京 100049)
病毒在地表环境中无处不在,是世界上数量最多的生物实体。噬菌体是感染细菌、古菌等原核生物的病毒统称,由于土壤中原核微生物数量远高于真核生物,噬菌体被视为土壤中主要的病毒类群。有研究推测,全球病毒粒子的数量为4.80×1031,其中,在土壤中占比为10%、海洋水体中占比为2.7%[1]。相比于海洋等水生系统,土壤的异质性更高,其组成在空间和时间尺度上差异很大。因此,尽管土壤中噬菌体的丰度非常高,但对其生态功能以及作用机理的研究相对滞后[2-4]。近年来,以宏病毒组结合高通量测序技术分析土壤噬菌体丰度变化、群落组成、功能注释的研究逐渐增多,对土壤生态系统中的病毒,尤其是噬菌体的生态功能进行了一些探索,逐步揭开土壤噬菌体的神秘面纱。本文基于当前土壤噬菌体的研究现状,综述了土壤噬菌体的分类和研究方法,揭示土壤噬菌体对宿主群落结构、生命元素循环及重金属转化过程的调控机制,阐明土壤噬菌体的生态功能及环境意义,并对土壤噬菌体未来研究重点进行了展望。
1 噬菌体分类及研究方法
1.1 噬菌体分类
噬菌体是一类个体微小、结构简单,能够感染细菌、古菌等原核生物的病毒,其遗传物质包裹在被称为衣壳的蛋白质结构中。噬菌体所携带的遗传物质主要分为双链DNA(dsDNA)、单链DNA(ssDNA)、双链 RNA(dsRNA)和单链RNA(ssRNA) 4类。土壤环境中以携带双链DNA的噬菌体为主[1,5]。
噬菌体形态各异,包括球状、丝状、杆状、棒状、水滴状和多面体等,如图1所示[6]。其中,有尾噬菌体有一条附着在衣壳上、可参与宿主识别和感染的蛋白质尾巴[7-8]。有尾噬菌体占已知噬菌体的90%以上,根据其尾部的形态特征,可再细分为肌尾、长尾和短尾噬菌体。有尾噬菌体可侵染的宿主十分广泛,涉及厚壁菌门Firmicutes、拟杆菌门Bacteroidetes、放线菌门Actinobacteria、蓝细菌门Cyanobacteria、变形菌门Proteobacteria、广古菌门Euryarchaeota等多个门类[9]。
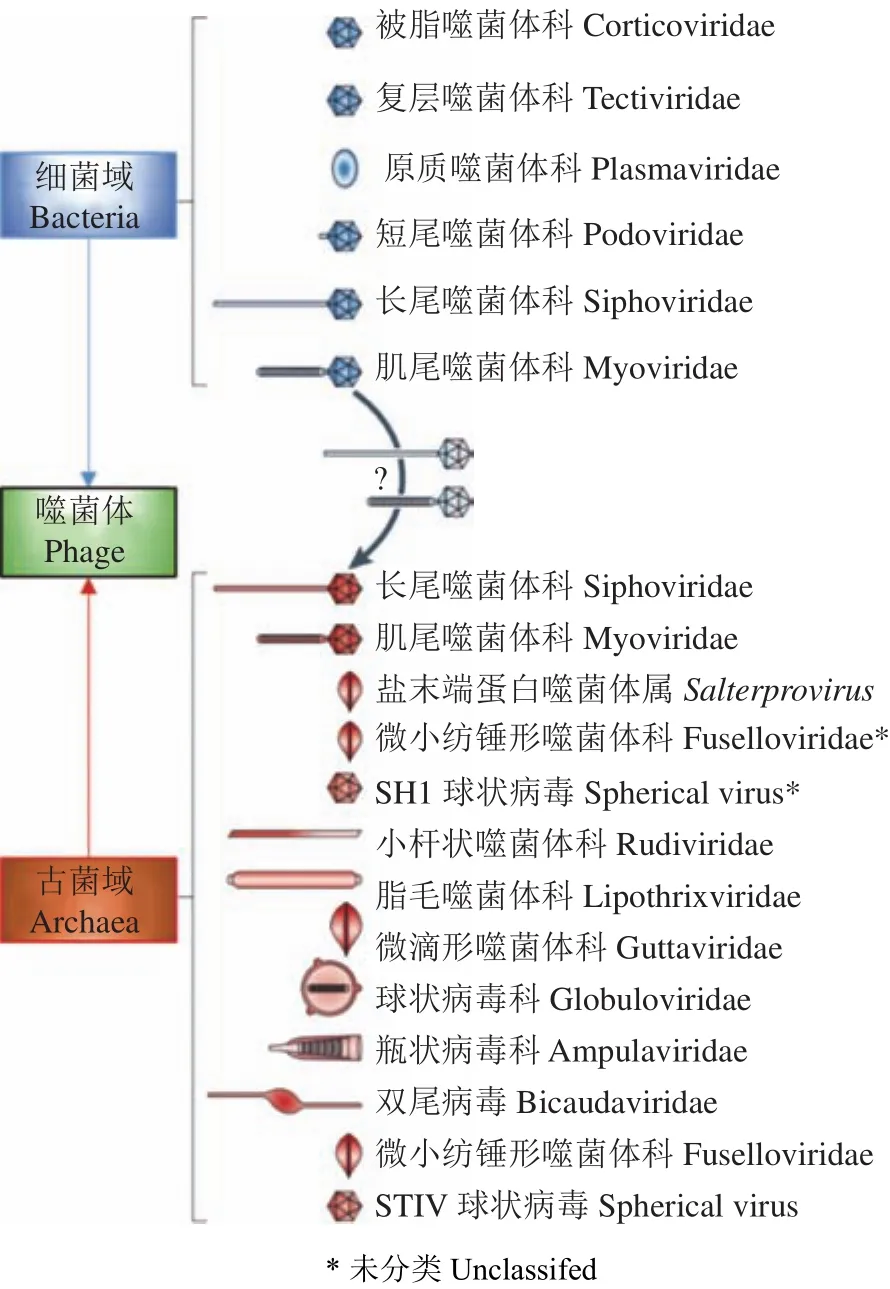
图1 侵染细菌域和古菌域的dsDNA噬菌体分类及形态示意图Fig.1 Classification and morphological diagrams of dsDNA phages infecting bacterial and archaeal domains
由于噬菌体自身缺乏代谢功能,不能独立繁殖,与合适的宿主细胞接触并侵染是其繁殖的唯一途径[10]。噬菌体生命周期根据其侵染周期作用于宿主细胞的方式,主要分为裂解循环(常见于烈性噬菌体和温和噬菌体)和溶原循环(常见于温和噬菌体)两大类:裂解循环是指噬菌体侵染宿主细胞后破坏宿主原有代谢功能和结构,进行大量子代噬菌体的增殖、装配,最终使宿主细胞裂解的过程;溶原循环是指噬菌体侵染宿主菌后并不立即进行增殖,而是将其核酸整合至宿主菌基因组中并跟随宿主核酸的复制而复制,最终随细胞的分裂进行繁殖的过程[11]。随后,又发现了慢性循环和假溶原循环[12-13]。慢性循环常见于丝状噬菌体,可以在宿主细胞内不断复制并释放,却不会裂解宿主细胞[14]。假溶原循环的噬菌体在侵染宿主细胞后,自身既不复制,也不裂解宿主,处于“休眠”状态[15-16];这种现象常见于营养受限,宿主细胞难以进行DNA复制或蛋白质合成的环境中。
土壤的异质性限制了土壤噬菌体的分布和活动。pH、含水量、土壤类型、细菌丰度和群落组成结构等非生物和生物因素都会对噬菌体群落结构、丰度等造成不同程度的影响,进而影响其生态功能。在一定的诱导条件下,烈性噬菌体与温和型噬菌体可发生转换。有研究表明,前噬菌体(Prophage)仍然保留了启动裂解循环的能力[17]。
1.2 噬菌体的研究方法
受限于大部分环境微生物的不可培养性,依赖于宿主细胞进行繁殖的病毒多样性研究受到了较大的阻碍。由于没有通用的标记基因,早期对病毒群落及功能的研究常使用特定病毒家族中高度保守的标记基因设计简并性引物,以评估其系统发育关系。此外,随机扩增多态性DNA标记(RAPD)[18]作为对未知序列基因组进行多态性分析的技术,被学者们应用于土壤中病毒多样性的研究中。近年来,使用宏基因组、宏病毒组等组学方法研究土壤噬菌体逐渐成为热门。这些方法可以大量获得相关种群的遗传信息,且不受病毒培养的限制。土壤宏基因组的研究依赖于从整个宏基因组数据中鉴定出病毒序列[19-21]。但宏基因组数据中通常包含高比例非病毒序列,这些序列往往在下游分析中需要去除。研究表明,对富集后的噬菌体进行宏病毒组分析,获得的目标序列多样性远高于直接对土壤环境DNA进行宏基因组分析[22]。因此,对噬菌体进行洗脱、纯化和富集成为了土壤噬菌体研究的重要一环[23]。总的来说,噬菌体的研究方法主要包括洗脱、纯化、浓缩、核酸提取、数据分析等[24],具体流程如图2所示。
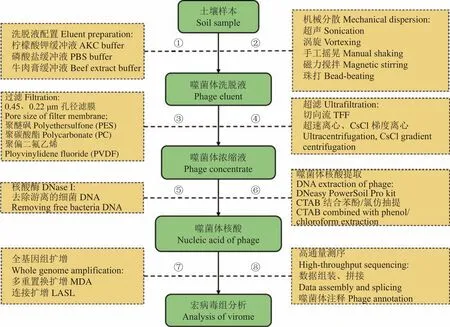
图2 土壤噬菌体洗脱、富集、核酸提取流程图Fig.2 Flowchart of elution, enrichment and nucleic acid extraction of soil phages
1.2.1 噬菌体的分离、富集与核酸提取 如何高效地洗脱吸附于土壤颗粒表面的噬菌体,是对土壤噬菌体进行分析的第1步。Göller等[25]对镁盐缓冲液、柠檬酸钾缓冲液、10%的牛肉膏、牛肉膏改良的磷酸盐缓冲液等4种病毒洗脱液的洗脱效率进行了比较。有研究表明,缓冲液中盐成分的主要作用是调节pH,作为病毒颗粒的稳定剂[26]。牛肉浸膏中的蛋白质成分提供了噬菌体的结合位点,使噬菌体更容易从土壤颗粒的吸附中剥离。
切向流过滤法(Tangential-flow filtration,TFF)和聚乙二醇沉淀法 (Polyethylene glycol,PEG)是从洗脱液中纯化、富集噬菌体的常用方法[24,27-28]。TFF是一种液体流动方向与过滤方向垂直的方法,通过不断地回流过滤,达到去除细菌等杂质、截留和浓缩病毒粒子的目的。PEG则是通过破坏噬菌体外壳蛋白质分子表面的水化层使其发生沉淀的方法。脱氧核糖核酸酶(DNase I)常用于去除浓缩液中的游离核酸以降低原核生物的核酸干扰。通过选择各类核酸提取试剂盒[29-30]以及甲酰胺或CTAB结合苯酚/氯仿抽提法[27]可以完成噬菌体核酸提取。
获得的噬菌体核酸浓度若未达到文库构建的要求,则还需要对其进行随机扩增。常见的随机扩增技术包括多重置换扩增技术[31]和连接扩增技术[32]等。值得注意的是,这些扩增技术的扩增产物通常会出现偏好[33],并不能有效地还原土壤中真实的噬菌体群落组成。因此,如何不依靠扩增手段获得足量噬菌体核酸被认为是前处理步骤中的一个研究重点。
1.2.2 土壤宏病毒组的分析方法 第2代测序技术是目前进行宏病毒组研究的主要测序技术[34-35]。面对测序下机后获得的海量短序列,分析过程主要包括数据质控、短序列组装、病毒序列鉴定、序列注释、相对丰度计算等内容。如何从海量数据中成功鉴定出病毒序列是后续解析病毒信息的关键。Chen等[31]利用宏病毒组学的方法研究了病毒群落与抗生素耐药基因的关系,测序下机后的原始数据首先使用Trimmomatic[36]去除Illumina平台的FastQC序列中的接头,对原始双端序列进行低质量过滤;再使用Bowtie2[37]软件与细菌、核糖体数据库比对去掉宿主序列,使用Megahit[38]对宏病毒组数据中剩余的Clean reads进行序列组装;利用VIBRANT[39]鉴定病毒序列,采用CD-HIT[40]获得非冗余病毒序列集,保留长度10 kb以上的序列进行下游鉴定及注释;最后,以95%相似度、80%最小覆盖度为筛选条件进行聚类,获得病毒操作分类单元(vOTUs),并进行后续的病毒物种分类、病毒功能注释等工作。
此外,ViromeQC[41]、Kraken2[42]、VirSorter[43]、Prodigal[44]、DRAMv[45]等软件也是近年来各国学者开发并用于环境病毒宏基因组数据解析的软件。ViromeQC是评估非病毒序列污染程度的软件,它可以基于3种微生物分子标记的丰度(SSU-rRNA、LSU-rRNA及31个原核单拷贝基因标记)对病毒组整体序列给出病毒富集得分(Enrichment score);Kraken2是对有效序列进行物种注释预测的软件,其数据库包括古菌、细菌、真菌和病毒,其中,病毒参考序列主要来源于 RefSeq complete viral genomes/proteins,基于k-mer精确比对,并采用LCA法鉴定物种;VirSorter是DNA和RNA病毒的识别工具,常用以识别宏基因组数据拼接后的病毒序列;Prodigal是对细菌和古生菌基因组进行蛋白编码基因预测的软件,其缩写源于 Prokaryotic dynamic programming genefinding algorithm,表示原核生物基因预测的动态规划算法;DRAMv是 DRAM的病毒模式,它建立了识别病毒编码的辅助代谢基因(Auxiliary metabolic genes, AMG)的规则;DRAMv可与Prodigal联用,首先使用Prodigal鉴定所获得病毒序列的开放阅读框 (ORF),然后对获取的ORF集合进行功能注释。
Roux等[46]发布了“未培养病毒基因组(Uncultured virus genomes,UViGs)的最少信息标准” (图3),为学术界提供对病毒进行宏基因/病毒组分析的参考标准。该标准包括UViGs的回收、病毒序列的鉴定、组装质量评估、分析与注释、分类和宿主预测等多方面。采用该标准将有助于改善公共数据库中非培养病毒组的报告,进而有助于全球病毒领域的系统探索。
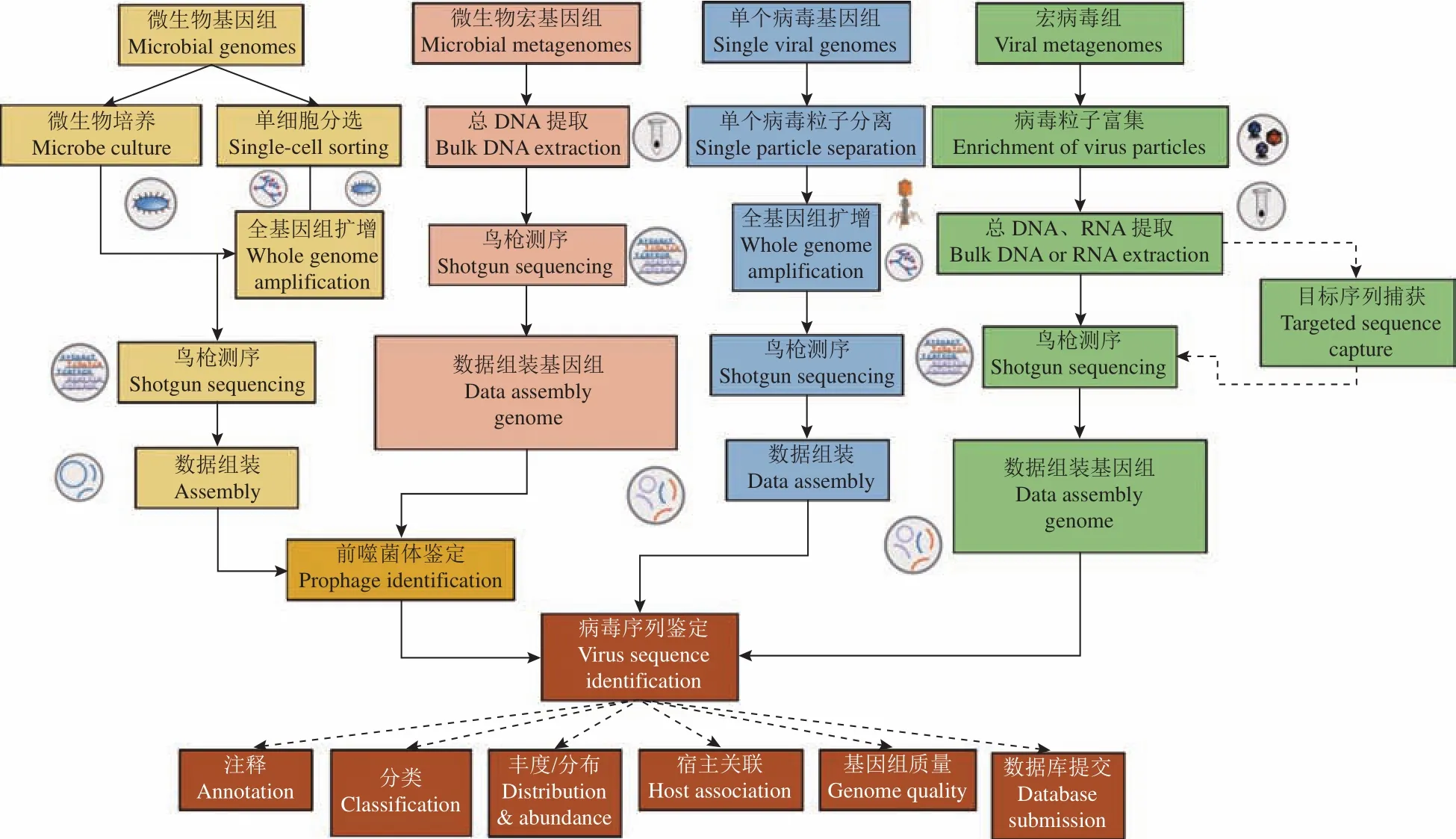
图3 未培养病毒基因组的识别Fig.3 Identification of uncultured virus genomes (UViGs)
2 土壤噬菌体的生态功能解析
关于噬菌体在水体环境中的研究已开展多年。研究发现,海洋中病毒颗粒(VLPs)的数目与丰度都十分惊人,且与生态系统中的微生物死亡关联密切[47-48]。据估计,海洋水体中20%~40%的细菌种群都会受到噬菌体裂解的影响。除水体系统以外,土壤[49]、哺乳动物胃肠道[50]、极端环境(沙漠、极地[19])、甚至是古代样品(木乃伊[51]等)环境中,病毒、噬菌体都随处可见。因此,噬菌体的感染无处不在,影响了微生物群落结构、物种进化以及细菌与其他生物体之间的相互作用。
尽管土壤噬菌体数量巨大,但由于提取难度大,对土壤噬菌体的认知尚刚刚起步。研究表明,土壤噬菌体的生态功能包括自上而下调节微生物群落结构、侵染宿主微生物导致其裂解、加速生命元素循环、加速微生物群落更替以保持其活力、促进微生物间的基因水平转移[52]、携带辅助代谢基因辅助宿主菌生长等[53]。
针对烈性噬菌体的研究发现,噬菌体群落的变化与其对应宿主的物种丰度密切相关,且能对宿主丰度变化产生迅速、积极的响应[54-55]。Chevallereau等[56]和Thingstad[57]研究表明,烈性噬菌体受宿主群落密度的影响,符合“杀死优胜者(Kill-the-winner,KtW)”模型,抑制、裂解生态系统中作为主导的一些细菌群落,从而使丰度较低的群落占比提高。这种调控主要是通过降低优势物种的增殖从而保持细菌群落的多样性。该模型下,温和噬菌体被认为与宿主更多的是产生互利共生行为,因此对细菌种群动态的影响有限。近年来也有研究发现,温和噬菌体可以将自身基因整合至宿主细胞中,放弃快速增殖,避免与其他病毒或噬菌体竞争,最终产生了随着宿主细菌丰度增加,病毒与细菌比例显著下降的现象,噬菌体的这一生长过程符合“搭乘优胜者(Piggyback-the-winner, PtW)”模型[12]。在细菌快速生长期间,病毒与宿主细菌整合获利更多,同时获得了更好的生长条件,是一种“聪明”的寄生行为。总的来说,KtW和PtW 模型并不冲突,应将其结合以充分了解噬菌体-宿主之间的相互作用,进而发掘噬菌体在环境中的生态学意义[58]。
噬菌体对其宿主微生物群落结构的控制,会进一步影响土壤系统的生态功能,如图4所示[59]。当宿主细菌群落介导生态系统中生命元素循环时,这些细菌的丰度、群落结构、功能和代谢将受到噬菌体的裂解影响,最终影响土壤中的养分周转;当宿主为重金属转化相关功能菌时,噬菌体的裂解将影响功能群落相关基因的整体表达,从而调控土壤的解毒功能[60]。此外,土壤生态功能的改变也会对微生物群落结构产生反馈作用。以噬菌体疗法进行病害防治为例,噬菌体通过裂解宿主病原菌,减少土传病害的传播,促进作物的生长[61];而作物生长产生的大量根际热区,如植物根系、真菌菌丝、蚯蚓的影响区域,有利于营养、水分的汇集,从而产生微生物的活动热点,为噬菌体与宿主进行基因交流、转导提供条件,最终形成噬菌体-宿主共同进化的发育模式[62]。
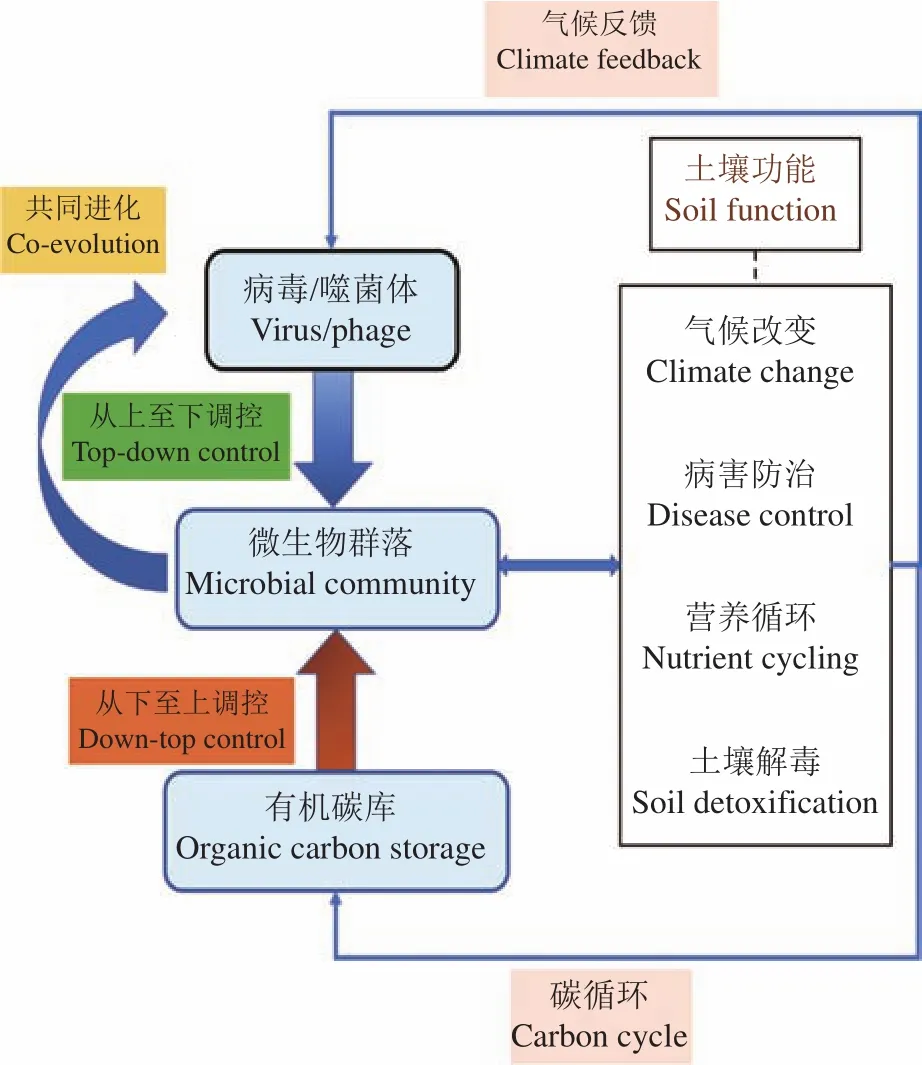
图4 噬菌体调控土壤微生物概念模型Fig.4 The conceptual model for regulating soil microbes by phages
2.1 影响生命元素循环
土壤微生物驱动着全球生命元素循环。土壤有机质是最大的陆地有机碳、氮储库,也是其他营养元素的主要来源。活跃的土壤微生物通过代谢功能推动土壤有机质周转,死亡的土壤微生物细胞内残余的化学成分不断积累并最终转化为土壤有机质,增加土壤中的有机质库存[63]。因此,噬菌体对微生物宿主的裂解是影响生命元素循环的重要推动力。噬菌体裂解微生物群落,释放出大量碳、氮、磷、硫等生命元素,并最终被其他细菌或植物所利用的循环过程被称为“病毒路径 (Viral shunt) ”[1,64-65]。这种裂解机制与外源性捕食者(如原生动物等)的捕食机制不同,外源性捕食者会将细胞中的营养物质带入更高营养级,从而降低营养物质的周转速率;噬菌体使细胞裂解后,促进了营养元素的累积、循环和转化,因此,有学者认为从元素循环的角度,噬菌体的裂解效应比原生动物等捕食者的捕食更加关键[60]。
土壤生态系统中,病毒路径影响生命元素地化循环的模型如图5所示。噬菌体群落对特定宿主专性裂解,细胞成分(细胞膜)、含氮化合物(氨基酸、葡萄糖)和各种胞外产物(EPS、酶、糖蛋白)被重新释放回土壤环境中,并作为有机质储存;子代噬菌体相比于宿主细菌富含氮、磷成分,会导致化学计量学上的差异,细菌细胞中有大约 10%的碳、15%的氮和 33%的磷营养转变成病毒颗粒成分,难以被其他生物利用,间接影响土壤系统中的养分循环[4];噬菌体裂解促进了微生物群落的更替,使大部分细菌都处于活跃的状态,代谢功能旺盛,有利于土壤功能群落长期保持活力并最终推动土壤有机质的周转。
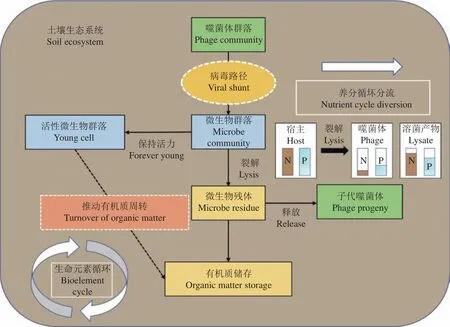
图5 病毒路径影响土壤生态系统示意图Fig.5 The diagram of viral shunt influencing soil ecosystem
Emerson等[19]对病毒如何影响永久冻土层中的微生物生态学和碳代谢过程进行了研究,结果表明病毒感染会不同程度地影响微生物对气候变化的反应;病毒编码的糖苷水解酶包括具有功能活性的甘露聚糖内切酶,病毒影响了该生态系统中复杂的碳降解,且这些病毒的丰度被视为甲烷动力学的重要预测因子。说明病毒可能会影响气候关键型土壤的生态系统功能,并对土壤碳循环具有多种潜在的贡献。Wei等[66]通过在灭菌土壤中接种不同浓度的土壤悬浊液,培养6周后,以CO2排放量以及16S rRNA和g23基因拷贝数作为指标,判断T4型噬菌体对土壤有机碳矿化的影响效应,结果表明土壤有机碳矿化受细菌种群大小的调节,而细菌种群可能受到了噬菌体自上而下的控制;通过杀死宿主细菌,减少了土壤中CO2的产生,进而降低了土壤有机碳的矿化。
除碳元素以外,生态系统中的噬菌体也可能在氮、硫、磷等生命元素循环中发挥重要作用。Braga等[67]研究了不同组合的噬菌体群落对土壤细菌群落和有效态氮的影响,并尝试量化噬菌体驱动的土壤细菌群落结构变化和功能表达,通过16S rRNA扩增子基因测序评估细菌群落的变化,比较活性噬菌体和高压灭活噬菌体的接种效果,结果表明噬菌体具有调节土壤功能的作用,噬菌体改变了土壤细菌群落结构和多样性,接种活性噬菌体有助于提高土壤铵浓度。Williamson等[59]提出,根际噬菌体对豆科植物-根瘤菌共生的影响可能会影响土壤氮含量。感染根瘤菌的噬菌体是大多数土壤的原生物种,这些噬菌体与宿主的相互作用决定了土壤中根瘤菌种群能力的强弱,包括有效固氮根瘤菌菌株的生存或死亡、结瘤的成功与否、以及最终的共生有效性和固氮率。Gao等[68]对不同深度尾矿样品的病毒群落多样性及其功能角色进行了研究,结果发现尾矿深度变化强烈影响了病毒群落的结构、多样性以及生活方式,深层尾矿病毒携带了同化性硫酸盐还原基因,揭示了病毒在尾矿硫循环中的重要作用。Kieft等[69]对噬菌体的硫辅助代谢生态学开展了相关研究,结果表明硫代谢微生物被噬菌体感染后,这些噬菌体可以编码辅助代谢基因(AMGs)以改变宿主细胞内的硫代谢过程;通过分析AMGs的丰度和表达等数据显示,噬菌体对淡水湖泊和海洋中硫、硫代硫酸盐的氧化有显著贡献,并对水热环境中硫浓度的变化有敏感的反应。
虽然噬菌体被认为可以改变微生物群落结构和功能组成[70],但这种影响的程度在不同土壤环境中有着较大的差异[71]。进一步研究生物和非生物因素如何影响病毒对微生物群落的调控作用,将有利于阐释噬菌体调控功能微生物群落的原理和机制。
2.2 影响重(类)金属转化
噬菌体对重金属的转化、解毒过程有着重要的影响[72]。噬菌体可以通过与微生物宿主间的水平基因转移 (Horizontal gene transfer, HGT)调控细菌对重金属的代谢过程以适应环境的变化[16]。Huang等[30]以不同程度铬污染的土壤为样本,研究了噬菌体与宿主微生物群落的共进化动态,结果显示铬污染对噬菌体以及细菌群落的丰度、多样性和组成均产生了不利影响;在环境压力的胁迫下,溶原性噬菌体的占比与铬污染水平呈正相关,噬菌体与细菌的共生作用增强;高铬污染土壤中,携带重金属抗性基因的噬菌体丰度增加,并显著地促进了重金属抗性基因的转导与传播,辅助调节微生物的解毒过程。 Tang等[29]在砷污染土壤中收集了耐砷细菌胞内的噬菌体,从其基因组中发现了砷抗性基因ArsC和砷甲基化基因arsM,且噬菌体砷功能基因的相对丰度与对应的土壤细菌群落中这些基因的丰度呈正相关;将富集后的噬菌体加入土壤中,其ArsC和arsM基因的相对丰度比对照土壤分别提高了122和575倍,证明噬菌体可以进行砷氧化还原过程的介导,协助砷还原,并在甲基化的功能微生物群落中发挥作用,改变土壤中砷的形态,调节土壤砷胁迫的状态。
生态系统中的噬菌体对重(类)金属循环具有潜在的调控作用。以砷的生物地球化学循环为例,它是由砷代谢微生物介导的一系列氧化还原反应组成,其微生物代谢机制的研究较为完善,主要包括As(V)还原、As(III)氧化和砷甲基化。从噬菌体生态学角度出发,噬菌体对砷污染土壤解毒功能的调控主要是通过以砷功能菌为宿主的各类噬菌体发生侵染,如烈性噬菌体裂解功能微生物细胞以控制微生物群落结构;温和噬菌体携带功能微生物宿主抗性、解毒基因进行水平基因转导,进而提高土壤微生物群落整体解毒水平,完成对土壤解毒功能的调控 (图6)[11]。
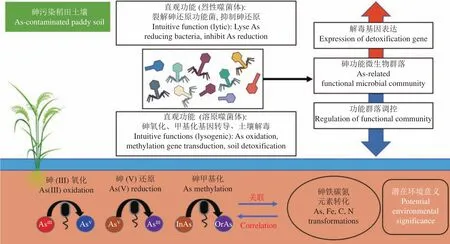
图6 噬菌体对土壤解毒功能调控示意图Fig.6 The diagram of regulating soil detoxification function by phage
实际上,如果将视野扩大到整个生态系统,以其他功能群落为宿主的噬菌体同样可能对土壤解毒功能起着潜在的调控作用。铁与砷的生物地球化学循环耦合关系最为密切,砷铁矿的转化是影响砷的生物可利用性的一个重要因子,铁在氧化条件下生成的Fe(III)矿物能够显著地固定砷,进而降低砷的生物可利用性。噬菌体与铁的关联同样十分紧密,如“铁”洛伊木马(Ferrojan horse)假说[73-74]所提出,部分有尾噬菌体可以利用其宿主的铁摄取机制,在噬菌体尾部复合铁络合物,吸引宿主细胞膜表面的铁受体与其强烈结合,提供噬菌体遗传物质进入细胞质的途径。噬菌体也有可能附着在铁矿物上,并随着矿物的形成、转化、溶解及一系列氧化还原反应而发生转移,改变噬菌体在土壤中的分布,加强与细菌群落间的接触与关联。Gao等[75]针对我国酸性矿山环境中病毒多样性和生态功能的研究结果表明,铁离子与病毒多样性和生态功能多样性呈正相关。因此,在进行噬菌体影响砷生物地球化学循环的研究中,影响铁氧化还原等相关功能的噬菌体对重(类)金属循环同样具有潜在的调控作用;除直观的研究目标微生物群落与对应噬菌体的关系外,与砷的地化循环过程耦合的其他元素(如铁、碳、氮、硫)相关的噬菌体发挥了何种作用也值得关注。
学术界对土壤噬菌体的生态功能和环境意义的认知也在不断发展。多年来,对噬菌体的研究关注重心放在了噬菌体-宿主的拮抗作用上,而最新的试验模型和理论进展表明,这种针对宿主变化、环境因素影响的感染策略的反馈是动态且持续的。Correa等[76]指出,噬菌体对宿主进行感染应当看作噬菌体与宿主不断对抗、互助等关联过程中的一个小单元。研究噬菌体与微生物的宿主关系,应扩大关注的周期,从更宏观的角度看待长期感染对种群、生态系统的作用。
3 展望
21世纪以来对海洋病毒生态学的重视,噬菌体对海洋生态系统的重要作用被逐步认知,推动了土壤噬菌体研究的开启。土壤噬菌体由于个体微小且无完整的细胞结构,在以往的研究中经常被忽视,土壤病毒领域的研究起步落后于海洋病毒领域近20年。土壤是一个固相、液相、气相组成的高度异质性的复合生态系统,这种空间结构创造了丰富的微生境,从而为细菌等微生物的发育提供了广泛的生态位。除了由土壤结构引起的空间异质性以外,农业活动如耕作产生的植物根际也被视作微生物的活动热点。对土壤噬菌体生态功能及环境意义的研究有待进一步深化,今后的研究重点可以关注如下几个方面:
1) 通用高效的土壤噬菌体提取方法。不同于海洋水生系统,不同类型的土壤理化性质差异巨大,目前仍缺乏针对不同类型土壤的通用高效的噬菌体提取方法。有效提取土壤噬菌体并获得足够量噬菌体核酸是深入研究的前提。
2) 裂解性噬菌体与温和噬菌体的共同关注。从土壤中洗脱的噬菌体绝大部分为裂解性噬菌体,以洗脱作为前处理工序的研究方法或弱化了温和型噬菌体的研究;而提取DNA后通过宏基因组识别前噬菌体则主要是针对温和噬菌体,且测序数据量要求较大。今后需要共同关注土壤中各类噬菌体的生态功能。
3) 土壤噬菌体与宿主之间的耦合关系。揭示环境因子、噬菌体群落、宿主群落三者之间的内在联系,从病毒生态学角度揭示噬菌体感染土壤微生物、原生动物的环境与生态效益,有助于深化对土壤噬菌体生态功能的认知,寻找通过土壤噬菌体调控生态系统功能的有效途径。
4) 噬菌体疗法。噬菌体疗法已被应用于如青枯病等植物病害的防治中。今后应重点研究噬菌体调控土壤微生物群落进而调控生命元素循环与污染物的脱毒转化,推动该方法在土壤污染治理、面源污染控制、土壤质量提升等方面的广泛应用。
