构建“免疫相关lncRNA基因对”模型预测未发生远处转移的原位肝癌预后及药物敏感性
2022-08-02李睿哲薛军帅杨龙山董兆如洪建国李涛王东旭
李睿哲,薛军帅,杨龙山,董兆如,洪建国,李涛,王东旭
(山东大学齐鲁医院 肝胆外科,山东 济南 250012)
原发性肝癌(primary liver cancer,PLC)排在最常见恶性肿瘤的第六位,肿瘤致死病因的第三位。尽管由于疫苗以及抗病毒药物的有效应用,东南亚国家的肝癌发病呈现下降趋势,但在欧洲、北美地区则呈现上升趋势,因此全球肝癌的发病形势依然严峻,寻求对肝癌的全方位防治尤为重要[1]。手术切除是肝癌获得根治的主要手段,然而初始可手术切除的肿瘤仅占到了15%~30%,多数患者在诊断后已处于中晚期,无法行一期根治性切除[2]。因此,系统治疗在肝癌的整体治疗中举足轻重。随着近几年来靶向、免疫治疗的进展,肝癌的系统治疗药物逐渐增多[3]。而基于这些药物的疗效,患者生存期不断延长的同时,晚期肝癌降期转化切除比例逐渐增多[4]。然而,能够在系统性药物的帮助下转化降期,进而获得根治性手术的肝癌主要局限在肝内,有明确的肝外扩散患者则希望渺然。同时,当前研究表明,肝癌转移离开肝脏后定植于机体其他器官组织,受限于不同的生长环境,肿瘤内的微环境与原位相比差异明显,药物治疗的敏感性也有所不同[5-6]。因此,针对未发生远处转移的肝癌,特异筛选出该类样本,探索其内微环境特点并发现能够与其性质相关的特异生物标志物有利于进一步提高治疗效果。
长链非编码RNA(lncRNAs)是一种长度超过200 bp的非编码RNA并广泛参与了机体细胞增殖、分化、调控等诸多生理过程[7-9]。当前研究表明lncRNAs与许多疾病的发生发展有关并且特定的lncRNAs对相关疾病的疾病特征有一定的预测价值,如肿瘤、心血管疾病、内分泌疾病等[8-9]。此外,lncRNAs可以作为在包括肝癌在内的多癌症中的良好预后生物标志物[10]。
1 资料和方法
1.1 研究数据纳入及整理
转录组数据准备:从XENA数据库(https://xenabrowser.net/datapages/)下载了TCGA-LIHC队列369例患者共421份样本的表达量数据,其中371份为肝细胞肝癌组织样本,50 份为正常组织样本,同时下载了GTEx数据库中的110 份正常肝组织样本。数据类型为TPM数据。LncRNA名称数据的提取和原始矩阵的所有RNA的名称匹配全部对照的是第23版本的GTF基因注释文件。临床数据准备:下载TCGA-LIHC队列和GETx数据库中肝脏的临床数据,去除有明确的转移M1或者转移状态不明确Mx的患者的表达量数据,保留未发生远处转移的M0患者的表达量数据进行后续分析。
1.2 免疫lncRNA“共表达基因对”的构建
提取免疫相关lncRNA:免疫基因集获取于ImmPort数据库(http://www.immport.org);通过免疫基因与lncRNA共表达分析鉴定出免疫相关性lncRNA;通过“limma”数据包提取正常组织和肿瘤组织中差异表达的lncRNA,过滤条件设定为FDR<0.05以及|logFC|>1。对获取的所有的lncRNA进行两者之间的表达情况比较,表达上升的基因对标记为“1”,反之为“0”。
1.3 “免疫lncRNA基因对”预后模型建立
获得的免疫lncRNA基因对与患者的生存数据整合。单因素Cox回归筛选出具有预后相关的候选基因对,P<0.05认为差异具有统计学意义。对单因素Cox回归筛选出的免疫LncRNA基因对运用LASSO回归模型进行进一步压缩筛选,并进行交叉验证。多因素Cox回归模型分析鉴定出的免疫lncRNA基因与患者的总生存OS关系。患者风险值计算公式为:
Riskscore=h0(t)×expCoefficient(Genei)×Exp(Genei)。h0(t)为基准风险函数,Coefficient(Genei)为多因素Cox回归分析得到的第i个基因的回归系数,Exp(Genei)为第i个基因的表达量。
1.4 预后风险模型(IRLP)与患者生存及临床因素的相关性
利用ROC受试者曲线绘制患者风险值对生存的预测图形,确定最优Cutoff值,根据Cutoff值区分患者为高风险组(H组)以及低分险组(L组)。同时,根据患者生存时间分为1、2、3 年生存率,分别计算AUC。根据患者风险分组情况对患者生存情况进行比较,并进行Log-rank检验。结合临床因素及患者风险值进行独立预后分析验证。
1.5 免疫浸润分析
免疫浸润分析数据库TIMER(http://timer.cistrome.org)下载肿瘤免疫细胞浸润数据。根据免疫相关基因的表达,利用多软件计算样本中的免疫细胞浸润丰度。利用Wilcoxon检验计算不同风险组免疫细胞的差异,P<0.05 认为差异具有统计学意义。相关性分析鉴定样本风险值与免疫细胞浸润的相关性。计算在高低风险组间免疫检查点相关基因的表达差异。
1.6 药物敏感性分析
pRRophetic算法根据GDSC细胞系表达谱和TCGA基因表达谱构建岭回归模型预测药物IC50。选取了在肝癌做过三期临床试验的相关药物索拉非尼(Sorafenib)、顺铂(Cisplatin)、阿昔替尼(Axitinib)、舒尼替尼(Sunitinib)、埃罗替尼(Erlotinib)、拉帕替尼(Lapatinib)并利用“pRRopheticPredict”工具包评估肝癌治疗中多种药物在不同风险组的敏感性。
2 结果
2.1 研究纳入样本及筛选流程
本研究的数据筛选流程在图1A 中展示。从XENA数据库(https://xenabrowser.net/datapages/)下载了TCGA-LIHC队列的421 份样本的表达量数据,其中371 份为肝细胞肝癌组织样本,50 份为正常组织样本,同时下载了GTEx数据库中的110份正常肝组织样本。数据类型为TPM数据。lncRNA名称数据的提取和原始矩阵的所有RNA的名称匹配全部对照的是第23版本的GTF基因注释文件。去除平均表达量小于0.5 的基因,肝癌样本中去除有明确的转移M1或者转移状态不明确Mx的患者的肝癌样本共106份,保留未发生远处转移的M0患者的肝癌样本265份进行后续分析。
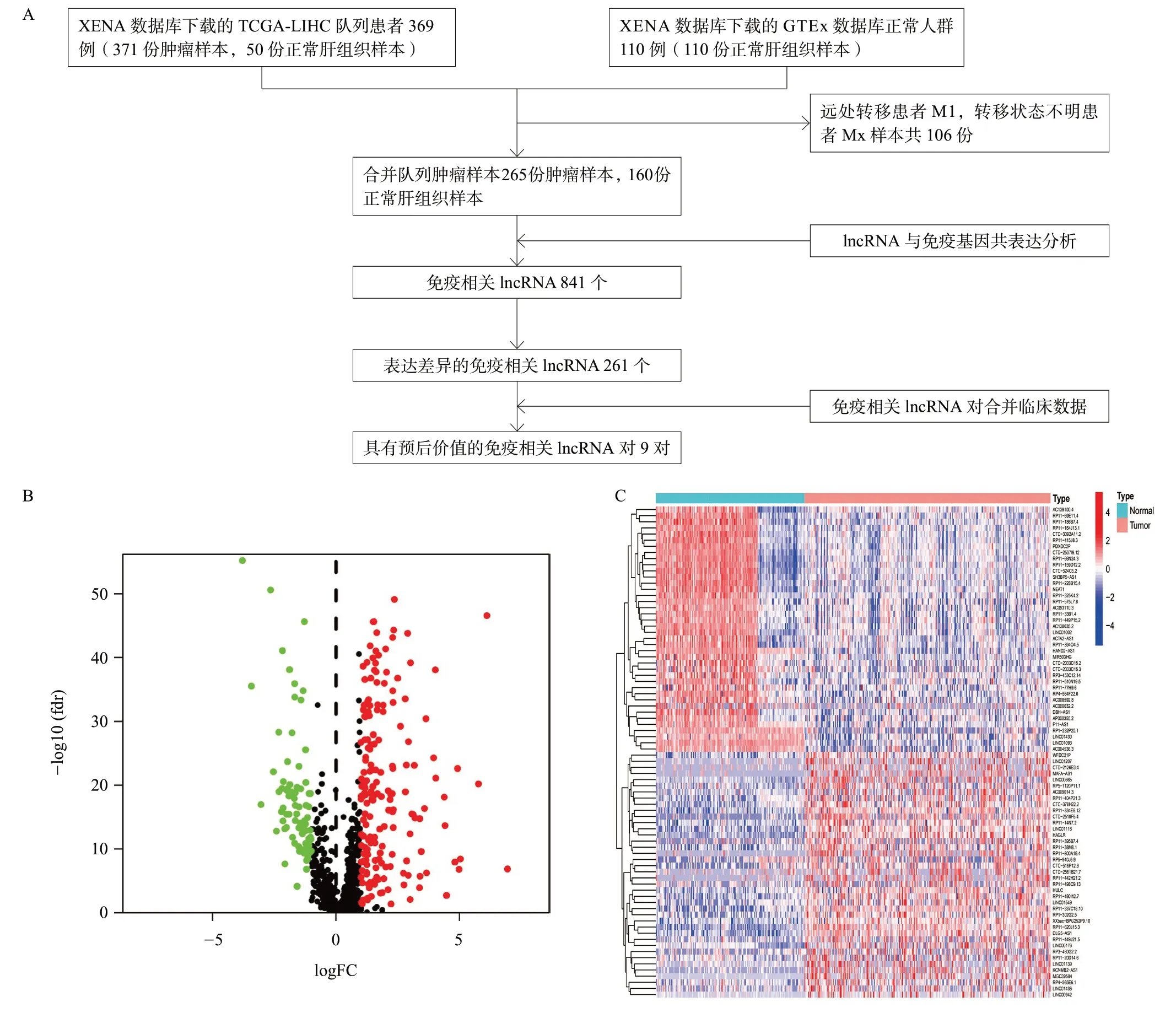
图1 流程图、差异分析火山图与热图
2.2 “免疫lncRNA基因对”模型(IRLP)构建
基于从免疫基因数据库ImmPort database(http://www.immport.org)下载的免疫基因,使用R软件从LIHC队列的表达矩阵中提取出1030 个免疫基因的表达矩阵。根据23版GTF基因注释文件从肝癌表达矩阵中提取出lncRNA的表达矩阵,我们使用共表达分析的方法找出与免疫基因表达相关的lncRNA为841个(相关系数>0.5),之后我们通过使用“limma”工具包找出在肝癌标本与正常肝组织中表达差异的免疫相关lncRNA 261个(图1B),肝癌组织与正常肝组织中上调、下调的差异表达的免疫相关lncRNA的热图见图1C。通过迭代循环和0或1个矩阵筛选,获得了17 835 对差异表达的lncRNA基因对。去除生存数据不完整的患者和生存时间为0 d的患者,剩余261 例肝癌患者进行后续的生存分析。通过单因素Cox回归分析根据筛选条件P<0.05的到2 456个差异表达的lncRNA基因对,表明该2 456个差异表达的lncRNA基因对与肝癌患者的预后相关,之后将其进行LASSO回归分析和使用交叉验证的方法进行迭代分析,并将其的筛选得到的lncRNA基因对进行逐步回归多因素Cox回归分析得到9 个预后相关的lncRNA基因对(AC009014.3|RP11-800A18.4、CTB-193M12.5|KB-68A7.1、CTC-518P12.6|RP5-1171I10.5、F11-AS1|RP5-940J5.9、KB-68A7.1|RP11-196G18.23、MIR503HG|RP11-498C9.15、MKLN1-AS|RP11-325K4.2、RP11-196G18.23|RP5-1171I10.5、RP11-498C9.15|RP13-104F24.2)(图2A),并根据其多因素Cox回归分析的相关系数来计算每个肝癌患者的风险打分,并建立免疫lncRNA基因对模型(IRLP),其模型公式为:
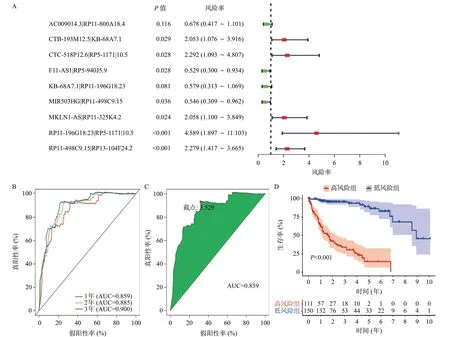
图2 免疫相关lncRNA基因对预后模型质量评估

2.3 “免疫lncRNA基因对”与患者的预后相关性分析
分析免疫lncRNA基因对模型(IRLP)的时间依赖性受试者工作特征曲线(ROC曲线),得到1、2、3年的曲线下面积(AUC值)分别为0.859、0.885、0.900(图2B),并且以1 年ROC曲线最大乔丹指数处的截止点的对应值作为截止值为3.529(图2C)。本模型1、2、3年的AUC值均大于0.850,表明IRLP预后模型中的9 个deirlncRNA基因对对于原位肝癌患者生存预测具有高度的敏感性和特异性。之后,我们根据选取的截止值3.529和每份肝癌患者对应的风险打分将患者分为高风险组(n=111)和低风险组(n=150)并画出K-M曲线,结果表明高低风险组的预后差异有统计学意义(P<0.001)(图2D)也表明了该9个差异表达的lncRNA基因对有预测肝癌患者预后的有效性。患者的风险曲线和生存时间随风险的分的分布见图3A、3B。风险曲线显示,高风险组HCC患者的病死率高于低风险组。排除无年龄、性别、分级和肿瘤分期信息的患者,对训练组的样本进行多因素Cox回归分析,以评估独立的危险因素。森林图显示,临床分期(HR1.500,95%CI1.131~1.988,P=0.005)和IRLP风险打分(HR1.074,95%CI1.055~1.094,P<0.001),差异有统计学意义(图3B)。在多因素回归分析中,IRLP风险打分(HR1.074,95%CI1.055~1.094,P<0.001)(图3B、3C)是影响HCC患者预后的独立危险因素。临床相关热图显示,风险打分与肿瘤分级、临床分期和T分期相关(图3D)。
2.4 免疫浸润分析
由于lncRNA与免疫相关基因相互关联,我们进一步探讨了该模型与肿瘤免疫微环境之间的关系。结果显示,模型中的高风险组与多种免疫细胞有相关性,见图4A。XCell基于标记基因,计算64 种免疫细胞和基质细胞相对富集分数,通过比较并制作箱式图,发现基质细胞分数与免疫微环境分数在低风险组中显著高于高风险组(图4B、4C)。CD8+T细胞在肿瘤免疫微环境中期重要作用,通过比较并制作箱式图发现CD8+T细胞在低风险组中显著高于高风险组(图4D)。以上表明低风险组的肝癌患者拥有较高的免疫浸润。之后,我们又分析了免疫调节基因包括免疫检查点在内的表达量在高低风险组的差异情况。结果表明,高风险组的免疫负性调节基因IL10RB、TGFBR1的表达量显著高于低风险组(P<0.05)(图5A、5B),潜在表明高风险组的患者有较强的免疫抑制的肿瘤微环境。
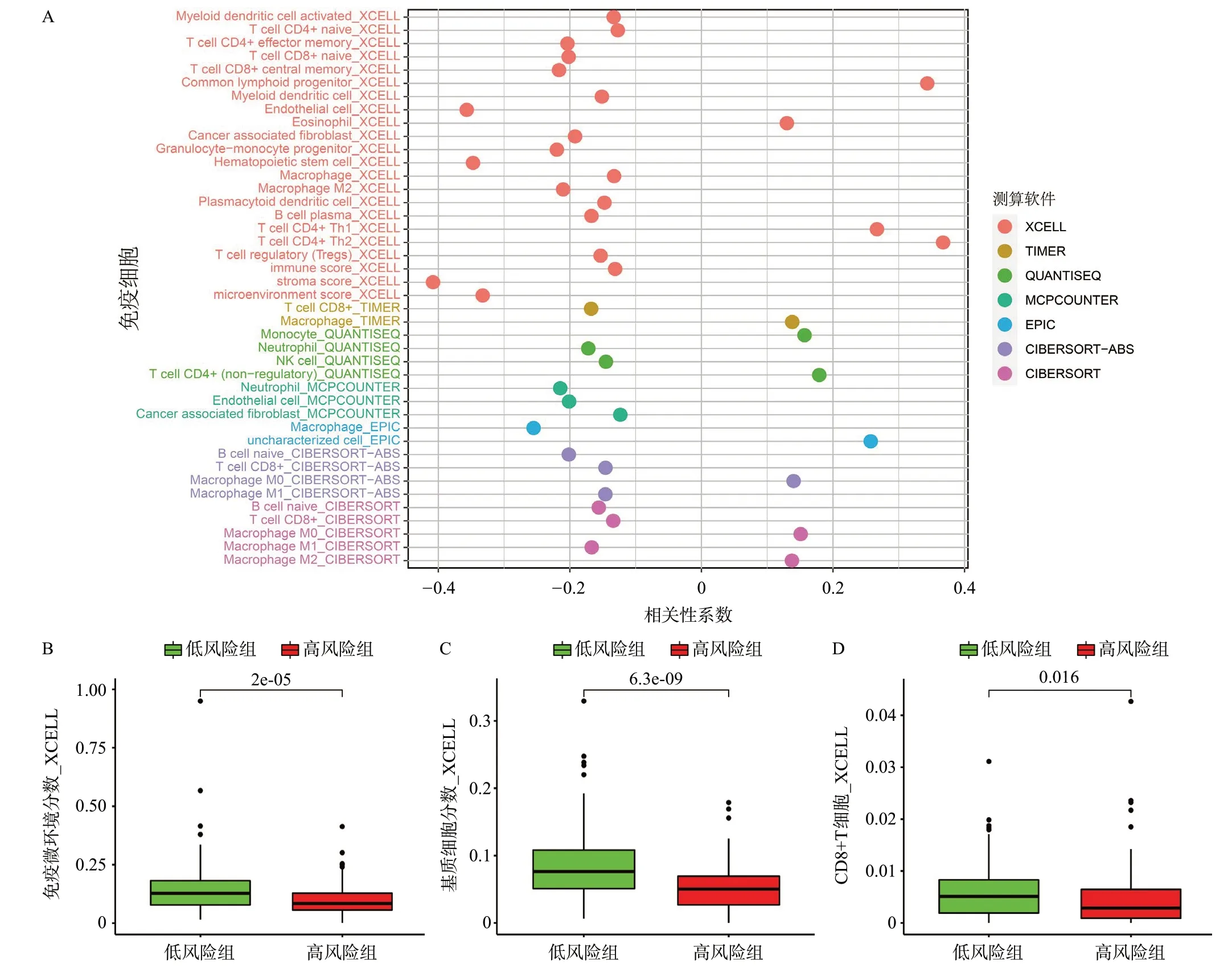
图4 免疫浸润分析
2.5 药物敏感性分析
药物敏感性分析显示,高风险组中厄罗替尼(P=0.0081)、阿昔替尼(P=0.0056)的IC50高于低风险组(图5C、5D),低风险组中的拉帕替尼(P=0.02)和临床肝癌一线用药索拉非尼(P=0.025)的IC50高于高风险组(图5E、5F)。结果表明,该模型可作为HCC治疗药物敏感性的潜在预测因子。
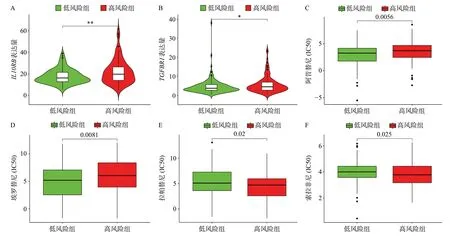
图5 免疫调节基因与药物敏感性分析
3 讨论
本研究中,我们在未发生转移的原位肝癌样本中筛选出免疫相关lncRNA基因对,并结合患者的生存状况建立了预测模型,该模型能够独立预测患者的生存状况,低风险组患者的生存获益显著优于高风险组(P<0.001)。同时,在肿瘤免疫微环境方面,高风险组表现出了更为突出的免疫抑制状态。而基于表达数据的药物敏感性分析表明了拉帕替尼、厄罗替尼、阿昔替尼以及顺铂在基于风险分组的患者中敏感性的差异,潜在为更加精准的应用相关药物提供了依据。
肿瘤数据库保存的样本涉及各个分期的肿瘤,当前大部分研究采用TCGA数据库数据进行分析时并没有对各个分期的肿瘤进行细致分类,这样导致样本中混杂了转移瘤,复发肿瘤以及原位肿瘤等多种情况。而针对性的区分肿瘤,能够避免肿瘤异质性的混杂,排除干扰,更加精确地筛选出靶标,从而建立预测价值更可靠的模型。
迄今为止,关于肿瘤中lncRNA的研究大部分集中在肿瘤发展和转移中的调控[11]。最近的研究表明,lncRNAs在肿瘤免疫的不同阶段发挥着至关重要的作用,包括抗原释放、呈递、免疫激活、免疫细胞迁移、肿瘤细胞的浸润等[11-12]。lncRNA在肿瘤免疫中的研究能够从一个新的角度揭示了肿瘤免疫中复杂的分子机制,潜在为肿瘤免疫治疗提供新的潜在靶点。该研究中,我们建立的模型能够很好的区分患者1、2和3年的生存获益状况;同时,该风险分值在包括肿瘤病理分级,临床分期的多因素下能够独立的预测患者预后。这表明该模型具有良好且稳定的预测能力。尽管研究纳入的数据是基因的表达量数据,然而,本研究采用基因对的组合模式,对数据内部之间进行比较,而不需要进行数据之间的校正,这样以来避免了不同样本间的数据偏差,有效降低了结果的偏倚[13]。
HCC中的肿瘤异质性明显。据估计,30%~50%的复发性或转移性肿瘤具有与原发肿瘤不同的克隆,22%~79%的同位置肿瘤具有克隆性差异,12%~66%的单个肿瘤具有肿瘤内异质性[6,14]。大量的肿瘤间和肿瘤内异质性使得生物标志物研究意义非凡,而这对于分子靶向治疗的开发和管理也至关重要。同时,肝癌早期免疫浸润丰富,肿瘤负荷较小;机体免疫并未完全失去战斗力,此时肿瘤内微环境特点与晚期肿瘤具有明显区别。因此充分了解此类肿瘤的免疫微环境的特征更有利于免疫治疗的开展,对早期应用免疫治疗更能提供相对精确的指导[15]。该研究基于XCELL计算了64 种免疫细胞和基质细胞相对富集分数,表明低风险组的肝癌患者拥有较高的免疫浸润,以及其免疫抑制情况较轻。在两组免疫调节基因的表达量差异情况上,高风险组的免疫负性调节基因IL10RB、TGFBR1的表达量显著高于低风险组,表明高风险组肿瘤逐渐进展衍生出更为广泛的免疫逃逸。
肝癌系统治疗近几年取得长足进步,包括索拉非尼、伦伐替尼等小分子靶向药物、纳武单抗等单克隆抗体在内的药物疗效令人欣喜[16-17]。除了上述药物被应用到肝癌一线、二线治疗外,还有一些潜在的药物也正在临床试验或临床前开发中[18]。拉帕替尼、厄罗替尼、阿昔替尼及顺铂均在肝癌开展过前瞻性的临床试验研究[19-21],虽然并没有统计学意义的结果,但其在特定类型或是状态下肝癌的疗效仍然值得关注。该研究中,对于不同风险组其药物敏感性具有显著不同,这潜在提示基于该模型下肝癌患者分组后或许可以更加精准的运用相关药物。
尽管本研究进行了严谨的设计,依然面对着一些问题。本研究中数据获取于TCGA数据库,其中肝癌TNM分期参考AJCC的指导原则,ⅣA期为没有肝外肿瘤的转移,仅有明确的淋巴结转移,ⅣB期患者为明确的肝外转移患者。因此本研究中ⅣA期患者数量较少,其临床相关性分析结果并不能完全说明问题。同时,对当前肝癌细胞系药物敏感性的模拟难以完全匹配机体肝癌的疾病特点[15,22]。不仅是在肝癌,在泛肿瘤中也是如此,肿瘤具有复杂的微环境特点,单独的肿瘤细胞离体模拟只能提供潜在参考价值。
总之,本研究利用未发生远处转移的原位肝癌样本构建了免疫相关lncRNA的基因对模型能够较好的预测患者生存状况,同时表明高风险的患者中免疫抑制状态明显,而不同的风险组对肝癌特定药物治疗敏感性不同。
