融合空间和多尺度特征的乳腺癌免疫组化Ki-67指数定量分析
2022-07-30熊学春吴焕文梁智勇
熊学春,吴焕文,任 菲,崔 莉,梁智勇,赵 泽
1中国科学院计算技术研究所,北京 100190 2中国科学院大学计算机科学与技术学院,北京 101408 3中国医学科学院北京协和医院病理科,北京 100730
国际癌症研究机构发布的2020年全球癌症统计数据显示,乳腺癌已成为女性最高发的恶性肿瘤(新增病例在所有肿瘤中占比11.7%),每年可导致68.5万病例死亡[1]。准确诊断是精准治疗的前提。Ki-67是乳腺癌诊断过程中常用的免疫组化标志物,是评估肿瘤细胞增殖活性的关键指标,Ki-67指数可作为乳腺肿瘤良恶性及其恶性程度的辅助诊断指标。一般情况下,乳腺癌的Ki-67指数高于10%,随Ki-67指数升高,肿瘤生长加快、组织分化能力降低,患者预后较差[2]。
目前临床工作中,Ki-67指数大多需病理医生通过显微镜对肿瘤细胞进行人工计数获得[3];部分研究通过浏览整张免疫组化染色的全视野数字图像(whole slide image,WSI),采用视觉评估法计算Ki-67指数[2]。上述方法工作量大、效率低、准确率差且具有主观性。2019年10月,国际乳腺癌Ki-67工作组(International Ki-67 in Breast Cancer Working Group,IKWG)召开会议,讨论了Ki-67免疫组化检测在乳腺癌诊断与治疗中的应用现状[4],指出基于机器学习的自动化评估可能有助于解决当前Ki-67免疫组化判读中的难题[2,4-5]。当前,深度学习在病理图像处理领域已取得了显著成效[6-8],自动化评估Ki-67指数亦取得重要进展。根据病理图像的处理方式,大致可分为两类:一类是基于图像处理技术,一类是基于机器学习和神经网络。Xing等[9]提出了一种基于全卷积网络的KiNet模型,对Ki-67细胞核进行检测和识别,并在胰腺神经内分泌肿瘤数据集中得到了良好的分类效果验证,曲线下面积(area under the curve,AUC)为0.724。Negahbani等[10]利用点标注的方式制作了SHIDC-B-Ki-67数据集,并提出一种用于Ki-67细胞检测和分类的模型,对免疫阳性细胞、免疫阴性细胞和淋巴细胞检测和分类的精确度为77.66%。Shete等[11]将基于染色强度的阈值化和分水岭分割方法相结合,依据细胞染色区域面积、细胞形态学特征、纹理特征等实现了对乳腺肿瘤细胞恶性程度自动化检测。Ko等[12]对图像进行染色归一化,分离染色区域后实现重叠细胞核分离,改善了不同实验室或环境中由染色变化引起的图像处理问题。但上述研究均是在单个图块上对Ki-67指数进行定量分析,并将部分区域定量分析的结果代替整张WSI的Ki-67指数。本研究提出一种智能化定量分析Ki-67指数的方法,可识别整张WSI中的Ki-67指数,提高Ki-67指数评估的准确性和客观性。
1 材料与方法
1.1 研究材料与数据集分组
回顾性纳入2020年1— 12月北京协和医院乳腺癌患者的病理切片。纳入标准:根据组织病理和免疫组化结果,确诊为乳腺浸润性癌;排除标准:切片质量不合格。
采用德国Leica公司Aperio AT2高通量切片扫描仪将病理切片以40倍率扫描为WSI图像。由2名病理科医生根据2019年IKWG制定的指南[4]对所有WSI中的Ki-67指数进行人工判读后,按5∶8的比例随机将WSI图像分为A、B两个数据集。2名病理科医生选取WSI中Ki-67阳性肿瘤细胞最集中且分布均匀的区域(热点区域,图1)对A数据集进行标注,用于WSI热点区域识别模型训练与测试;B数据集用于评估模型对Ki-67指数计算结果的准确性,并采用Bland-Altman法[13]对人工判读与智能定量分析结果进行一致性评价。

图1 乳腺癌Ki-67指数免疫组化病理图像标注示例A.热点区域;B、C.正常组织区域
本研究已通过北京协和医院伦理审查委员会审批(审批号:S-K311),并豁免患者知情同意。
1.2 方法
1.2.1 整体研究框架
本研究提出的Ki-67指数智能化定量分析方法主要包括2个部分(图2):(1)提出一种融合空间和多尺度特征的高精度乳腺癌WSI热点区域识别模型;(2)在识别出的热点区域内,40倍视野下随机选取每张WSI的10个视野进行自动细胞识别、分类、计数,计算10个视野下的Ki-67指数均值,为整张WSI的Ki-67指数。

图2 Ki-67指数智能化定量分析方法整体框架
1.2.2 图像预处理
由于WSI制作、扫描过程中可因抖动、电子脉冲干扰等因素的影响,导致图像失真、模糊,在分析前需进行预处理。结合实际情况和预试验比较结果,借助Opencv-Python库并采用高斯滤波对图像进行去噪、平滑图像、去除空白背景等处理[14]。由于在HSV(Hue,Saturation,Value)颜色空间中分析更方便,首先根据公式(1)将原始图像从RGB颜色空间转换至HSV颜色空间,然后基于Otsu算法[15]获取二值化的组织掩码,并进行形态学开闭运算,提取组织区域图像。
(1)
其中,R、G、B分别表示红色、绿色、蓝色,取值范围均为[0,255];H为色调,取值范围为[0,1];V为明度,取值范围为[0,255]。
1.2.3 提取热点区域掩码与图块
按7∶1∶2的比例将A数据集随机分为训练集、验证集和测试集。采用ASAP(Automated Slide Analysis Platform)软件对热点区域进行标注,然后提取热点区域掩码图像。用组织区域图像减去热点区域即可获取正常区域掩码图像。在2种图像内部,将原始WSI图像于40倍视野下随机裁剪为2000个 512×512像素的图块(patch)。在上述图块中随机选取50个,对其中的Ki-67阳性肿瘤细胞、阴性肿瘤细胞和非肿瘤细胞进行标注(Ki-67指数的计算仅包括肿瘤细胞,若不排除非肿瘤细胞,Ki-67指数将偏低),见图3。标注后,计算Ki-67指数[16]。
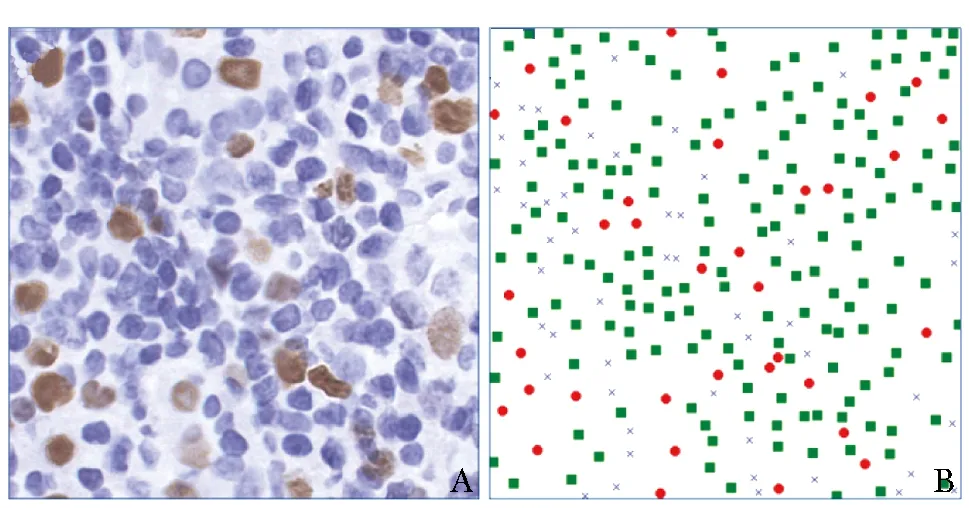
图3 热点区域内图块标注示例A.热点区域内随机选取的图块;B.标注结果,其中实心圆为Ki-67阳性肿瘤细胞,实心矩形为Ki-67阴性肿瘤细胞,“X”为非肿瘤细胞
1.2.4 热点区域识别
WSI可完全保留病理切片上的原始组织特征信息,对于数据量巨大且缺乏像素级的标注,病理医生可通过缩放、拖拽、平移图像的方式浏览整张WSI图像。在作出诊断决策时,需同时考虑单个区域相邻的信息和不同区域之间的相关性信息,最后确定整张WSI图像的Ki-67指数[8]。常用的病理图像分类深度学习方法,一般是将一张数万×数万像素的WSI切分为数万张图块,通过病理医生对热点区域的粗标注引入医学先验知识[17],此时数万张图块对应一个标签,然后采用VGG[18]、ResNet[19]、MobileNet[20]等卷积神经网络模型对图块进行分类[21]。此种分类方法会导致图块在整张WSI图像中的空间位置信息丢失,无法融合相邻图块的信息和空间位置信息。
针对该问题,本研究采用一种融合空间和多尺度特征的乳腺癌WSI热点区域识别方法,即条件随机场模型[22](图4A)。条件随机场模型是Lafferty等[23]于2001年提出的将最大熵模型和隐马可夫模型相结合的一种概率无向图模型。该模型在自然语言处理中的命名实体识别任务以及计算机视觉中的图像分割任务等领域均有广泛应用。
在概率无向图模型中,随机变量之间的关系较直观,易于理解。在实际应用时,目的是计算联合概率分布。为简化计算,假设将WSI中的图块设置为X1、X2、X3…Xn,若一个图块为热点区域,则其相邻图块为热点区域的概率很大(图4B)。进一步对图块的关系简化(图4C),并根据公式(2)计算模型的联合概率分布。

图4 图块之间的概率无向图模型示意图

×ψ2(X1,X4)]
(2)
1.2.5 模型构建与相关参数设置
经图像预处理获取热点区域和正常组织区域的图块后,将训练集和验证集图块输入至ResNet34预训练模型[19]进行特征提取。每个图块被编码为固定长度的Embedding,作为概率无向图模型中的节点进行空间特征维度建模。模型可输出每个图块的位置分布概率,用以判定特定图块是否位于热点区域。高精度乳腺癌WSI热点区域识别模型框架见图5。

图5 乳腺癌WSI热点区域识别模型整体框架图
硬件参数:Linux版本为CentOS 3.10.0~693.el7.x86_64,CPU版本为Intel(R)Xeon(R)Silver 4114 @ 2.20 GHz,显卡为NVIDIA GeForce RTX 2080 Ti(显存容量11 GB,显存位宽352 b,显存频率14 000 MHz),内存为 32GB 2666 MHz DDR4。软件环境:编程语言为 Python 3.6.5,相关的深度学习框架为Torch,CUDA及cuDNN环境分别为10.0和7.4,CPU环境运行下的编译器为GCC 4.8。Python计算包:Torchvision 0.4.2,Opencv-Python 4.5.1.48,Openpyxl 3.0.9,Openslide-Python1.1.2。模型训练时的参数:Batch Size 为32,优化器为Adam,学习率为0.001,损失函数为Cross Entropy,Epoch为30。
1.2.6 热点区域识别与肿瘤细胞自动计数
WSI输入经过训练的ResNet34热点区域识别模型,获取热点区域概率热力图,其中概率高于0.5的区域认定为热点区域。在热点区域内,40倍视野下随机选取10个视野进行Ki-67指数计算。首先对输入的RGB图像进行预处理,然后按照公式(3)将RGB值转换成光密度(optical density,OD)空间:
(3)


图6 肿瘤细胞计数方法
1.3 统计学处理
采用SPSS 23.0软件进行统计学分析。以病理科医生人工判读结果为金标准,计算模型对热点区域识别的准确率以及识别的Ki-67指数误差,误差在±0.2之间视为模型计算结果正确。采用Bland-Altman法评价人工判读和模型计算Ki-67指数的一致性。
2 结果
2.1 数据集
共入选符合纳入和排除标准的乳腺癌患者病理切片132张。其中A数据集 50张(训练集、验证集和测试集WSI图像分别35张、5张、10张,分别包含图块70 000个、10 000个、20 000个),B数据集82张。
2.2 热点区域智能化识别结果
训练后的ResNet34模型对测试集热点区域识别的平均准确率为81.5%。热点区域可视化结果见图7。

图7 热点区域可视化识别结果
2.3 Ki- 67指数定量结果
在热点区域识别的基础上,模型可根据选取的视野,自动对细胞进行分类、计数(图8),最终计算整张WSI中的Ki-67指数。以病理科医生人工判读结果为金标准,本研究该模型对B数据集82张切片Ki-67指数计算准确率为90.2%(74/82)。
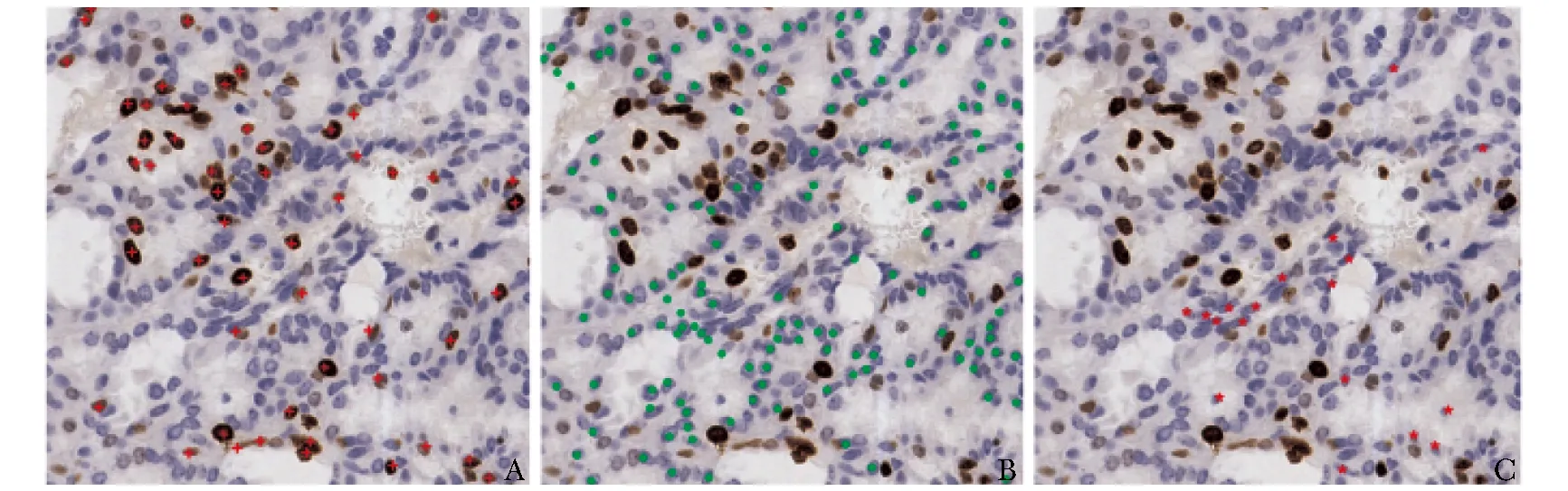
图8 模型对细胞分类计数结果
2.4 Ki- 67指数一致性评价


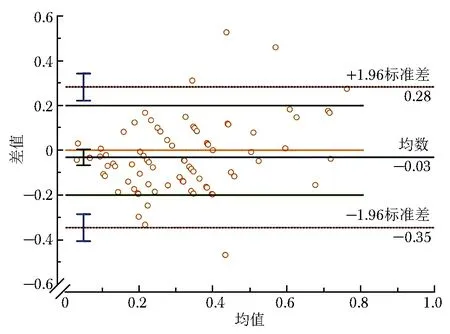
图9 Ki-67指数一致性评价结果的Bland-Altman图
3 讨论
乳腺癌免疫组化Ki-67指数在治疗方案制订和预后风险评估中占据重要地位,临床意义显著。目前在临床工作中,人工判读Ki-67指数工作量大,结果具有主观性,亟需探寻一种高效、易操作且准确率高的Ki-67指数评估方法。本研究基于ResNet34算法,首次构建针对乳腺癌WSI图像可自动识别并输出Ki-67指数的智能化判读系统,结果显示该方法对Ki-67指数计算结果的准确率为90.2%,与人工判读结果一致性较好,有望应用于临床,辅助临床决策。
组织病理是肿瘤确诊的金标准,病理科医生通过对病理切片进行观察,可为肿瘤诊断提供最重要的依据。免疫组化是组织病理学与细胞病理学的关键组成部分,在肿瘤诊断、分子分型、精准治疗等方面均具有极大的影响力,但实际应用过程中,受判读区域选择、计数细胞总和、计数方法等因素的影响,其结果准确性、可重复性方面仍存在诸多挑战。近年来,人工智能技术快速崛起,其在医学中的应用越来越广泛,医工结合、医工交叉已成为研究的新趋势[26]。但既往主要针对病理图像的图块进行研究,尚缺乏直接可识别整张WSI图像中Ki-67指数的方法[9-12]。本研究基于深度学习技术建立的整张WSI图像中Ki-67指数智能定量方法,在判读区域选择、计数细胞总和、计数方法3个方面均有明显改进,在保障了Ki-67指数判读结果的高准确率和可重复性的同时,可快速(识别一张WSI图像平均耗时60 s)输出结果,优势明显。
3.1 判读区域选择
由于肿瘤细胞具有较高的异质性,在乳腺癌免疫组化WSI图像中,Ki-67阳性肿瘤细胞多呈不均匀分布,存在明显的热点区域、冷点区域和边缘区域,不同区域内的Ki-67指数存在较大差异,其中以热点区域最高,边缘区域次之,冷点区域最低。因此,判读区域选取不同,必然导致Ki-67指数判读结果不一致,影响结果的准确性。IKWG建议,乳腺癌Ki-67指数免疫组化WSI中,若存在热点区域,则判读区域应选择在热点区域内[26]。本研究建立的融合空间和多尺度特征的高精度乳腺肿瘤WSI热点区域识别模型,对热点区域具有较高的识别能力(平均准确率为81.5%)且速度较快,结果重复性好,是Ki-67指数计算结果具有高准确率的前提和重要保障。
3.2 计数细胞总和
判读区域选定后,需对区域内细胞进行识别与分类,以计算Ki-67指数。对于细胞较少的区域,Ki-67指数判读结果稳定性差。部分国家的指南指出,应在热点区域计数200个肿瘤细胞进行Ki-67指数判读[27]。IKWG建议,免疫组化Ki-67指数定量分析中,每个区域内以计数1000个肿瘤细胞为宜,至少不应低于500个肿瘤细胞。本研究Ki-67指数智能化量系统在计算Ki-67指数前,首先对区域内肿瘤细胞进行计数,若Ki-67阳性肿瘤细胞数量不足500个,则需重新选择判读区域,因此保障了结果的准确性、稳定性和客观性。
3.3 计数方法
显微镜下人工阅片对肿瘤细胞进行分类,易发生“误数”或“漏数”的现象,导致计算结果不准确。本研究构建的模型可程序化自动分类与计数,不仅结果准确(准确率为90.2%)、省时高效,且避免主观因素的影响。Bland-Altman法一致性评价结果显示,人工判读与模型计算Ki-67指数的一致性良好,提示该智能定量方法对Ki-67指数判读结果的误差较小,可达到人工阅片水平,再次验证了其具有准确性高和可重复性好的优势。
本研究局限性:(1)受限于样本量,虽然模型对Ki-67指数的评估具有较高的准确性,但模型仍可能存在过拟合现象;(2)缺乏多中心数据对模型的性能进行外部验证。
综上,本研究提出的融合空间和多尺度特征的乳腺癌免疫组化Ki-67指数智能定量分析方法,可快速、准确、客观对Ki-67指数进行判读,辅助病理科医生完成乳腺癌诊断,提升了工作效率,为乳腺癌患者进行精准分子分型和个体化治疗提供科学可信的理论依据和技术支持。
作者贡献:熊学春负责对人工智能分析计算方法流程的实现及论文撰写;吴焕文负责病理图像收集、标注及论文撰写;任菲负责选题构思、论文修订;崔莉负责分析方法技术指导;梁智勇负责病理诊断流程制定、结果评测;赵泽负责智能分析方法指导、深度学习相关方案设计、论文修订与审核。
利益冲突:所有作者均声明不存在利益冲突
