基于矩阵计算的组织病理学图像压缩储存算法
2022-07-30何睿琳刘子妤杨欣怡李晓燕
何睿琳,刘子妤,杨欣怡,李 晨,李晓燕
1东北大学医学与生物信息工程学院,沈阳 110819 2中国医科大学肿瘤医院/辽宁省肿瘤医院病理科,沈阳 110042
医学图像是现代医学主要诊断依据之一,组织病理学图像作为医学图像的重要组成部分,对切实了解患者病情具有极为重要的意义。然而,由于组织病理学图像数据庞大且复杂,需在有限的存储空间中实现对大量医学信息的完整保存,即在高压缩率下,同时保证高质量、具有完整有效信息的图像重建。在众多数字图像处理方法中,矩阵计算应用较为广泛,一般由计算机增强、特征提取、腐蚀膨胀及分割等操作步骤组成,其中主成分分析法(principal component analysis,PCA)和奇异值分解法(singular value decomposition,SVD)是更具优势的图像压缩方法[1]。采用PCA对图像进行降维处理,在减小数据量、简化数据分析的同时,不丢失原矩阵图片的主要特征;采用SVD对矩阵图片进行分解,选择其中的奇异值进行重构,可获得原矩阵图片的主要信息[2]。
本研究基于PCA与SVD两种矩阵算法,对低、中、高分化的宫颈癌组织病理学图像进行压缩存储,对比分析压缩重建前后的图像质量,提出针对不同分化程度的组织病理学图像压缩存储的最优途径。
1 材料与方法
1.1 组织病理学图像来源
本研究为回顾性分析,以辽宁省肿瘤医院病理科资料库2019年1—10月宫颈癌组织病理图像作为研究对象,包括低、中、高不同分化程度的宫颈癌组织免疫组化染色图像及HE染色图像各10组(每组10张,共600张),分类均经病理诊断证实。以上图像均由全视野数字图像(whole slide image,WSI)剪裁获得,既可作为独立视野显微图像使用(直接进行压缩储存),又可作为WSI局部使用。作为WSI局部使用时,首先需将WSI图像剪裁至所需大小,然后对每张图像进行压缩储存,在需要使用WSI图像时,再将压缩后的图像进行还原拼接。
本研究已通过辽宁省肿瘤医院伦理审查委员会批准(审批号:202229),并豁免患者知情同意。
1.2 PCA图像压缩储存法
1.2.1 基本原理
PCA是利用正交变换的方法,将从图像中提取的原向量经过线性变化处理变换为新向量,并将计算分析出的新向量根据方差大小进行排序。因此,在正交变换中,总体方差不变,第一主成分计算结果是方差最大的向量组,第二主成分是次一级向量组,以此类推,可获得n个变量,即n个主成分,且新获得的主成分之间无相关性[3]。一张组织病理学图片往往具有较多维度,利用PCA实现了对图片的降维处理,在缩小其存储空间的同时,保存了图片中的大量信息。

1.2.2 压缩储存步骤
假设样本为
(2)将转化好的标准化矩阵进行计算、分析,得出相关系数矩阵。
(3)由相关系数矩阵求解其特征方程。通过计算可得出m个特征根,并使用sort函数对指定区间所有数值进行排序。利用数据,求出特征值和特征向量。
(4)计算贡献率。贡献率是主成分与各个分量方差总和的比值。累计贡献率则是所选取的主成分的贡献率的总和,累计贡献率的大小代表了数据的一种可靠性,且两者成正比关系,累计贡献率越大,所得出结论的可靠性越强;累计贡献率越小,图片损失的信息量越大[4]。通常在利用PCA对图像进行处理时,累计贡献率一般不低于85%,采用这一标准确定选取主成分的个数,能够作出较为科学的评价。
1.2.3 图像储存改进
本研究在以上PCA的基础上,对图像进行3种方式的压缩处理改进:(1) 将彩色图像转化为灰度图像,将图像数组矩阵转化为样本矩阵,并计算样本和变量的个数。计算并提取前p个主成分,根据系数矩阵重建样本矩阵,再将样本矩阵转化回图像数组。(2) 分块处理。利用im2col函数实现图像块重排列,再利用col2im对图像进行恢复。以自定义大小作为卷积核,对输入图像进行分割处理,形成新的矩阵。将图像块转换成矢向量,并计算图像块灰度均值。计算取出最大n个特征值,将自定义维度映射到n维,最后利用col2im重建图像。(3) 图像融合。提取图片主成分,融合第一、二主成分实现图像压缩。
1.3 SVD图像压缩储存法
1.3.1 基本原理
SVD是高等数学线性代数中的一种算法,可有效提取矩阵代数特征[5],主要应用对象为正方矩阵,在信号处理中有着极为广泛的应用。其从双线性函数f(x,y)=xTAy,a∈Rn×n着手,通过利用线性变换,x=Uζ,y=Vη,带入到双线性函数中,得到f(x,y)=ζTSη,其中S如公式(1.1)所示。
S=UTAV
(1.1)
由公式(1.1)可知,当满足矩阵U和矩阵V均为正交矩阵时,二者之间的选择存在(n2-n)的自由度。若通过这些自由度将矩阵S的对角线元素不置零,其余元素均置零,则矩阵S便成为对角矩阵且S=∑=diag(σ1,σ2,σ3,…,σn)。接着用U和VT各自左乘和右乘公式(1.1)。利用矩阵U和矩阵V的正交性,可得A如公式(1.2)所示。
A=U∑VT
(1.2)
1.3.2 压缩储存步骤
若一幅图像有n×n个像素,则利用n×n的矩阵A代表n×n个像素,对矩阵A进行奇异值分解,可知A=U∑VT,并将所得的奇异值由大到小排序。选取前k个奇异值逼近原图像,即可通过k×(2n+1)个值逼近原图像n×n个数值。可求得压缩比例,如公式(1.3)所示。
(1.3)
利用SVD的具体操作步骤如下:
(1)首先对输入图片进行灰度化处理,并计算输入图像尺寸;
(2)将原图像的灰度值转换成double类型;
(3)进行奇异值分解,并提出对角矩阵的对角线元素,得到一个向量,并计算最大、最小奇异值;
(4)保留前n个最大奇异值,并对其他奇异值置零,将向量转化为对角矩阵;
(5)还原图像,计算压缩比。
1.3.3 图像储存改进
本研究在以上SVD的基础上,对图像进行2种方式的压缩处理改进:(1) 在传统SVD的方法上,将图片uint8改为double类型,double更适合运算,uint8是为了节省存储空间。首先进行奇异值分解,提出对角矩阵对角线元素,获得向量。保留选定的奇异值,计算压缩比,若为矩形矩阵,则用0填满。(2)利用colormap和imagesc函数进行计算,获取当前色图,并以图像的方式显示矩阵。
1.4 图像质量评价指标
重建图像的还原度从以下两个角度进行考量:
(1)峰值信噪比(peak signal-to-noise ratio,PSNR)。PSNR是应用最为广泛的图像客观评价指标,基于对应像素点之间的误差进行图像评价,如公式(1.4)所示。PSNR高于40 db说明图像质量极好,十分接近原图像;>30~40 db说明图像质量较好,可察觉部分失真;20~30 db说明图像质量差;20 db以下则图像质量不可接受。
(1.4)
(2)结构相似度(structural similarity,SSIM)。SSIM是一种质量评价方法,可用于衡量两张图片相似性指标。SSIM取值范围为[0,1],值越大,表明图像结构失真越小。
1.5 统计学处理
所有数据应用SPSS 23.0软件进行描述性统计学分析,PSNR和SSIM以均数±标准差表示。
2 研究结果
2.1 PCA测试结果
利用PCA对图像进行压缩储存预试验,当主成分p分别为1、18、36、53时,图像压缩比分别为539.68、29.98、14.99、10.18,累计贡献率分别为99.35%、99.98%、99.99%、99.99%。
将主成分p设为53,应用PCA处理低、中、高分化宫颈癌组织免疫组化染色和HE染色病理图像各10组(每组10张)。结果显示,低、中、高分化宫颈癌组织免疫组化染色图像PSNR均值分别为43.84±0.43、43.27±0.25、43.71±0.49,压缩图像SSIM分别为0.964±0.004、0.963±0.006、0.965±0.005;HE染色图像PSNR均值分别为43.41±0.78、42.95±1.03、43.52±0.69,压缩图像SSIM分别为0.953±0.010、0.949±0.015、0.960±0.007。压缩前后的图像对比见图1,2。
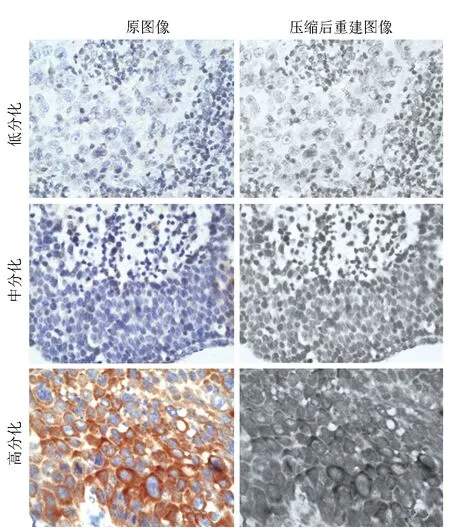
图1 不同分化程度宫颈癌组织免疫组化染色图像经主成分分析法压缩前后图像
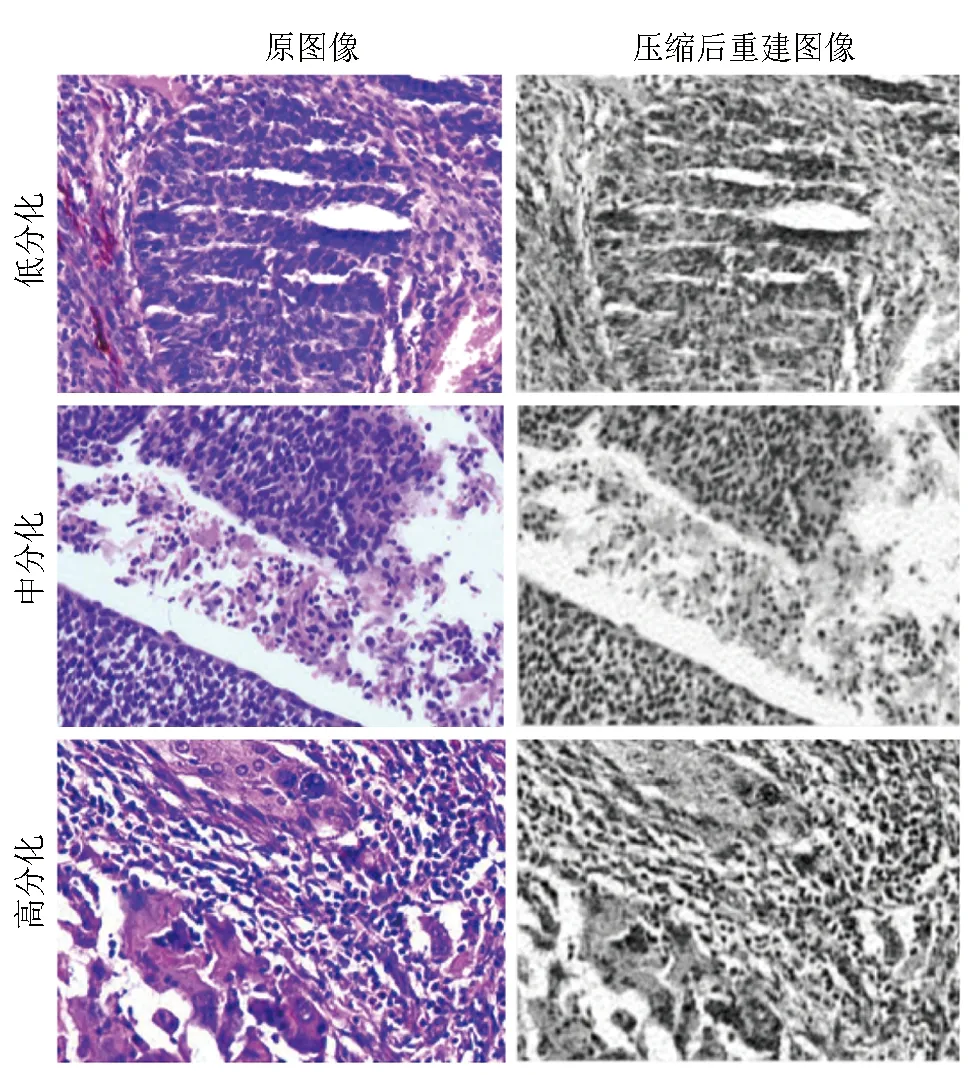
图2 不同分化程度宫颈癌组织HE染色图像经主成分分析法压缩前后图像
2.2 SVD测试结果
利用SVD对图像进行压缩储存预试验,当奇异值分别为32、48、64、108、128、200时,图像压缩比分别为47.99、32.00、24.00、14.22、10.00、7.68。
设定奇异值为128,应用SVD处理低、中、高分化宫颈癌组织免疫组化染色和HE染色病理图像各10组(每组10张)。结果显示,低、中、高分化宫颈癌组织免疫组化染色图像PSNR均值分别为39.89±1.69、38.20±2.19、40.90±0.50,压缩图像SSIM分别为0.949±0.006、0.938±0.011、0.955±0.004;HE染色图像PSNR均值分别为40.31±0.98、39.46±1.59、40.77±1.67,压缩图像SSIM分别为0.965±0.006、0.943±0.010、0.969±0.005。压缩前后的图像对比见图3,4。
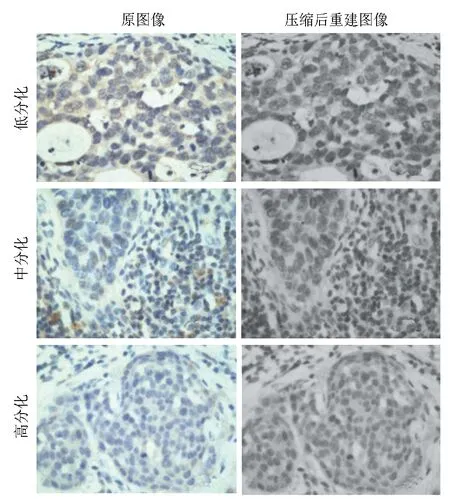
图3 不同分化程度宫颈癌组织免疫组化染色图像经奇异值分解法压缩前后图像
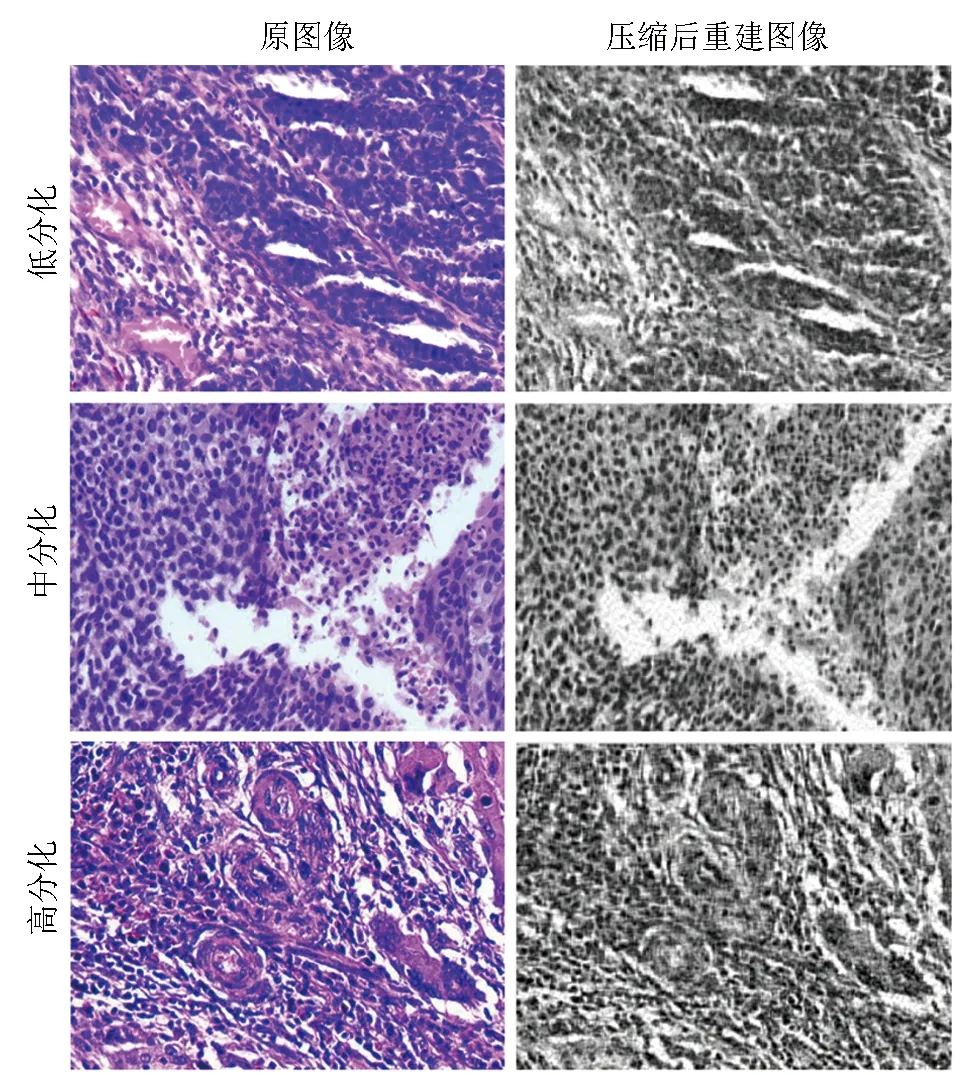
图4 不同分化程度宫颈癌组织HE染色图像经奇异值分解法压缩前后图像
3 讨论
本研究采用PCA和SVD两种矩阵算法对不同分化程度的宫颈癌组织免疫组化染色图像及HE染色图像进行压缩储存处理,解决了病理学图像数据庞大、图像包含信息量巨大、占用大量存储空间等问题。经PSNR和SSIM分别评价后,显示两种压缩储存算法在图像质量和结构保真度方面均较好。
近年来,随着数字病理学的发展,传统病理诊断瓶颈逐渐被打破,在一定程度上解决了医疗资源地域分布不均等问题,极大提高了病理诊断效率及诊断准确性。但同时,随着医学图像数量及尺寸(分辨率提高)的增加,对于有效存储和便捷传输方法的需求亦不断提高[6]。压缩后的图像需要保留精细的结构信息[7],而正是对图片质量的高要求,需要更大存储容量的设备,因而产生了额外的昂贵成本[8]。因此,高效的影像学数据压缩、传输不仅对于疾病远程诊断和监测十分重要,且是降低医院运营成本的重要举措。纵观所有高阶数据处理方法,PCA或类PCA是广泛使用的图像压缩方法[9]。在矩阵中,SVD可在不影响图像质量的前提下,实现图像重建的目的[10]。
PCA矩阵计算方法是先将图像数据进行分块,将每一块作为一个样本向量,再通过对多个样本构成的矩阵提取主成分[11],其去相关性好,根据图像本身数据,制订变换矩阵,其计算方式是在最小均方差状态下的正交变换,几乎适用于各种医学图像的压缩存储处理。主成分的贡献率越大,证明此主成分的比重越大,所包含的信息量越多。故本研究选择主成分p=53,此时压缩比为10.18,在保证运算速度的同时,使累计贡献率最大化,即图中有效信息的可检测性大,图像质量较优。
在利用矩阵计算方法对图像进行存储处理的过程中,矩阵的奇异值分解是一种重要的处理手段。自1970年由Golub和Reinch提出矩阵计算方法SVD以来,SVD已成为解决图像矩阵计算方面的有效解决工具[12]。其优点在于图像奇异值的稳定性较为出色,例如施加一些小的干扰,医学图像的奇异值不会发生较大的变动,且SVD所展现出的并非视觉特性,而是医学图片蕴含的固有属性[13-14],是在不改变医学图像原本矩阵度量的条件下,分析计算出有效秩,再在特定情形下给出图片矩阵秩的最佳逼近效果。通过利用图像的固有矩阵结构,对图像对应的数据矩阵进行图像再压缩。重构时,首先选择合适的奇异值及奇异值向量还原数据矩阵,再重构图像,这样可简化数据,极大降低图像所占用的资源[10]。本研究选择的最大奇异值为128,压缩比为10.00,此时可最大程度保留原图像信息,是SVD系数最优值。
本研究采用PSNR和SSIM针对图像压缩后重建质量进行分析评估。PSNR是基于对应像素点间的误差,即基于误差敏感的图像质量评价,其数值越大,图像失真越小。SSIM是一种全参考的图像质量评价指标,分别从亮度、对比度、结构三方面度量图像相似性,其数值越大,图像失真越小,且在图像相似度的评价上优于PSNR。本研究综合衡量两种指标,压缩免疫组化图像时,PCA算法相对于SVD算法质量更高且更稳定,波动的标准差在0.5以内,并与原图相比,SSIM近乎为1。压缩HE染色图像时,SVD 可在很大程度上保证继承原图像的大量信息,重建所得图像与原图像SSIM高于PCA算法。本研究发现,宫颈癌分化程度对组织图像压缩具有一定影响,相较于低、高分化,中分化宫颈癌组织图像压缩重建后SSIM稍低,推测原因:低、高分化宫颈癌一般具有典型的组织学形态,图像像素分布较为集中,因此压缩后重建效果较好。
综上,PCA和SVD两种算法均具有出色的图像压缩和重建功能,其中PCA技术稳定性相对更高,为解决病理图像存储难题提供了解决方案,值得临床进一步推广应用。
作者贡献:何睿琳负责结果分析、论文初稿撰写;刘子妤、杨欣怡负责临床试验;李晨、李晓燕构思论文框架、审核并修订论文。
利益冲突:所有作者均声明不存在利益冲突
