牛科物种FEZF2基因分子特征、功能预测及进化分析*
2022-07-28保志鹏涂兴调张自芳谷丽娇范新阳钱林东苗永旺
保志鹏,涂兴调,张自芳,谷丽娇,范新阳,钱林东,苗永旺
(1.云南农业职业技术学院 畜牧兽医学院,云南 昆明 650212;2.云南农业大学 动物科学技术学院,云南 昆明 650201)
FEZ家族锌指蛋白2 (FEZ family zinc finger protein 2,FEZF2)又称前脑胚胎锌指样蛋白2(forebrain embryonic zinc finger-like protein 2),是锌指蛋白超基因家族的重要成员,包含6个C2H2锌指结构和1个甘氨酸重复区域[1-5]。FEZF2作为一种转录激活/抑制因子,参与神经元发育的转录调控[1-4]。研究发现:动物神经元发育通过下游细胞因子的表达与机体免疫关联[2,5-7]。奶牛乳腺炎的易感性与信号素5A (semaphorin 5A,SEMA5A)的转录活性下降而导致的免疫反应受损有关[2]。乳腺炎可诱导FEZF2基因在乳腺中表达,导致SEMA5A的表达上调,进而诱导与机体免疫相关的TNF-α和IL-8等多个基因表达[2]。在乳腺炎中,FEZF2均能诱导SEMA5A表达上调,但在易感奶牛中其表达水平低于乳腺炎抗性牛[2]。在奶牛中发现:FEZF2基因编码产物的多个甘氨酸残基重复区存在12G重复和13G重复2种等位基因,13G型的FEZF2等位基因降低了SEMA5A的表达水平,导致13G/13G基因型奶牛对乳腺炎易感,发病率是12G/12G 基因型奶牛的2倍[8]。FEZF2基因位于普通牛第22号染色体,包含6个外显子和5个内含子,编码序列 (coding sequence,CDS)全长为1 380 bp (登录号:NM_001038198.2);水牛FEZF2基因定位于第21号染色体,包含9个外显子和8个内含子,CDS全长也为1 380 bp (登录号:XM_045163982.1)。魏伟等[5]对河流型和沼泽型水牛群体FEZF2基因的检测发现:河流型水牛中存在c.165G>A,属于同义替换,且河流型与沼泽型水牛的FEZF2基因多个甘氨酸残基重复编码区都为13G型。张广乐等[6]研究发现:FEZF2基因在12个水牛组织中都有表达,其中在大脑的表达量最高,该基因可能在水牛中作为一类核内转录因子广泛参与基因的表达调控过程。FEZF2分别位于山羊的第22号、绵羊的第19号染色体上,它们都含有5个外显子和4个内含子,编码区长度与普通牛和水牛基本一致。
牛科家养动物包括普通牛、水牛、牦牛、绵羊和山羊等,具有奶用、肉用和役用等多种用途,在全球畜牧业生产中发挥着重要作用。FEZF2作为一种转录因子,参与神经元的发育,可能与动物的先天免疫和奶牛的乳腺炎紧密相关[1-4],但目前有关牛科物种FEZF2基因的研究还十分有限。基于此,本研究从NCBI和Ensembl数据库下载牛科家畜FEZF2基因及其编码蛋白序列,并以下载的非牛科动物的同源序列为对照,采用生物信息学和比较基因组学方法,对牛科主要家畜FEZF2基因的结构、编码蛋白的氨基酸组成、理化特征、结构、进化关系、参与的生物学路径及分子功能等进行深入的比较分析,旨在解析牛科物种FEZF2基因的分子特征、基本功能及进化关系,为深入研究FEZF2基因调控动物免疫的机制和牛科家养动物的抗病育种提供参考。
1 材料与方法
1.1 FEZF2序列及其注释文件的获取
用于数据分析的水牛、普通牛、瘤牛、杂交牛、牦牛、山羊和绵羊等牛科物种的FEZF2基因及其编码蛋白序列以及用于比较分析的一些非牛科动物的同源序列,均从NCBI数据库(https://www.ncbi.nlm.nih.gov/)和Ensembl数据库(http://asia.ensembl.org/index.html)下载。经初步比较分析,过滤不完整的序列或可能存在错误的序列后,用于本研究的序列数据信息见表1。用于数据分析的各物种基因组注释GTF文件由NCBI基因组数据库(https://www.ncbi.nlm.nih.gov/genome/?term=)下载得到。

表1 FEZF2基因序列的来源Tab.1 Sequence source of FEZF2 gene
1.2 基因结构、一致性及差异分析
从NCBI数据库下载各物种FEZF2基因的核苷酸序列,然后用ORF Finder程序(https://www.ncbi.nlm.nih.gov/orffinder/)通过开放阅读框(open reading frame,ORF)查找确定每条序列的CDS,进一步利用Lasergene软件包(DNAStar Inc.,USA)中的MegAlign程序分析各物种序列的一致性;FEZF2基因的碱基组成和核苷酸序列差异采用MEGA 7[9]进行计算或输出。从NCBI数据库下载牛科家畜及常见非牛科动物的基因组注释GTF文件,使用TBtools软件[10]对每个物种的FEZF2基因转录本进行信息完整化处理,进一步使用在线软件Gene Structure Display Server (http://gsds.gao-ab.org/)对每个物种的基因转录区域结构进行可视化。
1.3 分子特征、进化关系及生物学过程和分子功能
从NCBI数据库下载FEZF2基因的氨基酸序列,利用MegAlign程序分析各物种的序列一致性;采用MEGA 7[9]分析氨基酸序列差异并构建FEZF2蛋白氨基酸序列的系统发育树;将FEZF2蛋白氨基酸序列提交至MEME Suite网站(https://meme-suite.org/meme/)获得其基序结构信息;将各物种蛋白序列提交至NCBI的Conserved Domains数据库(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)获得保守结构域。采用TBtools程序整合基序和保守结构域的分析结果。序列一致性、基序和保守结构域分析所使用的序列与基因结构分析使用的序列相对应。
FEZF2蛋白的理化特征与疏水性、信号肽和跨膜结构域采用在线软件ProtParam (https://web.expasy.org/protparam/)、ProtScale (https://web.expa sy.org/protscale/)、SignalP 5.0 server (http://www.cbs.dtu.dk/services/SignalP/)和TMHMM 2.0 server (http://www.cbs.dtu.dk/services/TMHMM/)分别进行预测;蛋白质亚细胞定位及其二级和三级结构分别采用在线软件ProtComp (http://linux1.softberry.com/berry.phtml)、SOPMA (http://npsa-pbil.ib cp.fr/)和SWISS-MODEL (http://swissmodel.expasy.org/)进行分析。与FEZF2蛋白相互作用的蛋白、FEZF2参与的生物学过程和分子功能分别使用在线工具STRING (https://string-db.org/)和InterPro (http://www.ebi.ac.uk/interpro/search/sequencesearch)进行分析。
2 结果与分析
2.1 牛科物种FEZF2基因序列分析
2.1.1 编码区碱基组成、序列一致性与差异
由表2可知:牛科物种间FEZF2基因编码区的碱基组成相似,与非牛科物种的编码区碱基组成差异较大。在牛科物种中,表现为牛属和羊属动物的属内一致性更高,水牛与牛属和羊属动物有一定差异。由表3和图1可知:在FEZF2基因编码区序列一致性上,牛科物种间序列一致性在97.8%~100.0%之间,序列差异性较小;牛科与非牛科哺乳动物序列一致性在88.5%~98.2%之间,序列间差异相对较大;牛科物种与鸡的序列一致性仅在71.8%~72.1%之间,序列差异性最大。值得注意的是,牛科物种FEZF2基因CDS第313~351位核苷酸之间存在1个编码连续甘氨酸的序列区,该区域存在长度多态性,有12G(编码12个连续的甘氨酸)和13G (编码13个连续的甘氨酸) 2种类型,两者在长度上相差1个密码子。除鸡外,非牛科物种在第303位核苷酸之后也存在这个连续编码甘氨酸的序列区,但该区域在非牛科物种间长度差异较大,与牛科物种也不一致。核苷酸位点c.247A、c.480T和c.537T是牛属动物与水牛和羊属动物相互区分的核苷酸位点;c.771T是水牛与牛属动物和羊属动物相互区分的核苷酸位点。

图1 牛科物种FEZF2基因核苷酸序列差异Fig.1 Nucleotide sequence differences of FEZF2 gene in species of Bovidae

表2 FEZF2基因编码区碱基组成Tab.2 Base composition in the coding region of FEZF2 gene
2.1.2 基因转录区的结构
由图2可知:牛科物种FEZF2基因的CDS长度由于在多个连续甘氨酸残基编码区存在长度多态性而略有不同。普通牛和瘤牛的FEZF2基因在该区域都存在12G和13G类型,前者比后者少1个密码子,其CDS长为1 377 bp;在水牛、牦牛、山羊和绵羊中,该区域只存在13G型,CDS长度均为1 380 bp。不同物种间FEZF2基因转录区结构不同,特别是非翻译区 (untranslated region:5′UTR和3′UTR)的长度及内含子长度不一致。在牛科物种中,除瘤牛的FEZF2基因包含2个选择性剪接转录本(它们主要在5′UTR存在差异)外,普通牛、水牛、牦牛、山羊和绵羊均只包含1个转录本。结合表4还可知:水牛FEZF2基因与其他牛科物种的转录区组成与结构差异较大,山羊和绵羊间的转录区组成与结构也相差较大;但牛科物种间FEZF2的CDS区结构组成与共线性关系较为一致,与其他非牛科物种相似性也较大。

表4 牛科和非牛科动物FEZF2基因结构的比较Tab.4 Comparison of FEZF2 gene structure between species of Bovidae and non-Bovidae
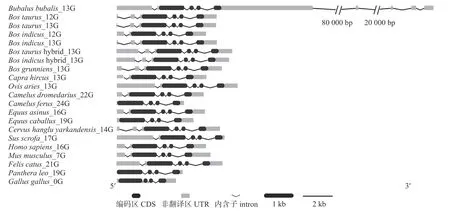
图2 牛科物种及非牛科动物的FEZF2基因结构Fig.2 Structure of FEZF2 gene in Bovidae and non-Bovidae
2.2 氨基酸组成、理化特征及结构分析
2.2.1 FEZF2蛋白的氨基酸组成
由表5可知:在氨基酸组成上,牛科物种间相似性较大,与非牛科动物(特别是鸡)的差异较大。在牛科物种中,水牛、普通牛、瘤牛和牦牛间FEZF2蛋白的氨基酸组成相似性较大,它们与山羊和绵羊在丙氨酸、天冬氨酸、谷氨酸、甘氨酸、蛋氨酸、苏氨酸和缬氨酸等氨基酸组成上存在一定差异。

表5 牛科物种与非牛科动物FEZF2氨基酸组成Tab.5 Amino acid composition of FEZF2 in species of Bovidae and non-Bovidae %
2.2.2 基本理化特征分析
由表6可知:牛科物种FEZF2的基本理化特征基本相似,都是亲水性蛋白质,但水牛属、牛属与羊属动物的属间有一定差异,主要表现在疏水性氨基酸与极性氨基酸的含量、不稳定指数和疏水性等方面。普通牛和瘤牛FEZF2的12G型和13G型除疏水性有一定差异外,其他指标较一致。牛科物种FEZF2的基本理化特征与人和家鼠的特别是与鸡的差异较大。
2.2.3 FEZF2蛋白的基序和结构域
由图3可知:除猪和鸡的FEZF2蛋白含有9个基序外,其他物种的FEZF2蛋白均含有10个基序。预测结果(表6)显示:牛科和非牛科动物的FEZF2蛋白都不含有跨膜结构和信号肽,但都含有1个COG5048保守结构域,由6个C2H2类型的锌指结构域组成(图3和图4),由基序3、2、9和1构成。牛科与和非牛科动物的FEZF2的COG5048保守结构域所在位置一致,在靠近多肽链的C端一侧(牛科动物在多肽链的AA288~440之间)。人、家鼠和猪除含有1个COG5048保守结构域之外,在AA385~441之间还含有1个SFP1超家族结构域;家鼠在AA317~339之间还有1个zf-H2C2_2结构域。

表6 牛科及非牛科物种FEZF2的基本理化特征Tab.6 Basic physicochemical characteristics of FEZF2 for species of Bovidae and non-Bovidae
2.2.4 二级结构组成与三级结构分析
由表7可知:水牛属和牛属动物与羊属动物的FEZF2氨基酸序列中形成各类二级结构的氨基酸数量有一定差异,牛科与非牛科动物差异更大。同源建模显示:构建牛科物种FEZF2蛋白的最佳模型为鼠锌指蛋白568模板(5wjq.1.C)。水牛、普通牛(12/13G)、瘤牛(12/13G)、牦牛和山羊的FEZF2蛋白三维结构与模板一致性为43%,覆盖率为37% (AA275~445之间);绵羊13G的三级结构与5wjq.1.C的覆盖率为37%,一致性为41%,缺少1个锌指结构;预测的模板覆盖区域在COG5048保守结构域区,三维结构显示该区域含有6个典型的锌指结构(绵羊含5个),与结构域分析基本一致。从三维结构来看,牛科物种的属间有一定差异;水牛与普通牛、瘤牛的13G和12G型FEZF2都有差异,但与普通牛13G型差异相对较小(图4)。

表7 形成各物种FEZF2蛋白二级结构的氨基酸数量占比Tab.7 Proportion of amino acids that form the secondary structure of FEZF2 proteins in different species %

图4 预测的牛科动物FEZF2蛋白三级结构Fig.4 Tertiary structure of FEZF2 protein predicted in Bovidae
2.3 亚细胞定位、生物学过程及分子功能
亚细胞定位分析表明:各物种FEZF2定位于细胞核内的支持分数值为10 (满分),表明FEZF2蛋白为在细胞核内发挥功能作用的蛋白质。生物信息分析表明:FEZF2蛋白主要参与轴突成束、细胞去分化、细胞群增殖的负调控、神经元分化的负调节、神经元命运的决定和神经元分化的正向调节等生物学过程。它能与染色质结合,具有DNA结合转录激活或抑制因子活性,能激活或抑制RNA聚合酶II转录的特定基因集转录;还具有能与蛋白编码基因顺式调控序列特异性结合的功能。
2.4 蛋白质互作分析
由图5可知:与各牛科物种的FEZF2蛋白相互作用的蛋白不完全相同,其中共有的互作蛋白主要为BCL11B、CUX1、CUX2、ETV1、FOXP2、KCNN2、NEUROG2、NR2E1、OTP、PAX6、SATB2、SLC38A11、SOX5、TBR1和ZFPM2等,主要为参与胸腺细胞发育过程中T淋巴细胞分化和存活的关键调节因子,参与控制大脑中神经元的增殖和分化的转录因子、转录激活或抑制因子,调节神经元的兴奋性钾离子通道,参与神经元分化的转录调节因子,参与调节视网膜发育核受体亚家族2组E成员,参与下丘脑神经内分泌细胞分化的骨同源盒蛋白,在眼睛、鼻子、中枢神经系统和胰腺的发育中起重要作用的转录因子,在核基质或支架相关区域与DNA结合的蛋白,参与软骨细胞分化和软骨形成转录因子,参与皮质发育(包括神经元迁移、板层和区域特征)以及轴突投射过程的转录抑制因子和具有锌指结构的转录调节因子等。

图5 牛科物种FEZF2蛋白质相互作用网络Fig.5 Interaction network of FEZF2 protein in species of Bovidae
2.5 氨基酸序列一致性与系统发育分析
由图6可知:各牛科物种FEZF2氨基酸序列的一致性在99.7%~100.0%之间;它们与非牛科哺乳动物FEZF2氨基酸序列的一致性在94.1%~99.3%之间;与鸡的序列一致性偏低,在78.9%~79.4%之间。各物种FEZF2氨基酸序列间的差异位点及其连续甘氨酸重复区的位置与差异见图7。

图6 牛科与非牛科物种FEZF2氨基酸序列一致性与分歧度Fig.6 Identify and divergence of FEZF2 amino acid sequences between species of Bovidae and non-Bovidae

图7 牛科与非牛科物种FEZF2氨基酸序列差异Fig.7 Amino acid sequence differences of FEZF2 among the species of Bovidae and non-Bovidae
由图8可知:在系统发育树上,普通牛、瘤牛、牦牛、中亚马鹿、水牛与山羊、绵羊等聚在一大支,支持率为99%;而人、马、驴、单峰驼、野骆驼、人、家鼠、猫和狮子聚在另一支。

图8 牛科和非牛科动物FEZF2的系统发育关系Fig.8 Phylogenetic relationships of FEZF2 in species of Bovidae and non-Bovidae
3 讨论
牛科家养动物是人类奶制品、肉类和畜力的重要来源,在畜牧业经济中占有十分重要的地位。近年研究表明:FEZF2参与动物的神经元发育,是与动物先天免疫和奶牛乳腺炎抗性紧密相关的重要候选基因[1-4,8],但目前对牛科物种FEZF2基因的研究主要针对普通奶牛,对其他牛科家养动物的涉及还十分有限[2-8]。阐明牛科物种FEZF2基因的分子特征、结构、功能及其与非牛科物种的差异,有助于深入理解FEZF2基因与动物神经发育和先天免疫的关联机制,也可为牛科家养动物奶用和肉用性状的抗病育种提供依据。为此,本研究从公共生物大分子数据库和基因组数据库下载了牛科家畜及用于比较的常见非牛科物种的FEZF2基因及其编码蛋白序列,采用生物信息学和比较基因组学方法进行深入的比较分析。结果表明:牛科物种FEZF2基因在CDS长度、碱基组成和CDS结构组成模式上具有较高的一致性;其编码产物的氨基酸组成、序列一致性、理化特征、基序、保守结构域和三维结构等虽具有一定的种属间差异,但一致性非常高,与非牛科动物的一致性也较高。系统发育分析也显示:牛科FEZF2蛋白序列聚集在一大支中,表明牛科家养动物的FEZF2蛋白有着较近的遗传关系。本研究表明:牛科FEZF2蛋白与非牛科哺乳动物具有相似功能,牛科家养动物间一致性更高。
有研究发现:FEZF2蛋白是一种含有C2H2型锌指结构域和连续甘氨酸重复区的转录因子,它能控制单胺能神经元的发育[1,11-12]。FEZF2缺陷的小鼠呈现出过度活跃的行为,揭示FEZF2基因在神经元发育中起作用[7]。SUGIMOTO等[2]报道FEZF2在普通牛大脑中强表达,在乳腺中弱表达。在泌乳期水牛中,FEZF2在大脑中的表达水平最高,在小脑、垂体、心、肺、脾和肾中有较高水平的表达,在乳腺、肌肉、小肠和十二指肠组织中有适量表达[6]。在奶牛中发现,乳腺炎能够诱导乳腺中FEZF2表达,进而上调细胞中SEMA5A表达,进一步引起与机体免疫反应相关的多个基因表达[2,8]。因此,FEZF2基因不仅与神经元发育相关,而且由于神经元发育和免疫之间存在串扰,导致其与先天免疫相关[2,8]。本研究显示:牛科动物FEZF2蛋白都是在核内发挥功能的亲水性蛋白,无信号肽序列及跨膜结构域,都含有1个COG5048保守的锌指结构域,其参与的生物学过程主要与神经元发育调节有关,分子功能主要与染色质结合、转录激活或抑制有关[12-13];与其相互作用的蛋白多为与神经元发育和先天免疫调节密切相关的转录因子或蛋白。结合其在序列和结构上的保守性,推测FEZF2蛋白参与牛科家养动物的神经元发育和与先天免疫相关的生物学过程。
SUGIMOTO等[2,8]研究表明:FEZF2基因中连续甘氨酸重复区的多态性与奶牛乳腺炎抗性相关,但这种多态性对产奶量、乳中脂、蛋白和非脂肪固型物产量的估计育种值无不利影响。该区域13G型等位基因导致奶牛对乳腺炎易感,而12G型等位基因使奶牛对乳腺炎具有抗性,这是因为12G型抗性等位基因促进SEMA5A表达的能力显著强于易感的13G型等位基因,即12G型等位基因引起的免疫应答水平高于13G型的等位基因[2,8]。进一步研究发现:易感的13G型FEZF2含有的甘氨酸重复区能与胰岛素样生长因子1受体(IGF1R)基因5′UTR中较长的胞嘧啶(C)伸展相结合,通过促进IGF1R的表达而抑制乳腺上皮细胞的自噬过程,进而介导对乳腺炎的易感性[3]。携带13G型的FEZF2与5′UTR中含较长C延伸的IGF1R的变异组合的奶牛更容易患乳腺炎[3]。本研究通过序列比对发现:哺乳动物的FEZF2蛋白都存在连续甘氨酸重复区,但各物种间甘氨酸重复的数量有差异。在牛科家养动物中,普通牛和瘤牛的FEZF2在该区域都存在12G和13G型等位基因,而在水牛、牦牛、山羊和绵羊中,该区域只存在13G型等位基因。前期魏伟等[5]对河流型和沼泽型水牛群体检测发现:这两类水牛的FEZF2都为13G型。在水牛、牦牛、山羊和绵羊中,FEZF2蛋白只存在13G型,它是否会导致这些牛科物种乳腺炎易感或先天免疫应答受损有待进一步深入研究。值得注意的是,截至目前,未见沼泽型水牛、牦牛、山羊和绵羊患乳腺炎的报道;奶用河流型水牛乳腺炎的发病率仅约4%,显著低于普通奶牛[14-16]。本研究发现:在氨基酸序列一致性、理化特性和结构上,各牛科物种的FEZF2虽然具有较高的一致性,但仍存在一定的种属间差异,这些差异是否与水牛、牦牛、山羊和绵羊对乳腺炎的抗性有关尚不清楚,因此,有必要通过群体水平的试验观察和进一步的细胞水平功能试验予以证实。
本研究通过序列比对,发现了一些具有种属特征的核苷酸或氨基酸差异位点,这些位点不仅反映了种属间FEZF2功能的差异,而且也可作为种属间相互区分的分子标记。
4 结论
牛科物种的FEZF2基因编码蛋白在氨基酸组成、序列一致性、理化特征和结构上虽然有种属间差异,但一致性较高。在牛科中,水牛、牦牛、绵羊和山羊FEZF2序列的连续甘氨酸重复区为13G型,而普通牛和瘤牛的同源区有12G和13G型。牛科动物FEZF2蛋白都是在核内发挥生物学功能的亲水性蛋白,都含有1个由6个锌指结构组成的COG5048保守结构域,参与的生物学过程主要与神经元发育调节有关,分子功能主要与染色质结合、转录激活或抑制有关。本研究揭示FEZF2蛋白可能参与了牛科物种神经元发育和机体免疫相关基因的表达调控过程。
