胃复春片治疗胃癌前病变作用机制的网络药理学研究*
2022-07-27朱秀芳邱硕程何竹涵王建欣杨继章
朱秀芳,邱硕程,杨 兰,何竹涵,王建欣,,李 超△,杨继章
(1.河北医科大学第一医院,河北 石家庄 050031; 2.河北医科大学药学院,河北 石家庄 050017; 3.清华大学附属北京清华长庚医院,北京 102218)
胃癌的发病率在所有癌症中位列第5,2020年全球死亡人数约76 万,居癌症总死亡人数的第4 位[1],远高于全球癌症死亡平均水平。胃癌的病理发展可经慢性非萎缩性胃炎、慢性萎缩性胃炎、肠上皮化生和异型增生的胃癌前病变多个阶段[2]。目前认为,针对其病因的一级预防难度较大。而胃癌前病变具有一定的可逆性[3],以其治疗作为胃癌二级预防的一部分,在胃癌的治疗中具有重要的现实意义。胃癌前病变属中医“胃脘痛”“胃痞”范畴,其病位在胃,其本在脾。脾虚基础上邪气乘袭,致气滞中焦、痰阻胃络是引起胃癌前病变的重要因素。健脾扶正兼顾理气、驱邪护正是其治疗原则。胃复春片由红参、香茶菜、麸炒枳壳组方,有健脾益气、活血解毒、祛邪功效,能有效抑制或逆转脾虚邪滞所致胃癌前病变的病理进展和“胃脘痛”“胃痞”的表证。但受限于中药成分复杂,其药理机制尚未完全阐明。为此,本研究中采用网络药理学和分子对接技术预测胃复春片治疗胃癌前病变的活性靶点、信号通路及分子基础,探讨其治疗胃癌前病变的作用机制,为其临床使用和深入开发提供理论支持[2]。现报道如下。
1 资料与方法
1.1 活性成分收集和靶点预测
活性成分收集:利用中药系统药理学数据库与分析平台(TCMSP,2.3 版)收集胃复春片的活性成分[ADME/T筛选参数设定为口服利用度(OB)≥30%,类药性(DL)≥0.18],并参考2015 年至2021 年香茶菜相关文献[4−9]进行活性成分补充。
靶点预测:利用TCMSP 预测胃复春片活性成分靶点,并采用Swiss Target Prediction 数据库(http:// www.swisstargetprediction.ch/)预测文献报道的活性成分潜在靶点,纳入Possibility >0.1 的靶点进行补充。通过Uniprot 数据库(https://www.uniprot.org/;更新于2020年12月15日)将蛋白靶点转换为基因靶点。
1.2 胃癌前病变疾病靶点收集
以“precancerous lesions of gastric cancer”“dysplasia of gastric mucosa”“intestinal metaplasia”为关键词,利用Genecards(https:// www.genecards.org,5.8 版,更新于2022 年1 月28 日),OMIM(https:// omim.org/ ,更新于2021 年12 月),DisGeNET(https://www.disgenet.org/,更新于2021 年5 月4 日),Drugbank(https:// go.drugbank.com/ ,5.1.8 版,更新于2021 年1 月3 日)数据库进行检索,收集胃癌前病变相关靶点,并整合去除重复基因。
1.3 活性成分-靶点-疾病网络建立
利用Cytoscape 3.7.1 软件构建中药−成分−靶点网络。将活性成分靶点与疾病靶点导入Venny(https://bioinfogp.cnb.csic.es/tools/venny/index.html,2.1 版)平台制作交集靶点韦恩图,并将交集靶点导入Cytoscape 构建成分−靶点−疾病网络。上述网络节点大小均以节点度(Degree)加权。
1.4 蛋白-蛋白相互作用(PPI)网络构建
将交集靶点导入Cytoscape,利用插件Bisogenet 3.0.0 进行分析和可视化,借助network analyzer 工具计算网络的拓扑参数,以节点度≥2倍中位数为条件筛选主要靶点一级PPI网络。以节点度≥54筛选主要靶点互作(二级PPI)网络,利用cytpNCA 工具对主要PPI 网络再次进行拓扑学分析,计算网络的介度(Betweenness)、紧密度(Closeness)、LAC、邻域连通性参数(Neighborhood Connectivity),以主要参数介度、紧密度、节点度同时位于前10%(即介度≥3 643.482、紧密度≥0.517 169、节点度≥223.6)筛选,得三级PPI 网络,最后以次要参数LAC、邻域连通性同时不低于其中位数(即LAC ≥32.63、邻域连通性≥50)筛选,得到核心靶点PPI网络。最后以节点度对节点的大小和颜色进行加权处理,对核心PPI网络进行优化,节点越大,颜色越偏向暖色,则说明其参与蛋白相互作用的环节越多。
1.5 富集分析
将得到的核心靶点导入Metascape(http://metascape.org/ gp/ index.html#/ main/ step1,更新于2021 年12 月18 日)进行基因本体论(GO)和京都基因与基因组百科全书(KEGG)通路富集分析。其中GO 分析包括细胞组分(CC)、生物过程(BP)、分子功能(MF),参数设置,P<0.01、最小计数(Min Overlap)为5、最小富集(Min Enrichment)>1.5。以Kappa 评分作为层次聚类的相似性指标,将相似性>0.3的子树条目视为一个簇,以簇内统计学意义最大的项来代表该簇。
1.6 分子对接
将核心靶点与中药小分子配体相匹配,从PubChem数据库中下载核心靶点所对应的分子配体2D 结构,导入Chem 3D(20.0.0.41版)软件,利用其构建配体的3D结构并进行最小结合能优化,保存为mol2 文件。利用PDB 数据库(http:// www.rcsb.org/)下载核心靶点蛋白受体的3D 结构,通过PyMOL 2.5.2 软件对靶点蛋白3D 结构进行去水、去配体处理。将处理后的受体导入AutoDock Tools 1.5.7软件,进行加氢处理,保存为pdbqt文件。于AutoDock Tools中设置配体受体结合的Grid Box参数,以AutoDock Vina 1.1.2 软件进行分子对接,通过PyMOL软件将结果可视化并标注配体受体的结合关系。
2 结果
2.1 成分分析及成分-靶点网络
共收集活性成分50 个,其中红参18 个,香茶菜26 个,枳壳6个。汇总得活性成分潜在靶点512个,见表1。中药−成分−靶点网络见图1,共包含550 个节点、1 859条线。

图1 中药-成分-靶点网络Fig.1 Traditional Chinese medicine-components-target networks

表1 胃复春片活性成分及其靶点Tab.1 Active components and targets of Weifuchun Tablets
2.2 靶点收集及活性成分-靶点-疾病互作网络
共获得胃癌前病变活性靶点2 093 个。韦恩图发现208 个共有靶点(见图2)。成分−靶点−疾病网络图有246个节点和347条线(见图3)。节点度较大的5个活性成分分别为XCC1,XCC13,ZK2,ZK3,A,面积越大的成分参与的生物功能越多,在胃癌前病变的治疗中发挥的作用越大。
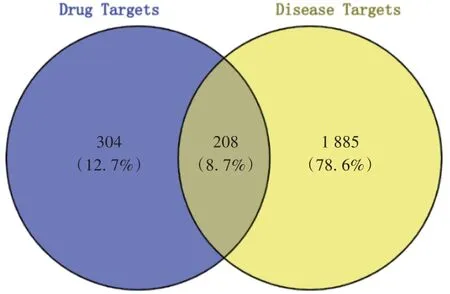
图2 活性成分-疾病靶点韦恩图Fig.2 Wayne diagram of active components-disease targets

图3 成分-靶点-疾病网络图Fig.3 Network of components-targets-disease
2.3 胃复春片治疗胃癌前病变PPI 网络
依次可得含7 180 个节点、172 088 条线的一级PPI网络,含1 858 个节点、79 667 条线的二级PPI 网络,含133 个节点、3 162 条线的三级PPI 网络,含66 个节点、1 109 条线的核心靶点PPI 网络。筛选流程见图4,节点度加权优化后核心靶点PPI 网络见图5。其中NPM1,
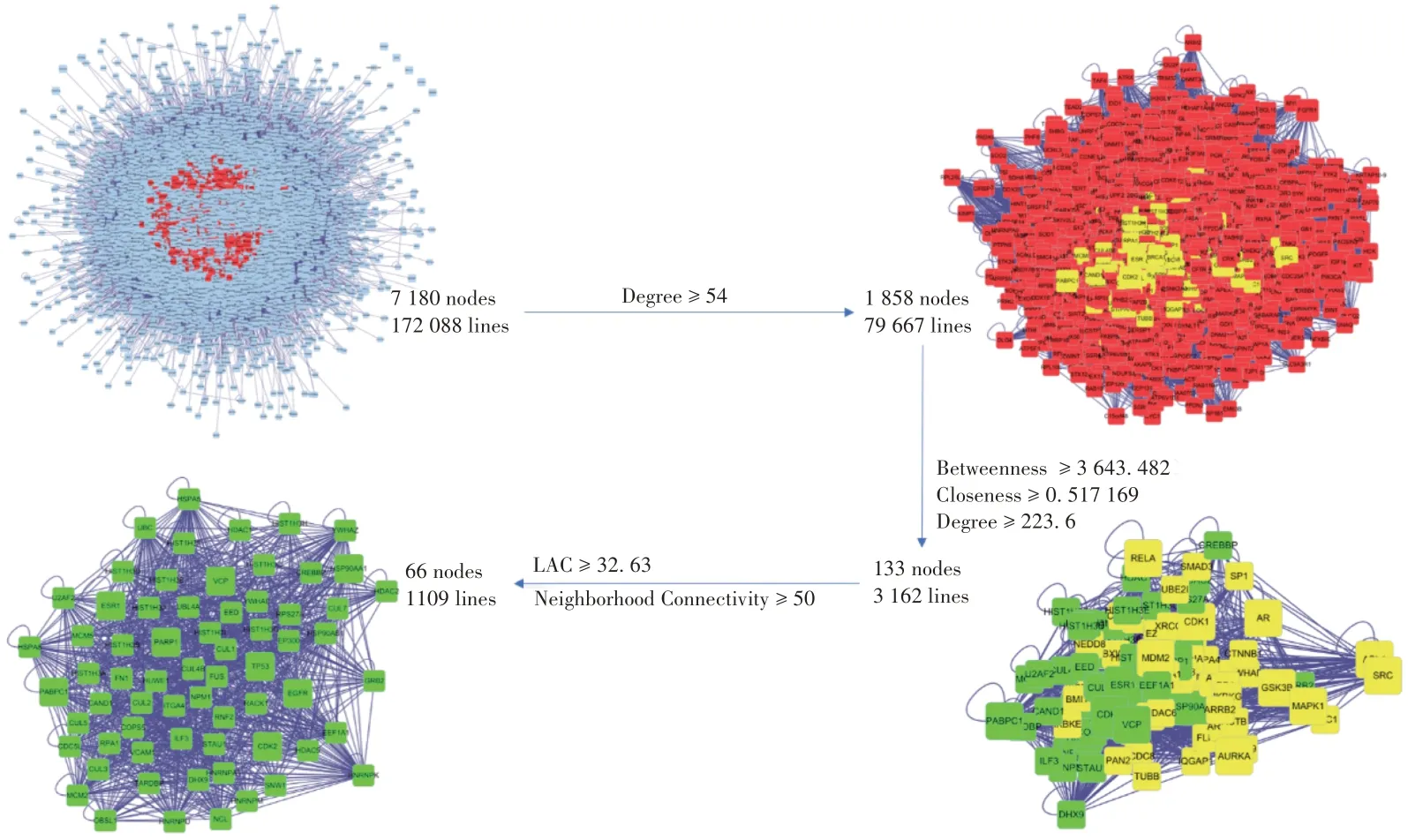
图4 核心PPI网络筛选流程Fig.4 Flow chart of screening PPI network of key targets
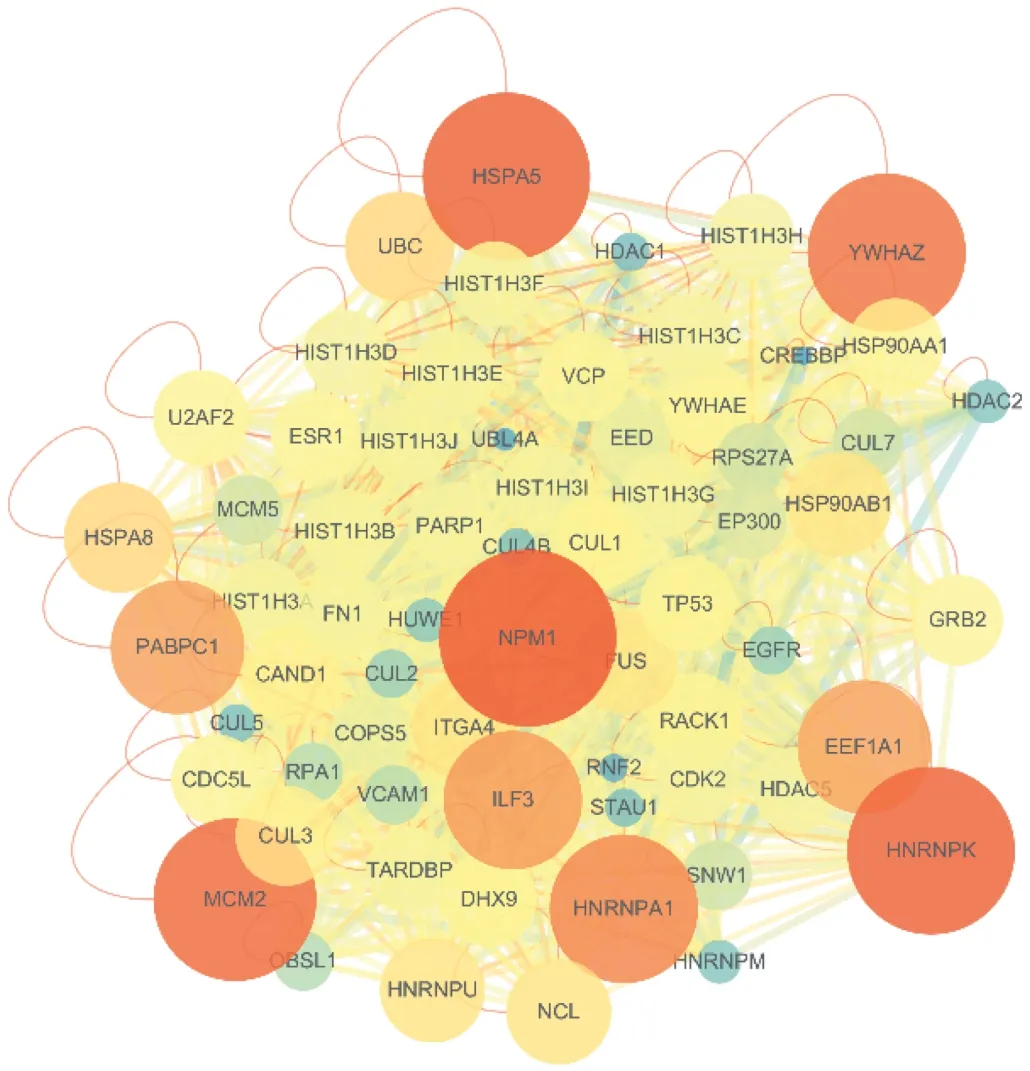
图5 核心靶点PPI网络Fig.5 PPI network of key targets
HSPA5,HNRNPK,YWHAZ,MCM2,HNRNPA1,PABPC1,ILF3,EEF1A1在PPI中发挥的作用较大。
2.4 GO 功能富集分析和KEGG 通路富集分析
GO 功能富集分析中,BP 涉及499 个条目、36 个簇,参与包括基因沉默的调控、mRNA 代谢过程的调节、细胞对DNA 损伤刺激的反应、生物节律过程、病毒过程、mRNA 代谢过程的负调控、内在凋亡信号通路等过程(图6 A);CC 涉及57个条目、16个簇,涵盖核染色体、核糖核蛋白复合体、转移酶复合物、富ficolin −1颗粒腔等(图6 B);MF 涉及87 个条目、21 个簇,包括与细胞黏附分子、与泛素蛋白连接酶、与蛋白质结构域(特异性)、与转录因子、与组蛋白脱乙酰基酶、与蛋白激酶等的结合(图6 C)。KEGG 通路富集分析中,94 条通路、11 个簇富集有潜在靶点,涉及乙醇成瘾(Alcoholism)、细胞周期(Cell cycle)、前列腺癌(Prostate cancer)、剪接体(Spliceosome)、PI3K −Akt 信号通路(PI3K −Akt signaling pathway)、泛素介导的蛋白水解(Ubiquitin mediated proteolysis)等通路(图6 D)。这些通路与多种癌症和细胞异常增殖与表达有关。
2.5 分子对接
最终选取ESR1,HSP90AA1,PARP1,VCP,EGFR,TP53,PABPC1,CDK2 这8 个核心靶点作为蛋白受体和其对应的14个小分子作为配体,并对其进行分子对接。配体与受体间氢键以黄色虚线表示,并标注连接处的蛋白残基名称。配体受体间结合活性最好的前11 对相互作用结果见表2,对接结果见图7。
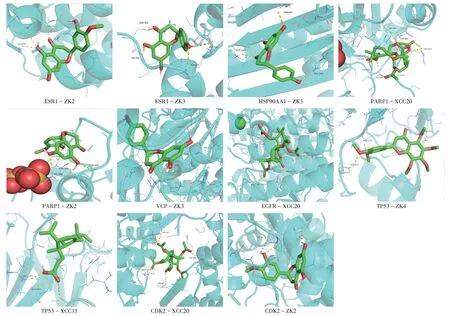
图7 分子对接结果Fig.7 Results of molecular docking

表2 分子对接配体受体相互作用Tab.2 Interaction of molecular docking ligand receptor
3 讨论
胃癌的发生是一个连续恶化的病理过程,目前最经济有效的方式是在胃癌前病变阶段进行治疗并逆转疾病发展[10]。而西医对此多以对症治疗和抗幽门螺杆菌治疗为主,并无特异性化学药物治疗方案。中医治疗理念多注重整体观念及辨证论治,以中医理论为依据采用中医药对胃癌前病变进行干预获得临床广泛认可[11−12]。胃复春片为治疗胃癌前病变常见中成药,虽其疗效已获证实[13],但受限于复杂的化学成分体系,传统的药理学方法很难找到其作用的靶点和通路[14],临床应用缺乏理论支持。网络药理学以系统生物学为基础,能建立起活性成分、靶点分子、生物功能间的复杂网络,从而预测可能的药理作用机制[14]。分子对接技术借助大数据分析,通过高性能计算准确地模拟小分子配体与蛋白受体间的互作关系,在药物设计领域应用广泛[15]。本研究中借助网络药理学联合分子对接的方法,建立成分−靶点−疾病网络,探究胃复春片治疗胃癌前病变的靶点通路关系,并通过分子对接技术,进一步在分子−蛋白互作层面模拟其结合活性。
本研究中经数据库筛选和靶点预测,最终得到活性成分50 个,与胃癌前病变相关靶点208 个,交集靶点主要为胃复春片中的3,4 −secoisopimara −4(18),7,15 −triene −3 −oic acid、chrysosplenol D、hesperetin、naringenin 和β−sitosterol。经PPI网络分析,其药理作用与NPM1,HSPA5,HNRNPK,YWHAZ,MCM2,HNRNPA1,PABPC1,ILF3,EEF1A1等核心靶点有关。经GO 功能和KEGG通路富集分析,胃复春片治疗胃癌前病变时与核染色体、核糖核蛋白复合体、转移酶复合物等作用,经基因沉默的调控、mRNA 代谢过程的调节、DNA损伤刺激的调节等过程与细胞黏附分子、泛素蛋白连接酶、蛋白质结构域等结合,通过乙醇成瘾、细胞周期、前列腺癌、PI3K −Akt 信号通路等通路发挥疗效。此外分子对接结果显示,其小分子活性成分能与HIS及GLY等多个核心靶点蛋白残基单位结合,有较好的结合活性,进一步证实胃复春片在分子−蛋白相互作用层面的药理作用机制。
PI3K 在包括胃癌、前列腺癌在内的多种恶性肿瘤中起重要的信号转导作用。PI3K/ AKT 信号通路的激活,可促进癌症细胞增殖、生长和迁移等环节,与胃黏膜上皮细胞异型增生有密不可分的关系[16]。WANG等[2]曾通过网络药理学联合体外实验验证的方法,证明抑制HRAS −PI3K −AKT 通路能抑制下游MDM2 及p21等蛋白的表达,从而抑制异常表达的细胞停留在G2/M期。此外,通过抑制剪接体通路、破坏剪接体活性,可抑制Wnt 相关通路的蛋白表达。而现有研究表明,包括Wnt/β −catenin 在内的Wnt 相关通路在胃癌中呈高表达[17−18]。虽目前尚未有以抑制乙醇作用通路来探究其对胃癌前病变或胃癌的抑制作用,但近年来,乙醇在胃食管癌中的研究结果表明,乙醇成瘾通路的激活或成为胃癌前病变发展中的一个重要环节[19]。
目前,胃复春片治疗胃癌前病变的药理学研究多以改善胃黏膜微生态入手[20]。本研究中利用网络药理学方法预测了胃复春片在胃癌前病变治疗中的潜在靶点和作用通路,并首次结合分子对接技术,模拟了活性成分与靶点蛋白的结合活性,为探究中药胃复春片在胃癌前病变治疗中的药理机制提供了可靠参考。由于网络药理学方法是基于系统生物学大数据分析来预测其可能的药理机制,不同的算法和模型均有可能对其准确性产生影响,故本研究存在一定的局限性,后续仍需进一步进行动物实验加以验证。
综上所述,经本研究结果可预测,胃复春片在胃癌前病变治疗中,以chrysosplenol D,hesperetin 等多种活性成分,作用 于NPM1,HSPA5,HNRNPK,YWHAZ,MCM2,HNRNPA1,PABPC1,ILF3,EEF1A1 等核心靶点,通过PI3K −Akt 信号通路、乙醇成瘾、细胞周期、前列腺癌、剪接体等通路,实现其调控细胞异常增殖、生长、凋亡的功能,从而发挥治疗胃癌前病变的作用。
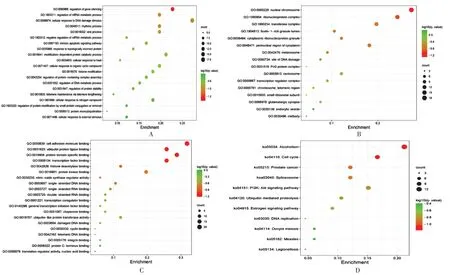
A.GO −BP B.GO −CC C.GO −MF D.KEGG信号通路图6 富集分析结果A.GO −BP B.GO −CC C.GO −MF D.KEGG signaling pathwayFig.6 Results of enrichment analysis
