一种基于遗传算法的PPI网络全局比对算法
2022-07-06陈悦,陈璟,2
陈 悦,陈 璟,2
1(江南大学 人工智能与计算机学院,江苏 无锡 214122) 2(江南大学 江苏省模式识别与计算智能工程实验室,江苏 无锡 214122)
1 引 言
随着高通量筛选技术和计算机技术的发展,人们可获得的PPI(protein-protein interaction,PPI)数据[1]与日俱增,对这些数据进行有效的分析有助于对同源蛋白质、蛋白质功能模块和蛋白质功能预测等的研究.网络比对是研究PPI网络数据的一个重要方法,它通过匹配不同PPI网络的节点,在源网络和目标网络的节点之间形成一种映射关系.
目前已经提出的成对网络全局比对的经典算法,大体可分为两类:2步算法和基于目标函数的搜索算法.2步算法的第1步是计算两个输入网络的节点相似性,第2步是根据第1步计算的相似性得分将网络比对问题转化为最大权重的二部图匹配问题.基于目标函数的搜索算法首先提出一个目标函数,然后利用启发式搜索方法去优化目标函数.
IsoRank[2]算法是全局比对算法中的先驱,首先使用PageRank算法基于节点拓扑相似性和序列相似性计算两个网络任意节点对之间的相似度,然后使用贪心算法匹配相似性高的节点对得到比对结果.GRAAL[3]算法首次提出用度标签相似性作为节点的拓扑相似性指标.PROPER[4]算法假设蛋白质序列的高相似度代表着功能的高相似度,因此,PROPER首先匹配序列相似度高的节点对,然后在该结果的基础上逐步完善比对结果.SPINAL[5]算法基于局部邻域匹配构建初始相似性矩阵并由此得到粗粒度的比对结果,继而使用种子扩展和局部搜索的方法得到细粒度结果.PSONA算法[6]提出一种基于permutation的粒子群优化算法来搜索生物网络的最优比对.MeAlign算法[7]将遗传算法和局部搜索算法相结合来寻找最优比对结果.MAGNA[8]算法随机产生初始种群并利用遗传算法迭代得到最优比对结果,MAGNA++[9]是MAGNA的一个扩展,选取拓扑指标作为目标函数,相较MAGNA取得了更好的拓扑特性,并提供了图形界面.MAGNA++的比对结果随着种群大小和迭代次数的增加而变好,但其目标函数收敛慢,且MAGNA++使用拓扑指标作为目标函数,导致其比对结果生物质量低.
为在比对结果的拓扑特性和生物特性上取得均衡的高指标,本文提出一种新的网络比对算法NABG,主要贡献如下:
1)基于节点在网络中的重要性得分计算节点对的拓扑相似性,使得网络中较为重要的节点更可能被比对上;
2)提出一种结合节点对相似性得分和边的保守性的目标函数,在迭代比对结果的过程中同时优化节点和边;
3)改进遗传算法的初始化、选择和交叉的过程,减少后代种群对初始种群的依赖,加快种群的收敛速度.
2 算法介绍
2.1 问题定义
在分析PPI网络数据时,通常会将其抽象成图模型[10]:将蛋白质抽象为网络节点,蛋白质间的相互作用关系抽象为边.两个PPI网络分别用无向图G1= (V1,E1) 和G2= (V2,E2)表示,其中V1、V2表示网络中的节点集合且|V1|≤|V2|,E1、E2表示网络中的边集合.成对PPI网络一对一映射的全局比对即在两个网络之间找到一种映射关系f,使得G1网络中的每个节点u唯一地比对到G2网络中的某一节点v,即v=f(u).
2.2 网络节点拓扑相似性
网络中通常会包含一些拓扑结构重要性高的节点,比如瓶颈节点和枢纽节点.节点的结构重要性一般用去掉该点后引起网络结构上的变化程度来衡量.在生物分子网络中,一个蛋白质节点在功能上的重要性也可以通过去掉该节点后引起的网络功能或者有机体适应度上的变化程度来衡量.在生物的生存或者繁殖过程中不可或缺的蛋白质/基因被称为必须蛋白质/基因,而拓扑重要性高的节点更可能成为必须蛋白质[11].在PPI网络中,一个节点的功能重要性被认为与其在网络中的拓扑结构重要性有关[11].
必须蛋白质节点在拓扑结构和功能上重要性高,这些节点变异的速度较慢,通常会更具有保守性.因此,NABG采用最小度启发式算法[12]计算网络中节点和边的重要性得分NIS(u),EIS(u,ui),并基于此计算节点对的拓扑相似性得分T(u,v).
为充分挖掘网络节点的拓扑信息,本文在计算节点对拓扑相似性得分时同时考虑节点与其相邻边的重要性得分:
(1)
公式(1)中,节点u,v分别是网络G1、G2中的节点,N(u)表示节点u的邻居节点集合,Max(V)表示网络中NIS(u)+∑ui∈N(u)EIS(u,ui)的最大值.
节点和边的重要性得分NIS(u),EIS(u,ui)计算方式如下:
NIS(u)的初始值设置为0,若在网络中存在边(u,ui),则EIS(u,ui)的初始值设置为1,否则设置为0.从度为1 的节点开始,到度为10的节点结束,删除当前度最小的节点u,并更新其相邻节点和边的权重,如公式(2)、公式(3)所示:
当|N(u)|=1时:
∀ui∈N(u),NIS(ui)=NIS(ui)+EIS(u,ui)
(2)
当|N(u)|>1时:

(3)
2.3 网络间节点对相似性
蛋白质的序列信息是获取生物信息的一个重要来源,而网络拓扑结构和蛋白质序列可能成为获取生物信息的互补信息[13],因此NABG在节点对的相似性得分S(u,v)中同时引入节点对的拓扑相似性得分T(u,v)和生物相似性得分B(u,v):
S(u,v)=αB(u,v)+(1-α)T(u,v)
(4)
其中α(0<α<1)控制节点对拓扑相似性得分和序列相似性得分的权重,B(u,v)表示节点对(u,v)的归一化bit-score值,即从输入的序列相似性文件中读取相应数值并进行归一化处理.
2.4 目标函数
NABG提出一种新的目标函数:由节点对的相似性得分和边正确性[13](Edge Correctness,EC)得分构成,同时优化节点对相似性和边的保守性,达到优化比对结果的拓扑特性和生物特性的目的.
源网络的边与目标网络的边比对上,称为保守边.EC是保守边与源网络边数的比例.
(5)
(6)
其中,|E1|表示G1网络中边的个数,|f(E1)|表示保守边的个数.A表示一个比对结果,F(A)表示比对结果A的目标函数得分,S(u,v)表示节点对(u,v)的序列相似性得分.
2.5 遗传算法应用
本文受MAGNA++算法启发,将比对结果类比为种群个体,将比对结果的优化过程类比为种群进化过程.设置种群大小为p,则种群集合为{A0,A1,...,Ap-1}.
2.5.1 种群初始化
1)初始化A0: 将两个网络中的任意节点对按照相似性得分S从大到小排列,优先比对相似性得分高的节点对,直到G1网络中的每个节点都被唯一地比对上.
2)初始化{A1,A2,...,Ap-1}: 随机产生p-1个比对结果.
2.5.2 种群适应度评估
NABG将网络比对的目标函数类比为种群个体的适应度函数,并依据适应度值为种群个体排序.
2.5.3 选择、交叉
保留上一代种群p/2个较优的个体作为下一代样本,并选择上一代种群的第i(0≤i
NABG使用Saraph[8]提出的交叉函数产生后代个体.该交叉算子利用Knuths正则分解和循环分解算法利用两个父代个体交叉产生一个子代个体,保证子代个体可以继承两个父代个体几乎各一半的特性.
2.6 算法流程
算法1.NABG算法
输入:源网络G1、目标网络G2,序列相似性文件,种群大小p,迭代次数阈值n_gen
输出:比对结果A
Begin
1.使用公式(2)、公式(3)计算节点和边重要性得分NIS、EIS;
2.使用公式(1)、公式(4)计算节点对相似性得分S(u,v);
3.根据步骤2的节点对相似性得分初始化种群{A0,A1,...,Ap-1};
4.使用公式(6)计算种群个体适应度值F(A)并排序;
5.选择、交叉产生下一代;
6.重复步骤4、步骤5直到目标函数收敛或者迭代次数达到最大阈值.
End
NABG算法流程如图1所示.
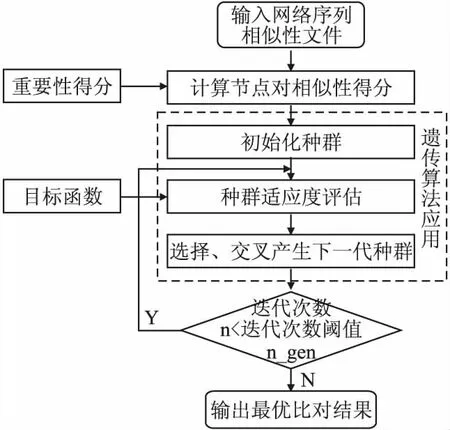
图1 NABG算法流程图Fig.1 Flow chart of NABG algorithm
3 实验结果和分析
3.1 实验数据
本文在真实网络数据和合成网络数据上分别进行了3组实验.真实网络数据是来自Isobase[14]数据库的3种真核生物的网络数据:秀丽隐杆线虫(Caenorhabditis Elegans,CE)、黑腹果蝇(Drosophila Melanogaster,DM)和酿酒酵母(Saccharomyces Cerevisiae,SC).合成网络数据来自NAPAbench[15]数据库.几种生物网络的节点和边信息见表1、表2.

表1 真实网络数据Table 1 Real network data

表2 合成网络数据Table 2 Synthesis network data
3.2 评价指标
3.2.1 拓扑指标
1)边正确性EC
边正确性是保守边与源网络边数的比例.EC无法惩罚稀疏网络到密集网络的比对.
2)诱导保守子结构得分ICS
诱导保守子结构(Induced Conserved Structure,ICS)得分计算保守边和诱导边的比例,诱导边是源网络中比对上的节点形成的集合在目标网络中所含有的边的数量.ICS无法惩罚稠密网络到稀疏网络的比对.
(7)
其中,G2(f(V1))表示G2中比对上的所有节点,|E(G2(f(V1)))|表示G2的诱导子网络的边数.
3)对称子结构得分S3
对称子结构得分(Symmetric Substructure Score,S3)是EC和ICS的结合,既能惩罚稀疏网络到密集网络的比对,又能惩罚稠密网络到稀疏网络的比对.
(8)
3.2.2 生物指标
生物网络比对的目的是寻找具有生物学意义的比对结果,在某种程度上,比对结果是否具有生物功能一致性相较拓扑保守性更为重要.
1)平均归一化熵MNE
平均归一化熵(Mean Normalized Entropy,MNE)是归一化熵(Normalized Entropy,NE)的平均值.MNE的值越小,表示比对结果的生物一致性越高,比对质量越好.
(9)
其中,d是节点对中蛋白质被注释的GO注释数量,pi是被GOi注释的蛋白质与所有被注释的蛋白的比例.
2)特异性Specificity
成对网络比对中,特异性(Specificity)是指被同种类型的GO项注释的蛋白质对占所有比对上的被注释的蛋白质对的百分比.
3)GO一致性GOC
基因同源一致性(Gene Ontology Consistency,GOC)是基于比对节点的GO一致性来衡量比对结果生物特性的常用方法.
(10)
其中,GO(i)表示节点i被注释的GO项集合,ai表示与节点i比对上的节点.
3.3 实验结果及分析
NABG使用参数α(0<α<1)平衡节点对的拓扑与生物相似性得分.如图2所示,在CG网络上对比不同的α取值产生的比对结果.当α>0.4时,生物指标MNE和Specificity趋向收敛,变化不大;α=0.5时拓扑指标EC、ICS和S3取得最优值,因此本文在后续实验过程中参数α取0.5.

图2 参数α实验Fig.2 Experiments on α
为衡量NABG算法比对结果的质量,本文使用真实网络CE-DM,CE-SC,DM-SC和合成网络CG、DMC、DMR各3组实验数据进行实验并分别与MAGNA++、PROPER和SPINAL算法的比对结果比较分析.NABG算法中α=0.5,p=5000,n_gen=3000,MAGNA++、PROPER和SPINAL算法的参数设置参照原文.
3.3.1 与MAGNA++结果比较分析
NABG与MAGNA++都是基于遗传算法的成对生物网络比对算法,且NABG在迭代过程中使用了MAGNA算法的交叉算子,因此本文首先与MAGNA++算法进行对比实验.MAGNA++算法的比对结果随着物种数目以及迭代次数的增加而变好,本文以CG合成网络为例,将NABG(p=5000)分别与MAGNA++初始物种p为5000和p取默认参数15000时产生的比对结果比较分析.
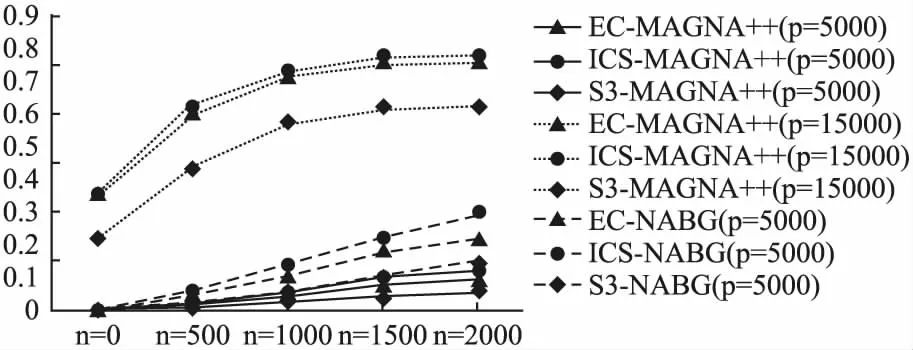
图3 NABG与MAGNA++拓扑指标得分Fig.3 Score of NABG and MAGNA++ on topological metrics
在拓扑指标上,分别依据EC、ICS、S3指标对比各算法在迭代了n(n=0,500,100,1500,2000)次后产生的比对结果,如图3所示.在生物指标上,分别依据MEN和Specificity指标对比实验结果,如图4、图5所示.
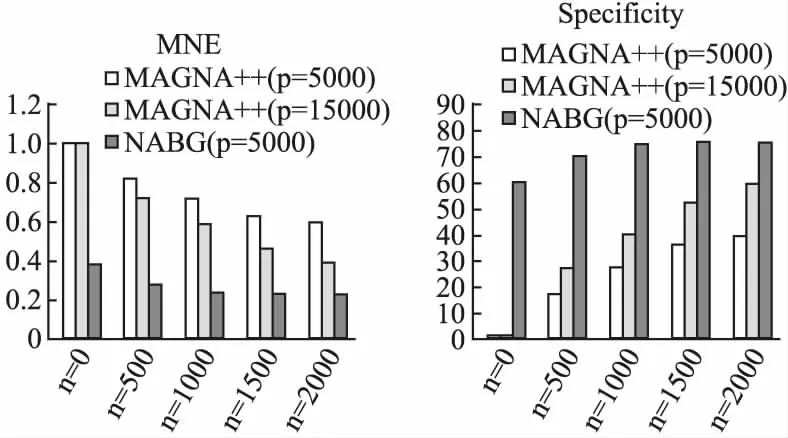

实验表明,NABG和MAGNA++算法比对结果的拓扑质量和生物质量随着迭代次数的增加而提高,并且NABG算法的收敛速度快于MAGNA++.在各项拓扑指标以及生物指标中,NABG(p=5000)的比对结果明显优于MAGNA++(p=5000)和MAGNA++(p=15000)的比对结果.
3.3.2 合成网络实验比对结果分析
4种算法在合成网络上的比对结果如表3所示,分别依据EC、ICS、S3、MNE、Specificity 5项指标对比4种算法.表3中指标最高的数值用黑体标出,次高的数值用下划线标出.从表3中可以看出,MAGNA++算法和PROPER算法在各项指标中表现最差.在拓扑和生物指标上,NABG的结果在各组实验中均取得较好的结果,与SPINAL不相上下.

表3 在合成网络上的实验结果Table 3 Experiments on synthesis networks
3.3.3 真实网络实验比对结果分析
在真实网络上,分别依据EC、ICS、S3、GOC这4项指标对比4种算法,各项指标对比结果如表4所示.表4中指标高的数值用黑体标出,次高的数值用下划线标出.在CE-DM,CE-SC,DM-SC 3对物种的实验结果中,MAGNA++算法的表现最差,SPINAL算法的拓扑指标较好而生物指标很差,PROPER算法生物指标较好而拓扑指标较差.NABG算法的生物指标优于PROPER算法,并且拓扑指标与SPINAL算法较为接近.

表4 在真实网络上的实验结果Table 4 Experiments on real networks
3.3.4 比对结果生物学意义分析
当一个节点对不存在公共的GO注释项时,这个节点对被认为不具有功能相似性[16].节点对被注释的公共GO项越多,比对越具有生物学意义[16,17].为了进一步分析各算法比对结果的生物学意义,本文比较了各算法产生的比对结果中包含c(c>0)个公共GO注释的节点对数目,如图6所示.在CE-SC和DM-SC两组实验中,NABG产生的具有生物学意义的节点对明显多于其它3种算法,在CE-DM实验中,NABG比对结果优于MAGNA++和SPINAL,节点对数目在c=4时少于PROPER.实验表明,NABG算法产生的比对结果相较其他算法更具生物学意义.

图6 公共GO注释项Fig.6 Common GO terms
综合合成网络和真实网络的实验结果来看,4种比对算法中,MAGNA++算法表现最差;SPINAL算法在合成网络中表现最优,但在真实网络中,其生物质量较差;PROPER算法在合成网络中不占优势,在真实网络中生物质量提升但拓扑质量较差;NABG算法在合成网络和真实网络的实验中,其比对结果的拓扑指标和生物指标均能保持均衡的高指标且更具有生物学意义.
4 结束语
本文提出一种新的成对PPI网络全局比对算法NABG,针对PPI网络比对结果难以同时取得好的拓扑特性和生物特性这一问题,在计算节点对的相似性得分时,基于节点和边的重要性得分计算节点对的拓扑相似性,引入生物序列信息,使得在拓扑结构和功能上重要的节点更可能被比对上.NABG算法利用节点对的拓扑和生物信息初始化种群;在种群优化过程中,利用包含节点相似性和边保守性的目标函数优化比对结果.实验表明,NABG可以在拓扑指标和生物指标上取得均衡的高指标,且比对结果具有生物学意义.
