单细胞组学数据库的研究进展
2022-07-01蔡浩洋
蔡浩洋
(四川大学 生命科学学院 生物资源与生态环境教育部重点实验室, 四川 成都 610064)
在过去,被广泛接受的一个观点是细胞个体特征与群体特征具有一致性.因此,研究人员的关注点在于研究对象的宏观水平而非其细胞的个体水平.研究者们得到的数据通常是一群细胞中某种信号的平均值,或者是其中占优势数量的细胞的信号值.然而,近年的研究逐步揭示出单个细胞的个体特征具有明显的异质性[1-2],继而研究人员将关注点从群体水平转移到单细胞水平,单细胞测序技术应运而生.在近10多年里,二代测序、显微镜和微流控技术的改进促使具有单细胞分辨率的各种复杂数据集迅速增加[3-4],在肿瘤学、免疫学、发育生物学甚至植物学研究领域,单细胞测序技术已经逐渐普及,极大地推动了不同生物学领域的研究.因而单细胞测序数据呈现指数级的增长,例如张泽民等[5]组建的“新冠肺炎单细胞中国联盟(SC4)”把单细胞测序技术应用在新冠病毒的研究中,共收集到196个新冠病人的284个样本,超过25 T近150万个细胞的单细胞转录组测序数据[5].随着海量的单细胞测序数据的产生,研究人员希望能通过整合和分析这些庞大的数据来挖掘有价值的信息,其中构建功能性数据库用于存储和分析这些数据成为一种高效的研究方法[4].迄今为止,单细胞相关数据库已经超过了20个,但如何有效检索和利用这些数据库是研究人员需要面对的重要挑战.
1 单细胞测序技术与大数据时代
细胞的功能是由基因表达的不同组合来决定的.自从发现细胞是生命的基本单位以来,研究人员一直试图根据其性质对细胞类型进行表征和分类[6].最初,细胞分类主要以其定位、形状和细胞成分为基础,细胞类型的定义在很大程度上取决于显微镜的改进.之后,免疫组织化学、流式细胞荧光分选技术(FACS)和荧光原位杂交(FISH)促进了标记基因(marker gene)在细胞分类中的应用[6].这些技术揭示了形态相似的细胞之间的异质性[7].单细胞基因表达分析的最新进展为大幅提高细胞识别率和分类准确性提供了可能.其中单细胞测序技术最吸引人的应用之一是解码复杂的细胞异质性,并创建不同组织或器官中所有细胞类型的参考图谱[8-9].例如,高通量单细胞qPCR[10-12],单细胞流式细胞术[13],单细胞转录组测序[14-18]都能以高分辨率研究细胞的异质性.除此之外的其他方法,如单细胞基因组分析[19-22],表观基因组分析[23-30],原位分析[31-33]等,为在单细胞水平上研究细胞表型和细胞行为提供了可能.
自2009年Tang等[17]发表第一篇关于单细胞转录组测序技术的文章以来,这项技术得到了迅速的发展与应用.单纯地对几个细胞进行测序已经不再满足科研需求,迫切需要一次性对几千甚至几万个细胞同时进行测序.随后,McCarroll等[34]在《Cell》上发表了基于微滴包裹单细胞和捕获磁珠技术的Drop-Seq方案,标志着单细胞转录组测序进入高通量时代.之后许多大规模低成本的单细胞测序技术被开发出来,例如,Cyto-Seq[35],Smart-seq3[36]等.自此,单细胞测序技术相关实验中分析的细胞数据量呈指数增长,超过了摩尔定律[37].庞大的数据给研究人员提供了宝贵的资源,同时也提出了挑战.在美国国家生物技术信息中心(NCBI)上查找近10年跟单细胞测序技术相关的文献已超过5 000篇(图1)[38],产生的单细胞测序数据量也逐渐增加.一方面单细胞技术领域的迅猛发展使得单细胞数据量激增,另一方面对于如何处理这些数据的方法也在不断增加.从Zappia等[39]的分析中可以看到用于处理单细胞数据的工具已超过1 000个,并且还在持续增加中,其中R和Python为主要的分析语言.
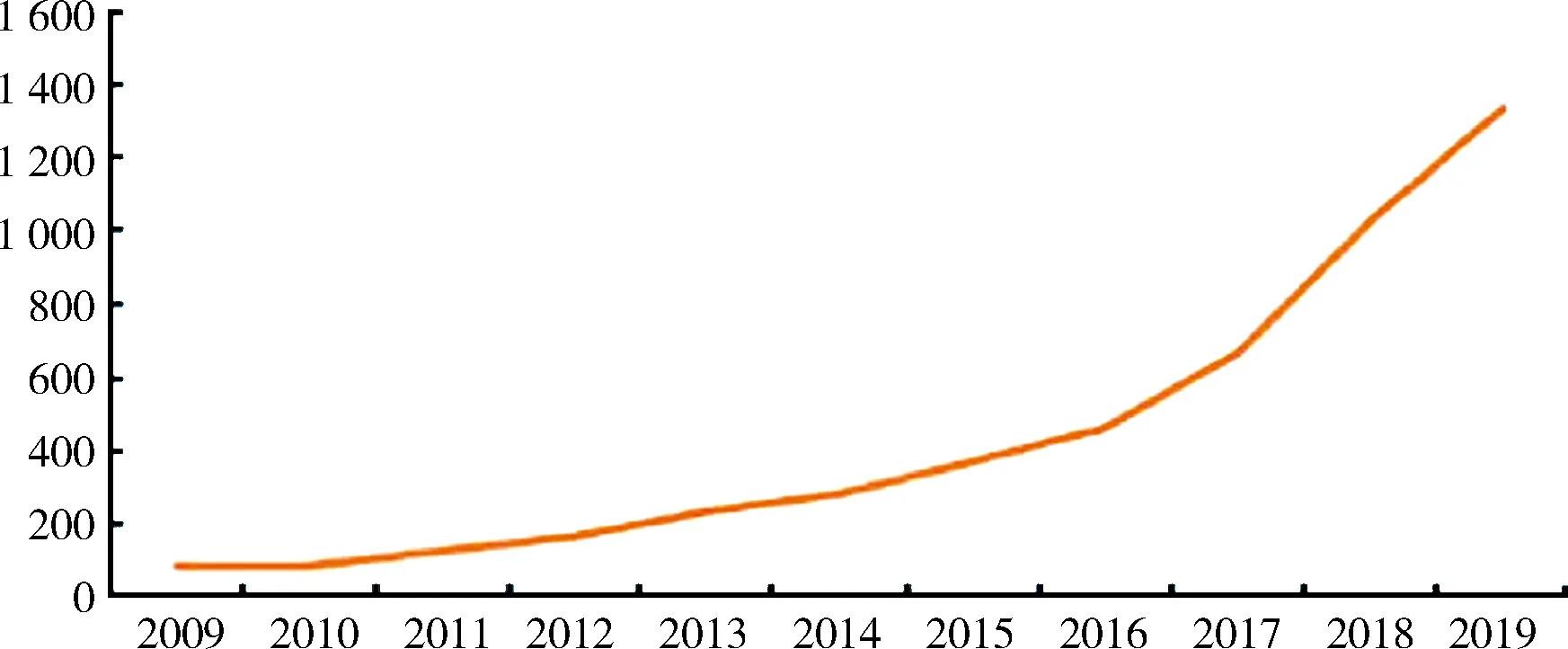
图1 单细胞研究文献发表数量增长趋势
自2012年以来,生物学大数据的挖掘和利用逐渐成为生命科学研究的前沿领域,单细胞测序技术的出现进一步加快了海量数据的产生,这些数据满足大数据的一般特性,即数据量大、速度快、类型多、价值高和真实性高,单细胞相关的大数据为各领域研究人员提供了宝贵的资源和机遇.
2 单细胞测序数据库
面对庞大的单细胞数据,构建功能性的数据库以统一整合和分析已有数据是一项迫切的需求.目前关于单细胞的数据库已经超过了20个,涵盖了转录组、基因组和表观基因组等,所涉及的物种以人和小鼠为主,组织器官超过200种,数据产生平台或技术包括10X Genomics、Smart-seq2、Fluidigm C1、Drop-seq等.本节主要介绍目前较有代表性的单细胞组学数据库(以下简称为单细胞数据库).
2.1 不同物种来源的数据库该类数据库的数据来源于已经发表的单细胞数据分析文献,并进行统一标准的处理.这些数据的测序物种包括人、小鼠、斑马鱼、果蝇、拟南芥等.大部分数据库的数据是以人和小鼠为主,有4个数据库包含除人和小鼠以外的物种,具体的数据库信息列在表1中.
2.1.1以人类和小鼠为研究对象的数据库 1) Mouse Cell Atlas[7].Mouse Cell Atlas数据库主要收集的测序数据来源于小鼠.目前已经更新到2.0版本,该数据库利用Microwell-seq对40多种组织器官和超过40万个细胞进行测序.Microwell-seq是一种高通量低成本的单细胞RNA测序技术,使用该技术获得小鼠各个组织器官的细胞聚类并做细胞类型定义,构建了小鼠细胞图谱.并且用户可根据组织器官进行搜索,展示各群的标志基因,同时网络图展示各基因之间的关系.Mouse Cell Atlas数据库有比较全面的小鼠单细胞测序数据,在小鼠相关研究领域具有重要的地位.
2) scRNASeqDB[40].scRNASeqDB数据库是一个收集和管理已经公开发表的人类单细胞基因表达数据集的数据库.该数据库覆盖了71个人类细胞系(或细胞类型)以及8 910个样本.同时数据库还提供了不同状态下细胞中基因表达的详细信息,以及基因表达的可视化图像、Gene Ontology和pathway等特征.该数据库有助于研究人员在广泛的生物学和医学领域对人类单细胞的基因表达进行研究.
3) CellMarker[41].CellMarker数据库通过收集已发表的文献,整理出人的158个组织/亚组织的467个细胞类型的13 605个标志基因,以及小鼠的81个组织、亚组织的389个细胞类型的9 148个标志基因,为人类和小鼠组织中的各种细胞类型提供全面而准确的细胞标记资源.CellMarker数据库提供交互式界面,用于浏览、搜索和下载不同组织的不同细胞类型的标记,给研究人员在定义细胞类型方面提供了宝贵的参考信息.
2.1.2除人和小鼠外以其他物种为研究对象的数据库 1) Single Cell Portal[42].Single Cell Portal数据库是由Broad institute建立的,旨在为研究人员扫除单细胞分析障碍,加速单细胞研究.该数据库所包含的物种除人和小鼠以外还包括斑马鱼、果蝇、原鸡、食蟹猴、猕猴、野猪、树鼩等9个物种.此外,数据库提供按研究项目和按基因检索,可通过物种、细胞类型、疾病类型、组织器官等选项搜索,以找到相应的研究项目.每个项目都提供相关的文献信息,并且可以在可视化界面进行数据挖掘.
2) Single Cell Expression Atlas(SCEA)[43].SCEA数据库收集整理了来自多个物种和不同实验条件下的原始单细胞测序数据,使用统一标准的方法重新分析数据,使得数据具有交叉可比性,并且该数据库将分析结果以用户友好的界面呈现出来.通过基因搜索,研究人员可以快速了解其感兴趣的基因在不同物种的单个细胞水平上的表达模式.SCEA数据库整合了18个物种的数据,涵盖了动物、植物、真菌以及原生动物,其中动物包括人类、小鼠、果蝇、血吸虫等10个物种,植物包括拟南芥、水稻、番茄以及玉米4个物种,真菌包含酵母,原生动物包含伯氏疟原虫以及恶性疟原虫.
2.2 收集特定研究领域数据的数据库
2.2.1肿瘤学数据库 1) CancerSEA[44].癌细胞的高度异质性是癌症研究和治疗中的主要挑战.单细胞测序技术为以单细胞分辨率破译癌细胞的各种功能状态提供了前所未有的机会,且癌症单细胞测序数据已经大量积累.CancerSEA描绘了一个癌症单细胞功能状态的图谱,涉及来自25种癌症类型的41 900个癌症单细胞的14种功能状态(包括干细胞、侵袭、转移、增殖、上皮细胞-间充质转化(EMT)、血管生成、凋亡、细胞周期、分化、DNA损伤、DNA修复、缺氧、炎症和沉默).数据库支持以基因名称、肿瘤类型和功能、基因集3种方式进行搜索,提供了丰富的数据检索方法.CancerSEA还在泛癌症、特定癌症类型和单个癌症类型单细胞数据集中提供了功能状态相关的PCG/lncRNA序列.
2) CancerSCEM[45].CancerSCEM数据库包括了28项研究和20种人类肿瘤类型的208份癌症样本,并且对每一个样本进行了统一标准的分析,包括对数据进行细胞类型注释、功能基因表达分析、细胞通讯和生存分析等.数据库采用统一的标准分析,因此可以比较不同癌症类型之间的细胞成分和许多功能分子的表达,此外友好的界面适合非生物信息学研究人员挖掘有价值的信息.同时该数据库还提供在线分析功能,可以分析不同细胞类型之间某些基因的表达情况,以及基因之间的相互作用.
3) TISCH[46].TISCH数据库整合了27种癌症类型的76个高质量肿瘤数据集中近200万个细胞的单细胞转录组数据,是一个专注于肿瘤微环境的大规模的管理数据库.数据库将所收集到的数据统一进行标准分析,允许在不同细胞类型、患者、组织来源、治疗,以及不同癌症类型之间进行系统比较.研究者可以在TISCH数据库中可视化、搜索和下载多种与肿瘤微环境相关信息,快速全面地进行肿瘤微环境的探索.
2.2.2发育生物学数据库 1) Human Cell Landscape(HCL)[47].HCL数据库收集的资源非常丰富,它旨在绘制完整的人类单细胞图谱.HCL数据库包括对60种人体组织样本和7种细胞培养样本进行Microwell-seq测序分析的数据.数据库包含了超过70万个单细胞,鉴定了人体102种细胞类型以及843种细胞亚型,系统性地绘制了跨越胚胎和成年2个时期的细胞图谱,涵盖了人体的8大系统.研究者可以在其中根据细胞分型、组织和基因进行搜索,数据库可以下载单细胞表达矩阵,进行自定义分析.研究者还可以上传自己的表达矩阵进行在线分析.
2) SCDevDB[48].SCDevDB是一个为研究细胞不同发育期间的单细胞基因表达谱而设计的数据库.该数据库收集了10个人类单细胞RNA-Seq数据集,并且将这些数据集拆分成176个发育细胞群,构建了24种不同的发育途径.研究者可在该数据库中根据发育的不同时期进行信息筛选,它提供每个发育途径中差异表达基因的列表,以及可视化分析结果.
2.2.3免疫学数据库 JingleBells[49].单细胞测序技术的发展加深了对免疫分化和激活过程的理解,JingleBells数据库将数据划分为免疫与非免疫类,收集了与120篇免疫相关文献以及182篇非免疫领域文献的单细胞测序数据集的原始数据,并用标准的分析流程处理数据,是一个标准化单细胞RNA-seq数据集的存储库,可供研究者下载数据用于后续分析.
2.2.4收集其他研究领域数据的数据库 1) SC2disease[50].SC2disease是一个人工收集并整合数据的数据库,能为研究者提供各种疾病的各细胞类型的基因表达谱.研究人员使用关于单细胞人类疾病样本文献中的数据,并根据疾病、组织和细胞类型整理数据.SC2disease包含946 481条数据,对应341种细胞类型、29种组织和25种疾病.数据库中的每个条目都包含不同细胞类型、组织和疾病相关健康状况之间差异表达基因的比较.SC2disease还提供了从基于单细胞的结果和基于全基因组关联分析(GWAS)的结果得出的疾病的易感基因.
2) KIT(Kidney Interactive Transcriptomics)[51].随着发表的有关肾脏单细胞文章的增加,研究人员对肾脏单细胞文章进行了整理,构建了一个肾脏单细胞数据集的在线分析数据库KIT.迄今为止(2021年12月)该数据库收录的数据来自于16篇关于肾脏的单细胞文献和RBK数据库.KIT数据库总共收录了130万个细胞,超过17种细胞类型,并且提供了根据基因名搜索以及在线分析的功能.

表 1 单细胞组学数据库及其功能特点

续表1 scRNA-tools--是一个专门收集用于单细胞测序数据分析软件的数据库https://www.scrna-tools.org/[52]Single CellExpression Atlas(SCEA)18个物种229项研究,5 978 348个单细胞数据提供非常丰富的数据资源,可通过标记基因来检索细胞,选择感兴趣的数据集、物种等查看聚类等基本信息https://www.ebi.ac.uk/gxa/sc/home[43]Single CellPortal(SCP)9个物种409项研究,超过1 800万个单细胞数据库分为按项目搜索以及按基因搜索,同时可视化相关信息,也可选择细胞类型、器官、物种、疾病等进行检索https://singlecell.broa-dinstitute.org/single_cell[42]TISCH人类27种癌症类型近200万个细胞是一个关于肿瘤微环境的单细胞数据库,该数据库允许在不同细胞类型、患者、组织来源等方面之间进行系统比较,可视化分析结果http://tisch.comp-genomics.org[47]CancerSCEM人类20种人类肿瘤类型,208份癌症样本是一个收集肿瘤单细胞测序数据的数据库,分析了多个公共测序数据中基因的表达情况,还分析了细胞表面受体-配体、细胞间互作网络等https://ngdc.cncb.ac.cn/cancerscem[45]ScMethBank人类和小鼠29种细胞类型和2种疾病模型是一个收集单细胞全基因组DNA甲基化数据的数据库,可通过样本、基因、甲基化区域进行搜索,并有可视化界面https://ngdc.cncb.ac.cn/methbank/scm/[57]
2.3 单细胞数据分析工具数据库scRNA-tools[52]随着单细胞测序数据的激增,用于分析单细胞测序数据的工具也呈现出增长的趋势.scRNA-tools是一个收集单细胞测序数据分析软件的数据库.该数据库记录了从2016年以来每年新增的分析软件供研究人员选择.截止目前已收集了1 124个用于单细胞数据分析的软件.数据库提供单细胞测序数据分析工具的详细信息,用户可以查询常用工具的具体信息,还可以通过引用次数对工具进行排序,以及根据分析目的对工具进行分类,从而选择合适的处理工具.
3 单细胞数据库构建与应用
3.1 数据库的构建大部分生物学数据库的主要功能是通过统一方法整合庞大的数据以提供快速检索信息的平台.单细胞数据库的构建一般主要分为收集数据、处理数据以及数据展示3个步骤(图2).

图2 数据库构建基本流程
1) 收集数据.单细胞数据库中所收集的数据主要来源于已公开发表的文献中的数据集.在PubMed中以关键词“single-cell sequencing”“single cell”“single cell RNA sequencing”等词进行检索然后筛选出符合条件的文献再从中获得数据.数据集从GEO、GSEA、ZENODO等网站手动下载.
2) 处理数据.将下载的数据进行数据分析是数据库构建的核心功能,用不同的分析方法可能会得到不同的数据,使得数据库面向的用户群体会大不一样.单细胞数据库中数据所使用的单细胞分析流程主要为质控、细胞分群、差异表达以及细胞类型注释.之后不同的数据库根据不同的研究问题进行自定义分析,将分析得到的结果的集合有序地存入信息表中(MySQL、MongoDB等).
3) 数据展示.通常数据展示是通过搭建在线的网站以方便用户搜索并在网站上展示出相关信息.用户在网站的前端通过关键词(如细胞类型、基因名等)发出请求,后台程序以关键词在信息表中搜索并且将搜索到的结果在网站前端可视化展示,以供用户查看.
3.2 数据库的选择由于对细胞异质性研究的兴起,单细胞技术被广泛用于回答发育生物学、神经科学、肿瘤学和免疫学的许多基本问题[58].单细胞转录组测序技术使得对生物变异进行详细研究成为可能[59].迄今为止,单细胞数据库已超过20个,如何选择合适的数据库进行信息检索是研究人员面对的首要问题.
1) 根据研究对象选择数据库.研究人员需根据不同的研究对象选择不同的数据库进行信息检索.如研究对象是人类,则可选择收集人类单细胞数据的数据库HCL、ScRNASeqDB等,在这些数据库中能根据细胞类型、组织、器官等关键词搜索相应的标志基因以及基因表达谱.CellMarker作为一个专门收集标志基因的数据库,也能根据细胞类型搜索标志基因.大部分数据库都提供可视化的功能,能直观地显示研究者所需要的信息.然而,不同的数据库处理数据的方法可能是不相同的,因此要求研究者基于所研究的问题选择合适的数据库进行分析.
2) 根据研究领域选择数据库.除了通过研究对象选择数据库,还需要根据研究领域选择合适的数据库.单细胞技术在肿瘤学、免疫学、发育学等领域上都得到了飞速发展,单细胞数据库也在这些研究领域收集数据,因此单细胞数据库也可分为与肿瘤相关的数据库CancerSEA、CancerSCEM、TISCH,与发育相关的数据库SCDevDB和免疫相关的数据库JingleBells,以及其他领域的数据库(图3).

图3 单细胞数据库分类
专业数据库和综合数据库是2类最常见的数据库类型,并且各有优势.例如,scREAD是一个专门针对阿尔兹海默症而建的单细胞数据库,对于阿尔兹海默症的研究很有帮助.在研究肿瘤方面的内容时,尽管HCL、scRNASeqDB等数据库也能检索到部分信息,但是选择肿瘤相关的单细胞数据库CancerSEA以及肿瘤微环境相关的单细胞数据库TISCH,可以得到更丰富的信息.
3.3 数据挖掘研究人员可通过对数据库中的数据进行搜索并进行再次整理,对某一方面的信息进行挖掘.例如,若想研究肿瘤细胞中细胞凋亡的机制,在肿瘤数据库CancerSEA中搜索跟细胞凋亡有关的信息,数据库返回的14种癌症类型里都有细胞凋亡相关的数据,点击查看更多的信息,可以发现在这些数据集里有一些显著的差异表达基因,研究者可根据这些基因进行更深层的探索,或者挑选部分基因进行实验验证.
总之,单细胞数据库给研究人员带来许多便利,这些数据库集合了各种研究数据以及分析结果,研究者只需在数据库中进行搜索就能找到重要信息,而无需进行原始数据收集和分析,节省了大量时间.
4 总结与展望
生物学中最基本的问题之一是哪些类型的细胞以功能协调的方式形成不同的组织和器官.单细胞测序技术的发展使得研究人员可以在单细胞分辨率研究细胞表型和细胞行为,其发展潜力巨大.目前关于单细胞测序数据的数据库各有优势,但各数据库之间有数据交叉重复的部分,会造成一定的信息冗余.不同的数据库所用的整合数据的方法也不相同.然而,迄今为止,尽管大量的数据集仍在持续产生,但可供研究人员使用的数据库仍然不够丰富.综合数据库,如scRNASeqDB、PanglaoDB等对文献中的单细胞表达数据集进行整合,其中大部分来自人类和小鼠样本.关注除人和小鼠以外物种的数据库目前只有4个.此外由于收集的数据来源不同、平台不同、实验不同等因素,给整合数据带来了困难,大部分数据库对于数据的处理一般只包含细胞聚类和差异基因表达谱的初步分析.
目前关于肿瘤的单细胞数据库如CancerSEA、CancerSCEM等在肿瘤单细胞研究中应用广泛.尽管CancerSEA是2018年发布的第一个以单细胞分辨率分析癌细胞不同功能的数据库,但它只包含41 900个癌细胞的14种功能状态,而没有考虑肿瘤微环境中的免疫细胞或基质细胞.至于HCL数据库虽然构建了人类单细胞图谱,但是完整的细胞图谱还应该整合更多的信息,如空间信息、多组学数据以及群体分析等.正如CellMarker数据库被研究人员常用来查找不同细胞类型所对应的标志(marker)基因,SignatureDB能查到关于免疫细胞-B细胞的信息,但这些数据库中也有冗余信息(例如在CellMarker数据库里也能查到B细胞的部分信息).不同的数据库由于收集的数据来自于不同的文献,数据整理的方法不一致,数据信息也无法完全对应.Mouse Cell Atlas数据库里能查到关于小鼠的组织器官以及对应的标志基因,但这些信息和CellMarker中的信息并不完全一致.这也给研究人员在筛选合适的数据方面增加了工作量.迄今为止缺少一个整合数据库的标准准则.因此根据已有数据库各自的功能特点,期待未来的单细胞测序数据库应具有以下3条特征.
1) 更具综合性.新方法的发展和新技术的进步推动了生物学的蓬勃发展.随着单细胞测序技术应用的持续推广和更新,单细胞测序数据类型将会越来越多,所涉及的物种种类、组织、器官、细胞和基因也会增加,使数据的整合变得更复杂.数据库所收录的数据应该更加全面与综合.另外,仅仅利用单组学进行研究已经不能满足研究人员的科研需求,单个组学数据只能解释某一层面的问题,无法从多个角度进行深入探索.近年来,单细胞多组学整合研究成为新的趋势,研究人员可以从多个组学的角度验证和分析数据并结合实验验证结果.因此,单细胞数据库里的数据不再仅仅局限于基因组或转录组的数据,还将包括表观基因组学、代谢组学等更加综合的单细胞数据.如ATAC-seq数据、蛋白质数据、FISH数据等.
2) 更具时效性.据《单细胞行研报告》[38],自2009年到2019年的10年间,单细胞研究相关文献发表数量增长了约14倍.尤其是自2013年至2021年,单细胞测序技术发展迅猛,新旧数据的更迭给研究人员选择适用的数据带来一定困难.因此,未来的单细胞数据库应及时更新数据信息,从新产生的大量的数据中及时梳理出有价值的数据以更新数据库,使数据库能跟上单细胞研究领域的发展进度,给研究人员提供及时并有价值的信息.
3) 更具健壮性.单细胞数据库以网站框架为主体,以便科研人员查找信息.海量的数据给网络服务提出了不小的挑战,当研究人员在使用在线数据库进行搜索时,能快速查找到正确的搜索信息并进行展示是关键的用户体验.目前所发表的单细胞数据库的构建框架基本是由PHP+MySQL+HTML的形式,但是查询的数据如何存储,以何种表格存储,如何构建查询方式等,是由构建数据库的研究人员设计的.面对数据库中如此庞大的数据,如何设计数据的存储模式和查询方式,如何更新数据等问题关系到数据库整个架构的健壮性.
总之,单细胞研究领域有巨大的发展潜力,有关单细胞数据的数据库的发展也需要与时俱进.在大数据时代,庞大的数据量给单细胞数据的整合带来了机遇,同时也带来了信息筛选的挑战.能满足上述3条特征的单细胞数据库将会极大地促进该领域的研究和发展.
