猎蝽科昆虫线粒体基因组的比较研究
2022-06-24赵婉清李巧李莹杨柳郑茜之张虎芳
赵婉清 李巧 李莹 杨柳 郑茜之 张虎芳









摘要:线粒体基因组是系统发育常用的分子标记,本研究旨在对猎蝽科昆虫的线粒体基因组进行比较研究,着重分析基因重排、碱基组成、密码子使用偏好性等特征,并构建基于13个蛋白编码基因的系统发育关系。在GenBank数据库下载猎蝽科11亚科30属代表物种的线粒体基因组序列,通过Geneious 8.0.4抽提各编码基因的序列。利用MEGA 7.0统计碱基组成、氨基酸和核苷酸序列信息位点、起始密码子、终止密码子、相对密码子使用频率(RSCU)等序列基本信息。另外,利用DnaSP 6.0计算蛋白编码基因的非同义替换率(Ka)和同义替换率(Ks),并通过Ka/Ks值比较基因的核苷酸进化速率。通过jModelTest选择进化模型,在MrBayes 3.2.2软件中构建基于贝叶斯法(BI)的系统发育树。结果表明:猎蝽科线粒体基因组均呈共线性结构,没有发现大面积的基因重排现象,只有部分序列中rrnS和trnT基因编码方向与原始序列编码方向相反,trnR和cytb基因出现多拷贝的现象。猎蝽科线粒体最常使用ATN起始密码子和TAA终止密码子。BI系统发育树对猎蝽科各亚科之间的关系进行了一定的解析,光猎蝽亚科(Ectrichodiinae)和猎蝽亚科(Reduviinae)为并系群,真猎蝽亚科(Harpactorinae)和锥猎蝽亚科(Triatominae)为单系群。本研究为猎蝽科昆虫的线粒体基因组进行了比较分析,为后续研究该科系统发生关系奠定了科学基础。
关键词:猎蝽科;线粒体基因组;比较基因组学;系统发育;分子标记;基因重排;碱基组成;密码子使用偏好性
中图分类号: S433.3文献标志码: A
文章编号:1002-1302(2022)12-0034-08
收稿日期:2021-11-23
基金项目:山西省高等学校科技创新项目(编号:2019L09841);忻州师范学院院级科研基金(编号:2018KY04)。
作者简介:赵婉清(1989—),女,山西灵石人,博士,主要从事昆虫分子系统学研究。E-mail:zhaowanqing@vip.163.com。
通信作者:张虎芳,博士,教授,主要从事昆虫分子系统学研究。E-mail:zh_hufang@sohu.com。
线粒体具有独立的基因组,且在真核生物细胞内广泛存在。线粒体基因组结构简单、排列紧密、进化速率较快、能够独立复制、编辑效率高,是系统发育进化、种群遗传学、生物地理学等常用的标记基因[1-3]。随着生物测序技术的迅速发展,GenBank数据库中的昆虫线粒体基因组序列越来越多,为昆虫线粒体的比较分析以及分子系统学研究提供了新的方向和依据。昆虫线粒体基因组一般包括37个编码基因(13个蛋白编码基因、22个tRNA基因和2个rRNA基因)和一段控制区[4]。对近缘物种进行比较线粒体基因组学研究可以为物种的分类和系统发育关系奠定理论依据。
猎蝽科(Reduviidae)隶属于半翅目(Hemiptera)异翅亚目(Heteroptera),广布于世界各地,种类较多,是异翅亚目中的一个大科[5]。该科多为捕食性昆虫,某些种类可以捕食褐飞虱、草地贪夜蛾、斜纹夜蛾、烟青虫等害虫,是农林业中重要的天敌昆虫,对该科昆虫的研究具有一定的科学和经济意义[6-9]。猎蝽科世界已知6 000余种,我国记录13个亚科400余种[5]。以往对猎蝽科系统发育的研究主要是基于少量分子数据,而对线粒体基因组的研究较少[10]。
本研究选取猎蝽科11亚科30属的代表物种,进行该科线粒体基因组的比较分析和系统发育研究。对线粒体基因组的排列、碱基组成、序列信息位点、密码子使用情况等进行比较分析,并基于贝叶斯法(Bayesian inference,BI)构建猎蝽科的系统发育关系。
1 材料与方法
1.1 序列获取
本研究选取猎蝽科11亚科30属的代表物种进行研究,通过NCBI中的GenBank数据库(https//www ncbi.Nlm.nih.gov/)下载获得线粒体序列,详细的序列信息见表1。
1.2 数据分析
利用Geneious 8.0.4[11]软件从线粒体基因组序列中抽提出各编码基因,再利用SequenceMatrix[12]串联成所需的数据集。对序列进行比对时,蛋白编码基因需将核苷酸序列转换为氨基酸序列,保存比对后的核苷酸序列;RNA基因直接进行比对后保存核苷酸矩阵序列。在SequenceMatrix中分别将蛋白编码基因串联成13个蛋白编码基因(protein-coding genes,PCGs)、重链编码的蛋白编码基因(PCGs-J)和轻链编码的蛋白编码基因(PCGs-J)3个数据集。在MEGA 7.0[13]中统计碱基组成和序列信息位点,并计算同义密码子相对使用频率(RSCU)。在DnaSP 6.0[14]软件中计算蛋白编码基因的非同义替换率(nonsynonymous substitution rate,Ka)和同义替换率(synonymous substitution rate,Ks),进而统计每个基因的核苷酸进化速率,即Ka/Ks值。为了探究猎蝽科昆虫的系统发育关系,选取臭虫科1种温带臭虫(Cimex lectularius)作为外群,基于13个蛋白编码基因构建贝叶斯系统发育树。BI发育树在MrBayes 3.2.2[15]中运行计算,运行10 000 000代,每1 000代抽样1次,舍去收敛前的样本。利用jModelTest[16]预测进化模型,预测结果为GTR+I+G模型。
2 结果与分析
2.1 线粒体基因组全序列的结构
截至2021年10月,GenBank共收录了猎蝽科11亚科30属的昆虫线粒体基因组序列110条,为研究该科昆虫的比较线粒体基因组学研究奠定了良好基础。与半翅目其他昆虫类似,猎蝽科线粒体基因组共37个编码基因,其中13个蛋白编码基因、22个tRNA基因和2个rRNA基因,还包括一段非編码控制区。线粒体基因组长度范围为13 977~16 759 bp,最长的是伐猎蝽(Phalantus geniculatus),最短的是海南杆蝓猎蝽(Ischnobaenella hainana)。
2.2 基因重排现象
猎蝽科30属代表物种的线粒体基因组均呈共线性结构,没有发现大面积的基因重排现象。通过依次比较30条线粒体基因组,对个别出现重排的序列结构进行了比较作图分析。由图1可知,Agriosphodrus dohrni和Brontostoma colossus线粒体基因组中rrnS基因编码方向与原始序列编码方向相反;Dipetalogaster maximus线粒体基因组中trnT基因编码方向与原始序列编码方向相反;猎蝽Brontostoma colossus线粒体基因组中trnR基因有多拷贝现象,形成trnR-trnA-trnR的排列方式;Eratyrus mucronatus线粒体基因组中cytb出现多拷贝现象。
2.3 碱基组成和序列信息位点分析
已测得的猎蝽科昆虫线粒体基因组的碱基组成具有明显的AT偏倚性,且AT含量最高的是Polytoxus fuscovittatus(77.8%),最低的是Panstrongylus rufotuberculatus(68.1%)。对A、T、G、C各碱基的组成进行比较发现,所有猎蝽科昆虫中碱基A的含量均高于T的含量,AT-skew均大于0(0.09~0.19);G的含量均高于C的含量,GC-skew 均小于0(-0.27~-0.12)。
对13个蛋白编码基因的碱基组成进行分析,结果如图2、图3所示。atp8的(A+T)含量最高(75.54%),cox1的(A+T)含量最低(65.75%)。位于J链的蛋白编码基因(PCGs-J),(A+T)含量为69.93%,且碱基A与T的含量相同,GC-skew为-0.15。位于N链的蛋白编码基因(PCGs-N),(A+T)含量为74.57%,AT-skew为-0.37,GC-skew为0.26。
13个蛋白编码基因的核苷酸和氨基酸序列的信息位点分析结果见图4和图5。cox1的保守性在核苷酸和氨基酸序列中均较高(47.63%和63.98%),而atp8基因的保守性较差(14.03%和9.20%)。蛋白编码基因的核苷酸串联序列中,保守位点有3 624个,变异位点有7 527个,简约信息位点有6 362个。在氨基酸串联序列中,保守位点有387个,变异位点有3 303个,简约信息位点有3 007个。
2.4 密码子使用情况
在猎蝽科线粒体蛋白编码基因中,最常使用ATN起始密码子,但也有一些特殊情况,如nad1、nad4L和nad5也会使用GTG和TTG为起始密码子。在ATN起始密码子中,ATG的使用频率较高,如atp6和nad4的起始密码子全部为ATG(图6)。对于终止密码子的使用,密码子TAA的使用频率最高,TA的使用频率最低。在cox1、cox2、cox3和nad5中,以T结尾的情况较多(图7)。
本研究统计了相对密码子使用频率(RSCU),使用频率较高的密码子为UUA(L)、GUU(V)和UAU(Y),使用频率相对较低的为CCG(P)、CUC(L)和CUG(L)(表2)。在同义密码子中,第3位以A/U结尾的密码子比G/C结尾的密码子使用频率高。
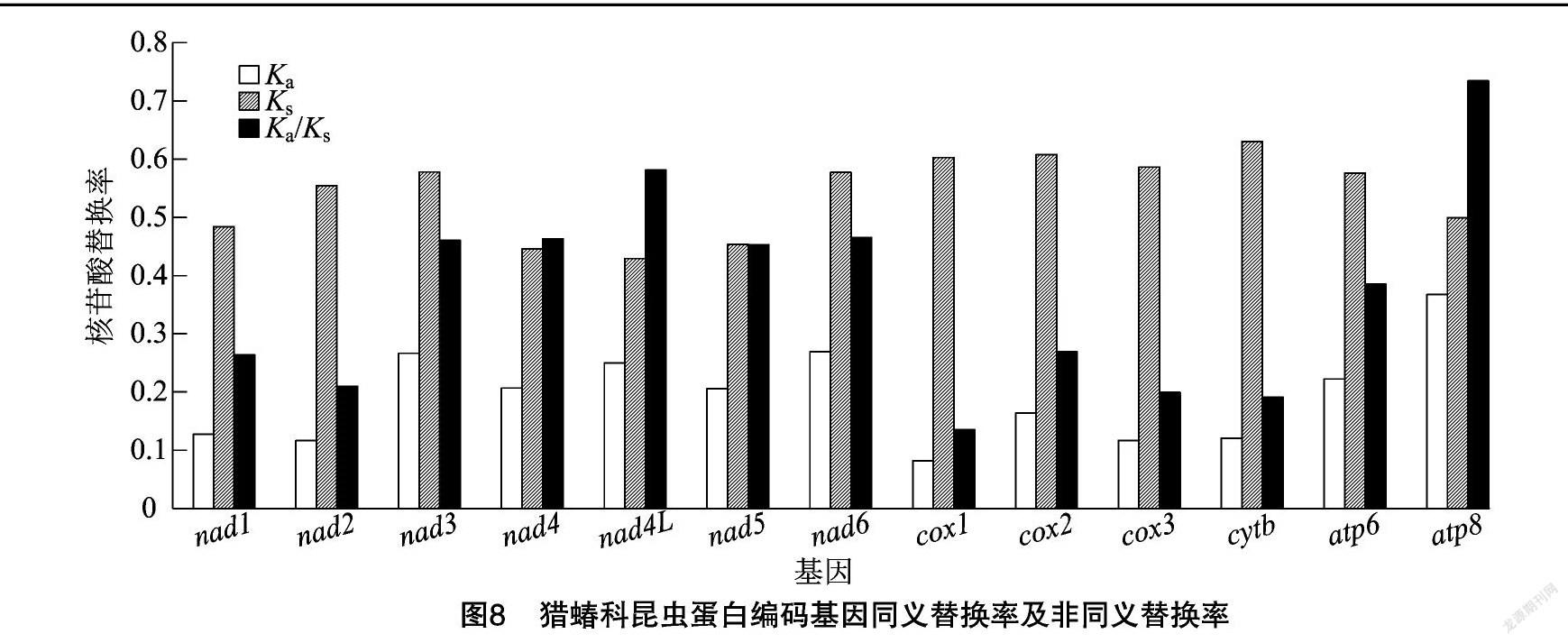
2.5 蛋白质编码基因进化速率
蛋白编码基因的核苷酸Ka、Ks及Ka/Ks值的统计结果见图8。所有基因的Ka/Ks值均小于1,表明受到了纯化选择的作用。其中,atp8的进化速率最快,cox1的进化速率最慢,具体的排列顺序为atp8>nad4L>nad6>nad4>nad3>nad5>atp6>cox2>nad1>nad2>cox3>cytb>cox1。
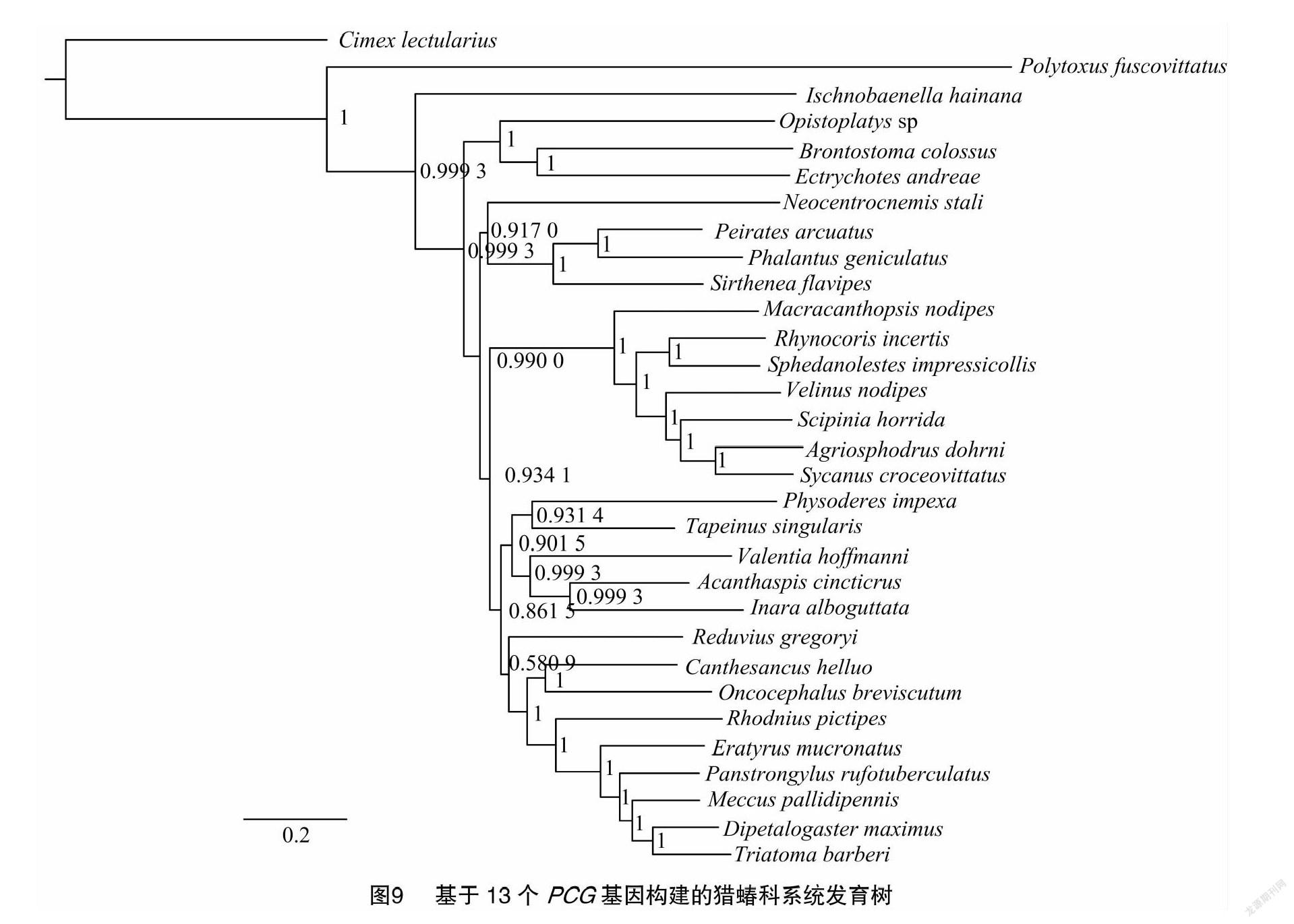
2.6 系统发育分析
以臭虫科作为外群,基于13个蛋白编码基因序列构建了猎蝽科30属的BI系统发育树(图9)。结果表明,猎蝽科各亚科的关系得到了一定的解析,该科基部分支为盲猎蝽亚科(Saicinae),光猎蝽亚科(Ectrichodiinae)和猎蝽亚科(Reduviinae)為并系群,真猎蝽亚科(Harpactorinae)和锥猎蝽亚科(Triatominae)为单系群。锥猎蝽亚科(Triatominae)和细足猎蝽亚科(Stenopodinae)为姐妹群关系,盗猎蝽亚科(Peiratinae)和新猎蝽亚科(Centrocnemidinae)为姐妹群关系。
3 讨论与结论
一直以来,果蝇(Drosophila yakuba)的线粒体基因组是公认的昆虫线粒体基因组的原始排列方式[17]。共线性分析结果显示,猎蝽科昆虫线粒体基因组的排列方式比较保守,大多数与原始排列方式一致,只存在少数基因重排现象,以基因编码方向与原始序列编码方向相反和基因多拷贝现象为主。其中,rrnS和trnT基因的编码方向出现与原始序列编码方向相反的情况,trnR和cytb基因均多拷贝了1次。
猎蝽科线粒体基因组长度范围为13 977~16 759 bp,具有明显的AT偏倚性,A+T含量为68.1%~77.8%,且碱基A的含量均高于T,碱基G的含量均高于C。13个蛋白编码基因中, cox1的保守性较高,而atp8基因的保守性较差。同时,采用Ka/Ks值比较基因进化速率结果也表明,atp8的进化速率最快,cox1的进化速率最慢,与先前的相关研究结果[18]一致。13个蛋白编码基因的Ka/Ks值均小于1,可能是受到了纯化选择的作用[19]。
在密码子的使用上,除偏好使用的ATN起始密码子外,猎蝽科线粒体蛋白编码基因也会以GTG和TTG作为起始密码子。对于终止密码子来说,除完整的TAA和TAG密码子使用频率较高外,不完整的TA和T在猎蝽科蛋白编码基因中也很常见。这些不完整的终止密码子会在转录形成mRNA后转变成完整的终止密码子[20]。同义密码子相对使用频率结果显示,以A/U结尾的密码子使用频率高于G/C结尾的,与昆虫线粒体基因组的高AT特性一致。
基于贝叶斯算法构建的BI系统发育树对猎蝽科各亚科的关系进行了一定的解析,结果显示猎蝽亚科为并系群,这与Weirauch选取162个形态特征对猎蝽科进行的支序系统学分析结果[21],以及赵广宇采用COI基因构建的系统发育树结果[10]一致。本研究中真猎蝽亚科和锥猎蝽亚科为单系群,与Carayon等的研究结果[22]相同。绒猎蝽亚科和光猎蝽亚科的姐妹群关系没有得到验证,与2个亚科线粒体基因组序列较少有很大关系。另外,由于猎蝽科昆虫的形态特征差异显著,尚无严格的分类系统,高级阶元间的关系非常混乱。随着猎蝽科被报道的线粒体基因组序列越来越多,系统发育的研究也将迎来新的进展。
本研究选取了猎蝽科11亚科30属的代表物种进行比较线粒体基因组学研究,对线粒体基因组的重排现象、碱基组成、密码子使用情况和蛋白编码基因的核苷酸进化速率进行分析,并基于13个蛋白编码基因构建了猎蝽科昆虫的BI系统发育树。猎蝽科线粒体基因组呈共线性结构,没有发现大面积的基因重排现象,只出现了极个别的多拷贝和基因编码方向与原始序列编码方向相反的现象。在蛋白编码基因中,cox1的保守性最高,进化速率最慢;而atp8基因的保守性最差,进化速率最快。BI系统发育树对猎蝽科各亚科的关系进行了一定的解析,为之后的系统进化研究奠定了一定的基础,但是由于该科昆虫本身差异显著、分类系统混乱,尚需进一步的深入探究。
参考文献:
[1]Zhao W Q,Zhao Q,Li M,et al. Comparative mitogenomic analysis of the Eurydema genus in the context of representative Pentatomidae (Hemiptera:Heteroptera) taxa[J]. Journal of Insect Science,2019,19(6):20.
[2]Cameron S L. Insect mitochondrial genomics:implications for evolution and phylogeny[J]. Annual Review of Entomology,2014,59:95-117.
[3]Jacobsen M W,da Fonseca R R,Bernatchez L,et al. Comparative analysis of complete mitochondrial genomes suggests that relaxed purifying selection is driving high nonsynonymous evolutionary rate of the NADH2 gene in whitefish (Coregonus ssp.)[J]. Molecular Phylogenetics and Evolution,2016,95:161-170.
[4]Wu Y F,Shi A X,Yang H L,et al. The complete mitochondrial genome of the assassin bug Sycanus croceovittatus (Hemiptera:Reduviidae)[J]. Mitochondrial DNA(Part B Resources),2020,5(3):2561-2562.
[5]黄 霞. 广西猎蝽科昆虫分类研究[D]. 桂林:广西师范大学,2007.
[6]侯峥嵘,孙贝贝,刘先建,等. 大红犀猎蝽对草地贪夜蛾3龄幼虫捕食功能反应[J]. 植物保护学报,2020,47(4):852-858.
[7]张 鑫. 红彩真猎蝽的饲养及其对褐飞虱的捕食作用研究[D]. 广州:仲恺农业工程学院,2017.
[8]苏湘宁,邓海滨,朱丹荔,等. 红彩真猎蝽对斜纹夜蛾幼虫捕食行为及室内扩散能力的研究[J]. 中国烟草学报,2016,22(5):111-119.
[9]邓海滨,王 珍,陈永明,等. 紅彩真猎蝽对斜纹夜蛾和烟青虫的捕食功能反应[J]. 广东农业科学,2012,39(13):107-109.
[10]赵广宇. 猎蝽科昆虫条形码研究[D]. 北京:中国农业大学,2014.
[11]Kearse M,Moir R,Wilson A,et al. Geneious basic:an integrated and extendable desktop software platform for the organization and analysis of sequence data[J]. Bioinformatics,2012,28(12):1647-1649.
[12]Vaidya G,Lohman D J,Meier R. SequenceMatrix:concatenation software for the fast assembly of multi-gene datasets with character set and Codon information[J]. Cladistics,2011,27(2):171-180.
[13]Kumar S,Stecher G,Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,2016,33(7):1870-1874.
[14]Librado P,Rozas J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25(11):1451-1452.
[15]Ronquist F,Huelsenbeck J P. MrBayes 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics,2003,19(12):1572-1574.
[16]Posada D.jModelTest:phylogenetic model averaging[J]. Molecular Biology and Evolution,2008,25(7):1253-1256.
[17]王丹陽,王予彤,于良斌,等. 绿眼赛茧蜂线粒体基因组全序列测定和分析[J]. 昆虫学报,2020,63(8):1028-1038.
[18]Yuan M L,Zhang Q L,Guo Z L,et al. Comparative mitogenomic analysis of the superfamily Pentatomoidea (Insecta:Hemiptera:Heteroptera) and phylogenetic implications[J]. BMC Genomics,2015,16:460.
[19]Wang J,Zhang L,Zhang Q L,et al. Comparative mitogenomic analysis of mirid bugs (Hemiptera:Miridae) and evaluation of potential DNA barcoding markers[J]. PeerJ,2017,5:e3661.
[20]Cao S Y,Wu X B,Yan P,et al. Complete nucleotide sequences and gene organization of mitochondrial genome of Bufo gargarizans[J]. Mitochondrion,2006,6(4):186-193.
[21]Weirauch C. Cladistic analysis of Reduviidae (Heteroptera:Cimicomorpha) based on morphological characters[J]. Systematic Entomology,2008,33(2):229-274.
[22]Carayon J,Usinger R L,Wygodzinsky P. Note on the higher classification of the Reduviidae,with the description of a new tribe of the Phymatinae[J]. Revue de Zoology et de Botanique Africaines,1958, 57:256-281.
