贝莱斯芽孢杆菌LG37全基因组测序分析及无机氮代谢相关候选基因的筛选
2022-06-22刘广鑫董晏君赵丽娟邓益琴程长洪马红玲江建军郭志勋
刘广鑫,董晏君,赵丽娟,邓益琴,程长洪,马红玲,江建军,冯 娟,郭志勋,林 蠡
1. 中国水产科学研究院南海水产研究所/农业农村部南海渔业资源开发利用重点实验室,广东 广州 510300
2. 肇庆市水产技术推广中心,广东 肇庆 526040
3. 仲恺农业工程学院 动物科技学院/广东省水环境与水产品安全工程技术研究中心/广州市水产病害与水禽养殖重点实验室,广东 广州 510225
高密度的养殖模式中,池塘中残余的饲料蛋白和水生动物的排泄物等均含有大量的含氮化合物。这些累积的含氮化合物在养殖水体中被慢慢分解,并产生氨基酸,氨基酸再被微生物脱氨产生氨氮,最终养殖水体的氮源会以分子氨 (NH3-N)、离子铵、亚硝态氮、硝态氮、氮气 (N2) 及有机氮化物 (R-NH2) 的形式以动态平衡的方式存在[1-3]。而NH3和之间可相互转化:,当水体 pH值和温度升高时,水体中NH3的比例增加。NH3和对经济动物组织、器官有直接毒害作用,是造成机体免疫力下降并导致疾病爆发的关键因素之一[4-5]。通过绿色、高效的生物方式降低水体中无机氮的浓度达到水质净化的目的,是提高水生动物成活率的主要方法之一。
芽孢杆菌 (Bacillusspp.) 作为异养微生物,是一种广泛存在于自然界中,并能够高效利用无机氮实现自身生长、增殖的革兰氏阳性菌,也是水产养殖领域中研究最多、应用最广泛的菌种之一[6]。随着研究的深入,芽孢杆菌同化无机氮的代谢途径逐渐被挖掘,同化代谢过程中涉及的多种特异性功能蛋白的作用被发现,例如特异性感应蛋白、转运蛋白、氧化还原蛋白、同化蛋白等,但其作用机理尚不清晰[7]。
全基因组测序 (Complete genome sequence) 是指将生物的基因组进行随机打断、测序和拼接,从而获得完整基因组序列的技术,通过序列分析和功能基因注释 (包括NR、KEGG、eggNOG、GO和CARD等),以便从基因水平深入了解菌株表型的机理[8-9]。目前,随着生物技术和信息学的有机结合,全基因组测序技术已经发展成为一种高效、准确的信息获得手段,在生物研究领域中应用广泛,例如肠道菌群分析、微生态群系组成、特异性表型分析、功能基因的预测及核心基因的挖掘等[10-13]。
利用芽孢杆菌降低水体中无机氮的浓度具有绿色、持续、无二次污染等特点,被学者和养殖户普遍认可[14]。尽管芽孢杆菌同化无机氮的效果得到了很好验证,但其代谢途径和特异性功能蛋白等仍有待深入解析和验证。贝莱斯芽孢杆菌 (B. velezensis) LG37筛选于华中农业大学教学实习基地养殖池塘中,前期研究发现LG37可通过无机氮作为唯一氮源的最小培养基进行培养,并具有高效同化无机氮的能力[7]。基于此,本研究结合三代PacBio RS II和二代 Illumina HiSeq 2000测序技术,对 LG37进行全基因组测序分析,并对获得的数据进行NR、KEGG、eggNOG、GO、CARD数据库注释、分析,旨在深入揭示LG37在无机氮代谢过程中的关键功能基因和代谢通路信息,为其在无机氮代谢研究和水产养殖中的应用提供参考信息和科学依据。
1 材料与方法
1.1 实验材料
1.1.1 菌株
贝莱斯芽孢杆菌LG37为实验室于华中农业大学教学实习基地水产养殖池塘中筛选获得。
1.1.2 相关试剂和仪器设备
LB (Luria-Bertani) 培养基;细菌基因组 DNA提取试剂盒,天根生化科技 (北京) 有限公司。Pac-Bio RS II (Pacific Biosciences, Menlo Park, CA, USA)。
1.2 实验方法
1.2.1 LG37 基因组 DNA 提取
利用LB固体培养基对LG37进行活化培养,挑取长势良好的单菌落接种于LB 液体培养基,培养至对数期时,通过离心的方式对培养菌液进行集菌及DNA提取,DNA 提取的具体操作流程根据天根生化试剂盒说明书进行。利用NanoDrop 2000/2000c (Thermo Scientific, Wilmington, DE, USA) 对提取、纯化的LG37基因组质量进行分析。其DNA(OD260/280) 值在 1.8~2.0,质量浓度≥20 ng·μL−1可用于后续的建库测序。
1.2.2 系统进化树分析
将扩增获得的 LG37 16S rDNA 序列与 NCBI中的芽孢杆菌16S rDNA序列运用DNAMAN软件进行比对分析。应用MEGA 6.0软件对LG37和下载的16S rDNA进行遗传进化分析。其中序列比对采用Clustal W 多重比较法进行,基于邻接法 (Neighbor-joining) 构建系统进化树,确定菌株的进化分类[15]。
1.2.3 文库构建和库检
采用三代 PacBio RS II结合二代 Illumina HiSeq 2000测序的方法对LG37进行全基因组测序,其完成单位为上海欧易生物医学科技有限公司[16]。经质检合格的 DNA 样品采用 PacBio RS II single-molecule real-time (SMRT) 测序技术 (Pacific Biosciences, Menlo Park, CA, USA) 对其进行全基因组测序。然后通过分级基因组装配(The hierarchical genome-assembly process, HGAP) 对随机打断的测序读段进行装配,以期获得超长的测序读段。接下来使用二代测序技术Illumina HiSeq 2000进行测序[17],并利用 Bowtie2 (v2.3.0) 将 Illumina 基因片段与组装的SMRT-contigs进行比对[18],从而获得完整的环状全基因组序列信息。用Glimmer 3.02进行基因预测和注释[19],用tRNAscan-SE进行tRNA genes的预测和筛选[20],用RNAmmer进行rRNA 的预测分析[21]。
1.2.4 本地 Blast+功能基因比对
通过NCBI下载本地Blast+,即“基本局部对比搜索工具”(Basic Local Alignment Search Tool, https://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/)。首先对于基因组测序获得的蛋白注释信息进行模型建库,然后将NCBI (https://www.ncbi.nlm.nih.gov/)和 UniProt (https://www.uniprot.org/) 数据库相关信息进行下载、比对、筛选和分析,获得无机氮代谢相关候选基因,并注释其GO分子功能[22]。
2 结果
2.1 LG37 基因组组装
通过三代 PacBio RS II结合二代 Illumina HiSeq 2000测序平台对LG37 进行全基因组测序分析(表1)。97 382 个平均长度 3 016 nt的测序读段被最终拼装成一条重叠基因组序列,长度为3 949 250 bp。通过测序比对分析,最终获得一条 3 929 697 bp的环状染色体基因组,GC含量为46.5%,总基因数3 967 个 (图1)。其中蛋白编码基因 3 854 个,编码区域长度为 3 495 864 bp,占总长度的 89.0%,GC含量为47.3%。RNA 基因113个,其中tRNA基因86个,rRNA基因27个。基因间隔区域长度433 833 bp,占总长度的11.0%。该LG37菌株无载体。
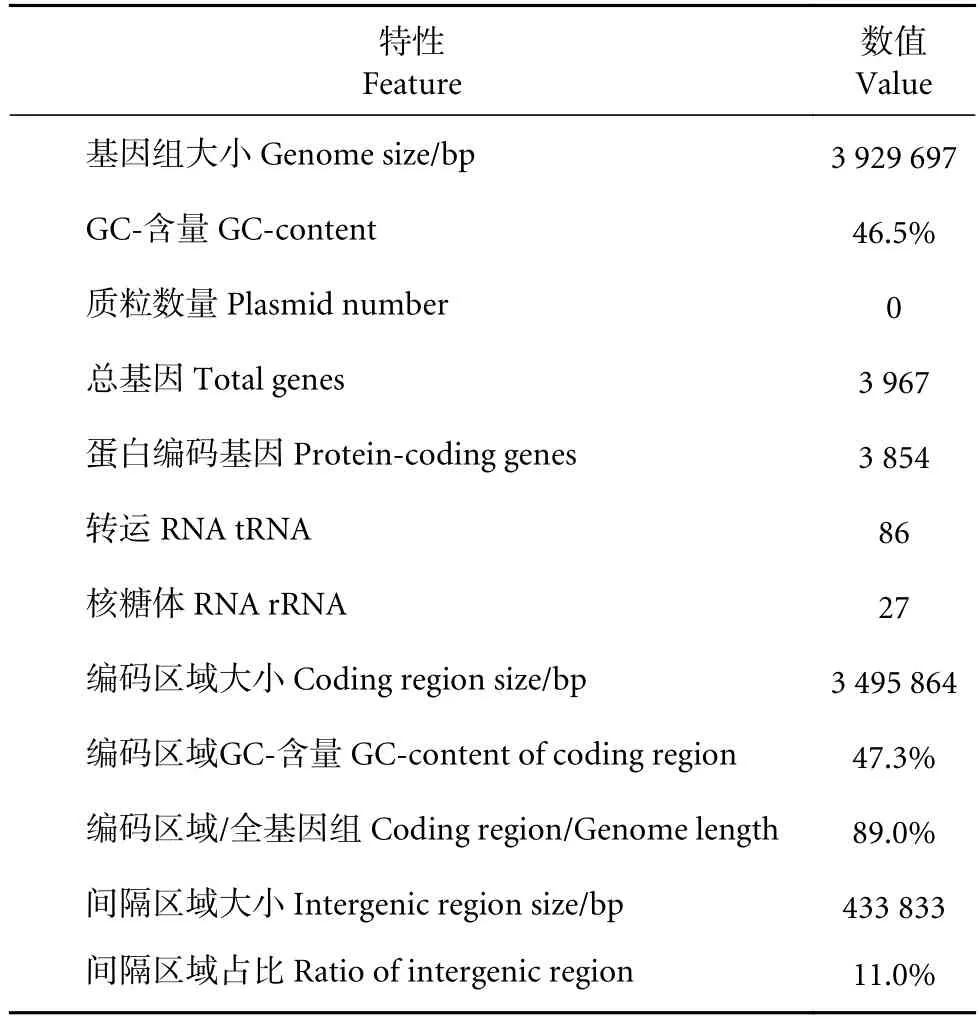
表1 贝莱斯芽孢杆菌LG37 基因组特性Table 1 Genome features of B. velezensis LG37 strain
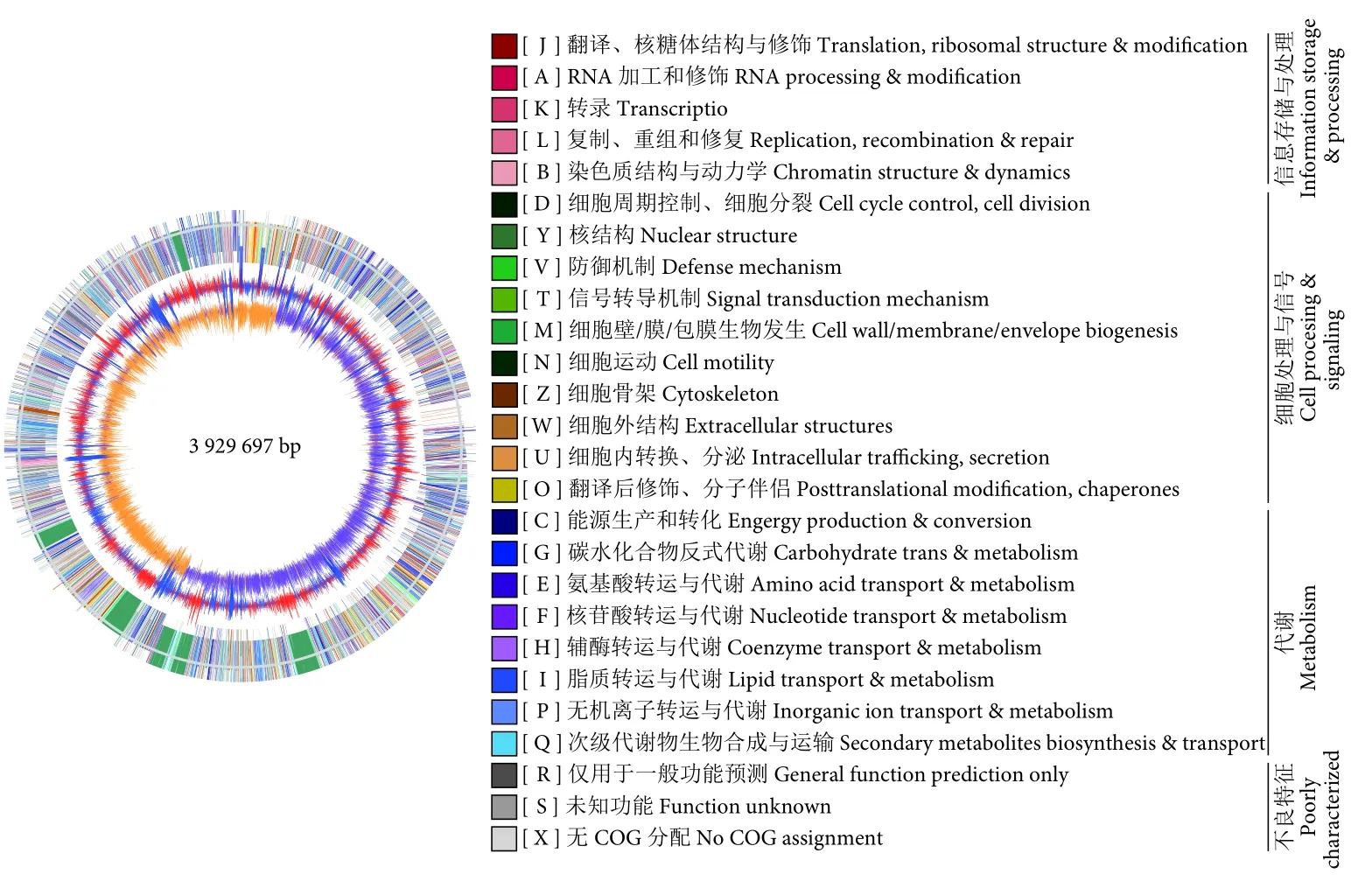
图1 LG37 环状基因组图谱Fig. 1 Circular genome map of B. velezensis LG37 strain
2.2 LG37 系统进化树分析
经全基因组测序结果显示,其16S rDNA基因序列长度为 1 445 bp。通过 NCBI→Blast→Nucleotide Blast在线分析软件比对数据库中已收录的16S ribosomal RNA sequences,并结合基因组测序数据匹配分析。结果表明,筛选的芽孢杆菌菌株为水源贝莱斯芽孢杆菌 (B. velezensis),菌株命名为LG37。将LG37菌株与NCBI数据库中进化关系较近的芽孢杆菌的16S rDNA序列进行比对,并通过MEGA 6.0软件中的邻接法构建系统发育树 (图2)。发现 LG37 与B. velezensisFZB42 亲缘关系最近,16S rDNA序列相识度高达100%。
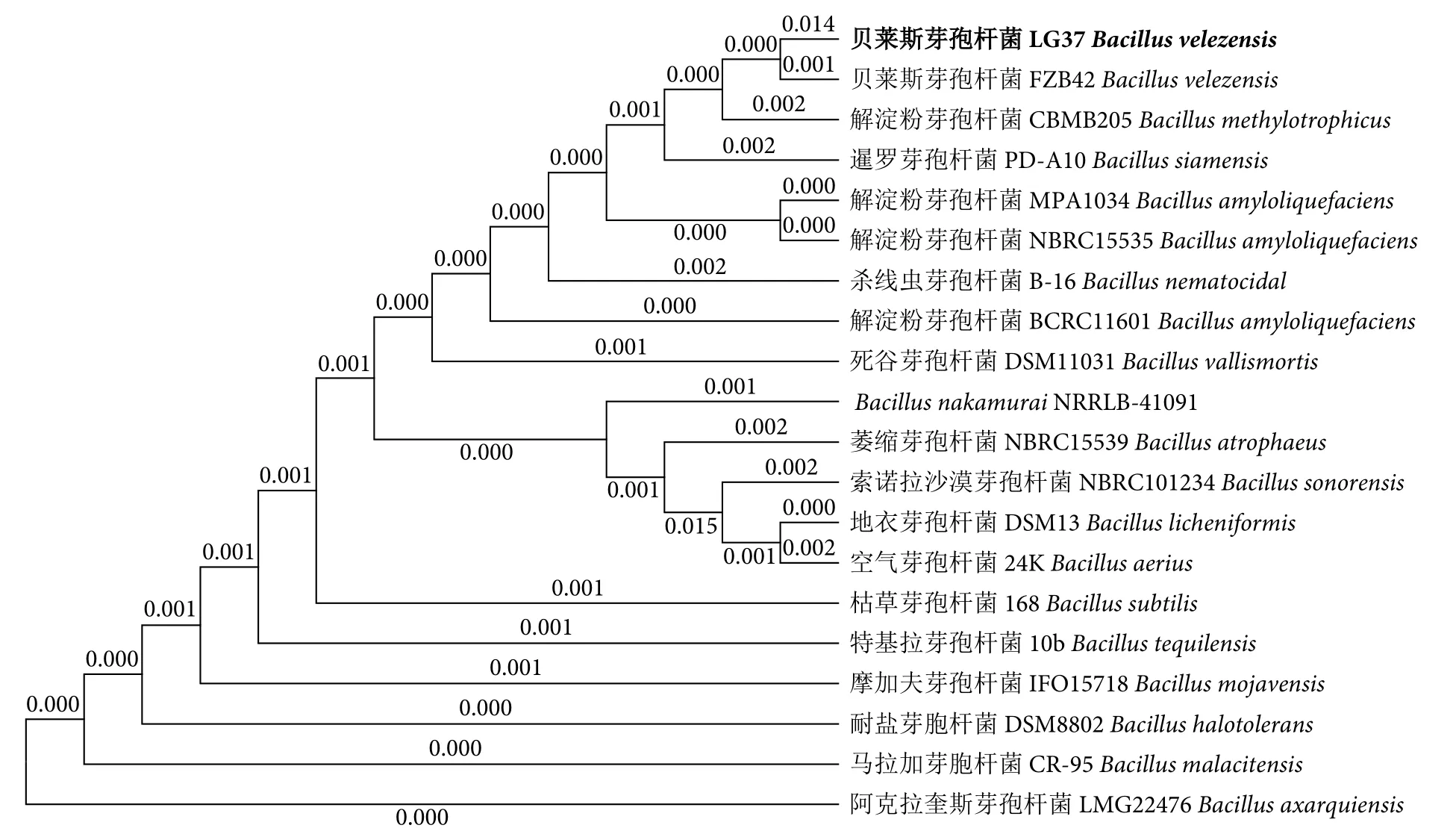
图2 贝莱斯芽孢杆菌基于16S rDNA 序列构建的系统进化树Fig. 2 Neighbor-joining tree of B. velezensis LG37 based on 16S rDNA sequences
2.3 LG37 基因功能注释
对全基因组测序获得的蛋白编码基因,通过搜寻 NCBI的 nr库、KEGG (Kyoto Encyclopedia of Genes and Genome) 蛋白数据库以及 SEED 蛋白数据库进行基因功能注释分析,利用CDD数据库进行 COG (Clusters of Orthologous Groups) 分类,并通过KEGG数据库构建代谢通路。全基因组测序共获得3 854个蛋白编码基因,具有明确生物学功能的蛋白编码基因3 057个,具有KEGG的ortho-log蛋白编码基因2 108个,具有COG分类的蛋白编码基因2 890个。所有蛋白匹配的同源蛋白来源于 75 个物种,其中B. velezensisFZB42 比例最高(78.4%)。
2.3.1 COG 功能分类
LG37完整基因组的蛋白编码基因的氨基酸序列数据与测序所得数据进行匹配获得蛋白序列注释,并根据功能进行COG class聚合分析。COG分类的蛋白编码基因共2 890个,排名前3的分别为:General function prediction only 324个,Cell motility and secretion 312 个,Coenzyme metabolism 283 个,共占比 31.8% (图3)。
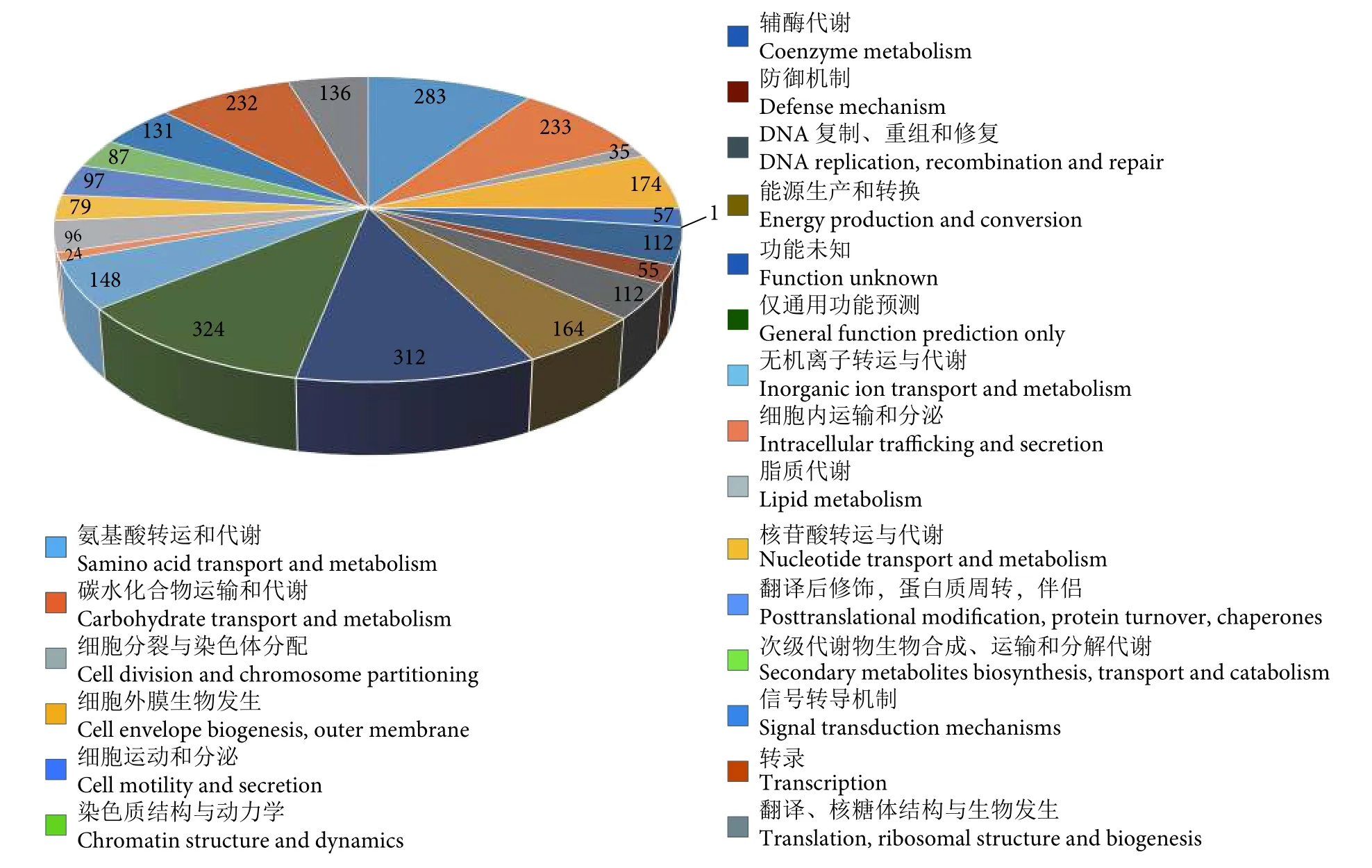
图3 COG功能分类Fig. 3 COG classification
2.3.2 KEGG 代谢通路分析
LG37全基因组测序获得的氨基酸数据经过蛋白注释后,与KEGG数据进行比对分析,将目的基因注释蛋白与想匹配的蛋白功能注释信息相结合,获得目的基因的翻译蛋白信息。具有KEGG的ortholog蛋白编码基因2 108个,共匹配到160个KEGG代谢通路中。其中排名前6的KEGG通路匹配基因数分别为:碳水化合物代谢(Carbohydrate metabolism) 和氨基酸代谢 (Amino acid metabolism) 各 157 个,膜转运 (Membrane transport) 104个,辅酶因子和维生素的代谢(Metabolism of cofactors and vitamins) 93 个,信号转导 (Single transduction) 90 个,核苷酸代谢 (Nucleotide metabolism) 82 个,共计 683 个,占总数的32.4%。其中涉及到氮代谢相关的KEGG通路主要集中在GO的Cell Metabolism中,包括:氨基酸代谢,核苷酸代谢,辅酶因子和维生素的代谢,能量代谢 (Energy metabolism),其他次生代谢产物的生物合成 (Biosynthesis of other secondary metabolites and amino acid metabolism),详见图4。
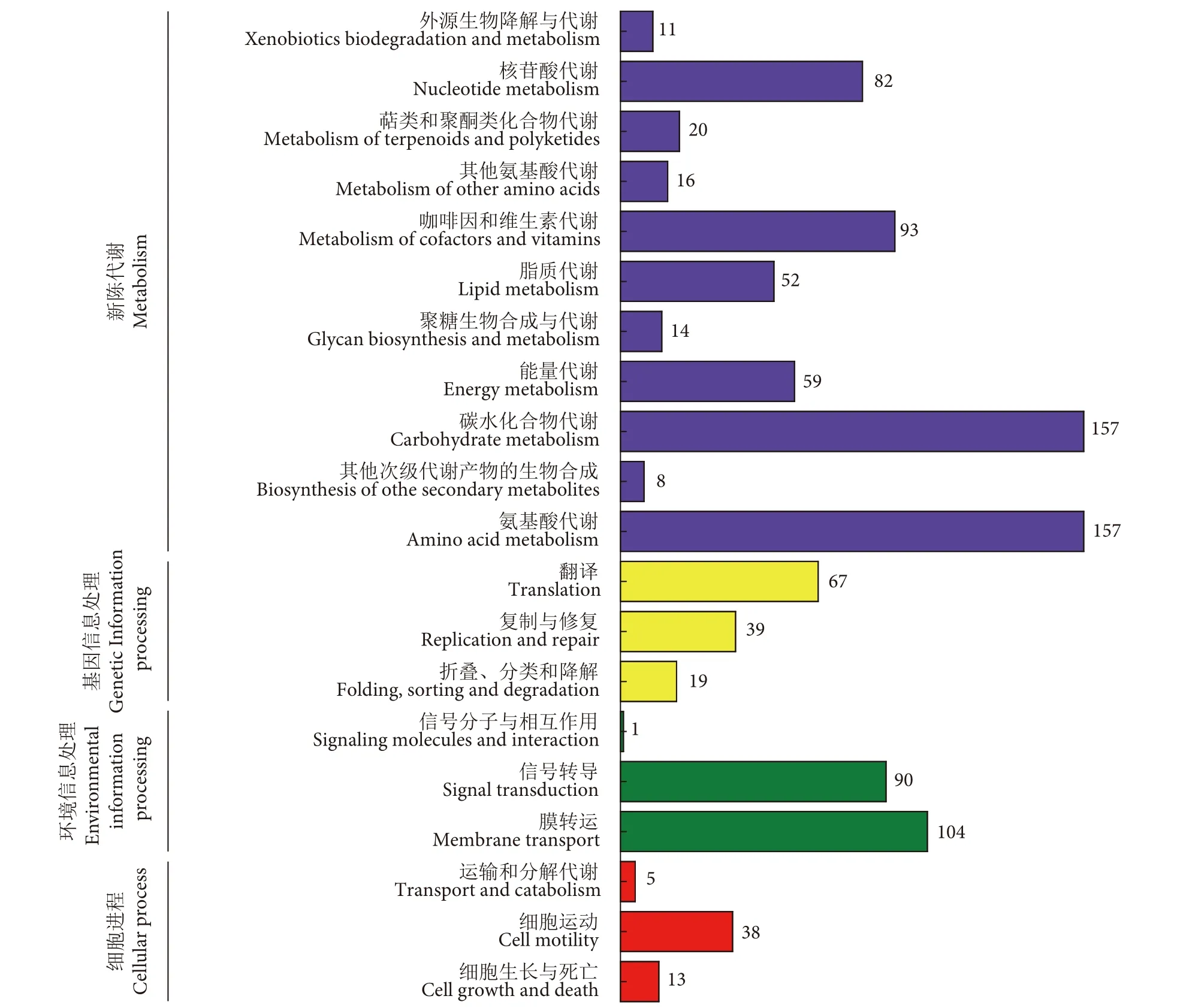
图4 LG37基因功能注释KEGG代谢通路Fig. 4 Gene KEGG pathway classification map of LG37
LG37同化无机氮的主要代谢通路为氮代谢(ko00910),通过基因组测序分析,共筛选得到18个基因被匹配至氮代谢通路中 (表2)。其中涉及的Pathway ID及匹配的通路基因分别参考 https://www.genome.jp/pathway/map00910和图5。
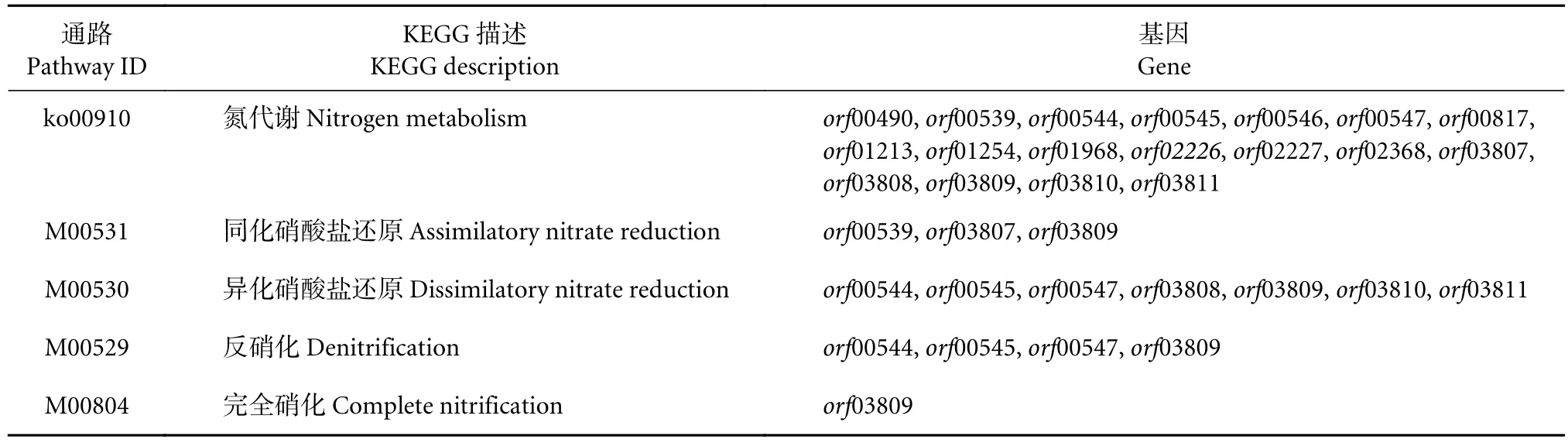
表2 LG37 基因组氮代谢通路及其相关基因Table 2 Related genes of nitrogen metabolism pathways of LG37
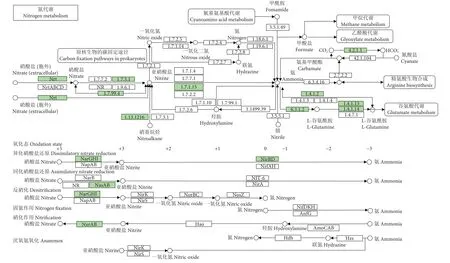
图5 氮代谢通路(ko00910)Fig. 5 Nitrogen metabolism (ko00910)
2.4 无机氮代谢相关候选基因的筛选
测序数据分析结果显示,共有18个基因涉及到氨氮代谢通路中。然后利用本地Blast+将测序获得的全基因组信息与NCBI和UniProt数据库中无机氮代谢相关数据进行比对分析,额外筛选出无机氮代谢候选基因76个。其中共筛选出转运蛋白编码基因7个,氧化还原蛋白编码基因13个,调节蛋白编码基因5个,结合蛋白和转录调控因子编码基因8个,同化蛋白编码基因6个及其他无机氮代谢相关基因 (表3)。

表3 无机氮代谢候选基因Table 3 Candidate genes of inorganic nitrogen metabolism
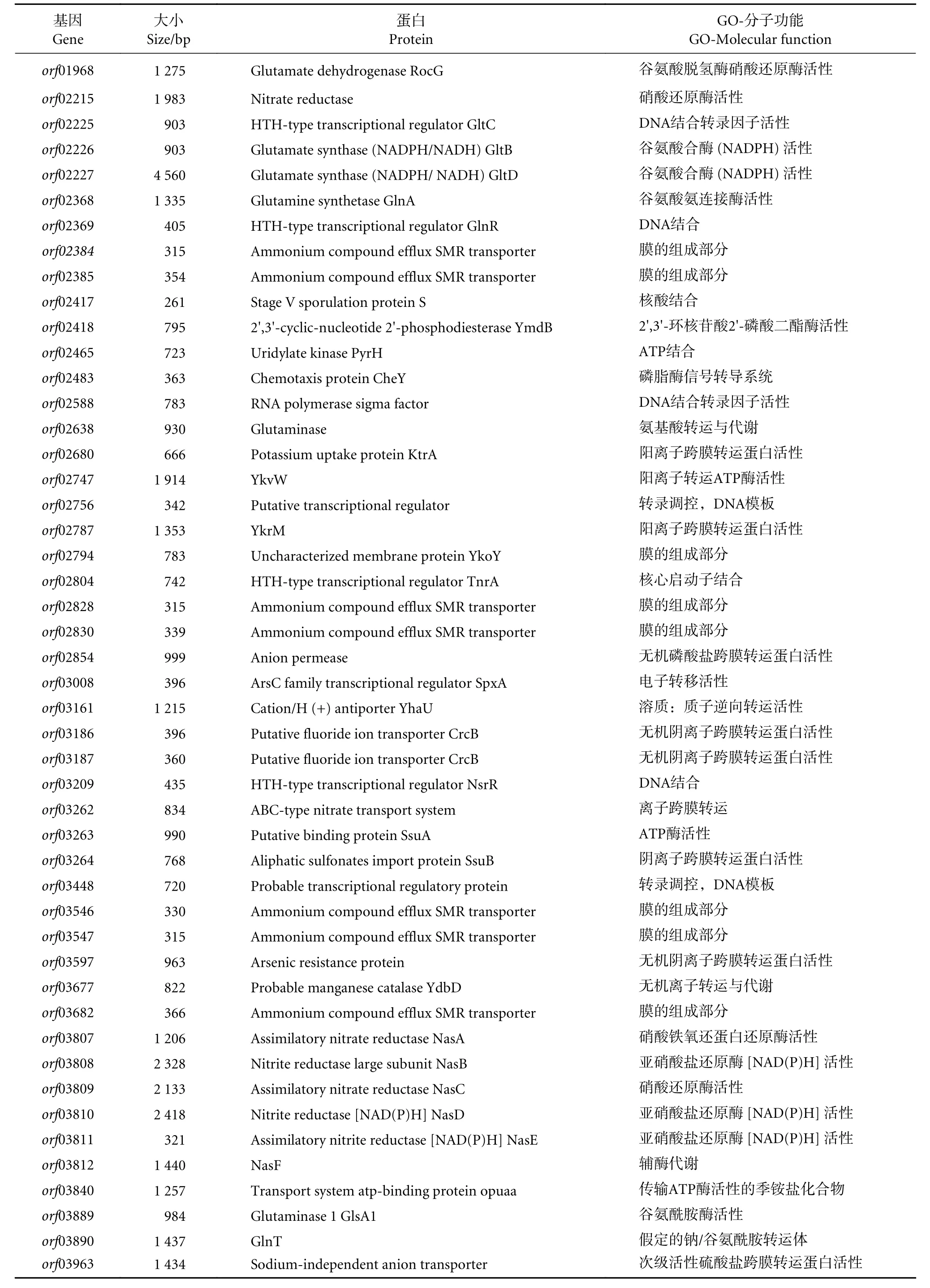
续表3 Ato be continued
3 讨论
近年,随着高密度养殖等模式的逐步完善,我国水产养殖单位面积产量呈逐年上升趋势,但高产出的同时也伴随着饲料营养的大量投入,其每天产生氨氮的量可根据公式初步计算[23-25]:Q=F×R×0.144 [Q为氨氮产生的总量;F为配合饲料投喂总量 (kg·d−1);R为饲料中蛋白质所占比例;式中蛋白氮含量约16%,0.144是指90%的被经济动物吸收的氮作为氨和尿素氮排出 ]。养殖水体中由于4种无机氮之间可相互转化,且因水体中多种理化指标及微生物等多重作用因素而处于动态平衡状态,因此,虽然水体中NH3和对水生动物有毒害作用,但不能仅降低NH3和的浓度,而应在降低NH3或浓度的同时改良水体中的微生物菌落组成和水体中总无机氮含量[2-3],实现降低无机氮浓度的目的。
贝莱斯芽孢杆菌不仅可降低无机氮的浓度,而且对养殖水体中病原微生物的生长、繁殖起到很好的抑制效果。雷阳等[26]在对虾养殖池塘中筛选获得一株可高效同化氨氮的芽孢杆菌,后经鉴定并命名为贝莱斯芽孢杆菌FS017。Zhu等[27]筛选获得一株贝莱斯芽孢杆菌CPA1-1,可以很好地降低养殖水体中氨氮和亚硝态氮的浓度。同时,贝莱斯芽孢杆菌可对副溶血弧菌 (Vibrio parahaemolyticus)、哈维氏弧菌 (V. harveyi)、嗜水气单胞菌 (Aeromonas hydrophila) 等常见水生动物病原菌起到很好的抑制效果[28]。表明贝莱斯芽孢杆菌在水产养殖领域具有较高的应用价值,因此对其无机氮同化机理进行研究非常必要。
为从基因水平探讨贝莱斯芽孢杆菌同化无机氮的机理,本研究对LG37的基因组进行了测序分析。结果显示,该菌株基因组序列全长 3 929 697 bp,经过基因功能注释,共有3 057个蛋白具有明确的生物学功能,所有蛋白匹配的同源蛋白来源于75个物种,其中贝莱斯芽孢杆菌FZB42比例最高(78.4%)[29]。通过基因注释和本地Blast+的筛选、分析,3 057个功能蛋白编码基因中,与NCBI和UniProt数据库中记录的无机氮代谢相关基因相匹配的共有94个,约占功能基因的3.1%。其中涉及多种特异性无机氮同化功能蛋白的编码基因,包括感应蛋白 (GlnK)、转运蛋白(MnrA、NarT和NrgA)、氧化还原蛋白 (NasABD、NarIGHK)等相应编码基因[7],其通路下游涉及的同化蛋白和调节蛋白等不仅参与无机氮的同化代谢,同时也参与有机氮的同化代谢。本结果表明LG37同化无机氮的代谢途径与有机氮的代谢途径之间,仅在通路上游的感应、转运、氧化还原至氨的相关节点的功能蛋白上具有特异性,而对氨下游涉及的氮化合物合成代谢相关途径中,不论氮源是无机氮还是有机氮,其代谢途径均一致,涉及的蛋白的功能和作用也一致。
氮元素在微生物新陈代谢过程中必不可少[30],进化过程中为适应环境因子的变化和氮源的多样性,微生物对氮元素的同化代谢已逐渐发展成为多元、互补、交叉及全局性的补偿性代谢网络途径,进而实现对不同种类氮源的利用[31-33]。微生物在代谢过程中涉及的相关功能蛋白,其在基因组上的编码基因或基因簇往往处于相临或相近的位置[34-35],以此提高微生物的代谢效率,实现营养物质的高效利用,同时降低自身的能量损耗[36-37]。本研究发现,orf00316—orf00630和orf03597—orf03963两个区段基因翻译的蛋白功能主要涉及无机氮的转运和氧化还原,并最终生成NH3或说明该区段内的基因翻译蛋白主要行使的功能是微生物对环境中无机氮的感知、转运、氧化还原、吸收同化等;而筛选的其他基因翻译的蛋白在无机氮或有机氮的代谢途径中均行使相应的功能。
综上,贝莱斯芽孢杆菌LG37基因组序列的解析为深入研究其同化无机氮的关键功能基因提供了参考依据,为进一步研究其无机氮的代谢途径提供了理论数据。后续将聚焦于orf00316—orf00630和orf03593—orf03963基因区段的研究,即LG37感知、转运、氧化还原等无机氮同化途径。
