基于扬麦158/CI12633重组自交系群体的籽粒千粒重QTL分析
2022-06-16郭元世梅佳罗德祥涂军陆健康厉婕侯运章吕乐城周淼平
郭元世 梅佳 罗德祥 涂军 陆健康 厉婕 侯运章 吕乐城 周淼平



摘要 利用高产广适亲本扬麦158和抗纹枯病种质资源CI12633构建的重组自交系(RIL)群体为研究对象,采用ddRAD-seq技术开发SNP标记并构建遗传连锁图,结合RIL群体2018—2020年的千粒重表型数据进行千粒重相关QTL的定位。结果表明:RIL群体的千粒重存在超双亲分离,且基本符合正态分布,RIL群体家系间及年度间的差异均达极显著水平;2018—2020年共检测到5个QTL位点,分别位于1A、4A、6A、6B和7D染色体,单个QTL可解释9.70%~21.80%的表型变异,其中位于4A和6B染色体的2个QTL位点可在不同年份重复检测到,可能为主效QTL,可用于分子标记辅助选择育种。
关键词 小麦;重组自交系;千粒重;QTL
中图分类号 S512.1 文献标识码 A 文章编号 0517-6611(2022)11-0098-03
doi:10.3969/j.issn.0517-6611.2022.11.025
开放科学(资源服务)标识码(OSID):
Analysis of QTLs for the Thousand-gran Weight in a Recombiannt Inbred Lines Population from Yangmai158/CI12633
GUO Yuan-shi,MEI Jia,LUO De-xiang et al
(Jiangsu Choosan Co.,Ltd.,Nanjing,Jiangsu 221500)
Abstract The objectives of the present study were to detect QTLs of thousand grain weight using ddRAD-seq technology to develop SNP markers and construct genetic linkage map in a recombinant inbred lines population derived from a cross between Yangmai158 and CI12633,and evaluated for thousand grain weight at three years under organic management.The results showed that the thousand grain weight of the recombinant inbred lines has super parental separation,and basically conforms to the normal distribution.The differences between families and years of RIL population have reached a very significant level.Five QTLs for TKW were mapped on chromosomes 1A,4A,6A,6B and 7D.QTL showing proportions of 9.70%-21.80% of phenotypic variance explained in at least two environments were located on chromosomes 4A and 6B, which may prove useful for MAS for improvement of the thousand grain weight in bread wheat.
Key words Wheat;RIL population;Thousand-grain weight;QTL
普通小麦 (Triticum aestivum L.)是全球種植面积最广的粮食作物之一,全世界约有40%的人口以小麦为主粮[1]。据国家统计局数据,我国年均小麦播种面积和产量分别为2 338万hm2和13 425万t,其中小麦播种面积自2016年以来呈下降趋势,而小麦总产量因新品种、新技术的应用而稳中略增[2]。小麦作为分蘖成穗特性作物,其单位面积产量由单位面积穗数、每穗粒数和千粒重决定,即产量“三要素”,其中千粒重的遗传率最高,且与穗数和穗粒数的相关性小,因此,在不能扩大种植面积的情况下,提高小麦单产是提高小麦总产及保障粮食安全的重要措施[3]。
近年来,通过不同遗传背景、环境条件等方式发现了大量关于小麦千粒重的QTL位点,但这些QTL位点因受遗传效应不稳定、解释率偏低等影响,只有较少QTL位点能通过开发相应分子标记用于小麦辅助育种[4-9]。该研究利用长江中下游高配合力农艺亲本扬麦158和抗纹枯病资源CI12633构建充足自交系(RIL)群体,并利用双亲间存在多态性的SSR标记和采用ddRAD-seq技术开发的SNP标记,构建高密度连锁图谱。通过连续3年测定扬麦158、CI12633和94个RIL家系的千粒重,进行小麦千粒重的QTL分析,以期获得有效的QTL位点并筛选出可直接用于小麦辅助育种的分子标记。
1 材料与方法
1.1 试验材料及田间设计
群体以高产稳产高配合力品种扬麦158(江苏里下河地区农业科学研究所提供,千粒重约44 g)为母本,抗纹枯病种质资源CI12633(江苏省农业科学院种质资源与生物技术研究所提供,千粒重约28 g)为父本,杂交配组后采用连续单粒传法于2018年构建了94个F8代RIL家系。
94个RIL群体及双亲于2018—2020年种植于江苏中江种业国家杂交水稻育种创新基地,试验采用随机区组设计,3次重复,每个材料单行播种,行长1 m,行距25 cm, 每行50粒。田间管理同当地大田。
1.2 RIL群体遗传连锁图的构建
RIL群体的基因型分析和遗传连锁图的构建参照文献[10],构建的遗传连锁图含有31个连锁群,均可分配到相应的染色体,总遗传距离2 510.7 cM,含有分子标记3 355个,标记间平均遗传距离为0.75 cM。
1.3 千粒重调查和数据分析
成熟期每材料随机剪50穗混脱并晾晒至安全水分,随机调查1 000个完整籽粒并称重,以各家系每年3次重复的平均值为千粒重表型值进行数据分析。数据分析采用EXCEL和MATLAB软件。
1.4 QTL定位 结合遗传连锁图[10]和各家系的千粒重,用软件MapQTL 4.0先进行QTL区间作图,然后利用筛选的辅因子进行多QTL作图,并增加LOD值判断是否存在QTL。
2 结果与分析
2.1 双亲及RIL群体千粒重的平均值及表型变异
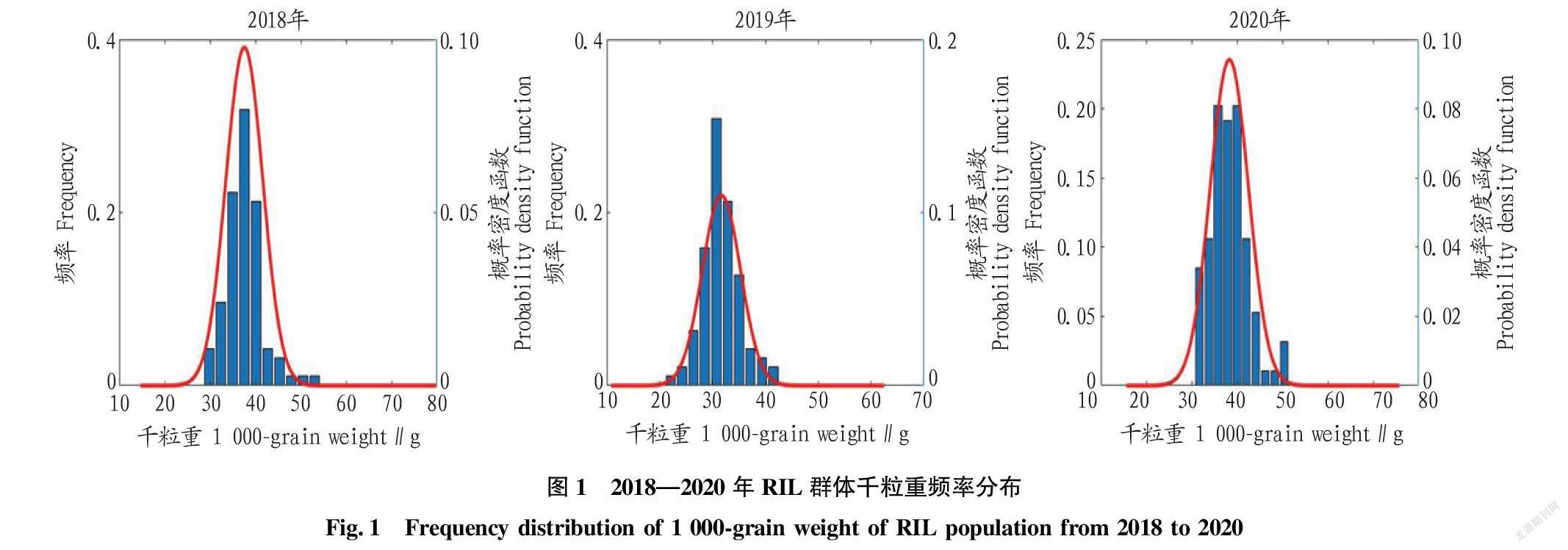
由表1可知,母本扬麦158千粒重为42.17~48.78 g,父本CI12633的千粒重为23.41~ 32.25 g,双亲差异明显。RIL群体3年的千粒重均值均介于双亲之间,而在2018年和2020年RIL群体的千粒重均存在双向超亲分离。RIL群体3年的千粒重频率分布见图1,可以看出千粒重基本呈正态分布,说明小麦千粒重为数量遗传性状。经方差分析可知,RIL群体家系间、不同试验年份间千粒重的差异均达到极显著水平。
2.2 籽粒千粒重的QTL分析
利用构建的遗传连锁图谱和2018—2020年RIL群体表型数据进行QTL分析,共检测到8个与籽粒千粒重相关的QTL位点,其中2018年检测到1个位于6B染色体的QTL位点,该位点加性效应值为1.42,可解释11.5%的表型变异;2019年检测到4个分别位于1A、4A、6A和6B染色体的QTL位点,加性效应值分别为1.25、1.29、2.87和1.60,可分别解释11.0%、9.7%、13.0%和18.4%的表型变异;2020年共检测到3个分别位于4A、6B和7D染色体的QTL位点,加性效应值分别为1.41、1.45和3.92,可分别解释10.8%、11.5%和21.8%的表型变异(表2)。检测到增加粒重的QTL位点均由亲本扬麦158提供,QTL染色体分布见图2。
3 讨论
该研究发现的6B染色体上位于80.83~84.45 cM的QTL在2018—2020年研究结果中均可检测到,LOD值最高为4.15,且贡献率均在10%以上。经比较前人研究结果发现,吴旭江等[6]利用扬麦9号和CI12633构建的RIL群体也在6B染色体定位到稳定的QTL,其解释率达25.61%~31.39%,该位点(Xmag3298.1~GPW222)与定位到的QTL位点相近。WU等[7]利用Yanda1817和Beinong6构建的RIL群体在6B染色体上定位了3个与小麦千粒重相关的QTL,其中与该研究定位到的QTL相近的 QTgw.cau-6B.3 可在3個不同环境下被监测到,且在该位点及相邻位点还存在控制籽粒长度的QTL。另外,Blanco等[8]、Tsilo等[9]、Ramya 等[11]分别在6B染色体(标记区间分别为CA594434d~wPt-3581、xWpt0052和Xgwm889~Xgwm1486)检测到与千粒重相关的QTL,且相关位点与该研究检测到的QTL位点相近。因此,可以推断该研究中定位的6B染色体上的千粒重QTL为稳定主效QTL,可用于分子标记辅助选择育种。
该研究发现的4A染色体上位于75.04~75.25 cM的QTL在2019和2020年结果中均可检测到,2年贡献率分别为9.7%和10.8%。经比较前人研究发现, Zou等[12]利用一个RIL群体(Attila×CDCGo)定位到一个与该研究位点相近的主效QTL (标记区间wsnp_Ex_c7899_13416443~ wsnp_Ex_rep_c97236_84366722) ,2年LOD值分别为5.8和9.3,而Maphosa等[13]在4A染色体wPt-0023标记附近定位到的QTL与该研究获得的QTL位点相近,其LOD值达3.6,均说明该位点附近存在主效稳定QTL。Liu等[14]在4A染色体与该研究相近位点定位到2个与千粒重相关的QTL(标记区间分别为Xwmc491~Xbarc1052和Xbarc28b~Xcau195),且该位点附近还定位到与穗粒重相关的QTL,说明该QTL可能通过影响千粒重从而影响单穗粒重。以上研究均说明该研究中定位于4A染色体上的千粒重QTL为稳定主效QTL,可用于分子标记辅助选择育种。
该研究中在7D染色体上定位到的一个QTL位点,其LOD值达到5.54,贡献率为21.8%,可能为一个主效QTL,但该QTL未能在2018和2019年监测到,这可能与该基因受环境影响较大有关。Mir等[15]在7D染色体短臂上(Xgwm295~Xgwm437)定位到一个与该研究相近(xwmc463~xgdm67)的QTL,且该位点的LOD值达到20.14,这与Huang等[16-18]获得的QTL位点相近(均在Xgwm295~Xgwm437),说明该位点可能是一个主效QTL,但由于受环境影响较大,需要进一步精细定位并确定其利用价值。
.
参考文献
[1]
GUPTA P K,MIR R R,MOHAN A,et al.Wheat genomics: Present status and future prospects[J].International journal of plant genomics,2008,2008:1-36.
[2] 刘志勇,王道文,张爱民,等.小麦育种行业创新现状与发展趋势[J].植物遗传资源学报,2018,19(3):430-434.
[3] WU W,LI C J,MA B L,et al.Genetic progress in wheat yield and associated traits in China since 1945 and future prospects[J].Euphytica,2014,196(2):155-168.
[4] ZANKE C D,LING J,PLIESKE J,et al.Analysis of main effect QTL for thousand grain weight in European winter wheat ( Triticum aestivum L.) by genome-wide association mapping[J].Frontiers in plant science,2015,6:1-14.
[5] ZHANG X Y,DENG Z Y,WANG Y R,et al.Unconditional and conditional QTL analysis of kernel weight related traits in wheat ( Triticum aestivum L.) in multiple genetic backgrounds[J].Genetica,2014,142(4):371-379.
[6] 吳旭江,臧淑江,程凯,等.扬麦9号/CI12633 RIL群体中控制小麦粒重QTL位点的初步分析[J].扬州大学学报(农业与生命科学版),2015,36(4):90-95.
[7] WU Q H,CHEN Y X,ZHOU S H,et al.High-density genetic linkage map construction and QTL mapping of grain shape and size in the wheat population Yanda1817 × Beinong6[J].PLoS One,2015,10(2):1-17.
[8] BLANCO A,MANGINI G,GIANCASPRO A,et al.Relationships between grain protein content and grain yield components through quantitative trait locus analyses in a recombinant inbred line population derived from two elite durum wheat cultivars[J].Molecular breeding,2012,30(1):79-92.
[9] TSILO T J,HARELAND G A,SIMSEK S,et al.Genome mapping of kernel characteristics in hard red spring wheat breeding lines[J].Theoretical and applied genetics,2010,121(4):717-730.
[10] 周淼平,姚金保,杨学明,等.小麦纹枯病抗性QTL分析[J].麦类作物学报,2020,40(5):554-559.
[11] RAMYA P,CHAUBAL A,KULKARNI K,et al.QTL mapping of 1000-kernel weight,kernel length,and kernel width in bread wheat ( Triticum aestivum L.)[J].Journal of applied genetics,2010,51:421-429.
[12] ZOU J,SEMAGN K,IQBAL M,et al.QTLs associated with agronomic traits in the Attila × CDC Go spring wheat population evaluated under conventional management[J].PLoS One,2017,12(2):1-20.
[13] MAPHOSA L,LANGRIDGE P,TAYLOR H,et al.Genetic control of grain yield and grain physical characteristics in a bread wheat population grown under a range of environmental conditions[J].Theoretical & applied genetics,2014,127(7):1607-1624.
[14] LIU G,JIA L J,LU L H,et al.Mapping QTLs of yield-related traits using RIL population derived from common wheat and Tibetan semi-wild wheat[J].Theoretical and applied genetics,2014,127(11):2415-2432.
[15] MIR R R,KUMAR N,JAISWAL V,et al.Genetic dissection of grain weight in bread wheat through quantitative trait locus interval and association mapping[J].Molecular breeding,2012,29(4):963-972.
[16] HUANG X Q,C STER H,GANAL M W,et al.Advanced backcross QTL analysis for the identification of quantitative trait loci alleles from wild relatives of wheat ( Triticum aestivum L.)[J].Theoretical and applied genetics,2003,106(8):1379-1389.
[17] SOMERS D J,ISAAC P,EDWARDS K.A high-density microsatellite consensus map for bread wheat ( Triticum aestivum L.)[J].Theoretical and applied genetics,2004,109(6):1105-1114.
[18] R DER M S,HUANG X Q,B RNER A.Fine mapping of the region on wheat chromosome 7D controlling grain weight[J].Functional & integrative genomics,2008,8(1):79-86.
