光皮桦BlBLH1基因的克隆、表达及互作蛋白筛选*
2022-06-15庄和必俞子承林二培黄华宏
庄和必 俞子承 林二培 黄华宏
(浙江农林大学 亚热带森林培育国家重点实验室 杭州 311300)
木材(次生木质部)的形成是一个涉及形成层细胞分裂、细胞伸长、次生壁大量沉积、细胞程序性死亡、心材形成等步骤的重要生物学过程,受到复杂的转录网络调控(Plomionetal., 2001)。已报道,BLH(BEL1-like homeodomain)和KNOX (KNOTTED-like homeodomain)与次生壁形成密切相关,它们均属TALE(Three amino-acid loop extension)基因家族。BLH亚基因家族起源古老,几乎存在于所有物种中(Bellaouietal., 2001),如在拟南芥(Arabidopsisthaliana)中有13个成员,水稻(Oryzasativa)中有14个成员(Arnaudetal., 2014, Hamantetal., 2010 , Mukherjeeetal., 2009)。BLH家族基因成员均具有一个高度保守的HD结构域以及HD上游的SKY和BEL结构域,这3个结构域对BLH蛋白功能的行使具有十分重要的意义(Beckeretal., 2002)。在拟南芥中,BLH6、BLH7和BLH10被发现可能冗余地调控植物次生壁形成(Liu, 2015),且BLH6能通过形成OFP4-BLH6-KNAT7复合物的形式参与拟南芥花序茎中管状分子的发育,进而调控拟南芥次生壁的形成(Liuetal., 2015)。表达分析还显示BLH7和BLH10在拟南芥的花序茎中高表达,BLH7可以与肉桂酰辅酶A还原酶(CCR1)启动子结合,可能与木质素的合成有关(Taylor-Teeplesetal., 2015)。此外,Ma等(2019)研究发现,在棉花(Gossypiumhirsutum)中也存在着与拟南芥类似的次生壁合成调控网络,棉花BLH家族成员(BLH1、BLH5和BLH6)可以与GhKNAT7和其他转录因子(OFP1、OFP4和MYB75)相互作用形成复合物,直接与木质素和纤维素生物合成基因的启动子结合,调控纤维次生细胞壁的形成。这些研究表明,BLH转录因子在植物次生壁发育等重要的生物学过程中具有重要调控作用。
光皮桦(Betulaluminifera)属桦木科(Betulaceae)桦木属(Betula)落叶乔木,是我国特有的优良速生用材树种(郑万钧,1983)。广泛分布于云南、贵州、四川、广西和浙江等省区。光皮桦具有生长快、童期短、材质优良等特点,其木材质地良好,供制各种器具; 树皮、叶、芽可提取芳香油和树脂,具有较高的经济价值。材性改良一直是光皮桦育种的主要目标之一。近年来,有关光皮桦木材形成相关的分子机制也取得了一些进展。如Cai等(2018)利用RNA-seq技术对光皮桦应拉木形成过程的基因表达谱进行了分析,鉴定获得了一批在应拉木形成过程中差异表达的基因,并筛选得到了18个与纤维素合成酶基因共表达的转录因子。Huang等(2014)通过原位杂交和表达模式分析发现在光皮桦的8个纤维素合成酶基因中有3个与次生细胞壁合成有关。俞子承等(2019)在光皮桦中克隆了一个木质素合成关键酶-咖啡酰辅酶A-O-甲基转移酶(CCoAOMT)基因,表达分析发现其在木质化茎段中优势表达,且随着木质化程度提高而逐渐增强,单核苷酸变异情况分析表明该基因在不同基因型中存在丰富SNP变异且在演化进程中主要受到了纯化选择压力。但是目前还没有BLH基因参与调控光皮桦木材形成的研究报道。
因此,基于光皮桦基因组序列,对光皮桦BLH基因进行了鉴定,克隆获得了BlBLH1基因,明确了其序列特征和进化关系,并分析了BlBLH1在光皮桦不同组织、器官和应拉木处理过程中的表达模式。同时,利用酵母双杂交技术,筛选获得了与BlBLH1互作的蛋白,且进一步利用BiFC验证了BlBLH1与3个KNOX蛋白的互作关系。这些结果将为深入研究BlBLH1基因在光皮桦木材形成中的功能和调控机制提供重要基础。
1 材料与方法
1.1 植物材料
采用2年生光皮桦无性系‘G49-3’作为基因克隆和表达分析的材料,所用植株均种植于浙江农林大学林木种质资源圃的温室大棚内,样品采集时间为2019年3月至7月。收集的材料包括根(R)、未木质化茎(S1)、木质化茎(S2)、嫩叶(YL)、成熟叶(ML)、雌花序(FI)和雄花序(MI)等7种不同组织器官。其中,根(R)取自无性系‘G49-3’的水培苗; 未木质化茎(S1)和木质化茎(S2)分别取当年生主枝上距枝顶端0~2 cm间和10~12 cm间的茎段; 嫩叶(YL)和成熟叶(ML)分别取自当年生主枝自上而下第2片和第5片; 雌、雄花序在3月中旬采集。应力木(Reaction wood)是指在外力作用下形成的弯曲树干或树枝 (刘一星等, 2012)。由于其具备的特殊材质,应拉木已成为研究阔叶树木材形成机制的重要模型之一。为了分析基因在应拉木形成过程中的表达,选取主干笔直、生长状况良好且较为一致的2年生无性系‘G49-3’植株,将其倾斜摆放,使其主干与垂直方向成45°,在处理30天、60天和90天时,剪取应力区茎段,将皮剥去后采集应拉木(tension wood, TW)及对照组(CK)的外层木质部,液氮速冻后保存于-80 ℃,用于检测基因在应拉木中的表达。植株取样的生物学重复至少3次。
1.2 基因鉴定与生物信息学分析
根据Hamant等 (2010)的报道,从NCBI(http://www.ncbi.nlm.nih.gov)下载13个拟南芥BLH蛋白序列。利用本地Blast软件将拟南芥BLH蛋白序列与光皮桦基因组序列(未发表)进行比对获得候选序列; 然后利用在线工具FGENESH(Softberry, http://www.softberry.com/berry.phtml)预测候选序列的基因结构并获得cds和蛋白序列,最后利用在线工具InterProScan(http://www.ebi.ac.uk/Tools/pfa/iprscan/)进一步分析候选基因的保守结构域完整性,确定编码BLH基因的序列。选择其中1个基因进行克隆验证,并利用DNASTAR软件包确定预测其ORF、编码氨基酸序列、相对分子量和等电点。利用DNAMAN软件进行多序列比对,MEGA7.0的邻接(neighbor joining,NJ)算法构建系统进化树。
1.3 RNA提取与基因克隆
使用PureLinkTMPlant RNA Reagent试剂盒(Thermo Fisher)提取不同组织器官及应拉木的RNA,利用1%琼脂糖凝胶电泳检测RNA完整性,利用Nanodrop测定RNA的纯度和浓度,合格的RNA样本冻存于-80 ℃备用。根据鉴定获得的基因序列,在5′和3′非翻译区设计引物BlBLH1-FF/FR,用于基因克隆,引物序列见表1。逆转录采用PrimeScript II cDNA第1链合成试剂盒(TaKaRa)进行,采用Ex Taq®酶(TaKaRa)进行PCR扩增,将扩增得到的目的片段连接到pEASY-T1 Cloning Kit(TransGen)上,转化大肠杆菌(Escherichiacoli)DH5α,挑取阳性克隆送南京金斯瑞生物科技有限公司测序。
1.4 荧光定量PCR
采用Prime ScriptTMRT reagent Kit反转录试剂盒(TaKaRa)进行逆转录合成cDNA。选择SYBR®Premix Ex-TaqTM试剂盒(TaKaRa)进行定量PCR,定量PCR在CFX96TM Real-Time PCR仪(Bio-Rad)上进行,程序如下: 95 ℃预变性3 min,95 ℃ 5 s,60 ℃ 30 s(此步完成时采集荧光信号),共40个循环。光皮桦BlEF1α作为内参基因(刘文哲等, 2016),采用2-ΔΔCt法处理数据。荧光定量PCR引物序列见表1。不同器官组织和应拉木的表达数据用Duncan法进行多重比较。
1.5 BlBLH1互作蛋白筛选
提取光皮桦‘G49-3’茎和叶的总RNA并反转录为cDNA,使用CloneMiner II cDNA Library Construction Kit试剂盒(Thermo Fisher)构建光皮桦茎叶cDNA文库,通过LR反应将cDNA片段重组至表达载体上(pGADT7)后转化Y187酵母感受态细胞,获得酵母杂交文库,计算滴度为5.23×107CFU·mL-1,总克隆数为2.09×108个,文库质量较高,可用于后续文库筛选实验。利用引物BlBLH1-F/R将BlBLH1基因构建至载体pGBKT7,将构建好的pGBKT7-BlBLH1载体转入酵母菌株Y2HGold,经毒性和自激活检测后用于文库杂交,杂交方法参照Matchmaker® Gold Yeast Two-Hybrid System(Clontech)试剂盒说明书进行。文库杂交后,提取阳性克隆质粒,转化大肠杆菌DH5α后挑单克隆进行测序,将测序获得的序列与NCBI数据库进行序列比对,确定互作的蛋白注释信息。针对筛选获得的BlKNOX4、BlKNOX9和BlSTM基因,分别利用引物BlKNOX4-F/R、BlKNOX9-F/R和BlSTM-F/R将其构建至pGADT7载体,并转化酵母菌株Y187,与pGBKT7-BlBLH1进行杂交,在酵母SD/-Leu/-Trp二缺培养基(以下简称DDO)、酵母SD/-Ade/-His/-Leu/-Trp/+X-α-Gal/+AbA四缺培养基(以下简称QDO/X/A)上筛选,验证它们的互作关系。酵母杂交所用引物均列于表1。
1.6 双分子荧光互补(BiFC)实验
利用引物BlBLH1-inF/R、BlKNOX4-inF/R、BlKNOX9-inF/R、BlSTM-inF/R分别PCR扩增出BlBLH1、BlKNOX4、BlKNOX9和BlSTM的ORF序列,采用ClonExpress®II One Step Cloning Kit试剂盒(Vazyme)将BlBLH1插入到pSAT1-cEYFP的多克隆位点中,将其余基因分别插入到pSAT1-nEYFP的的多克隆位点中,提取质粒,通过PEG介导转化拟南芥原生质体 (赖叶林等, 2020),利用激光共聚焦显微镜(卡尔蔡司LSM880)观察并拍照记录蛋白互作信号。

表1 本研究中使用的PCR引物Tab.1 PCR primers used in this study
2 结果与分析
2.1 BlBLH1基因的克隆
通过生物信息学的方法,在光皮桦基因组中鉴定获得BLH家族基因。选择其中1个进行克隆,经PCR和测序验证后将其命名为BlBLH1(GenBank登录号为MW835211)。序列分析表明该基因序列长度为2 128 bp,包括210 bp 5′UTR、88 bp 3′UTR和1 830 bp ORF,编码609个氨基酸,相对分子量为66.8 kDa,等电点为7.89。通过多序列比对发现,光皮桦BlBLH1与其他植物的BLH蛋白序列类似,都具有一个高度保守的HD结构域,以及HD结构域上游的SKY和BEL结构域(图1)。进一步的系统进化分析也表明,BlBLH1与拟南芥BLH1聚为一个分枝,有较近的亲缘关系(图2)。
2.2 BlBLH1基因的表达分析
通过定量PCR,对BlBLH1基因在不同组织、器官及应拉木形成过程中的表达进行了分析。结果表明,BlBLH1在不同组织器官中的表达存在显著差异,其在雄花序中的表达量最高,其次是雌花序,但在根和非木质化茎段中几乎不表达; 在茎段中,BlBLH1在木质化茎段(S2)中的表达则明显高于非木质茎段(S1)(图3A)。分析BlBLH1在应拉木形成过程中的表达,发现该基因在应拉木形成过程中的表达呈明显上升的趋势,在处理30天、60天和90天时的表达量分别为对照的1.44、3.30和2.56倍(图3)。综上,BlBLH1可能不仅参与了光皮桦花序等器官发育,也可能茎段木质化过程中起重要作用。
2.3 BlBLH1互作蛋白的筛选
利用酵母双杂交技术在光皮桦茎叶文库中筛选出了47个与BlBLH1较强互作的蛋白(图4A,表2)。基因注释的结果表明,这47个蛋白可大致分为3类: 第一类为结构蛋白,如叶绿素a/b结合蛋白、细胞色素b6-f亚基等; 第二类为酶,如L-抗坏血酸过氧化物酶(APX)、甲酸脱氢酶(FDH)等; 第三类为转录因子,如GATA、KNOX等转录因子,其中与BlBLH1互作的KNOX转录因子包括BlKNOX4、BlKNOX9和BlSTM(与拟南芥STM同源的光皮桦KNOX转录因子)3个成员(表2)。

图1 BlBLH1与同源BLH多序列比对分析Fig. 1 Amino acid residue alignment analysis of the BlBLH1 and Homologous BLHsBl: 光皮桦 Betula luminifera; Ptr: 毛果杨 Populus trichocarpa; Gs: 野大豆 Glycine soja.

图2 BlBLH1与拟南芥BLH的系统进化分析Fig. 2 Phylogenetic analysis of BlBLH1 and BLH proteins fromA. thaliana ATH1: ARABIDOPSIS HOMEOBOX 1.
BLH与筛选获得的3个KNOX蛋白互作验证结果表明,含有pGADT7-BlKNOX4、pGADT7-BlKNOX9和pGADT7-BlSTM的酵母菌株分别与含pGBKT7-BlBLH1的酵母菌株杂交后,在筛选培养基(QDO/X/A)上均呈现阳性(显蓝色),与文库杂交的结果一致,但BlSTM与BlBLH1的互作较弱(图4B)。
2.4 体内验证蛋白互作
利用BiFC技术进一步验证BlBLH1和3个KNOX蛋白在植物细胞内的相互作用。结果如图5所示,BlBLH1-cEYFP、BlKNOX4-nEYFP、BlKNOX9-nEYFP和BlSTM-nEYFP单独转化拟南芥原生质体后,并未显示黄色荧光信号; 而将BlBLH1-cEYFP与BlKNOX4-nEYFP、BlKNOX9-nEYFP和BlSTM-nEYFP分别共转拟南芥原生质体后,可以观察到BlBLH1和BlKNOX4、BlBLH1和BlKNOX9蛋白可以发生相互作用呈现黄色的荧光信号,但是BlBLH1-cEYFP 和BlSTM-nEYFP的组合未产生黄色荧光。这些结果说明BlBLH1与BlKNOX4,BlBLH1与BlKNOX9间存在互作,而BlBLH1与BlSTM蛋白在植物细胞内没有互作关系。
2.5 BlKNOX4、BlKNOX9基因的表达模式
为了进一步分析BlBLH1与BlKNOX4、BlKNOX9之间的关系,采用荧光定量PCR对BlKNOX4、BlKNOX9在不同组织器官和应拉木处理过程中的表达进行了检测。结果显示,BlKNOX4和BlKNOX9在不同组织器官中的表达趋势基本一致,

图3 BlBLH1在不同组织、器官和应拉木形成过程中的表达分析Fig. 3 Expression analysis of BlBLH1 in different tissues, organs and tension wood formation标有不同小写字母的样品表示在0.05水平差异显著。R: 根; YL: 嫩叶; ML: 成熟叶; FF: 雌花序; MF: 雄花序; S1: 未木质化茎段; S2: 木质化茎段; TW: 应拉木; CK: 对照。Different small letters above columns indicate significant difference at 0. 05 levels. R: Root; YL: Young leaf; ML: Mature leaf; FF: Female inflorescence; MF: Male inflorescence; S1: Non-lignified stem; S2: Lignified stem; TW: Tension wood.

表2 BlBLH1文库杂交阳性克隆比对结果①Tab.2 The BLAST results of BlBLH1 yeast library hybridization positive clones

续表2 Continued

图4 酵母双杂交部分阳性克隆鉴定及验证Fig. 4 Detection and verification of the positive clones from yeast two hybridA:部分阳性克隆鉴定; B:BlBLH1与3个KNOX蛋白的互作结果验证。A: Detection of partial positive clones; B: Verification of the interactions between BlBLH1 and 3 KNOX proteins. LamT: 阴性对照; 53T: 阳性对照; 图4A中不同数字代表不同的阳性克隆; 图4B数字代表稀释倍数,如1∶10表示稀释10倍。LamT: Negative control; 53T: Positive control; Different numbers in Fig. 4A represent different positive clones; Numbers in Fig. 4B represent dilution ratio, such as 1∶10 for dilution ratio.

图5 BiFC验证BlBLH1与3个BlKNOX 蛋白的相互作用(Bar=10 μm)Fig. 5 Verification of interactions among BlBLH1 and three KNOX proteins by using BiFC (Bar=10 μm)第一列为拟南芥叶片原生质体细胞的YFP荧光信号,第二列为叶绿体自发荧光,第三列为明场下的拟南芥原生质体,第四列为YFP荧光、叶绿体自发荧光和明场的叠加。The first column is yelow fluorescent signal of YFP in Arabidopsis protoplast cells, the second column is chloroplast spontaneous fluorescence, the third column is the bright-field image of Arabidopsis protoplast, and the fourth column is the merged image of YFP fluorescence, chloroplast spontaneous fluorescence and bright-field.
在木质化茎中表达量最高,其次是非木质化茎,在雌花中居于第3位,在其他器官中表达量较低; 在应拉木处理过程中,BlKNOX4先上调表达,然后在90天恢复至对照水平;BlKNOX9的表达也呈现先上升后下调的趋势(图6)。BlKNOX4和BlKNOX9在茎段和应拉木中表达规律与BlBLH1基本一致,这表明BlKNOX4、BlKNOX9在茎段和应拉木中可能与BlBLH1存在共表达的关系,推测共同参与了光皮桦木材形成的过程。
3 讨论
不同植物BLH基因家族成员在次生细胞壁的生物合成及纤维发育调控的时空表达有所差异。已报导,拟南芥BLH1优势表达在种子和茎中 (Kimetal., 2013)。而棉花GhBLH部分成员的表达则在棉纤维发育第20和25天时达到相对较高水平 (Maetal., 2019)。浙江红山茶(Camelliachekiangoleosa)CcBLH6的表达水平和果实木质化过程呈正相关 (Yanetal., 2021)。使用cDNA微阵列技术检测到加拿大松(Pinuscanariensis)PcBLH1(Contig04961) 在次生生长的顶芽中优势表达(Chanoetal., 2021)。本研究测定了光皮桦应拉木形成过程中BlBLH1的表达变化,发现有明显上调趋势,并且木质化(S2)茎段高于非木质化茎段(S1),推测BlBLH1参与了光皮桦茎的木质化过程。
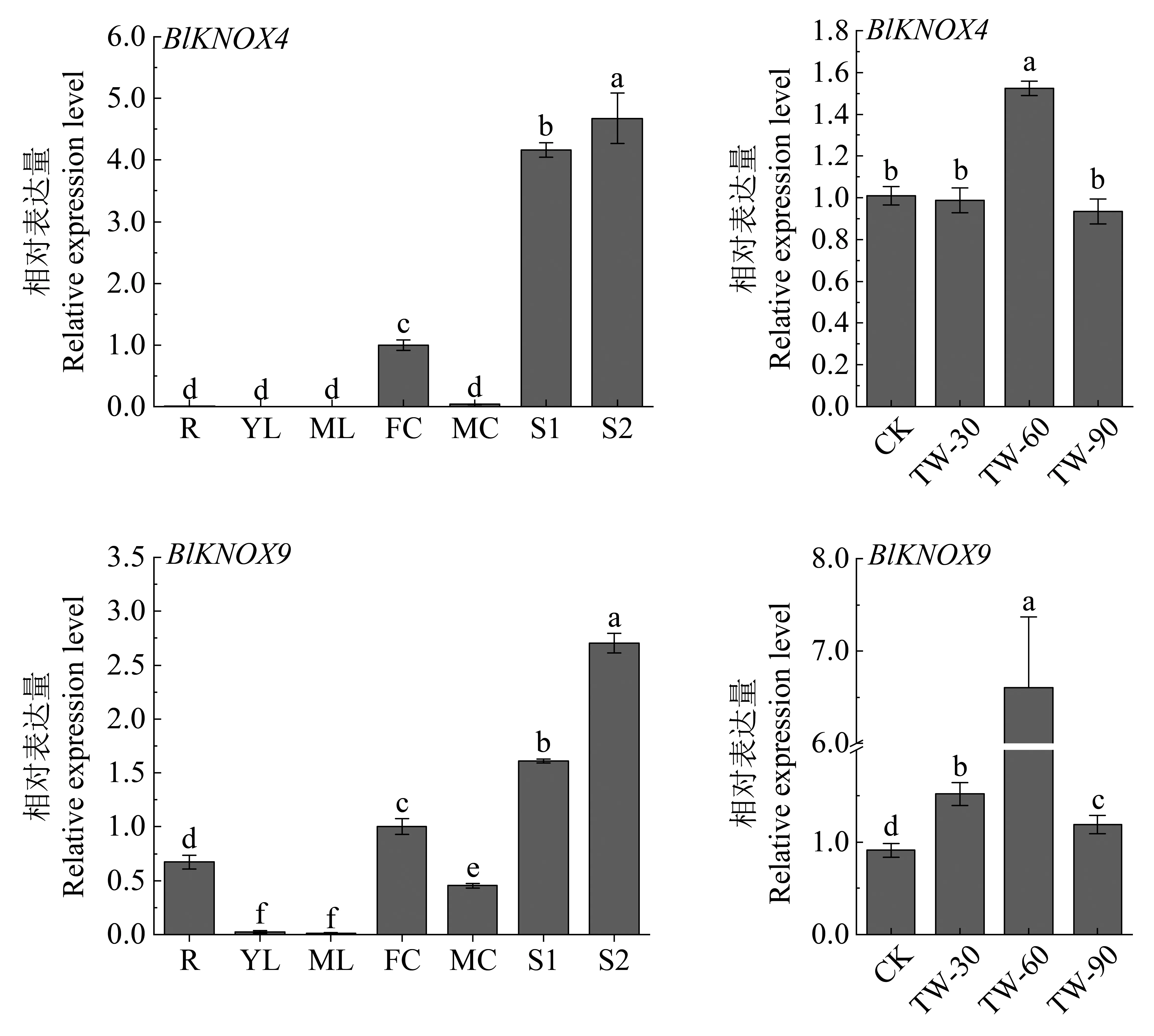
图6 BlKNOX4和BlKNOX9在不同组织、器官和应拉木处理过程中的表达分析Fig. 6 Expression analysis of BlKNOX4and BlKNOX9 in different tissues, organs and treatment process of tension wood标有不同小写字母的样品表示在0.05水平差异显著。R:根; YL: 嫩叶; ML: 成熟叶; FF: 雌花序; MF: 雄花序; S1: 未木质化茎段; S2: 木质化茎段; TW: 应拉木; CK: 对照。Different small letters above columns indicate significant difference at 0. 05 levels. R: Root; YL: Young leaf; ML: Mature leaf; FF: Female inflorescence; MF: Male inflorescence; S1: Non-lignified stem; S2: Lignified stem; TW: Tension wood. CK:Control.
BLH和KNOX、OFP蛋白之间的互作是调控植物生长发育的重要途径 (Bhattetal., 2004 , Coleetal., 2006 , Hackbuschetal., 2005)。例如,BLH3与OFP1互作调控拟南芥营养期到生殖器的转化 (Zhangetal., 2016) 。在拟南芥茎尖分生组织中,PNF/PNY能够和KNOX家族的STM以及BP相互作用,形成BLH-KNOX二聚体 (Smithetal., 2003)。BLH1和KNAT3互作形成二聚体,通过直接靶向ABI3来调节ABA介导的发育过程 (Kimetal., 2013)。此外,在拟南芥、棉花等多种植物中的研究表明,部分BLH和KNOX蛋白的互作是调控植物次生壁发育重要机制。例如,BLH6与KNAT7互作,能抑制REV(REVOLUTA) 基因的表达进而抑制拟南芥次生壁的形成 (Liuetal., 2014)。Ma等(2019)研究发现,棉花BLH家族成员(GhBLH1、GhBLH5和GhBLH6)可与GhKNAT7、GhOFP1、GhOFP4、GhMYB75等转录因子形成复合物,与木质素(GhCAD5、GhCOMT1等)和纤维素生物合成基因(GhCESA4/7/8)的启动子结合,形成次生细胞壁的调控网络。笔者也发现,BlBLH1与2个BlKNOX蛋白存在互作关系,且在茎段木质化和应拉木形成过程中协同表达来调控木质素、纤维素等的形成,这为深入研究BlBLH1在木材细胞壁成分合成及结构功能提供了重要线索。通过酵母文库杂交还筛选到了F-box、WD40、PAL、XTH等与BlBLH1互作的蛋白,但这些蛋白与BLH的互作关系及其调控功能却均未见报道。
4 结论
本研究在光皮桦中分离获得BlBLH1基因,其编码609个氨基酸,具有典型的HD、BELL和SKY结构域,在进化上虽与BLH1聚为一类,但在表达上有差异。BlBLH1在雌花、雄花中优势表达,并与BlKNOX4、BlKNOX9存在蛋白互作,在木质化茎及应拉木形成过程中有相似表达趋势。
