侵染贵州火龙果的蟹爪兰X病毒和火龙果X病毒基因组序列差异和遗传变异
2022-06-06郑乾明王小柯马玉华
郑乾明,王小柯,马玉华
(贵州省农业科学院果树科学研究所,贵阳 550006)
【研究意义】火龙果(Hylocereusspp.)是仙人掌科(Cactaceae)量天尺属(Hylocereus)多年生果树,近年来在我国台湾、广西、广东、贵州、云南和海南等省种植,具有较好的经济效益和生态效益。火龙果主要受甲型线状病毒科(Alphaflexiviridae)马铃薯X病毒属(Potexvirus)病毒[1-5]及近期发现的花椰菜花叶病毒科(Caulimoviridae)杆状DNA病毒属(Badnavirus)病毒侵染[6]。分离和分析病毒基因组全长序列,有助于了解其遗传变异和多样性,为病毒检测和防控奠定理论基础。【前人研究进展】侵染火龙果的马铃薯X病毒属病毒主要包括仙人掌X病毒(CactusvirusX,CVX)[1-2,4-5,7-10]、蟹爪兰X病毒(ZygocactusvirusX,ZyVX)[3-5,11]、仙人指X病毒(SchlumbergeravirusX,SchVX)[3-5, 12]和火龙果X病毒(PitayavirusX,PiVX)[5]。CVX在我国台湾[1-2]、贵州[4-5]和海南[7,10]等省以及韩国[8]和美国[9]均有报道,但对ZyVX和PiVX侵染火龙果的研究较少。ZyVX最早于2004年在蟹爪兰(Zygocactusspp.)中分离获得B1分离物基因组全长序列(GenBank登录号:AY366208)[13],此后于2008年在巴西火龙果中检测到[3],后来在台湾火龙果中报道P39分离物基因组全长(GenBank登录号:JF930326),在贵州火龙果中报道GZ分离物基因组近全长序列[11]。ZyVX为单分体正义单链RNA病毒,含有5个开放阅读框(Open Reading Frame,ORF)[11],ORF1编码依赖RNA的RNA聚合酶(RNA-dependent RNA polymerase,RdRP),ORF2-4分别编码TGB1-3蛋白(Trige gene block 1-3),ORF5编码外壳蛋白(Coat protein,CP)。PiVX于2011年在我国台湾省首次报道P37分离物基因组序列(GenBank登录号:JF930327),此后仅在仙人掌科锦绣玉属(Parodia)金晃(P.leninghausii)报道NI分离物编码CP的ORF序列(GenBank登录号:KY581589)[14]。近期,在贵州省61份火龙果样品中检测PiVX发生率为96.72%[5]。由此可见,ZyVX和PiVX均普遍侵染火龙果。【本研究切入点】目前报道的ZyVX和PiVX基因组全长序列较少,无法开展病毒基因组序列差异和和系统进化分析,不清楚其遗传变异和多样性。因此,采用RNA-seq对病毒感染火龙果样品进行高通量测序和分析,组装获得ZyVX和PiVX基因组序列。【拟解决的关键问题】获得ZyVX和PiVX基因组序列全长,开展序列变异、基因重组和系统进化分析。
1 材料与方法
1.1 材料
样品于2017年8月采集自贵州省罗甸县和镇宁县火龙果果园。于火龙果盛果期采集茎表面表现黄化褪绿、斑驳和畸形等疑似病毒病症状的样品,置于冰盒带回实验室。用无菌水清洗后晾干,切取茎组织在液氮中速冻。所有样品均在-80 ℃超低温冰箱保存。
1.2 RT-PCR检测
检测ZyVX和PiVX的引物,总RNA提取、反转录PCR、RT-PCR扩增和电泳等步骤参见文献[4]和[5]中的方法。
1.3 RNA-seq分析
筛选获得6份含有ZyVX和PiVX的样品,使用去核糖体RNA试剂Ribo-ZeroTMrRNA Removal Kit(Epicentre公司)去除样品中的寄主核糖体RNA。剩余的总RNA片段化,然后合成第一链和第二链cDNA。经产物末端修复,连接测序接头,片段大小选择和PCR扩增等步骤完成测序文库构建。利用Illumina HiSeq 4000进行双末端各150 bp测序,每份样品测序数据量约15~20 Gb。测序获得的raw read进行过滤,每份样品产生的clean read利用Trinity软件单独组装[15],获得的contig序列使用Blast程序在NCBI非冗余核酸和蛋白质数据库检索和注释。
1.4 序列分析
ORF预测使用ORF Finder程序(https://www.ncbi.nlm.nih.gov/orffinder/),核苷酸序列翻译成氨基酸使用在线ExPASy程序(https://web.expasy.org/translate/),核苷酸及氨基酸的序列一致性计算使用Lasergene软件(DNASTAR,USA)中的MegAlign程序。
1.5 重组和系统进化分析
基因重组分析使用RDP4程序,首先利用ClustalW进行序列的多重比对,然后使用RDP、GENBCONV、BootScan、Maxchi、Chimaera和SiScan等6种算法共同分析。在有4种以上算法支持的情况下,同时P<10-6时,认为存在明显的重组。
从NCBI下载CVX(GenBank登录号:AF308158),CVX-NTU(GenBank登录号:JF937699),SchVX-K11(GenBank登录号:AY366207),OpVX(GenBank登录号:AY366209)基因组序列,连同ZyVX和PiVX各分离物序列,利用ClustalW程序进行多重序列比对。在MEGA 7.0软件中利用邻接法构建系统进化树,Bootstrap检验设置为1000次重复。
2 结果与分析
2.1 ZyVX和PiVX贵州分离物的相关序列
对6份样品产生的clean read分别组装,产生的contig序列在NCBI网站中利用BLAST程序检索和注释(表1),共获得ZyVX相关贵州分离物序列4条,分别命名为ZyVX-GZ-RF、ZyVX-GZ-ZNF、ZyVX-GZ-ZNS和ZyVX-GZ-ZYP,去除poly A后,长度为6570~6600 bp,在其基因组5′末端缺失24~34 bp,3′末端缺失12~20 bp,覆盖参考基因组ZyVX-P39分离物基因组序列的99.2%~99.6%。共获得PiVX相关贵州分离物序列3条,分别命名为PiVX-GZ-RF、PiVX-GZ-ZNS和PiVX-GZ-ZYP,去除poly A后,长度分别为6649~6657 bp,在其基因组5′末端缺少14~22 bp,3′末端均完整,覆盖参考基因组PiVX-P37分离物序列的99.3%~99.8%。

表1 基于RNA-seq获得的ZyVX和PiVX贵州分离物基因组序列
2.2 基因组序列差异
将获得的ZyVX和PiVX贵州分离物与基因序列全长的分离物ZyVX-P39、ZyVX-B1、ZyVX-GZ[11]、PiVX-P37以及侵染火龙果的马铃薯X病毒属病毒CVX、CVX-NTU和SchVX-K11共同计算序列一致性结果(表2)表明,4条ZyVX贵州分离物之间的基因组序列一致性为96.7%~99.5%,与ZyVX-P39及ZyVX-B1一致性为95.2%~96.9%,与ZyVX-GZ一致性为91.4%~92.2%,与CVX、CVX-NTU和SchVX一致性为68.0%~73.6%。3条PiVX贵州分离物之间的基因组序列一致性为98.2%~98.5%,与PiVX-P37一致性为98.0%~99.1%。PiVX贵州分离物和ZyVX贵州分离物之间的序列一致性为68.6%~69.1%,与CVX、CVX-NTU及SchVX-K11序列一致性为68.9%~71.1%。
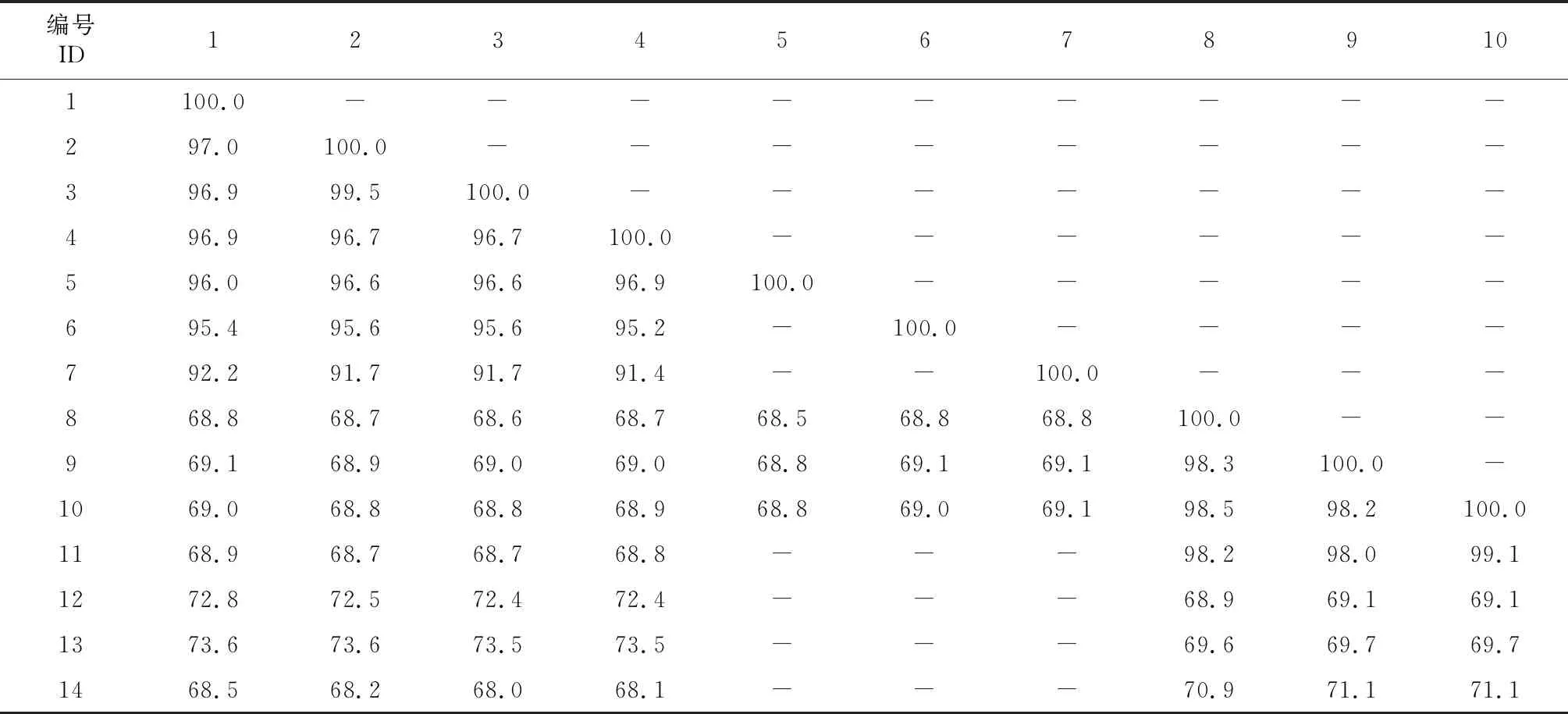
表2 ZyVX和PiVX贵州分离物及其他病毒的基因组序列一致性
2.3 ORF编码的氨基酸序列差异
从表3看出,ZyVX贵州分离物之间RdRP、TGB1、TGB2、TGB3和CP编码的氨基酸序列一致性分别为98.5%~99.8%、97.4%~98.7%、96.5%~100.0%、93.8%~100.0%和97.3%~100.0%。ZyVX贵州分离物与P39、GZ分离物RdRP编码的氨基酸序列一致性分别为98.1%~98.4%和97.0%~97.1%,低于ZyVX贵州分离物之间的序列一致性;与B1分离物RdRP和TGB1编码的氨基酸序列一致性较低,分别为97.2%~97.5%和93.9%~95.2%。PiVX贵州分离物之间RdRP、TGB1、TGB2、TGB3和CP编码的氨基酸序列一致性分别为98.5%~98.7%、99.6%~100.0%、98.2%~99.1%、100.0%和97.8%~99.1%,其与P37分离物各ORF编码的氨基酸序列一致性分别为98.2%~99.3%、99.6%~100.0%、98.2%~100.0%、100.0%和97.3%~99.6%。
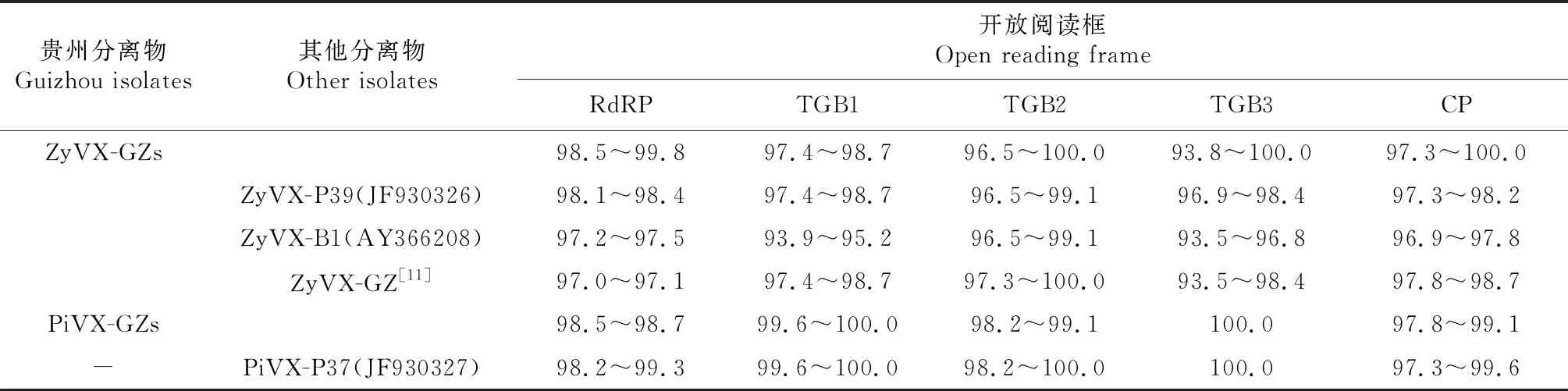
表3 ZyVX和PiVX贵州分离物ORF编码的氨基酸序列一致性
2.4 重组与系统进化分析
对ZyVX贵州分离物、P39、B1和GZ,PiVX贵州分离物和P37分别进行基因重组分析,均未发现明显的重组事件。将上述分离物基因组序列与CVX、CVX-NTU、SchVX-K11和OpVX共同进行系统进化分析结果(图1)表明,ZyVXs、PiVXs均与CVX、CVX-NTU、SchVX-K11和OpVX等明显分开;4条ZyVX贵州分离物与P39亲缘关系最近,然后与B1和GZ聚为一类,未见明显的遗传分化。3条PiVX贵州分离物与P37聚为一类,也未见明显的遗传分化。
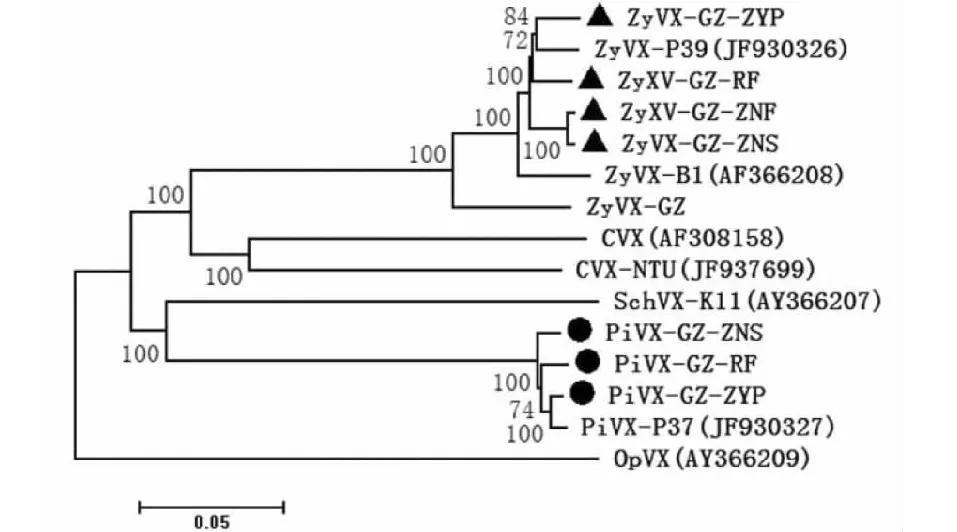
图1 基于基因组序列的ZyVX和PiVX贵州分离物系统进化树
3 讨 论
基于Illumina平台的RNA-seq广泛用于侵染园艺作物的病毒挖掘和鉴定,其具有读长较短、准确率高的特点[16]。测序文库的构建方式对后续结果至关重要,目前普遍采取富集mRNA、提取病毒双链RNA、提取小RNA和去除寄主核糖体RNA等方式构建测序文库[17-21]。其中,通过提取总RNA并去除寄主核糖体RNA后构建测序文库,近年来成功应用于油桃[22]、甜樱桃[23]、苹果[24]和茶树[25]等多年生木本园艺作物中的病毒鉴定。本研究对含有ZyVX和PiVX的火龙果样品进行RNA-seq分析,分别获得4条ZyVX和3条PiVX贵州分离物基因组近全长序列。相比于使用RT-PCR分段扩增的策略,使用RNA-seq分析能够快速获得大量分离物基因组近全长序列。尽管在其基因组5′和3′末端缺失若干核苷酸,但仍覆盖参考基因组全长的99.3%~99.8%。这些分离物基因组近全长序列均包含5个完整的ORF序列,能够开展ZyVX和PiVX群体的遗传变异分析。
本研究获得的4条ZyVX分离物之间基因组序列一致性为96.7%~99.5%,其与B1和P39分离物的序列一致性明显高于与GZ分离物。B1分离物于2004年在蟹爪兰(Zygocactusspp.)中分离获得基因组全长[13],蟹爪兰与火龙果属仙人掌科不同属植物。P39分离物于2014年在我国台湾省火龙果植株中分离获得基因组全长。GZ分离物于2017年利用RT-PCR分段扩增的策略,从贵州火龙果获得基因组近全长序列[11]。系统进化分析也表明本研究获得的ZyVX分离物与B1和P39分离物的亲缘关系较GZ分离物更近。研究表明,基因突变和重组是RNA病毒产生变异,从而适应寄主和环境的主要途径[26]。上述分离物均未发生明显的遗传重组,其基因组变异均为单核苷酸变异。这说明自2004 年首次报道ZyVX以来,其基因组序列在同科不同属种寄主中未发生明显的单核苷酸变异,但在近几年来发生一定程度的基因组单核苷酸变异。这可能与近年来火龙果大规模种植,同时获得的病毒群体数量较多有关。不过,从ORF编码的氨基酸序列一致性来看,其氨基酸水平上未出现明显的变异。
2015年来多次报道我国东南沿海地区进境火龙果种苗中检测到携带PiVX。最近利用RT-PCR检测多份贵州火龙果样品,PiVX检出率为96.7%[5]。PiVX普遍侵染火龙果,目前尚不了解其基因组序列的遗传变异。本研究获得的3条PiVX分离物基因组与首次报道的P37分离物间未发生明显的重组和遗传分化,变异均为单核苷酸变异。3条PiVX分离物与P37分离物基因组序列一致性为98.0%~99.1%,ORF编码的氨基酸序列一致性为97.3%~100%,说明目前PiVX分离物群体的遗传变异较小。
4 结 论
本研究分离4条ZyVX和3条PiVX贵州分离物基因组近全长序列,丰富了ZyVX和PiVX的群体数量。ZyVX和PiVX贵州分离物之间的基因组序列一致性分别为96.7%~99.5%和98.2%~98.5%。其变异均为单核苷酸变异,未发现基因重组和明显的遗传分化。因此,ZyVX和PiVX群体的基因组序列变异较小,有利于快速建立病毒检测体系,为研究病毒的流行、传播途径和防控奠定理论基础。
