水稻白叶枯病菌FabG同源蛋白生物信息学分析
2022-06-06马建荣余永红刘戈飞沈晓萌
马建荣,余永红,宋 卉,刘戈飞,沈晓萌
(广东食品药品职业学院,广州 510520)
【研究意义】黄单胞菌水稻致病变种(Xanthomonasoryzeapv.oryzea,Xoo)是水稻白叶枯病的病原菌,又称作水稻白叶枯病菌,属于变形菌纲,γ-亚纲,黄单胞菌科,黄单胞菌属[1]。Xoo可通过叶片或者叶鞘上的伤口或水孔等自然开口侵染水稻[2-3]。 病菌接触和吸附宿主细胞之后,通过细胞间隙进入维管束,在24~72 h快速繁殖,并分泌大量的胞外多糖堵塞水孔,水稻也因此而缺水枯萎[4]。Xoo属于排名前十的致病病原性细菌,在全世界范围内引起重大经济损失,亟需开发新的防控策略与方法[5]。 而随着分子生物学和生物技术的发展,Xoo全基因组已经测序完成,为控制水稻白叶枯病提供了新的思路[6]。【前人研究进展】Xoo在致病过程中产生众多的致病因子,包括具有抗氧化性的菌黄素[7-8]、胞外多糖(EPS)、脂多糖(LPS)、黏附素、生物被膜等,而这些致病因子的产生受到双组分调控系统、群体信号分子、转录调控因子GamR和Clp、III型分泌系统等多种方式调控。其中由DSF信号分子介导的群体感应系统通过多级分层调控网络,广泛调节致病相关基因表达,提高细胞解毒和抗氧化能力[9]。 野油菜黄单胞菌中DSF信号分子为不饱和脂肪酸结构,其合成前体来源于脂肪酸合成中间代谢产物[8,10-11]。细菌细胞中脂肪酸不仅是细胞膜的重要组成部分,还在细菌表面识别、宿主吸附、能量代谢、生物活性物质合成等多方面发挥作用[12-13]。 而细菌采用Ⅱ型脂肪酸合成(FAS II)系统从头合成脂肪酸,包括聚合、还原、脱水和再还原4个反应步骤,每步反应都由独立的酶催化完成,最终合成长链脂肪酸[14]。在FAS II中,3-酮脂酰ACP还原酶(FabG)催化第一步还原反应,为细菌脂肪酸合成的关键酶[15-16]。不同细菌中FabG广泛存在且相对保守,被认为是重要的抗菌药物筛选靶点[17]。 随着研究的深入,不同细菌中多个FabG同源蛋白被陆续报道,这些蛋白属于短链脱氢酶/还原酶(SDR)超家族,也具有类似Ser-Tyr-Lys的催化活性中心,但具有不同的生物学功能[8,10,18]。如野油菜黄单胞菌(Xanthomonascampestrispv.campestris)中FabG1是脂肪酸合成关键酶,对不同链长底物都具有较高催化活性;而FabG2仅对中长链底物3-酮脂酰ACP具有催化活性,特异性调节DSF信号分子合成;FabG3对不同链长底物也具有较低的催化活性,但特异性参与细胞色素菌黄素合成[8,10,18]。【本研究切入点】目前关于Xoo的脂肪酸合成机制报道较少,仅本实验前期研究发现其3-酮脂酰ACP聚合酶III (FabH)对多种底物都具有催化活性[19]。对Xoo中3-酮脂酰ACP还原酶的相关研究还未见报道。【拟解决的关键问题】本研究利用生物信息学方法,对Xoo基因组中6个大肠杆菌FabG(EcFabG)同源蛋白进行系统分析,重点包括物理化学性质、跨膜区与信号肽、亲疏水性等,并预测了这6个FabG同源蛋白的生物学功能,为深入研究Xoo的脂肪酸合成途径以及3-酮脂酰ACP还原酶多样性等方面提供了前期理论依据。
1 材料与方法
1.1 基因及蛋白信息的来源
在NCBI网站,在Xoo基因组中通过BLAST检索得到PXO_03085、PXO_00989、PXO_03629、PXO_02411、PXO_02878、PXO_02709共6个基因与EcFabG基因具有同源性,分别命名为XoofabG1~XoofabG6,其编码蛋白分别命名为XooFabG1~XooFabG6。
1.2 生物信息学分析
使用Clustal Omega以及ESPript将XooFabG1~XooFabG6与 EcFabG的氨基酸序列进行同源比对。使用Protparam tool分析XooFabG1~XooFabG6与EcFabG的基本理化性质。使用Predict protein对XooFabG1~XooFabG6与EcFabG进行二级结构的预测。使用Swiss model对XooFabG1~XooFabG6与EcFabG的三级结构进行预测[20]。使用Protscale分析XooFabG1~XooFabG6与EcFabG的亲水性。使用NetPhos Serverv 3.1预测分析XooFabG1~XooFabG6与EcFabG的磷酸化位点。使用Motif scan分析XooFabG1~XooFabG6与 EcFabG的翻译后修饰位点。使用TMHMM分析XooFabG1~XooFabG6与 EcFabG的跨膜结构域。 使用SignalP 5. 0 Server预测XooFabG1~XooFabG6与 EcFabG的信号肽。 使用String对XooFabG1~XooFabG6与 EcFabG进行蛋白质相互作用分析[21]。
2 结果与分析
2.1 Xoo基因组中编码FabG同源蛋白基因的基本信息
在Xoo中经过同源性检索得到PXO_03085、PXO_00989、PXO_03629、PXO_02411、PXO_02878、PXO_02709共6个基因编码蛋白与EcFabG具有同源性,将6个基因依次命名为XoofabG1~XoofabG6,所编码蛋白依次命名为XooFabG1~XooFabG6。根据NCBI及KEGG网站显示,6个基因长度在735~777 bp,6个蛋白质长度在244~258 aa(表1),基因和蛋白长度均无明显差异。
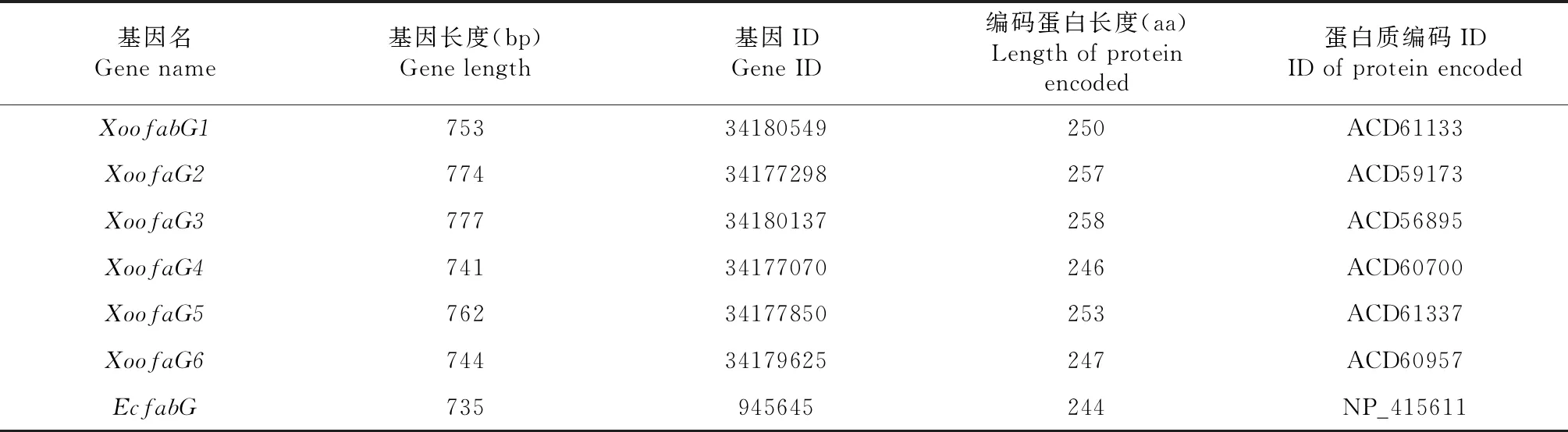
表1 Xoo基因组中FabG同源蛋白编码基因基本信息
如图1所示,6个基因分布在Xoo基因组中不同的位置:XoofabG1、XoofabG2处于未知功能的基因簇中(图1-a,图1-b)。XoofabG3显示为脂肪酸代谢基因,基因上游和下游为维生素和辅因子代谢基因,推测与生物素合成相关(图1-c)。XoofabG4显示为碳水化合物代谢基因,处于未知功能的基因簇中(图1-d)。XoofabG5显示为脂肪酸代谢基因,处于未知功能的基因簇中(图1-e)。XoofabG6(图1-f)和EcfabG(图1-g)标注为脂肪酸代谢基因,位于保守的脂肪酸代谢基因簇中。

a~f: XoofabG1~XoofabG6所处的基因簇; g: EcfabG所处的基因簇
2.2 蛋白序列同源比对
如图2所示,XooFabG1~XooFabG6与EcFabG的序列一致性分别为29. 06%、29. 44%、30. 54%、39. 26%、42. 26%和67. 21%。 EcFabG属于短链脱氢酶(SDR)家族,氨基酸残基Ser138-Tyr151-Lys155(图2中箭头所示)形成催化活性中心,活性位点入口处的氨基酸残基Arg129和Arg172(图2中三角形所示)对底物结合有重要作用[22]。只有XooFabG1、XooFabG2、XooFabG5和XooFabG6具有相同的Ser-Tyr-Lys催化活性中心,但XooFabG1、XooFabG2不具备活性位点入口处的精氨酸残基。蛋白序列比对结果发现XooFabG5和XooFabG6与EcFabG的序列一致性较高,且兼具活性位点入口处的氨基酸残基和Ser-Tyr-Lys保守活性位点,初步表明XooFabG5和XooFabG6可能具有与EcFabG类似的催化活性,而XooFabG3和XooFabG4可能不具有该活性;XooFabG1与XooFabG2可能具有酮基还原酶活性,但催化底物与FabG不同。另外,XooFabG1~XooFabG5含有半胱氨酸,有可能形成二硫键。
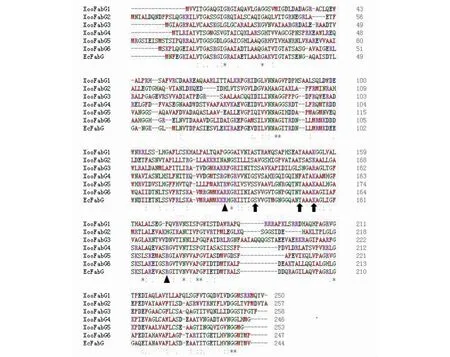
图2 XooFabG1~XooFabG6与EcFabG氨基酸序列比对
2.3 XooFabG基本理化性质
如表2所示,6个基因的蛋白之间长度差异不大,但是其他理化性质差别较大。不稳定系数大于等于40,系统判定为不稳定蛋白[23],因此XooFabG1为不稳定蛋白,XooFabG2~XooFabG6为稳定蛋白。XooFabG1和XooFabG3的pI与EcFabG差别较大。XooFabG1在稳定性和亲水性方面与EcFabG差异较大。XooFabG6与EcFabG理化性质相近。

表2 Xoo基因组中FabG同源蛋白基本理化性质
2.4 XooFabG二级结构及三级结构预测
如表3所示,各蛋白的二级结构组成百分比没有显著性差异,α-螺旋约占45%,β-折叠约占12%,其余为转角结构。使用Swiss model对XooFabG1~ XooFabG6与EcFabG蛋白三级结构[]进行预测并构建模型。 如图3所示,XooFabG1~ XooFabG6均为同源四聚体蛋白,三级结构由α螺旋、无规则卷曲和β折叠组成,其中α螺旋是主要的结构元件,与蛋白质二级结构中各元件所占比例相符。 所有蛋白的基本框架一致,α螺旋和β折叠形成相似蛋白质三级结构。 这些蛋白很可能属于同一个大的蛋白家族,因此具有相同的蛋白质框架,但因为在活性位点附近氨基酸的不同或结构上的微小差异,而具有不同的活性。
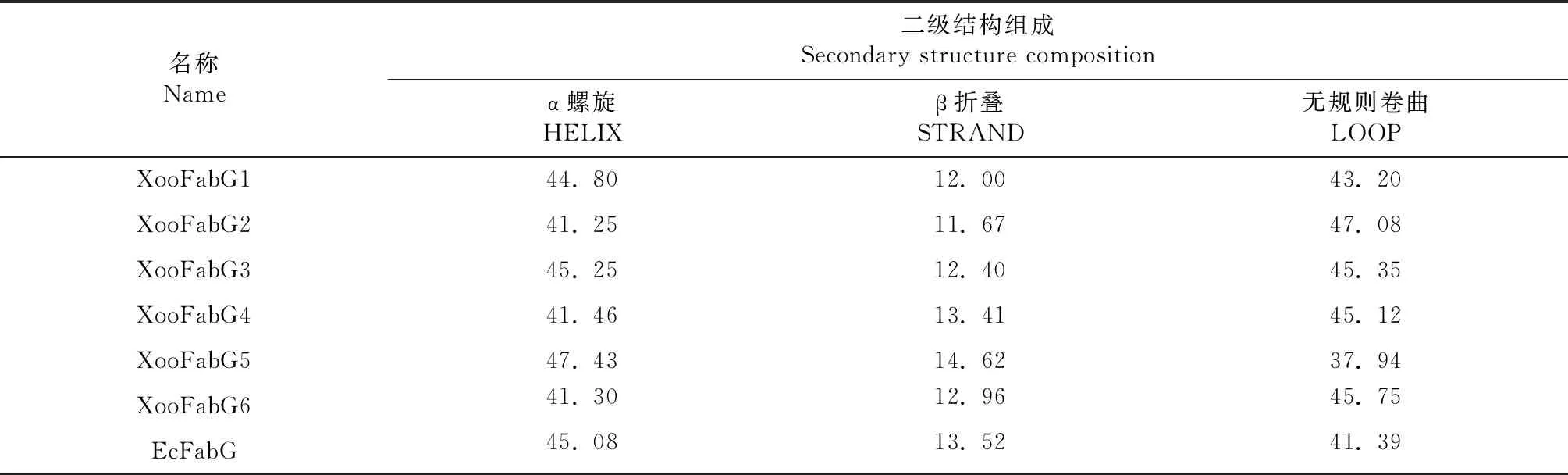
表3 Xoo基因组中FabG同源蛋白二级结构预测结果
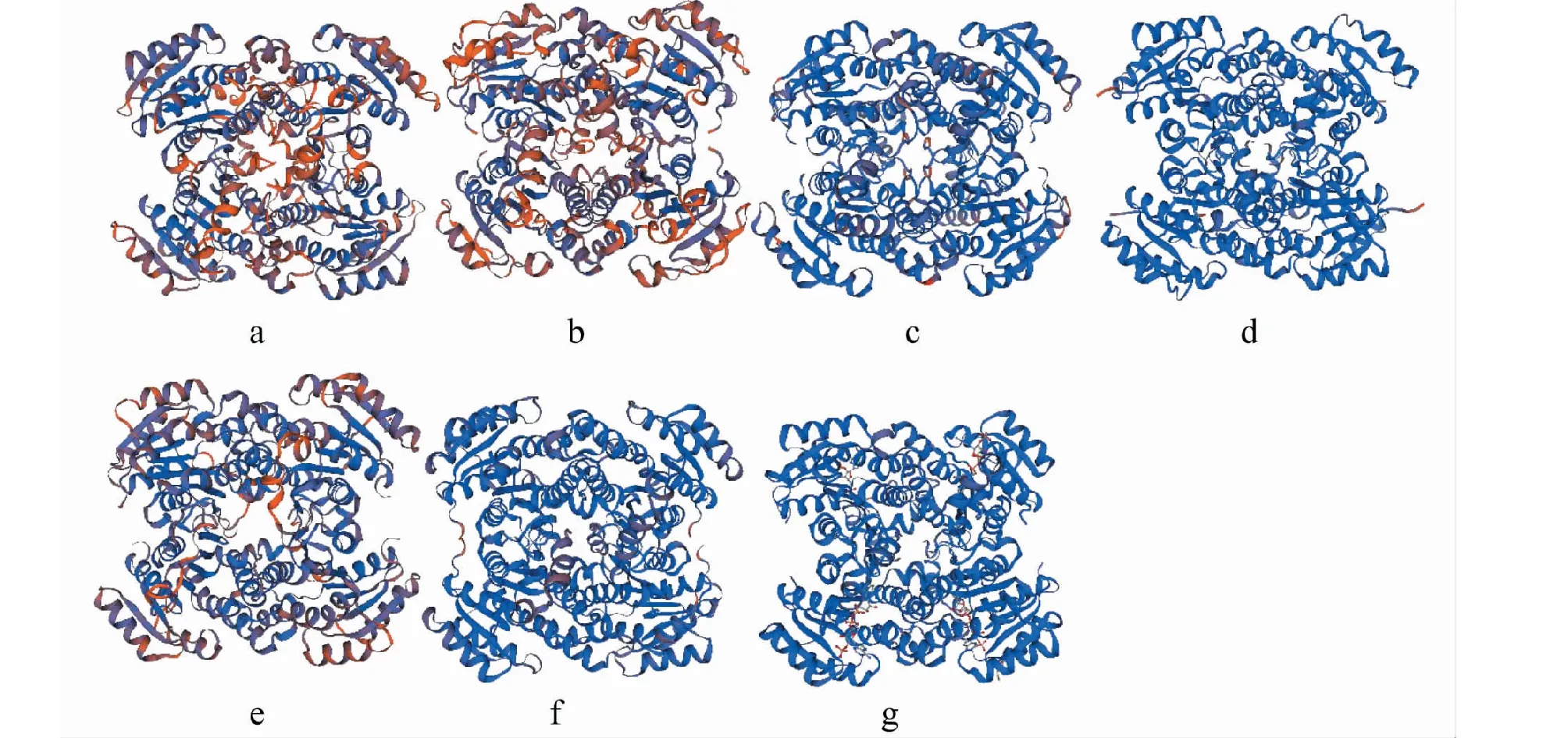
a~f:XooFabG1~XooFabG6的三级结构预测结果; g: EcFabG的三级结构预测结果
2.5 XooFabG亲水性分析
氨基酸的亲水性与蛋白质的构象相关,疏水性的氨基酸残基倾向位于蛋白构象的疏水内核,而亲水性氨基酸残基则多位于蛋白的亲水表面,因此亲水性上的差异,一定程度上影响各自的蛋白构象,进而造成功能上的差异。如图4所示,图中标出绝对分值大于2的强亲水疏水位置,EcFabG有2个强疏水区域及一个强亲水区域(图4-g)。与之相比,XooFabG1仅有1个强的疏水区域和1个强的亲水区域与EcFabG相对应,但还有1个强的亲水集团与EcFabG差别较大(图4-a)。XooFabG2仅有1个强的疏水区域与EcFabG对应,没有相应的亲水区域(图4-b)。XooFab3仅有1个强的亲水区域与EcFabG对应,没有相应的疏水区域(图4-c)。 XooFabG4仅有1个疏水区域和1个亲水区域与EcfabG相对应,但还有1个强的亲水区域与EcFabG差异较大(图4-d)。XooFab5仅有2个强的疏水区域与EcfabG对应,没有相应的亲水区域(图4-e)。XooFab6有2个强疏水区域及1个强的亲水区域与EcfabG相对应(图4-f)。 因此推测XooFab6具有与EcfabG相近的催化活性。
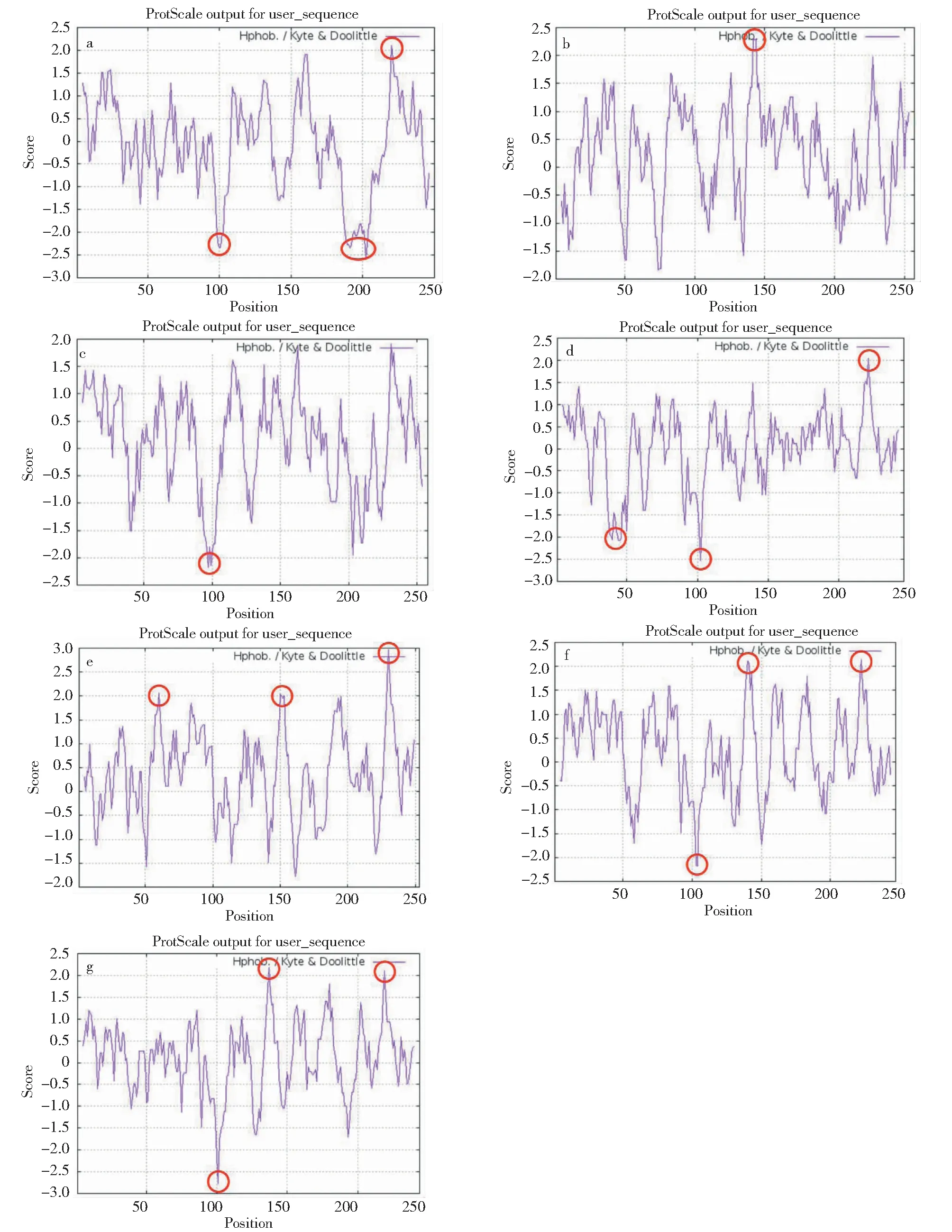
a~f: XooFabG1~XooFabG6亲水性分析; g: EcFabG亲水性分析
2.6 XooFabG蛋白磷酸化位点及翻译后修饰位点分析
如表4所示,只有XooFabG6与EcFabG在相近区域具有短链脱氢酶/还原酶家族特征与KR domain,并且在相近区域具有N-糖基化位点。虽然EcFabG和XooFabG6在N-糖基化位点、短链脱氢酶/还原酶家族特征和3-酮酰-ACP还原酶方面有诸多相似性,但在磷酸化位点方面有明显区别,EcFabG只有酪氨酸激酶II磷酸化位点(68-71),而没有蛋白激酶C磷酸化位点,XooFabG6只有蛋白激酶C磷酸化位点(120-122,229-231),而没有酪氨酸激酶II磷酸化位点。

表4 XooFabG1~XooFabG6与EcFabG磷酸化位点及翻译后修饰位点分析
2.7 XooFabG跨膜区域与信号肽预测
蛋白质的跨膜区域主要是指膜内在蛋白和细胞膜的膜脂相结合的部位,通过TMHMM分析XooFabG1~XooFabG6与EcFabG的跨膜区域,所有蛋白的跨膜螺旋数量为0(图5),说明这些蛋白均不是跨膜蛋白质,合成后在Xoo体内发挥作用。
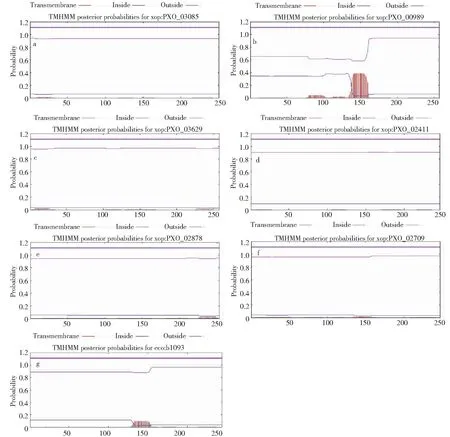
a~f: XooFabG1~XooFabG6跨膜结构域分析结果; g: EcFabG跨膜结构域分析结果
SignalP是一个信号肽及其剪切位点的预测工具,它采用一个神经网络来区分信号肽与非信号肽,另一个神经网络来识别剪切位点,其分析结果由以下数值组成:CS值是信号肽酶切位点分值,每个氨基酸都会有一个CS值,且处于剪切位点处的氨基酸CS值最高;Sec/SPI、TAT、Sec/SPII值是信号肽分值,信号肽区域的氨基酸该值较高;OTHER值表示以上4种情况以外的可能性。系统会综合分析各分值,从而得出可能性最大的一种推测。如图6所示,XooFabG1、XooFabG2、XooFabG5、XooFabG6与EcFabG一致,没有信号肽,为非分泌性蛋白,在菌体内发挥生理作用;而XooFabG3和 XooFabG4有信号肽的可能性较大。

a~f: XooFabG1~XooFabG6信号肽分析; g:EcFabG信号肽分析
2.8 XooFabG相互作用预测
在蛋白质相互作用预测图中(图7),XooFabG1、XooFabG2和XooFabG4的相互作用蛋白皆为未知蛋白,信息量较少,参考意义不大。XooFabG3、XooFabG5和XooFabG6相互作用的蛋白有很大的共性:都与FabH、FabD、FabG、FabA、FabZ以及未知蛋白PXO_02711、PXO_03137、PXO_02883、PXO_00990有相互作用关系。区别在于XooFabG5还与PXO_02882有相互作用关系,XooFabG6还与acpP和PXO_3455有相互作用关系。同时,XoofabG5与PXO_02883,PXO_02882为相邻基因;XoofabG6与fabD,PXO_02711,fabH,acpP位于同一基因簇,为相邻基因。XooFabG6与fabD存在基因融合现象,推测可能存在与同一读码框中。

a~f: XooFabG1~XooFabG6互作预测; g: EcFabG互作预测
EcFabG主要与其他脂肪酸合成途径中的关键酶相互作用,如FabF,FabI,FabB,FabH,FabD,FabZ,FabA和AcpP,在Eco中,EcFabG负责脂肪酸的从头合成。在Eco基因组中,EcofabG与fabD,fabH,fabF,acpP,plsX,rpmF的编码基因位于同一基因簇中,为毗邻基因,共同构成了脂肪酸合成基因簇,其中EcfabG与fabD存在基因融合的现象,推测因为两者在基因簇上位置相近,因此位于同一个开放阅读框中。而fabA,fabB,fabI和fabZ编码蛋白虽然参与脂肪酸的合成,但是它们位于离脂肪酸合成基因簇较远的位置。
综上所述,虽然XooFabG3、XooFabG5和XooFabG6在相互作用网络方面有很大共性,但是与EcFabG相比,只有XooFabG6在基因分布方面具有相似性。
3 讨 论
生物信息学分析在蛋白质理化性质、生物活性、相互作用等方面具有显著优势,能提高实验的成功率[25]。本研究通过生物信息学分析发现,XoofabG6位于保守的脂肪酸合成基因簇中,而该基因簇与已报道的多种细菌脂肪酸合成基因簇高度同源[18,26]。通过序列比对发现,与EcFabG类似,XooFabG6也具有Ser-Tyr-Lys催化活性中心且兼具活性位点入口处的精氨酸残基[24]。在亲水性分析方面,XooFabG6有2个强疏水区域及1个亲水区域与EcFabG相对应。同时,XooFabG6与EcFabG在类似位置存在短链脱氢酶/还原酶家族特征、KR domain以及N-糖基化位点。在蛋白质相互作用的分析中,XooFabG6与EcFabG类似,主要与脂肪酸合成途径中的关键酶相互作用。同时,XooFabG6与野油菜黄单胞菌FabG1的氨基酸序列一致性超过90%,因此推测XooFabG6具有3-酮脂酰ACP还原酶活性,催化脂肪酸合成循环的第一还原反应[10]。同时本研究发现,XooFabG1是一个不稳定蛋白,与EcFabG相比,它具有Ser-Tyr-Lys催化活性中心,但缺少活性位点入口处的精氨酸残基,在类似位置符合KR domain和短链脱氢酶/还原酶家族特征。 推测XooFabG1属于短链脱氢酶家族,具有3-酮脂酰ACP还原酶活性,但因为其不稳定性,很可能与细菌应激状态下的反应有关。XooFabG3在Xoo基因组中被标注为为脂肪酸代谢基因,亦与脂肪酸合成途径中的相关蛋白相互作用,具备Ser-Tyr-Lys催化活性中心,但不具备活性位点入口处的精氨酸残基。因此推测XooFabG3为短链脱氢酶,但与EcFabG相比,功能相差较大,根据其上下游基因功能推测其与辅因子和维生素的代谢相关。
XooFabG5具有Ser-Tyr-Lys催化活性中心且兼具活性位点入口处的精氨酸残基,具有KR domain,但其位置与EcFabG相比差异较大,因此推测其具有3-酮脂酰ACP还原酶活性,但因亲水性差异较大,其底物可能与EcFabG不同,类似于野油菜黄单胞菌中FabG2仅对长链底物有选择性[10]。
本研究分析发现,在Xoo中,XooFabG1、XooFabG5和XooFabG6都具有3-酮脂酰ACP还原酶活性,故推测XooFabG6催化脂肪酸合成循环的第一还原反应,是脂肪酸合成中的关键酶;XooFabG1可能与细菌的应激反应有关,在特定条件发挥作用;XooFabG5底物可能与EcFabG不同。这与郭巧巧等[27]在铜绿假单胞菌(Pseudomonasaeruginosa)中的发现一致,铜绿假单胞菌中有3个具有3-酮脂酰ACP还原酶活性的蛋白,其中PA2967为脂肪酸合成关键酶,而PA4389和PA4786也具有3-酮脂酰ACP还原酶活性,可能在不同碳源代谢过程中发挥作用。
4 结 论
本文通过生物信息学的方法,分析了Xoo中与大肠杆菌中EcFabG同源的6个蛋白XooFabG1~XooFabG6。结果表明,XooFabG6在理化性质、亲水性、信号肽和蛋白质翻译后修饰方面与EcFabG有较高的一致性,并且具备EcFabG中存在的Ser-Tyr-Lys催化活性中心和活性位点入口处的精氨酸残基,因此推测具有3-酮脂酰ACP还原酶活性,催化脂肪酸合成循环的第一还原反应。该结果为研究它们的生理学功能提供了前期证据,为开发以脂肪酸合成途径为靶点的抗水稻白叶枯病药物提供了理论依据。
