基于数据库挖掘5种胃癌干性基因在胃癌中潜在价值研究
2022-06-01余水红
余水红,马 军,钱 燕 ,龚 莉
(1.安庆医药高等专科学校科研中心;2.安庆市立医院普外科;3.安庆师范大学生命科学学院,安徽 安庆 246502)
胃癌 (Gastric cancer,GC) 是世界范围内发病率排名第五、死亡率排名第三的癌症,中国2018年约有39万胃癌新发病例和37万死亡病例[1]。近些年来随着对肿瘤分子生物水平研究的深入,分子靶向药物提高了胃癌患者生存期。但由于胃癌具有易复发、易转移及易耐药等特性,晚期胃癌患者术后5年总生存率仅约为10%。
胃癌干细胞是具有干细胞特性的癌细胞,与癌症的致癌、自我更新、复发、转移、耐药等特性紧密相关[2]。胃癌干细胞的研究可为胃癌治疗、提高患者生存质量及生存总期提供新的方案,研究表明富含亮氨酸重复序列G-蛋白偶联受体5 (leucine rich repeat -containing G-protei n coupled receptor 5,LGR5)、白细胞分化抗原分化簇第44号 (cluster of differentia tion44,CD44)、异常纺锤体样小头畸形相关基因 (abnormal spindle-like,microce phaly associated,ASPM)、三磷酸腺苷结合盒跨膜转运蛋白G超家族成员2 (ATP-binding cassette subfamily Gmember2,ABCG2) 及前列腺干细胞抗原 (prostate stem cell antigen,PSCA) 是胃癌干性的标志基因。LGR5可以通过激活胃腺癌细胞中的Wnt/catenin信号通路促进细胞增殖和侵袭[3],CD44表达与胃癌复发与患者死亡率直接相关[4],ASPM参与与胃粘膜癌变过程通路[5],ABCG2介导胃癌耐药机制[6],PSCA抑制胃癌细胞增殖[7]。
本文借助多个在线肿瘤数据库分析胃癌组织中5个典型干性基因的表达、变异及其与患者预后的关联,同时多方位评估干性基因相关的基因网络,并探讨其在肿瘤免疫中的作用,为LGR5、CD44、ASPM、ABCG2 及PSCA可能是胃癌诊断基因、治疗靶点基因及预后潜在候选基因提供研究。
1 材料与方法
1.1 材料数据库:UALCAN数据库(http://ualcan.path.uab.edu),Oncomine数据库(https://www.oncomine.org/),Kaplan-Meier plotter 数据库 (http://kmplot.com/analysis),cBioPortal数据库(http://www.cbioportal.org/),LinkedOmics 数据库 (http://www.linkedomics.org/login.php),webgestalt数据库(http://www.webgestalt.org/),TIMER数据库(https://cistrome.shinyapps.io/ timer/)。
1.2 方法
1.2.1 UALCAN数据库分析胃癌干性基因mRNA表达水平 在UALCAN数据库中进行TCGA数据集中分别检索LGR5、CD44、ASPM、ABCG2 及PSCA,于数据库选项中选择胃癌,分析5种干性基因在胃癌组织与正常组织中mRNA的表达。
1.2.2 Oncomine数据库分析胃癌干性基因DNA拷贝数水平 在Oncomine 数据库界面分别选择(1)Gene:LGR5、CD44、ASPM、ABCG2 及PSCA;(2)Analysis type:cancer vs.normal analysis;(3)Cancer type:gastric cancer;(4)Date type:mRNA and DNA;(5)临界值设定条件(Pvalue<0.05;gene rank:top 30%),分析LGR5、CD44、ASPM、ABCG2 及PSCA干性基因在胃癌组织与正常组织中DNA拷贝数。
1.2.3 Kaplan-Meier plotter数据库分析胃癌干性基因与患者预后的关系 在Kaplan-Meier plotter数据库中选择胃癌,基因框中同时输入LGR5、CD44、ASPM、ABCG2 及PSCA,分析比较患者样本高表达组及低表达组的生存预后。
1.2.4 cBioPortal数据库分析胃癌干性基因变异以及与患者预后的关系 在数据库类型中选择“TCGA database”,肿瘤类型选择胃癌,基因选择“LGR5、CD44、ASPM、ABCG2 及PSCA”,分析LGR5、CD44、ASPM、ABCG2 及PSCA在胃癌中的变异类型、变异数目及变异与预后的关系。
1.2.5 Linked Omics数据库分析胃癌干性基因及共表达基因网络 在Linked Omics数据库界面依次选择(1)肿瘤类型:选择结胃癌;(2)数据类型选择RNAseq;(3)数据属性:分别选择GR5、CD44、ASPM、ABCG2 及PSCA;(4)靶数据类型:选择RNAseq;(5)统计学方法:选择非参数检验。统计分析LGR5、CD44、ASPM、ABCG2 及PSCA干性基因的共表达基因,在火山图和热图中显示结果。
1.2.6 webgestalt数据库对胃癌干性基因正负相关基因进行功能注释及KEGG通路分析 在webgestalt数据库分别输入LGR5、CD44、ASPM、ABCG2 及PSCA相关的基因,对LGR5、CD44、ASPM、ABCG2 及PSCA干性基因正负相关的500个基因进行功能注释及KEGG通路。
1.2.7 TIMER数据库分析胃癌干性基因与免疫细胞的关系 在TIMER数据库界面“Gene Symbol”框中分别输入“LGR5、CD44、ASPM、ABCG2 及PSCAI”,“Cancer Types”选择“STAD”,其余为默认选项设,分析LGR5、CD44、ASPM、ABCG2 及PSCA干性基因在胃癌中的表达与免疫浸润的相关性,包括B细胞、CD4+T细胞、CD8+T细胞、中性粒细胞、巨噬细胞和树突状细胞以及肿瘤纯度。
1.3 统计学分析实验结果数据均来自以上数据库,采用系统默认的统计学方法,计算危险比(Hazard Radio,HR)及95%置信区间(Confidence Interval,CI),LongrankP<0.05检验差异,LongrankP<0.05表示差异具有统计学意义。
2 结果与分析
2.1 胃癌干性基因在胃癌组织中的表达异常表达的基因是诊断肿瘤潜在的重要分子指标,结果表明胃癌组织中LGR5、CD44及ASPM mRNA的表达高于相应的正常组织 (P<0.05),而ABCG2和PSCA mRNA水平在胃癌组织中的表达低于正常组织 (P<0.05) (图1A~1E)。Oncomine数据库中的数据表明LGR5、CD44、ASPM及PSCA的DNA拷贝数在肿瘤组织中明显高于正常组织 (P<0.05)(图1F~1I),而ASPM表达无统计学意义。因此LGR5、CD44、ASPM和PSCA的表达可作为诊断GC潜在的指标。

图1 干性基因在胃癌组织和正常胃组织中的表达注:A:LGR5 mRNA的表达;B:CD44 mRNA的表达;C:ASPM mRNA的表达;D:ABCG2 mRNA的表达;E:PSCA mRNA的表达;F:LGR5DNA拷贝数;G:CD44 DNA拷贝数;H:ASPM DNA拷贝数;I:PSCA DNA拷贝数
2.2 胃癌干性基因与胃癌患者预后的相关性肿瘤患者预后是评估基因重要性的指标之一,结果表明LGR5、CD44、ABCG2高表达组的总生存期明显短于低表达组 (HR>1,P<0.05),ASPM高表达组生存期明显长于对照组 (HR<1,P<0.05),而PSCA表达与胃癌患者预后的相关性无统计学意义(HR>1,P>0.05)(图2A~2E)。因此LGR5、CD44、ASPM及ABCG2可作为患者预后候选基因。

图2 干性基因的表达和胃癌患者总生存期的关系(Kaplan-Meier plotter)注:A:LGR5;B:CD44;C:ASPM;D:ABCG2;E:PSCA
2.3 胃癌干性基因在胃癌组织中的变异及类型基因变异是肿瘤发生发展重要的内在原因,结果显示在369例患者中有104例发生胃癌干性基因变异,LGR5、CD44、ASPM、ABCG2 及PSCA扩增性变异分别为7、18、3、1、21例,突变变异分别为12、7、35、6、2例,缺失变异分别为1、0、0、2、0例,mRNA上调分别为1、2、3、5、0例 (图3)。LGR5、ASPM及ABCG2变异类型最多的是突变变异,CD44及PSCA

图3 干性基因在胃癌中的变异(cBioPortal)
最多的是扩增性变异。ASPM变异可提高胃癌患者5年生存期,ABCG2变异不利于胃癌患者5年生存期,且具有统计学意义 (P<0.05,图4A、4B),其他基因没有统计学意义。因此ASPM和ABCG2的变异可能与胃癌的发生发展有密切的联系。

图4 ASPM及ABCG2变异对胃癌患者5年生存率的影响(cBioPortal) 注:A:ASPM变异与生存关系;B:ABCG2变异与生存关系
2.4 胃癌干性基因在胃癌中的共表达网络胃癌干性基因潜在功能的发挥,可能是与多种基因共同作用的结果,火山图显示有1804、3251、5095、2332、5554个基因(红点)分别与LGR5、CD44、ASPM、ABCG2和PSCA呈显著正相关 (FDR<0.01),而其中有477个、2279个、5352个、1843个、1659个基因 (黑点) 分别与之呈显著负相关(FDR<0.01);正负相关的前50个关键基因热图如图所示(图5A~5E)。结果表明这些正负相关基因参与的生物学过程有生物调节、代谢过程、刺激反应、多细胞生物过程等13类过程;细胞组分主要集中在细胞膜、细胞核、细胞溶质、膜封闭腔等21类;分子功能有蛋白结合、离子束缚、核酸结合等17类 (图6A~6C,图7A~7C)。通路分析结果表明干性基因及正相关的基因参与了驱动蛋白、蛋白逆向运输、脂肪消化吸收及有丝分裂前中期等过程 (表1);干性基因及负相关的基因参与了平滑肌收缩、细胞周期各阶段、肌肉收缩、磷酸代谢及幽门螺杆菌感染等过程 (表2)。富集分析有助了解LGR5、CD44、ASPM、ABCG2、PSCA及其相互作用基因在胃癌中的作用机理。
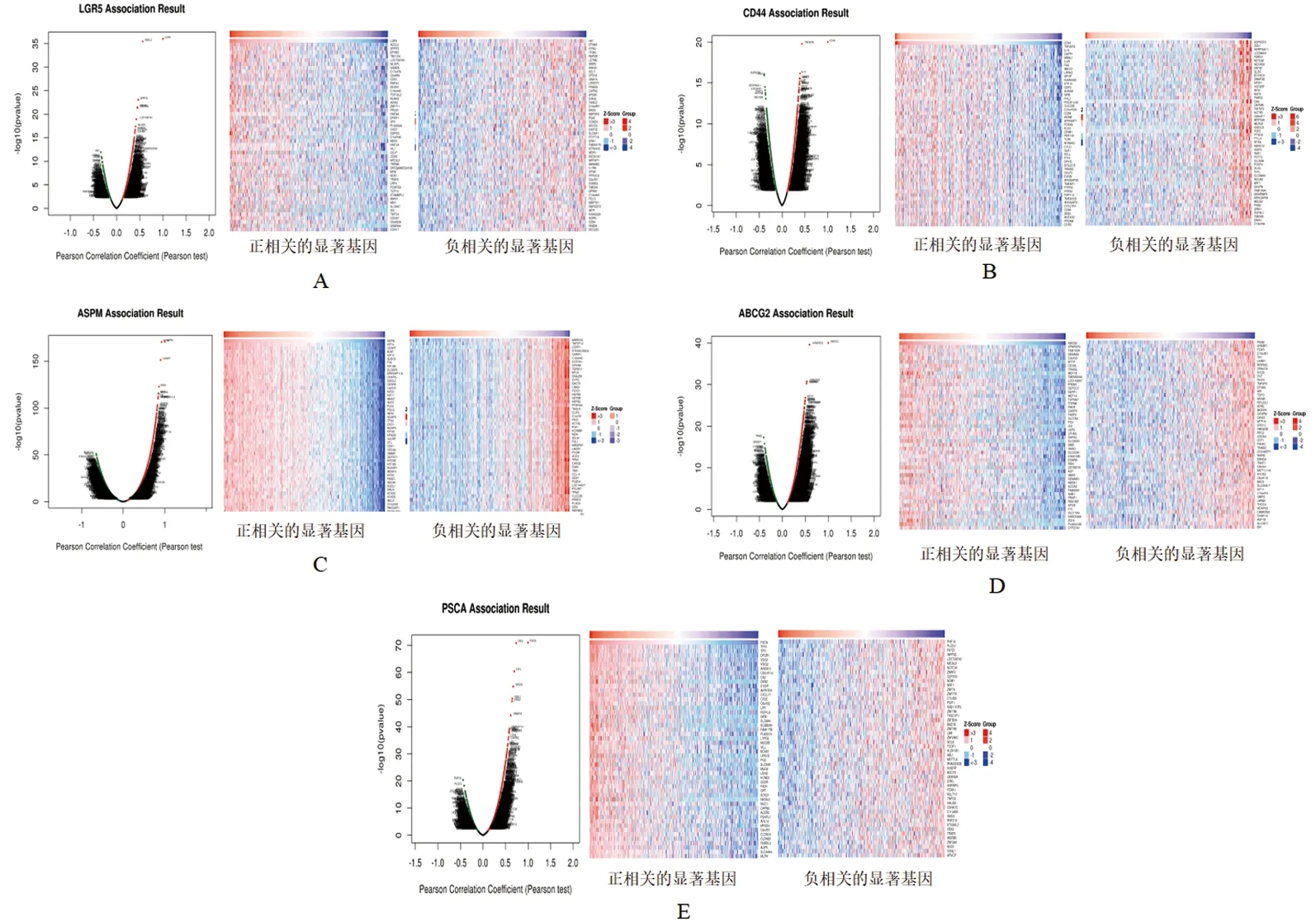
图5 胃癌干性基因共表达基因 (LinkedOmics)注:A:LGR5相关基因火山图与热图;B:CD44相关基因火山图与热图;C:ASPM相关基因火山图与热图;D:ABCG2相关基因火山图与热图;E:PSCA相关基因火山图与热图

图6 胃癌干性基因和正相关基因的GO功能注释(MetaScape)注:A:生物学过程;B:细胞组份;C:分子功能

图7 胃癌干性基因和负相关基因的GO功能注释(MetaScape)注:A:生物学过程;B:细胞组份;C:分子功能

表1 胃癌干性基因及正相关基因KEGG通路富集(MetaScape)

表2 胃癌干性基因及负相关基因KEGG通路富集(MetaScape)
2.5 胃癌干性基因在胃癌组织中与免疫细胞的关系肿瘤患者生存期与免疫细胞浸润相关,利用TIMER数据库分析胃癌干性基因LGR5、CD44、ASPM、ABCG2 及PSCA在胃癌免疫微环境中的表达(图8A~8E),结果显示这5个干性基因与肿瘤纯度的关系分别为无统计学意义、负相关、正相关、无统计学意义、负相关;LGR5、CD44及ASPM与B细胞的表达无统计学意义,ABCG2 及PSCA与B细胞的表达呈正相关;LGR5、ABCG2 及PSCA与CD8+T细胞的表达无统计学意义,CD44、ASPM与CD8+T细胞的关系分别为正相关、负相关;LGR5、CD44、ASPM、ABCG2与CD4+T细胞的表达呈正相关,PSCA与CD4+T细胞的表达无统计学意义;LGR5、CD44、ASPM、ABCG2 及PSCA与巨噬细胞的表达的关系分别为无统计学意义、正相关、负相关、正相关、无统计学意义;CD44与中性粒细胞及树突状细胞的表达呈正相关,ASPM呈负相关,LGR5、ABCG2 及PSCA均无统计学意义。巨噬细胞及树突状细胞高表达不利于胃癌者5年(60个月)生存,具有统计学意义(P<0.05,图9),而B细胞、CD4+T细胞、CD8+T细胞及中性粒细胞与患者5年生存的关系无统计学意义(P>0.05)。因此研究胃癌干性基因及免疫细胞可以为胃癌患者免疫治疗提供参考。
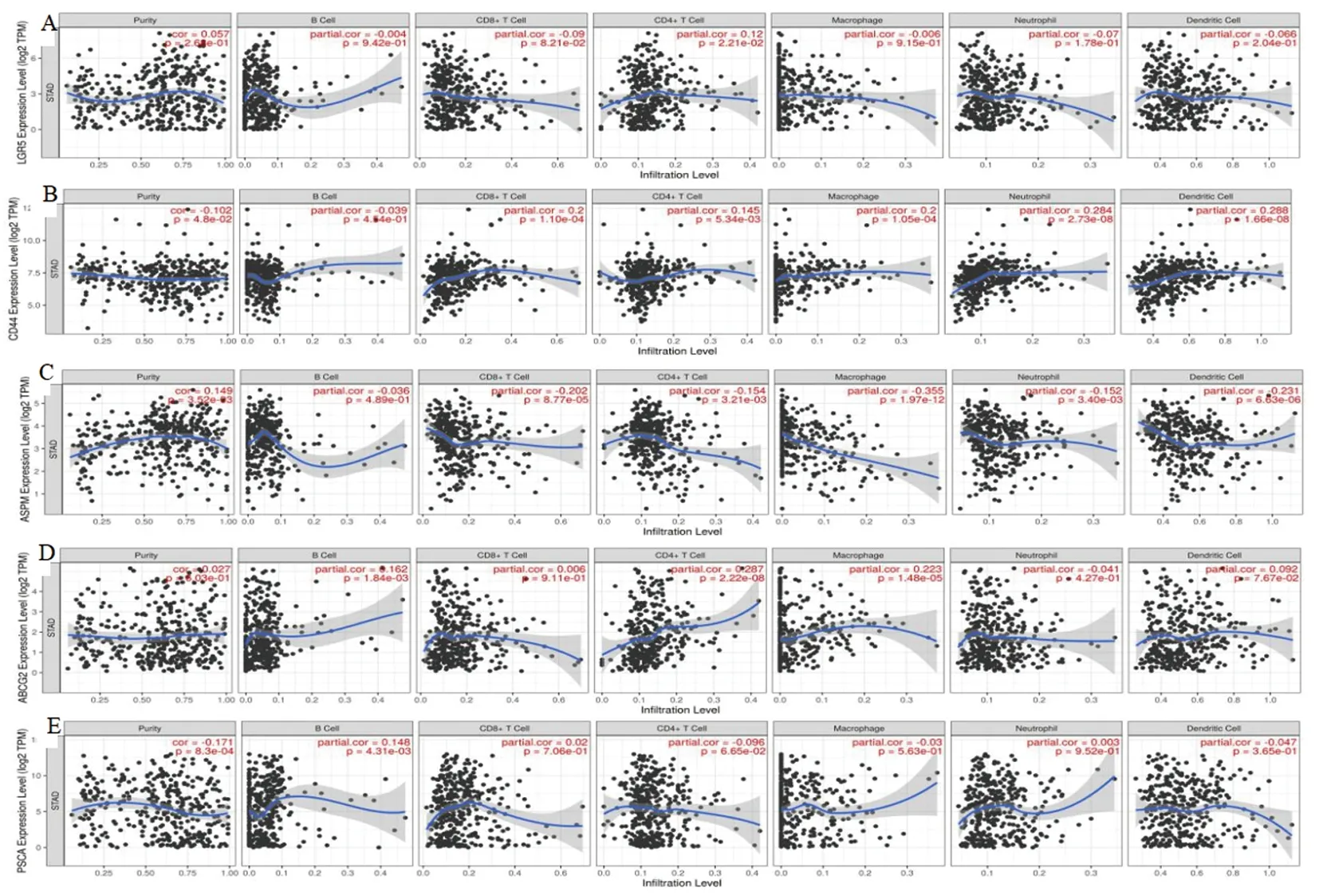
图8 干性基因与肿瘤免疫细胞间的相关性(TIMER)

图9 高表达干性基因 mRNA的B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、中性粒细胞及树突状细胞对胃癌患者5年生存率的影响(TIMER)
3 讨论
胃癌干细胞研究是胃癌治疗中的研究热点与重点,其中标志干性基因LGR5、CD44、ASPM、ABCG2 及PSCA在胃癌发生发展过程中起着至关重要的作用[5,10-17],有望成为胃癌治疗潜在的分子靶点来源。为能够详细了解LGR5、CD44、ASPM、ABCG2 及PSCA在胃癌中的潜在功能及其基因调控网络,本研究利用多个成熟的在线公共数据库对其进行生物信息学分析,以期为干性基因在胃癌治疗中的进一步研究提供参考。
对胃癌干性基因转录分析表明其mRNA水平及DNA 拷贝数明显异常于正常的胃癌组织,研究表明LGR5、CD44、ASPM 及PSCA在胃癌组织中mRNA相对表达与本文研究一致[5,7,18-19],其中LGR5增强胃癌细胞生长、迁移及耐药能力,CD44增强化疗或辐射诱导的细胞死亡的耐受性,ASPM参与胃粘膜癌变过程,PSCA抑制胃癌细胞增殖。而ABCG2在胃癌细胞中相对表达较高且介导胃癌的耐药机制[6],PSCA基因mRNA水平及DNA 拷贝数在胃癌组织中表现不一致,ABCG2及PSCA在胃癌中DNA 拷贝数可能更多与与肿瘤细胞分裂有关,从而影响肿瘤发展;而RNA可能更多与肿瘤细胞合成蛋白分子有关,从而影响肿瘤的发生。
对胃癌干性基因共表达网络分析表明,异常表达的干性基因及正相关基因参与了细胞有丝分裂前中期、脂肪消化吸收及蛋白运输等过程,负相关基因参与了细胞周期各阶段、磷酸代谢及幽门螺杆菌感染等过程,这些发现与胃癌治疗基础研究的分子途径基本一致[10,20],但脂肪消化吸收过程是如何影响胃癌发生发展需要基础研究深入探索。
基因变异是肿瘤发生发展的内在原因之一,其可以破坏基因、改变基因编码蛋白数量及种类、导致表型差异[21]。本研究发现ASPM及ABCG2变异类型最多的是突变变异,其中ASPM变异提高胃癌患者5年生存期,而ABCG2变异则缩短患者5年生存期。有研究表明胃癌中ASPM表达的改变导致其编码的蛋白功能受到影响,随之中间体功能出现缺陷使染色体分离的失败导致肿瘤发生发展[22]。ABCG2可以作为Gli1的功能执行基因,介导胃癌化疗耐药的发生,导致患者生存期缩短[23]。
肿瘤微环境是非癌细胞存在于肿瘤内部或肿瘤周围外部环境,免疫细胞是肿瘤微环境重要组成部分,对基因的变异或选择性表达具有重要的影响作用[24],本研究表明5种胃癌干性基因与肿瘤微环境中的不同免疫细胞密切相关,很可能参与肿瘤细胞的免疫逃逸和免疫抑制过程。
总之,本研究通过生物信息分析从多个层次探讨这5种干性基因在胃癌中的作用。它们不仅在胃癌发生发展、诊断治疗及患者预后方面非常重要,在免疫调节和免疫治疗中也具有重要作用,但这些发现仍需要进一步的胃癌基因组学及功能研究。
