基于mtDNA Cytb基因和D-loop区序列的陕西林麝遗传多样性分析
2022-06-01刘晓玮李瑞香王兴龙
刘晓玮,李瑞香,王兴龙,李 晓
(1.西北农林科技大学动物科技学院,杨凌 712100;2.动物疫病预防控制中心,临渭 714000;3.西北农林科技大学动物医学院,杨凌 712100)
林麝(Moschusberezovskii)在世界自然保护联盟红色名录中被列为濒危物种[1],是中国珍稀濒危物种[2],对于中国的经济发展有重要影响[3]。由成年雄性林麝分泌的麝香[4],不仅是中药中不可缺少的成分,而且在香水工业中也被广泛使用[5]。由于市场对麝香的高需求,大量的野生林麝被狩猎以供应麝香贸易,导致野生林麝的数量迅速减少[6]。为控制这种情况的持续发展并满足市场对麝香的需求,中国自1958年以来大力支持人工饲养林麝,并逐渐扩大规模[7]。林麝是一种胆小、警惕和孤独的动物[8],与野生林麝相比,在圈养环境中种群易患腹泻[9-10]、肺炎等疾病[11-12],同时遗传多样性可能会降低。因此,分析遗传多样性对林麝资源的保护和利用具有关键性作用[13]。
彭红元等[14]使用生物进化距离(NJ)、统计特征(ML)和离散特征(MP)3种构建进化树常用方法研究麝与鹿的亲缘关系,其中,系统发育进化树(Neighbor-Joining tree)利用树状分支图形可以直观地体现各物种间的亲缘关系。Su等[15]利用细胞色素b (cytochrome b,Cytb)基因序列研究表明,麝与鹿科有较近的系统发育关系。Guha等[16]基于线粒体Cytb基因部分片段分析了麝等各类群的系统发生关系,结果显示,麝与牛、鹿的关系更为密切。肖宇辰等[17]研究发现,麝科动物的演化史可能起源于北亚,以中国为中心,由北向南呈辐射状扩散[18]。目前麝属中有原麝(Moschusmoschiferus)、林麝(Moschusberezovskii)、马麝(Moschuschrysogaster)、黑麝(Moschusfuscus)、安徽麝(Moschusanhuensis)及喜马拉雅麝(Moschusleucogaster),了解它们之间的关系是进行有效保护的前提[19]。
Cytb基因和D-loop区是种群遗传多样性研究中常用到的2个分子标记。线粒体DNA(mtDNA)是研究物种遗传多样性的一种重要遗传物质,存在于真核生物中,在结构和功能上与核DNA有明显差异[20],为群体遗传学的研究提供了极大的便利[21]。mtDNA中的Cytb基因进化速度适中,一个较小的基因片段可以包含从种内到种间乃至科间的遗传进化信息,比较适用于生物系统进化关系的研究[22]。mtDNA中的非编码序列置换环(D-loop)是长度变异最大的区域[23],其序列变异不仅包括核苷酸之间的替换,还包括核苷酸之间的缺失、插入和串联重复[24]。本研究通过对陕西林麝样品mtDNACytb基因和D-loop区序列进行相关分析,探究陕西林麝的进化关系及遗传多样性,以期为进一步保护和利用遗传资源提供客观依据。
1 材料与方法
1.1 样品采集
选取陕西渭南市凤县林麝养殖场人工圈养的43只林麝,非损伤法采集毛发,存放于装有无水乙醇的离心管,用封口膜进行密封保护,分装编号后,-20 ℃保存备用。
1.2 主要试剂与仪器
DL2000 DNA Marker、核酸染料均购自北京聚合美生物科技有限公司;琼脂糖购自北京擎科生物科技有限公司;2×TaqPCR MasterMix购自北京康为世纪生物科技有限公司;Chelex-100试剂盒购自天津诺维莱博科技有限公司。
超纯水仪(Uitra Genetic)购自威立雅水处理技术(上海)有限公司;全自动高压灭菌锅(SQ810C)购自上海尔迪仪器科技有限公司;超微量分光光度计(DS-11)购自广州深华生物技术有限公司;PCR仪(S1000)和全自动凝胶成像分析系统(Geldoc XR+)均购自时代联想生物科技有限公司;电泳仪(JY-SPCT)购自北京君意东方电泳设备有限公司。
1.3 基因组DNA提取
按照Chelex-100试剂盒操作说明从林麝毛发中提取基因组DNA[25],用超微量分光光度计测定浓度和纯度,-20 ℃保存备用。
1.4 PCR扩增及测序
参考文献设计并合成Cytb基因[26]及D-loop区[3]引物,引物信息见表1。引物均由生工生物工程(上海)股份有限公司合成。
PCR反应体系40 μL:TaqDNA聚合酶20 μL,上、下游引物各2 μL,DNA模板6 μL,超纯灭菌水10 μL。PCR反应程序:95 ℃预变性 5 min;94 ℃变性30 s,56 ℃退火50 s,72 ℃延伸50 s,共35个循环;72 ℃延伸10 min;4 ℃保存。PCR扩增产物采用1.5%琼脂糖凝胶电泳检测,取鉴定正确的PCR产物送至北京奥科鼎盛生物科技有限公司进行纯化,并在Applied Biosystems 3730xl DNA Analyzer测序仪上双向测序。
1.5 数据分析
对测序峰图和碱基的准确性进一步核对,统计序列碱基组成,并利用NCBI数据库对序列进行 BLAST比对。利用ClustalX 2.0软件对所有序列进行整合比对,以获得群体中核苷酸多态性(SNP)位点。采用DNASP 5.10软件统计种群序列核苷酸多样性(Pi)、序列的单倍型数(H)、单倍型多样度(Hd)、平均核苷酸差异数(K)等参数;运用Mega 7.0软件计算Cytb基因和D-loop区序列不同单倍型间遗传距离,并构建Neighbor-Joining(NJ)系统进化树,对林麝群体(表2)的遗传多样性和进化关系进行评估。
2 结 果
2.1 PCR扩增
对林麝样品进行mtDNACytb基因和D-loop区PCR扩增,PCR产物用1.5%琼脂糖凝胶电泳进行检测,结果显示,Cytb基因和D-loop区扩增条带整齐明亮,大小分别为472和669~711 bp(图1、2),均与预期片段大小相符,符合测序要求。

M,DL2000 DNA Marker;1~5,Cytb基因PCR产物M,DL2000 DNA Marker;1-5,PCR products of Cytb gene图1 林麝Cytb基因PCR扩增产物电泳图Fig.1 Agarose gel electrophoresis of PCR amplification products of Cytb gene in Moschus berezovskii
M,DL2000 DNA Marker;1~5,D-loop区PCR产物M,DL2000 DNA Marker;1-5,PCR products of D-loop region图2 林麝D-loop区PCR扩增产物电泳图Fig.2 Agarose gel electrophoresis of PCR amplification products of D-loop region in Moschus berezovskii
2.2 Cytb基因和D-loop区序列碱基组成
林麝群体Cytb基因和D-loop区序列的碱基组成见表3。由表3可知,所有样品Cytb基因序列长度为472 bp,经NCBI系统BLAST查询后确定为Cytb基因;4种核苷酸T、C、A、G的平均含量分别为30.9%、18.8%、30.9%及19.4%,AT碱基含量为61.8%,GC碱基含量为38.2%,具有明显的碱基偏好性。林麝D-loop区序列全长在669~711 bp之间,不同个体间存在序列长度差异(表4);T、C、A、G的平均含量分别为30.1%、22.9%、32.1%及14.9%,AT碱基含量为62.2%,GC碱基含量为37.8%,具有明显的碱基偏好性(表3)。 D-loop区的A、T含量较高,C含量较低,群体的D-loop区比Cytb基因序列具有较高的AT含量及较低的GC含量。
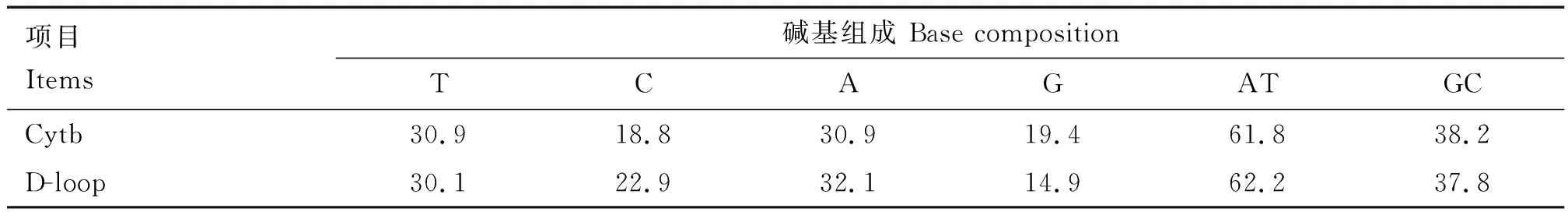
表3 林麝群体Cytb基因和D-loop区序列的碱基组成Table 3 Base composition of Cytb gene and D-loop region sequences in Moschus berezovskii population %
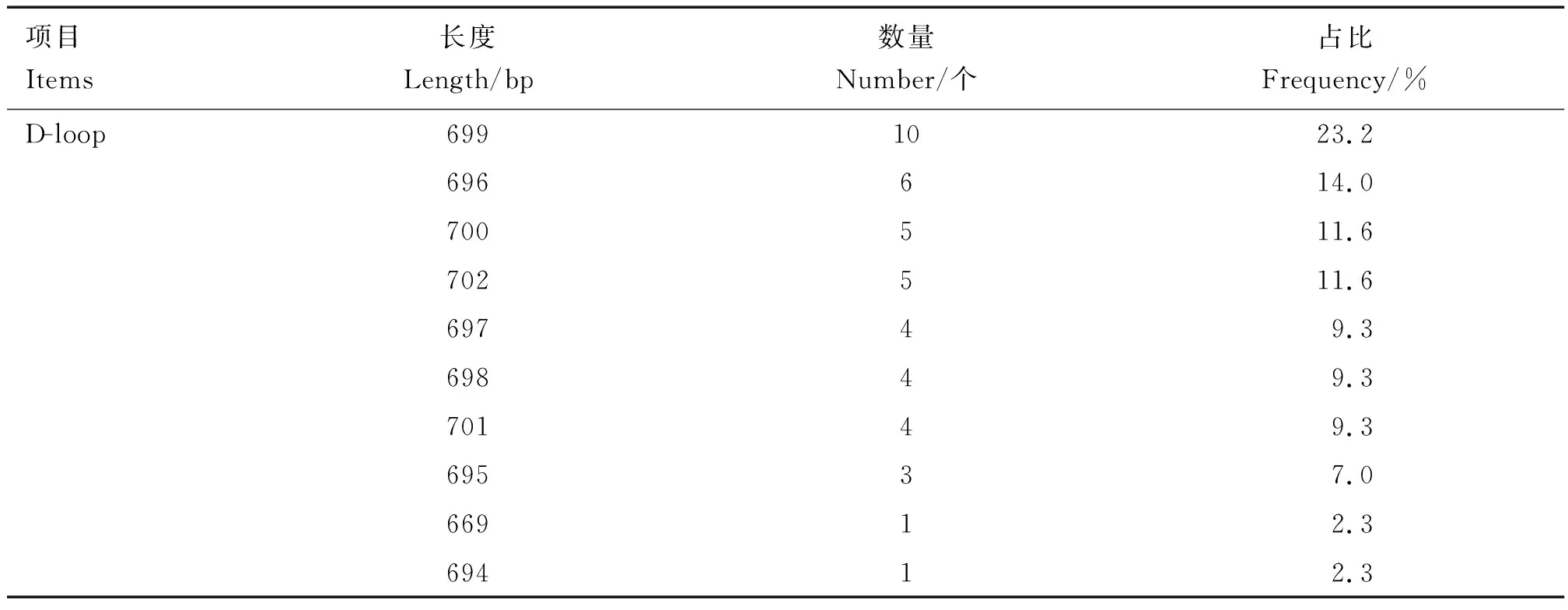
表4 林麝群体D-loop区序列长度信息Table 4 Length information of D-loop region sequence in Moschus berezovskii population
2.3 遗传多样性指数
利用ClustalX 2.0、DNASP 5.10软件对获得的Cytb基因和D-loop区序列进行分析,结果显示,Cytb基因和D-loop区序列分别有241和383个SNPs,其中241个SNPs中包含236个简约信息位点和5个单一信息位点,383个SNPs中包含347个简约信息位点和36个单一信息位点;单倍型数、单倍型多样度、核苷酸多样度等结果均为Cytb基因序列较高,林麝所有个体样本在Cytb基因和D-loop区分别检测出35和29个单倍型,单倍型比例分别为81.4%和67.4%,单倍型多样性分别为0.983和0.975;核苷酸多样度分别为0.28343和0.07707(表5)。
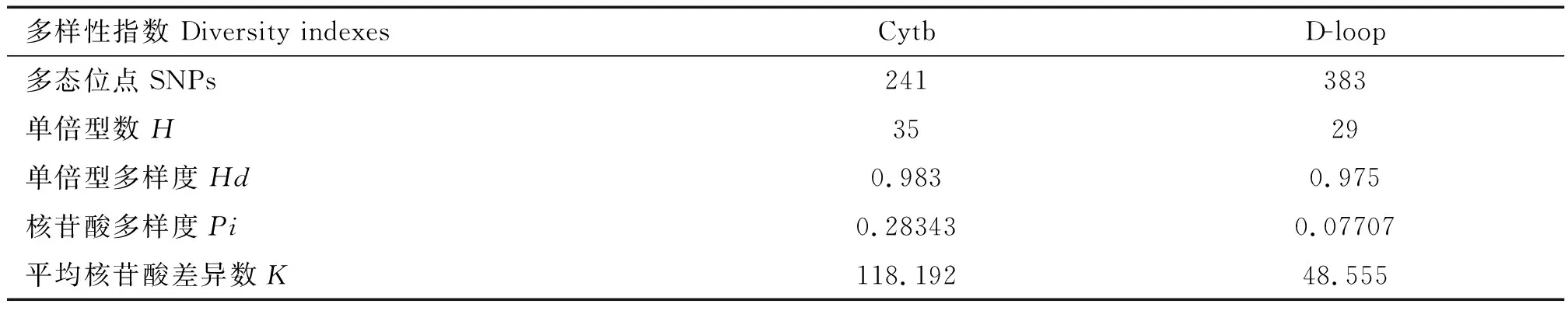
表5 林麝Cytb基因和D-loop区序列遗传多样性指数Table 5 Genetic diversity indexes of Cytb gene and D-loop region sequences in Moschus berezovskii
单倍型具体分布情况见表6。由表6可知,43条Cytb基因序列共定义35个单倍型(Hap1~Hap35),其中单倍型Hap12的数量最多,共有5个(11.63%),单倍型Hap3的数量为3个(6.98%);单倍型Hap5、Hap7的数量均为2个(4.65%),其余单倍型数量均为1个(2.33%);43条D-loop区序列共定义29个单倍型,其中单倍型Hap1的数量最多,共有5个(11.63%),单倍型Hap4、Hap13和Hap14的数量均为3个(6.98%);单倍型Hap3、Hap17、Hap19和Hap26的数量均为2个(4.95%);其余单倍型数量均1个(2.33%)。
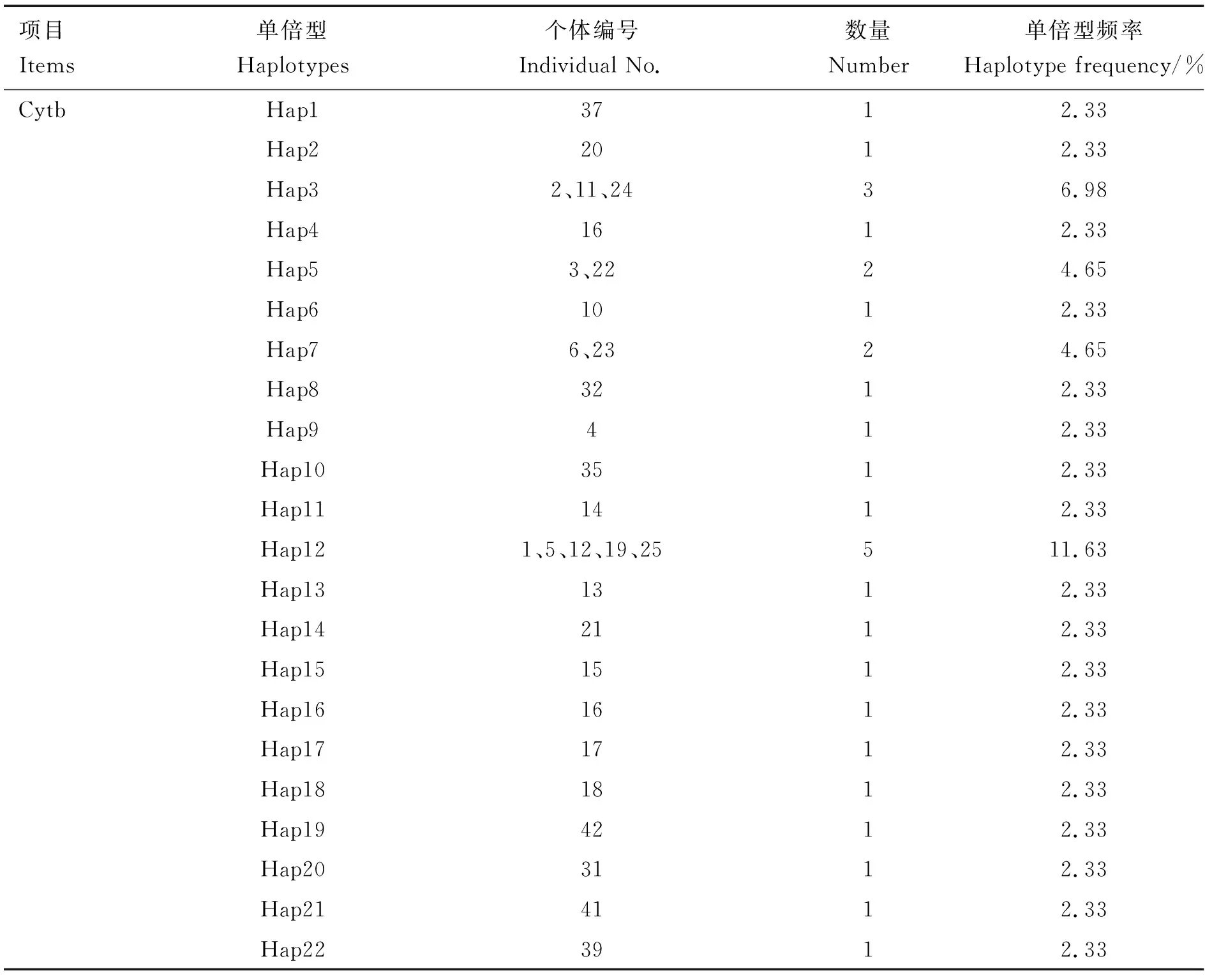
表6 林麝Cytb基因和D-oop区序列的单倍型分布Table 6 Haplotype distribution of Cytb gene and D-loop region sequences in Moschus berezovskii
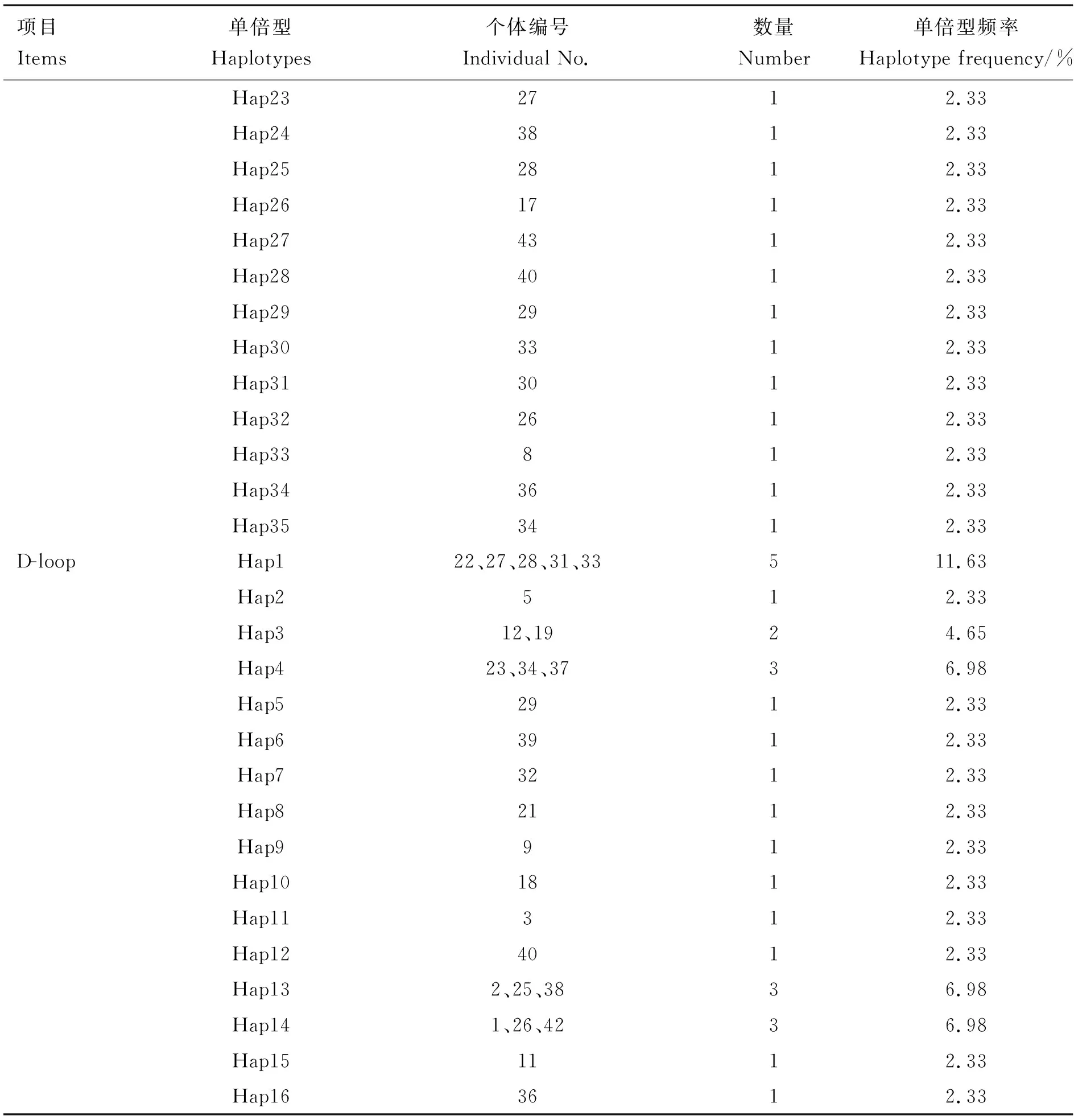
续表
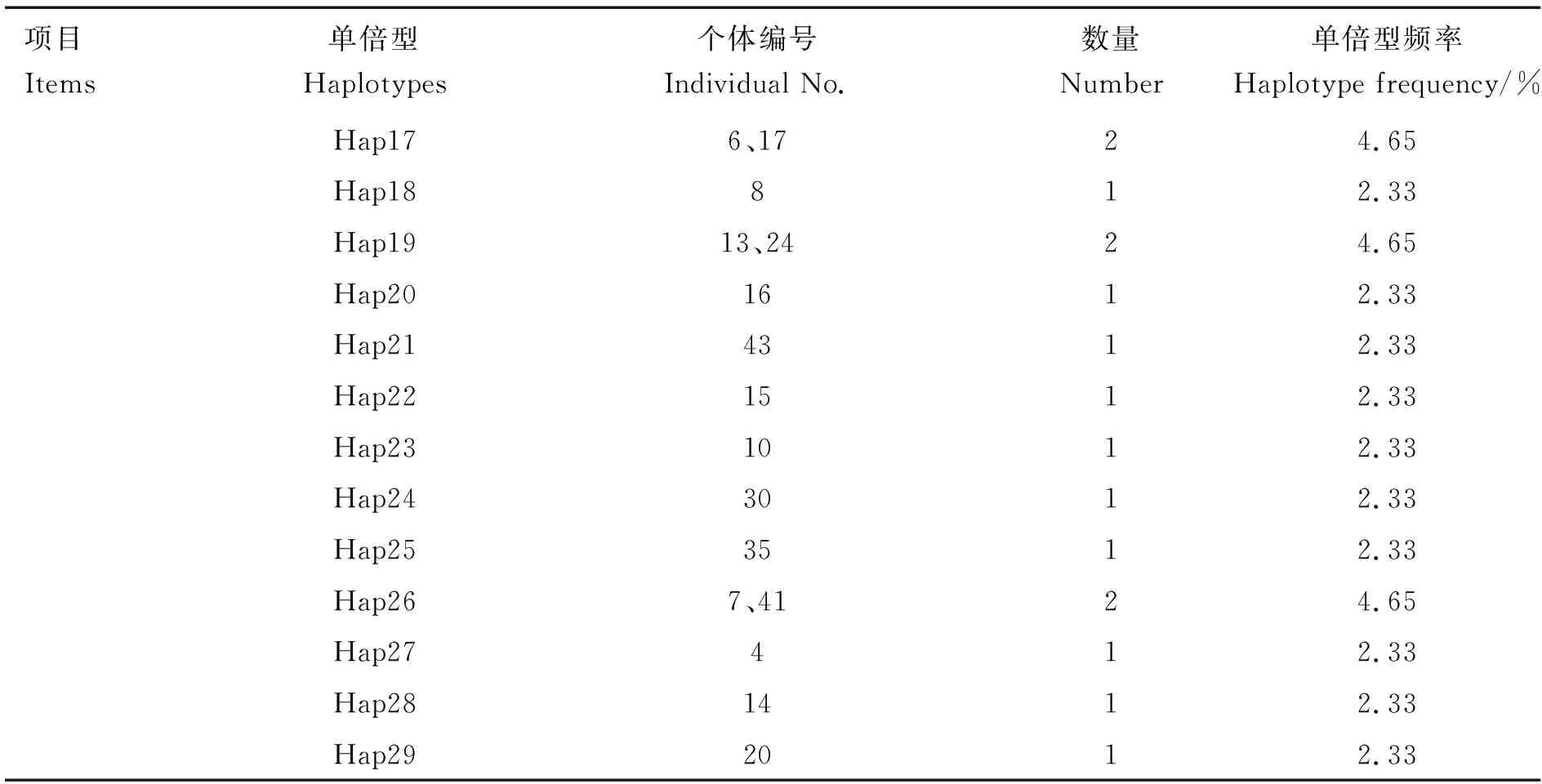
项目Items单倍型Haplotypes个体编号Individual No.数量Number单倍型频率Haplotype frequency/%Hap176、1724.65Hap18812.33Hap1913、2424.65Hap201612.33Hap214312.33Hap221512.33Hap231012.33Hap243012.33Hap253512.33Hap267、4124.65Hap27412.33Hap281412.33Hap292012.33
2.4 遗传距离
通过Mega 7.0软件分别计算林麝Cytb基因和D-loop区序列不同单倍型间的遗传距离,结果显示,Cytb基因35个单倍型之间的遗传距离在0.002~0.831之间,其中Hap6与Hap12的遗传距离最小,为0.002;Hap1与Hap13、Hap10与Hap13的遗传距离最大,均为0.831(图3)。D-loop区序列29个单倍型之间的遗传距离在0.006~1.342之间,其中Hap4与Hap17的遗传距离最小,为0.006;Hap9与Hap25的遗传距离最大,为1.342(图4)。
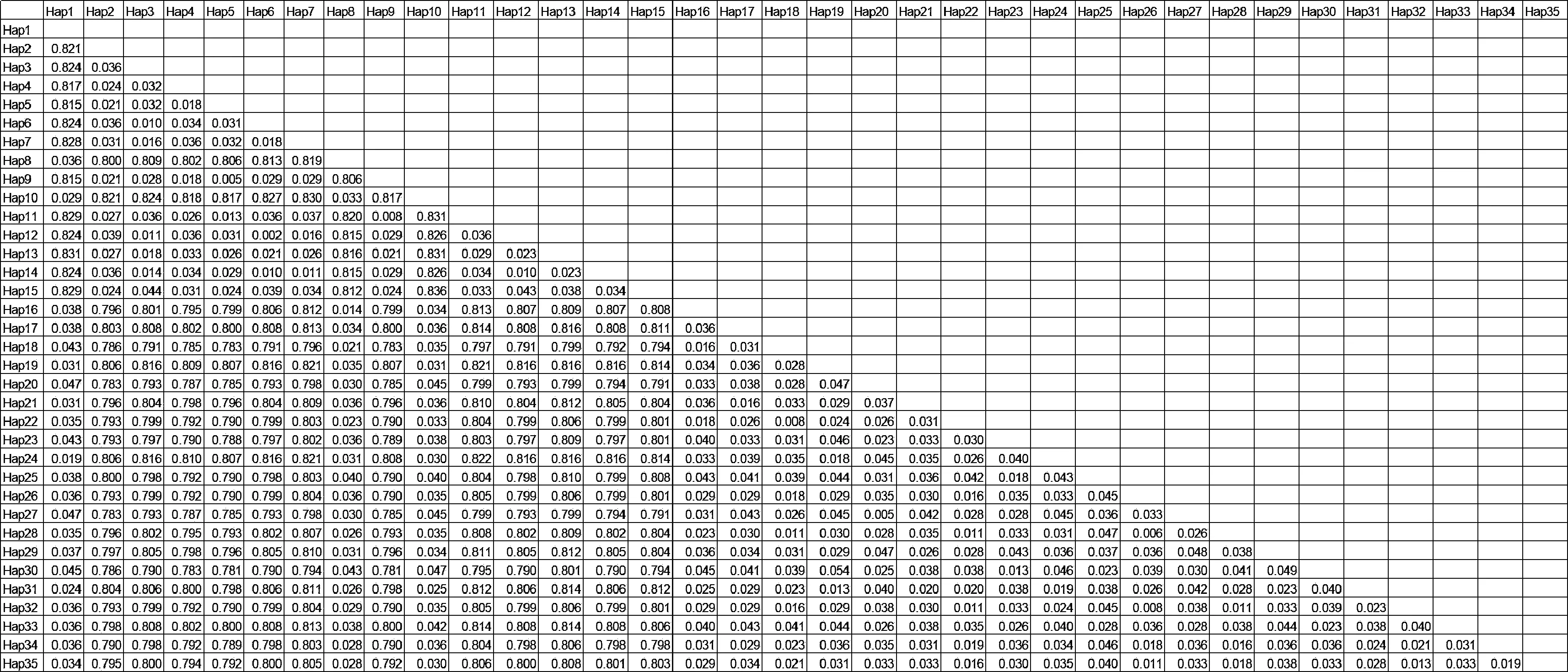
图3 林麝Cytb基因序列的单倍型遗传距离Fig.3 Haplotype genetic distance of Cytb gene sequence in Moschus berezovskii

图4 林麝D-loop区序列的单倍型遗传距离Fig.4 Haplotype genetic distance of D-loop region sequence in Moschus berezovskii
2.5 系统进化树构建
2.5.1 林麝Cytb基因和D-loop区序列单倍型系统进化树 利用Mega 7.0软件构建林麝群体的系统进化树,结果见图5、6。进化树分支上的百分比代表经过检验有多少个数位于此分支,也反映了进化树的置信度;标在分支上的数字为支长的具体数值。由图5、6可知,顶点上的百分数全部为100%(>70%),整体上具有可信度,基于Cytb基因,林麝所有个体可以被归为2个大分支,有较远的遗传距离,其中单倍型Hap1、Hap8、Hap10、Hap16~Hap35聚为一支,单倍型Hap2~Hap7、Hap9、Hap11~Hap15聚为一支;基于D-loop区,林麝所有个体可以被归为2个大分支,有明显较远的遗传距离,其中单倍型Hap9、Hap18聚为一支,其余单倍型聚为一支。与Cytb基因序列不同的是,大部分单倍型都处于一个分支,只有2个单倍型为另外一个分支,可能与D-loop区进化速度较快有关。
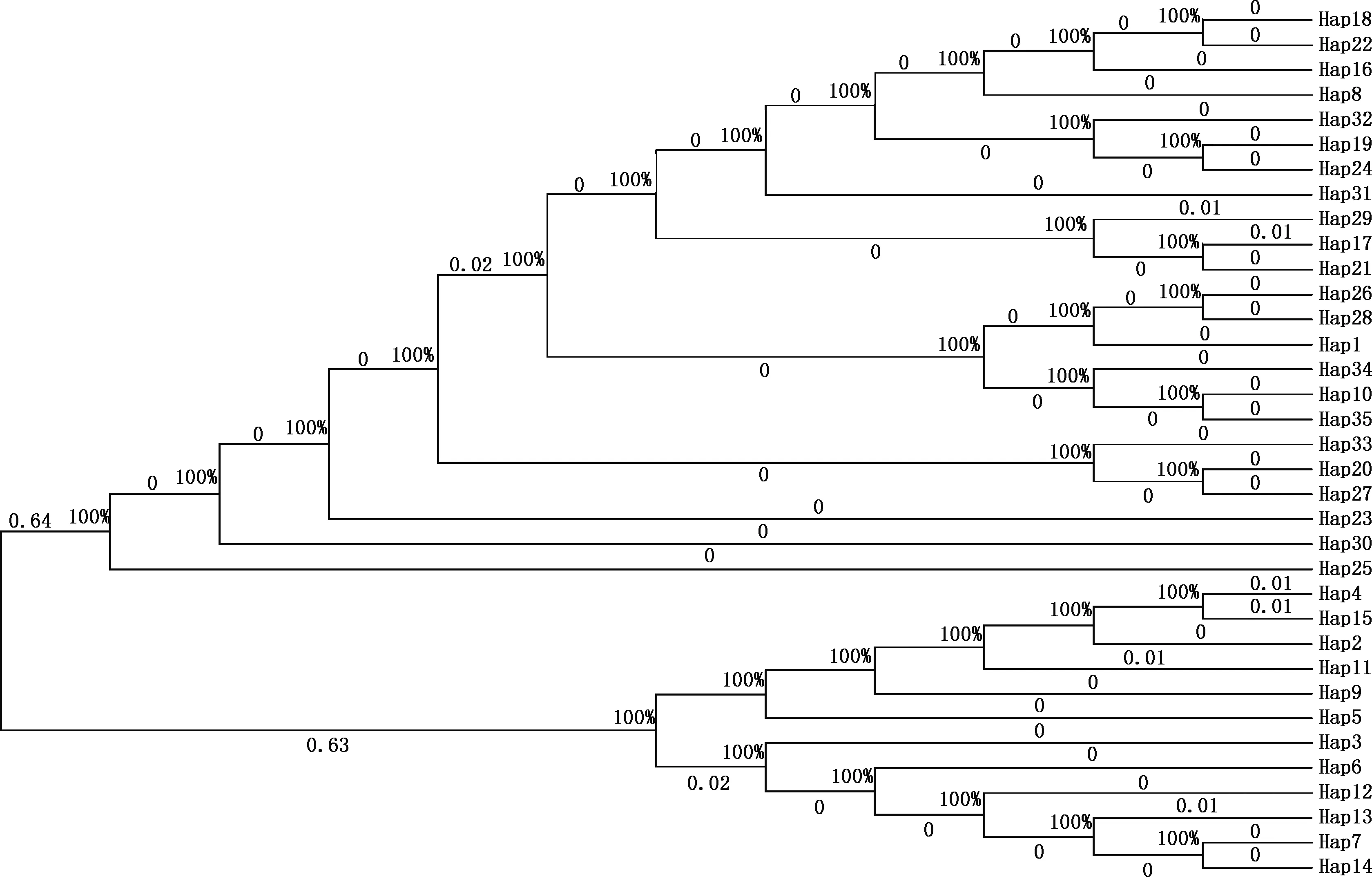
图5 林麝Cytb基因序列单倍型系统进化树Fig.5 Haplotype phylogenetic tree of Cytb gene sequence in Moschus berezovskii
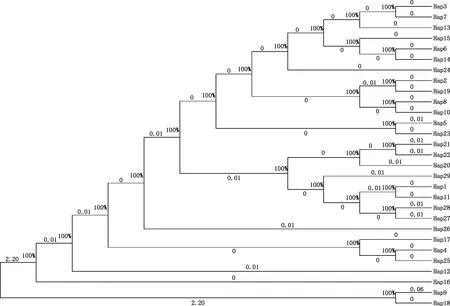
图6 林麝D-loop区序列单倍型系统进化树Fig.6 Haplotype phylogenetic tree of D-loop region sequence in Moschus berezovskii
2.5.2 麝属系统进化树 应用Mega 7.0软件构建麝属系统进化树,结果见图7、8。由图7、8可知,喜马拉雅麝、原麝、林麝、原麝、黑麝、安徽麝为6个独立种,林麝和原麝亲缘关系较近,林麝与其他麝属的亲缘关系较远。
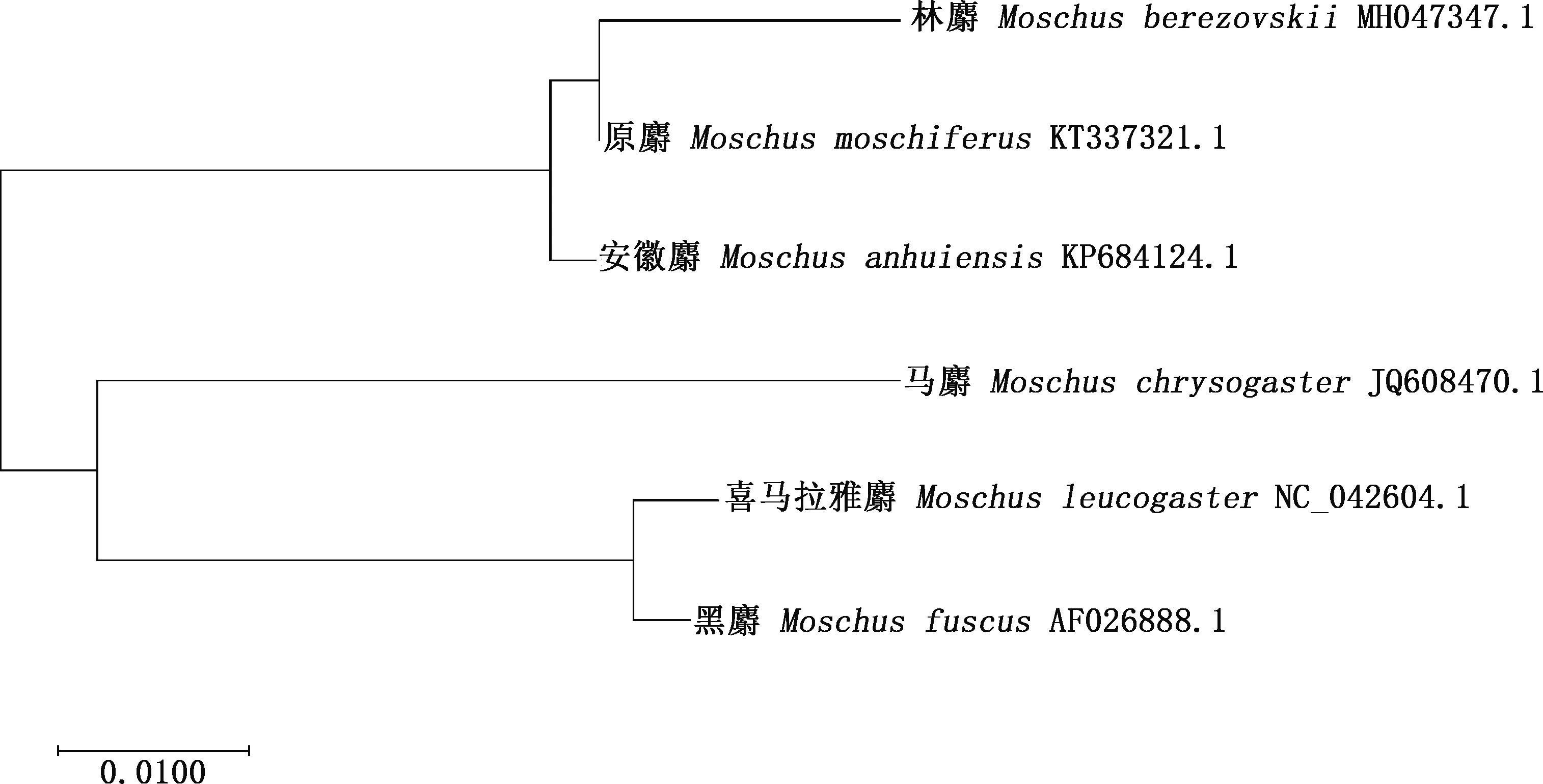
图7 基于Cytb基因部分序列构建的系统进化树Fig.7 Phylogenetic tree constructed based on partial sequences of Cytb gene
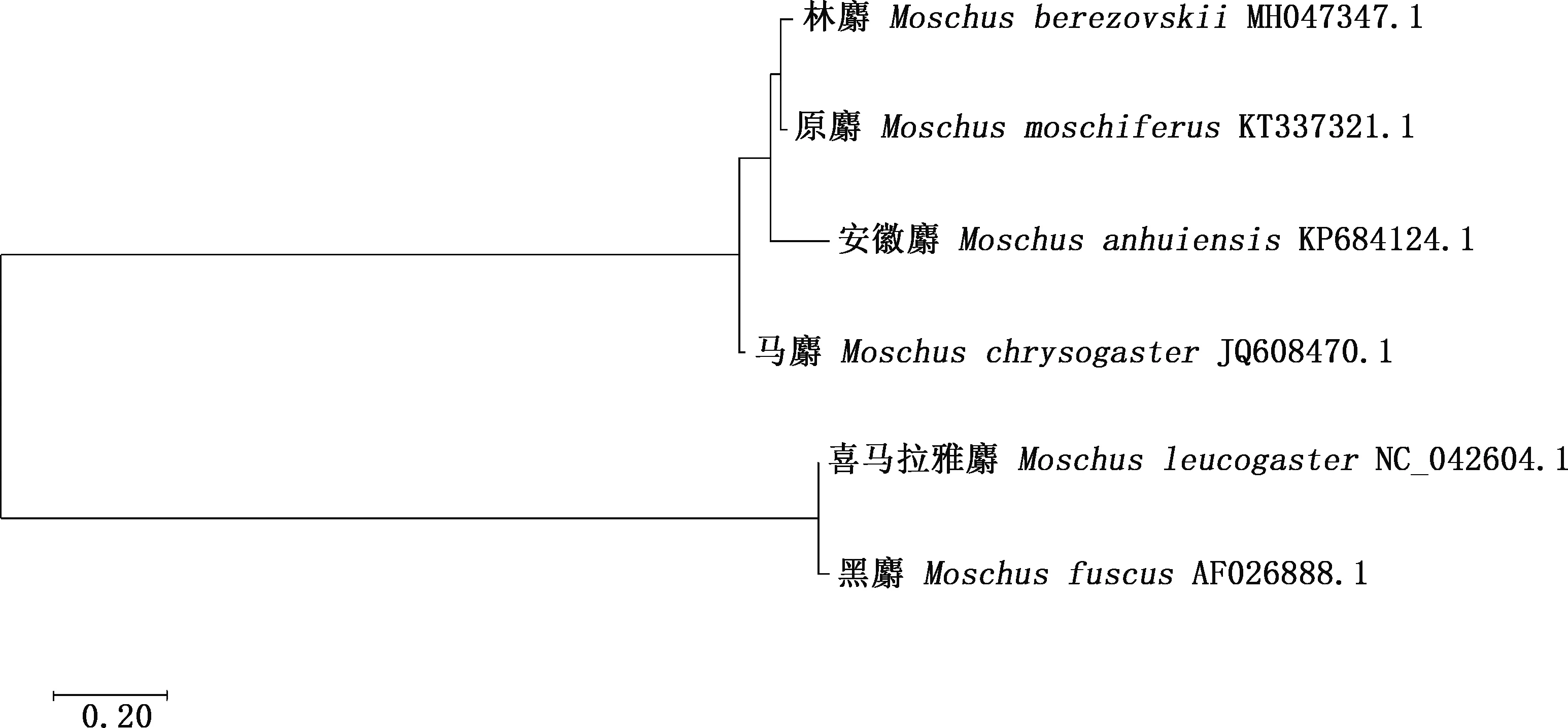
图8 基于D-loop区部分序列构建的系统进化树Fig.8 Phylogenetic tree constructed based on partial sequences of D-loop region sequence
3 讨 论
3.1 基于Cytb基因和D-loop区序列林麝的遗传多态性研究
本试验采用林麝自然脱落的毛发样本,避免对珍稀物种造成人为损伤。经过序列分析,本研究获得部分Cytb基因和D-loop区序列各43条。Cytb基因长度为472 bp,共检出241个SNPs位点,包含236个简约信息位点和5个单一信息位点,没有插入和缺失位点。 D-loop区序列长度为669~711 bp,其中10条长度为699 bp,占比23.2%;6条长度为696 bp,占比14.0%;长度为700和702 bp各5条,分别占比11.6%;长度为697、698及701 bp各4条,分别占比9.3%;3条长度为695 bp,占比7.0%;长度为669及694 bp各1条,占比2.3%。与冯慧等[13]对野生林麝D-loop区研究不同的是,本试验圈养林麝D-loop区序列共检出383个SNPs位点,包含347个简约信息位点和36个单一信息位点,均大于野生林麝,但样本时间与来源方式不同,需进一步采集野生和人工圈养林麝样本进行比较。
单倍型多样度和核苷酸多样度是衡量一个群体mtDNA变异程度的2个指标。单倍型多样度是指样本中随机抽取到2个不同单倍型的频率;核苷酸多样度是指序列间每个位点的平均核苷酸差异数。Grant等[27]研究显示,单倍型多样性以0.5为临界值,核苷酸多样性以0.005为临界值,二者的值越大,群体的多样性程度越高。本试验样本Cytb基因和D-loop区序列单倍型多样度分别为0.983和0.975,核苷酸多样度分别为0.28343和0.07707。单倍型多样度和核苷酸多样度都较大,群体的多态程度较高,遗传多样性较丰富。Hong等[28]认为,当核苷酸多样度在0.0015~0.0047时,群体的遗传多样性较低。本研究中,林麝Cytb基因和D-loop区序列的核苷酸多样度均>0.0047。藏羚羊的线粒体核苷酸多样度为0.0296[29],太平洋白鳍豚的线粒体核苷酸多样度为0.0165[30],同为珍稀哺乳动物,林麝的核苷酸多样度较高,再次验证其核苷酸多样度处于中上水平。整体来看,林麝种群遗传多样性呈现出高单倍型多样性和高核苷酸多样性特征。
3.2 基于Cytb基因和D-loop区序列林麝不同单倍型的遗传距离及系统进化树分析
单倍型间的平均遗传距离可以体现一个群体的遗传变异程度,即大多数哺乳动物的平均遗传距离值在0.01以上就可以认为这个群体变异程度大[28]。本试验结果显示,Cytb基因和D-loop区序列平均遗传距离分别为0.389和0.212,均>0.01,说明林麝群体的遗传变异较大。Cytb基因单倍型之间的最大遗传距离是0.831,远远小于D-loop区单倍型之间最大距离(1.342),这是由于线粒体不同区域的进化速率存在差异,Cytb基因作为线粒体编码基因,其进化速度相对较慢,D-loop区序列进化速率较快。
中国麝类有6种,其中,原麝分布在东北、华北地区;马麝分布在青藏高原及邻近各省;黑麝见于西藏和云南的少数地区;喜马拉雅麝几乎只分布在西藏部分地区;林麝数量多,分布在长江流域及以南各省区[31]。冯慧等[13]研究发现,原麝和林麝最先聚到一起,亲缘关系较近,与马麝、黑麝和喜马拉雅麝亲缘关系较远,本试验研究结果与其一致,同时增加了安徽麝这一麝属。安徽麝仅见于安徽西部大别山地区,是一种特有种。本试验结果显示,安徽麝与林麝、原麝亲缘关系较远,与马麝亲缘关系较近,符合章敬旗等[32]根据麝头骨数据所做的分析。基于Cytb基因构建的系统进化树中,林麝和原麝先聚在一起,然后是安徽麝,与马麝、黑麝和喜马拉雅麝相距较远;而基于D-loop区序列构建的系统进化树中,林麝和原麝最先聚在一起,然后是安徽麝与马麝,这一结果也与D-loop区序列变异速度较快、样本数量少有关;但相同的是,黑麝与喜马拉雅麝亲缘关系较近,为一个小分支,原麝和林麝为一个小分支,充分验证且丰富了6种麝属之间的亲缘关系,但由于采集林麝样本范围较小,还需更多样本进一步验证。通过对Cytb基因和D-loop区序列测定分析,在2个系统进化树中均可以发现林麝具有2个母系来源,这与冯慧等[13]报道的其他中国林麝群体研究结果一致。因此,要加强对现有林麝资源的保护力度,对人工圈养林麝进行科学管理,改善养殖环境,防止其遗传结构发生变化,从而有利于遗传资源的可持续发展。
4 结 论
本研究对陕西林麝毛发样本Cytb基因和D-loop区序列进行分析,发现陕西林麝群体呈现出高单倍型多样性和高核苷酸多样性特征,表现为较丰富的遗传多样性;进化上存在2个母系起源,同时进一步支持了林麝和原麝属于一个分支的观点。
