基于级联Dense-UNet和图割的肝脏肿瘤自动分割
2022-05-31邸拴虎赵于前曾业战
杨 振 邸拴虎 赵于前*③ 廖 苗④ 曾业战
①(中南大学自动化学院 长沙 410083)
②(中南大学湘雅医院肿瘤科 长沙 410083)
③(湖南湘江人工智能学院 长沙 410083)
④(湖南科技大学计算机科学与工程学院 湘潭 411100)
⑤(湖南工业大学电气与信息工程学院 株洲 412007)
1 引言
肝癌是全球范围内最常见的癌症之一,其致死率在所有癌症类型中排第2。据统计,2020年我国肝癌新发和死亡病例分别占全球肝癌新发和死亡病例的45.27%和47.11%[1]。肝癌的早发现、早诊断和早治疗对于提高患者生存率和生活质量起着至关重要的作用。在临床上,计算机断层扫描(Computed Tomography, CT)因其信噪比高、空间分辨率高和成像速度快等优点被广泛应用于肝脏肿瘤的诊断[2]。从CT图像中准确分割肿瘤组织是癌症治疗过程中的重要环节。医生可以通过评估肿瘤的大小、形状和位置,为患者制定合适的治疗方案。目前,在临床实践中,肿瘤组织的分割通常由放射科专家手动勾画完成,此过程非常繁琐、耗时且分割结果高度依赖操作者的经验和技巧。运用图像处理、人工智能等技术,实现腹部CT图像肝脏肿瘤的自动或半自动分割已成为当前学者研究的热点。
CT图像中,肝脏肿瘤通常具有灰度多样、形状不规则、与毗邻组织对比度低、边界模糊等特点,这些均给肿瘤的自动准确分割带来了很大困难。现有的肝脏肿瘤分割方法,主要分为阈值、区域生长、聚类、活动轮廓模型、图割、机器学习和深度学习等。阈值、区域生长和聚类具有易于实现、时间和空间效率高等优点,是医学图像分割中常用的方法。这类方法的主要缺点是仅利用图像灰度信息进行分割,易在肿瘤边界模糊的位置出现过分割,以及在灰度不均的肿瘤区域出现欠分割。活动轮廓模型和图割法在考虑图像灰度信息的情况下,引入了目标的形状信息,可有效提升分割精度[3,4]。
为了实现肝脏肿瘤的全自动分割,不少文献提出了基于机器学习的方法。Kadoury等人[5]提出了一种基于条件随机场和格拉斯曼流形(Grassmannian manifolds)的肝脏肿瘤自动分割方法,该方法首先在训练阶段学习肝脏和肝脏肿瘤组织的非线性灰度分布,然后采用高阶条件随机场对肝脏区域的肿瘤组织进行分割。Foruzan等人[6]首先运用支持向量机(Support Vector Machine, SVM)和分散数据逼近算法获取初始肿瘤边界,然后采用sigmoid边界模型对初始分割结果进行优化。基于机器学习的分割方法虽然可以实现肝脏肿瘤的自动分割,但该类方法通常需要人工挑选特征,且分割结果的好坏严重依赖特征选取的优劣。
近年来,深度学习以其在自动特征提取、非线性建模上的优势,被广泛应用于医学图像分割领域[7,8]。Li等人[9]利用混合密集连接UNet来分割肝脏和肿瘤。通过提取层内和层间特征,该网络在3DIRCADb测试集上获得的Dice值分别为0.95(肝脏)和0.65(肿瘤)。Jiang等人[10]提出了一种注意力混合连接网络,该网络结合了软硬注意机制、长短跳跃连接和级联网络,在肝脏和肿瘤分割中,获得的Dice值分别为0.95和0.62。深度学习方法依赖高性能的硬件平台和大量标记数据进行相关模型的训练,对于肝脏区域通常可以获得良好的分割效果,但对于灰度多样、形状不规则、边界模糊的肿瘤区域,其分割效果往往较差。
为了自动、精确分割CT图像中的肝脏肿瘤区域,本文提出一种基于级联Dense-UNet和图割的分割方法,该方法主要包括肿瘤感兴趣区域提取和肿瘤组织精确分割两部分。首先运用基于级联的Dense-UNet网络自动获取CT图像中肝脏肿瘤初始分割结果及包含肿瘤组织的感兴趣区域,然后根据初始分割结果,构建基于像素级特征的灰度模型和基于区域级特征的概率模型,并将其融入图割算法进一步精确分割感兴趣区域中的肿瘤组织。
本文方法主要具有以下优点与创新:(1)将Dense-Net思想融入UNet网络结构,提出一种Dense-UNet网络模型,并以级联的方式依次分割肝脏和肿瘤区域,实现肿瘤初始分割及感兴趣区域自动准确提取;(2)分别从像素级和区域级特征出发,建立可有效区分肿瘤与非肿瘤的灰度模型和概率模型,并将其融入图割能量函数的构建,达到准确识别肿瘤模糊边界,提高肿瘤分割精度的目的;(3)提出方法通过结合Dense-UNet和图割,可实现CT图像中肝脏肿瘤的自动精确分割,尤其对于低对比度、边界模糊的肿瘤,分割精度明显优于现有多种自动分割方法。
2 方法描述
2.1 基于级联Dense-UNet的肝脏肿瘤感兴趣区域提取
由于肝脏肿瘤通常具有灰度多样、边界模糊、形状不规则、位置不固定等特点,直接采用端到端的网络模型很难实现肿瘤组织的准确识别。针对该问题,本文提出一种Dense-UNet网络模型,并采用级联的方式自动获取CT图像中肝脏肿瘤初步分割结果及感兴趣区域。Dense-UNet是基于Dense-Net[11]和U-Net[12]的语义分割模型,整体结构类似于U-Net,包含了收缩路径、扩张路径和跳跃连接3部分。收缩路径通过卷积结构和池化结构来获取图像上下文信息;扩张路径利用上采样结构和卷积结构来实现肿瘤的精确定位;跳跃连接将底层信息和高层信息进行拼接,实现高维特征与低维特征的融合。与U-Net结构不同的是,Dense-UNet将DenseNet模型引入到U-Net网络中,这样将更有利于梯度的反向传播,使得网络更容易训练。
本文提出的Dense-UNet网络结构如图1所示,其中Dense Conv为卷积结构,Pool为池化结构,Up Sample为上采样结构。在卷积结构中,首先使用两个连续的卷积层提取特征图,然后将得到的两个特征图进行拼接,并通过重复卷积和拼接操作,充分提取图像特征;在池化结构中,分别采用池化和卷积操作降低特征图的尺寸,并将池化后的特征图与卷积后的特征图进行拼接,扩大网络感受野;在上采样结构中,采用反卷积扩充图像尺寸的同时降低通道数量,并将扩充后的特征图与收缩路径中相同尺寸的特征图进行拼接,获取更好的特征重建效果。

图1 Dense-UNet网络结构
在基于级联Dense-UNet的分割框架中,首先运用Dense-UNet分割CT图像中的肝脏区域,然后再次利用Dense-UNet从肝脏区域中提取肿瘤组织,如图2所示。在肝脏区域分割中,将CT原始图像和相应的肝脏手动分割结果作为训练数据进行网络训练。而在肿瘤区域分割中,为了减少腹部其他组织或器官的干扰,仅采用肝脏区域和相应的肿瘤手动分割结果进行训练,这样既可降低网络训练难度,又可提高肿瘤分割精度。此外,考虑到由CT图像中目标与背景所占比例差距大导致的类不平衡问题,本文采用Dice系数作为网络训练过程中的损失函数

图2 基于级联Dense-UNet的分割框架

其中,P和G分别为Dense-UNet网络预测和专家手动分割结果,|·|表示目标像素数目。采用Dice损失函数进行网络训练的本质是最大化网络预测结果的Dice系数,这与分割的真实目标一致。因此,采用Dice损失函数进行网络训练,可以使网络获得良好的分割效果。
图3(a)—图3(c)分别为从某一患者CT序列中随机挑选的第51, 80和109个切片的肝脏肿瘤感兴趣区域提取结果,其中红色曲线所示区域即为采用级联Dense-UNet模型获取的肝脏肿瘤初步分割结果,取初步分割结果的最小外接矩形即可获取肝脏肿瘤感兴趣区域,为了确保感兴趣区域包含完整的肿瘤组织,将外接矩形框的边缘适当向外延伸一定距离,本文取20像素,得到的肿瘤感兴趣区域如图3中蓝色方框所示。从图中可以看出,采用级联Dense-UNet模型虽然可以大致定位肿瘤区域,但却无法精确获取肿瘤边缘,尤其对于对比度低、边界模糊的肿瘤。

图3 从某一序列中随机挑选的CT切片肝脏肿瘤感兴趣区域提取结果示例
2.2 基于图割的肝脏肿瘤精确分割
为了精确提取感兴趣区域中的肿瘤组织,首先根据肿瘤初始分割结果,利用图像像素级和区域级特征,在感兴趣区域中分别构建可有效区分肿瘤与非肿瘤的灰度模型和概率模型,然后将其应用于图割算法的能量函数构建,并通过最小化能量函数实现肿瘤组织的自动精确分割。
2.2.1 基于像素级特征的灰度模型
单一组织或器官由于具有近似的密度,其在CT成像中的灰度范围往往较窄且呈正态分布[13]。假定肿瘤区域的灰度范围为[Imin, Imax],则基于像素级特征的灰度模型可构建为
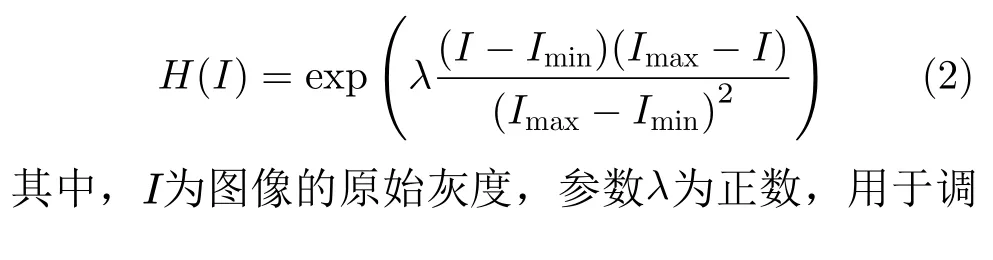


图4 原始肿瘤感兴趣区域示例

图5 灰度模型结果示例
2.2.2 基于区域级特征的概率模型
为了进一步区分肿瘤与非肿瘤,利用图像局部区域特征构建肝脏肿瘤概率模型,具体包括以下步骤:

2.2.3 图割分割
结合图像灰度和概率模型,利用图割算法精确分割感兴趣区域中的肝脏肿瘤组织。首先,在肿瘤


图6 概率模型结果示例

图7 图的构建示意图

3 实验结果与分析
3.1 实验数据及平台
本文采用LiTS[14]公共数据库作为Dense-UNet网络的训练集,采用3Dircadb[15]数据库作为测试集。LiTS是肝脏肿瘤分割挑战赛使用的数据集,共收集了131个来自不同医院和不同研究机构的腹部CT序列图像,其中118个序列的肝脏区域存在不同程度的肿瘤病变。CT序列切片平面像素数目为512×512,平面像素间距范围为0.55~1.0 mm,层间距为0.45~6.00 mm。3Dircadb数据库提供了来自欧洲不同医院的10位男性和10位女性患者静脉增强期的腹部CT序列图像,其中15个序列包含肝脏肿瘤,CT切片平面像素数目为512×512,平面像素间距范围为0.56~0.87 mm,层间距为1.0~4.0 mm。
实验硬件环境为Intel 酷睿i7 6700K,32 GB内存,NVIDIA GTX2080TI GPU,系统为Windows 7。Dense-UNet网络在Pytorch开源框架下实现,采用RMSprob优化器,学习率为0.001,动量系数为0.9。图割算法采用C++/Matlab混合编程。

图8 图割分割结果示例
3.2 评价指标
为了定量评价算法分割性能,采用Dice系数、精准率(Precision)和召回率(Recall)作为评价标准,具体定义为

其中,TP表示正确分割的肿瘤像素数目,FP和FN分别表示将背景误分割为肿瘤,以及将肿瘤误分割为背景的像素数目。与Dice系数相同,精准率与召回率的取值越高,表明算法的分割结果越接近金标准,算法分割性能越好。
3.3 损失函数比较与分析
考虑到肝脏及肿瘤在整个CT图像中占比较小,本文采用Dice损失函数进行网络训练。为了验证Dice损失函数的有效性,本文将其与均方损失函数进行了比较。均方损失函数计算公式为

其中,pk和gk分别为CT图像中第k个像素的Dense-UNet网络预测和专家手动分割结果,N为CT图像中的像素数目。
在级联Dense-UNet网络训练过程中,将LiTS数据集按5:1随机划分为训练集和验证集,分别采用Dice和均方损失函数作为目标函数,得到的训练损失值和验证准确率如图9—图10所示。均方损失函数计算图像中所有像素预测结果与金标准之间的均方差,对于背景区域占比较大的CT图像,其背景区域的预测对训练损失值影响较大,仅当背景区域被正确预测时,训练网络即可获得较小的损失值,而此时得到的网络模型通常难以准确预测目标区域。从图9—图10可以看到,采用均方损失函数训练肝脏及肿瘤分割网络时,随训练轮数增加,训练损失值均快速下降并收敛至0附近,但肝脏和肿瘤的验证准确率均未见明显提升,其值分别在0.18及 0.05附近波动。而Dice损失函数以最大化Dice系数为目标,只有当网络同时正确预测图像中的目标与背景时,训练损失值才会明显下降。从图中可以看到,采用Dice损失函数训练级联网络时,随训练轮数增加,训练损失值均显著下降,同时肝脏及肿瘤的验证准确率逐渐上升并分别收敛至0.94和0.70左右,表明即使肝脏及肿瘤在CT图像中占比较小,采用Dice损失函数,网络也可有效学习图像特征,实现肝脏及肿瘤的准确分割。

图9 肝脏分割网络训练过程

图10 肿瘤分割网络训练过程
3.4 结果与分析
3.4.1 分割结果定性与定量分析
图11给出了3Dircadb公共数据库部分切片的实验结果,其中图11(a)列为原始CT图像,图11(b)和图11(c)列中的绿色区域分别为运用级联的Dense-UNet(DU)和本文方法(DU+GC)得到的肝脏肿瘤分割结果,其中红色封闭曲线所示区域为专家手动分割结果。DU方法虽然能大致分割CT图像中的肿瘤区域,但由于不同切片中肿瘤灰度、形状、尺寸等特征差异较大,该方法难以获取精确的肿瘤边界,尤其在肿瘤边界较为模糊的区域容易出现欠分割或过分割。而本文方法(DU+GC)根据DU初始分割结果,构建可有效区分肿瘤与非肿瘤的灰度和概率模型,并将其用于图割能量函数构建,可从感兴趣区域中自动精确分割肿瘤。从图中可以看到,相较于DU,本文方法得到的结果更加贴近于专家手动分割结果。

图11 3Dircadb数据库部分切片分割结果示例
采用DU和本文方法(DU+GC)对3Dircadb公共数据库提供的15个包含肝脏肿瘤的腹部CT序列进行测试,并运用Dice, Precision和Recall 3个指标对分割结果进行评价,得到的均值±标准差如表1所示。可以看到,采用本文方法得到的所有评价指标均优于DU方法,尤其是Dice和Precision分别高出8%和5%,表明提出方法通过结合深度学习与图割,可有效提升肝脏肿瘤分割的精度。
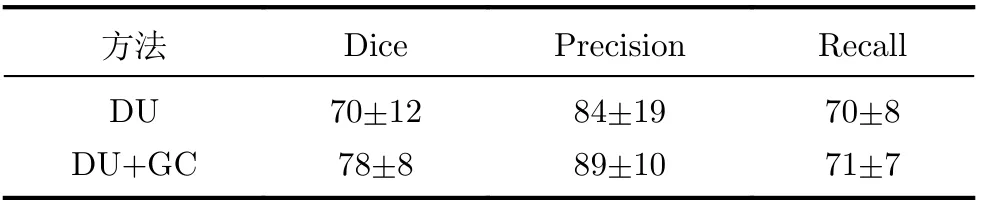
表1 本文方法(DU+GC)与DU的分割性能比较(均值±标准差)(%)
3.4.2 不同标注结果对肿瘤分割的影响
本文方法属于有监督学习方法,为了分析不同标注对分割结果的影响,本文分别采用半径r为2和4的圆形结构元素,对训练集中的肿瘤手动分割金标准进行膨胀和腐蚀,模拟人工标注过程中可能出现的过分割和欠分割现象,将处理后的分割结果输入网络进行训练,获得权重参数不同的网络模型,并结合图割算法对图像中的肿瘤区域进行分割。图12给出了采用不同标注结果进行网络训练得到的肿瘤分割结果,其中绿色曲线所示区域为运用级联Dense-UNet(DU)得到的肝脏肿瘤初步分割结果,蓝色曲线为采用DU+GC方法得到的最终分割结果,红色曲线为3Dircadb数据库给出的专家手动分割金标准。为了清晰显示分割结果,将图中青色和黄色方框所示肿瘤局部区域进行了放大显示。由于级联Dense-UNet以标注结果为学习目标,其分割结果容易受标注结果的影响,当标注结果存在过分割或欠分割时,采用级联Dense-UNet得到的肝脏肿瘤则会出现相应的过分割或欠分割现象。本文方法(DU+GC)将级联Dense-UNet预测结果作为肿瘤初始分割结果,利用图像像素级和区域级特征构建灰度和概率模型,并将其融入图割能量函数实现肿瘤区域的精确分割。从图12可以看出,本文方法通过结合图割算法可有效修正网络模型的分割结果,解决不同标注导致的过分割和欠分割问题,获得精确的肿瘤分割结果。

图12 采用不同标注进行训练得到的肿瘤分割结果
3.4.3 不同方法分割结果比较
为了进一步证实本文方法的有效性,将其与其他多种深度学习方法进行比较,包括 UNet[12],FCNs[16], DeepLabv3+[17], UNet 3+[18]和TransUNet[19]。这些网络均为目前医学图像分割领域应用最广泛、性能最好的深度学习网络。图13为采用不同方法得到的肝脏肿瘤分割结果,其中(a)列为原始图像,图13(b)–图13(g)列分别为采用UNet, FCNs,DeepLabv3+, UNet 3+和TransUNet和本文方法得到的肿瘤分割结果。图中绿色区域为算法的分割结果,红色封闭曲线所示区域为专家手动分割结果。可以看到,虽然UNet, FCNs, DeepLabv3+,UNet 3+和TransUNet均可大致识别CT图像中的肝脏肿瘤,但却无法获得精确的肿瘤边缘,尤其对于灰度异质、对比度低、边界模糊的肿瘤易出现过分割或欠分割。本文方法通过结合Dense-UNet和图割,可有效分割CT图像中不同大小、形状和位置的肝脏肿瘤,分割精度明显优于其他方法。表2为不同方法在3Dircadb公共数据库上的分割性能比较。从表中可以看到,本文方法可获得较高的Dice系数、准确率和召回率均值,表明提出方法的肝脏肿瘤分割精度明显优于其他方法,同时较低的标准差,表明所提方法具有较强的鲁棒性。
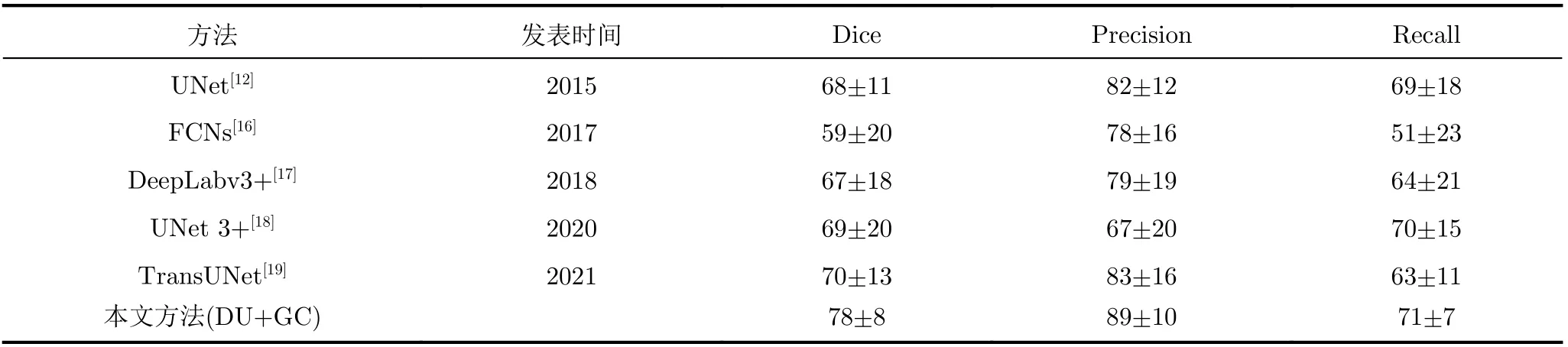
表2 本文方法与其他现有方法的分割性能比较(均值±标准差)(%)

图13 不同方法分割结果比较
4 结束语
本文针对腹部CT图像肝脏肿瘤灰度异质、形状多样、边界模糊等特点,提出了一种基于级联Dense-UNet和图割的肝脏肿瘤自动分割方法。该方法采用由粗到精的分割策略,首先运用基于级联Dense-UNet的深度学习网络模型获取肝脏肿瘤初始分割结果及包含肿瘤组织的感兴趣区域,该网络模型融合了DenseNet与UNet的优点,通过采用级联方式依次分割肝脏和肿瘤,可有效避免不相关器官与组织对于肿瘤分割的干扰、提高肿瘤识别的准确性;然后,根据初始分割结果,分别利用图像像素级及区域级特征,构建可有效区分肿瘤与毗邻组织的灰度模型和概率模型,并将其融入图割算法的能量函数构建,通过最小化能量函数实现感兴趣区域中肿瘤组织的精确分割。提出方法不需要任何的人工干预,可自动精确鲁棒分割腹部CT图像中边界模糊、对比度低,以及大小、形状、位置多样的肝脏肿瘤。为验证方法的有效性,本文采用LiTS作为训练集,并在3Dircadb公共数据集上进行了测试,得到的Dice系数、精准率与召回率的均值±标准差分别为78%±8%,89%±10%,71%±7%,明显高于其他多种自动分割方法,表明提出方法可自动有效分割CT图像中的肝脏肿瘤,准确性高、鲁棒性强。
