合成生物学与荧光成像技术
2022-05-14武伟红李炜张先恩崔宗强
武伟红,李炜,张先恩,崔宗强
(1 中国科学技术大学生命科学与医学部,安徽 合肥 230026; 2 中国科学院武汉病毒研究所,病毒学国家重点实验室,湖北 武汉 430071; 3 中国科学院生物物理研究所,生物大分子国家重点实验室,北京 100101)

合成生物学作为新兴领域,自概念诞生以来,多个国家、机构和国际组织将其评价为未来的颠覆性技术[1-2]。合成生物学技术是基于生物反应本质对生物组件进行改造,或者从头设计并构建出具有生物活性和特定生理功能的生物元件[3-7]。利用合成生物学技术开发荧光材料,发展生物分子定点标记方法[8-9],对细胞内特定生物分子进行高灵敏高分辨成像与示踪研究,能够直观精准地揭示细胞内关键分子事件及机制[10-11]。传统获得荧光材料的方法依赖于化学合成,常需要高温、高压、电离、有毒溶剂等条件,存在成本高、耗材大、环境污染等问题。而且,化学合成的外源荧光材料用于生物分子标记与成像,常存在生物相容性差、标记效率低、难以精准控制等问题[12-13]。而通过合成生物学构建细胞工厂,直接在细胞内合成特定荧光材料或探针[14-15],不仅可以有效避免传统合成方法中的剧烈条件和使用易燃易爆原料,还可以赋予这些材料或探针优越的生物相容性和稳定性[16-18]。通过合成生物学技术,也可以高效、精准和灵活地对细胞进行基因改造,对蛋白质[19]或核酸进行定点定量特异性标记[20]。基于合成生物学发展荧光探针与标记技术,结合高灵敏、高分辨成像和实时示踪[21]等手段,高效、精准成像细胞和活体内生物分子,深入解析生命活动过程,实现对机体的生理状态检测和对疾病的预防诊断和治疗[13,22-23]。本文将主要总结近年来合成生物学技术在细胞生物荧光成像方面的应用,并对该领域关键科学问题进行探讨和展望。
1 利用合成生物学技术合成新型荧光材料
针对不同的成像目标与模型,基于合成生物学技术,建立材料生物合成新方法[24],可以开发性能优良的荧光材料和探针。荧光成像最常见的荧光材料包括有机荧光染料分子、荧光蛋白和荧光纳米材料等[25]。其中,荧光纳米材料如量子点等具有优异的光学性能[26-27],在生物分子标记与成像中越来越广泛被应用[28-31]。合成生物学与纳米生物技术交叉融合,可以在活细胞中原位合成新型荧光纳米材料[32-34]。这些生物合成的新型荧光纳米材料具有稳定性高、生物相容性好的特点,易于进行后续生物修饰和标记。与生物矿化过程不同,生物合成荧光纳米材料是利用特定的生化反应途径得到的中间产物为制备无机晶体所需的前体,转运至特定的空间,实现无机纳米晶体的成核、生长及自组装[35]。活细胞合成纳米杂合生物活性材料是利用活细胞作为反应器:将细胞内高效专一的生物化学反应过程应用于纳米杂合生物活性材料的合成,将不同途径的合成反应过程在细胞进行操作耦合,使得细胞中不同时空的反应通过精确的调控网络按照设计目的进行。例如:趋磁细菌[36-38]利用细胞中的Fe3+部分还原形成Fe2+,并在铁蛋白中快速生成多种晶型的磁性纳米颗粒,用于趋磁细菌的核磁共振活体示踪成像和磁热治疗。
量子点作为一种荧光纳米材料,在可视化荧光成像研究中的优势尤为突出[39-41]。量子点具有宽的激发光谱和窄的发射光谱,且发射光谱根据粒径大小可控。相对于有机染料分子和荧光蛋白,量子点具有光稳定性好、不易漂白、表面可以进行各种特异性的化学修饰和偶联等优点。但是,化学合成的量子点往往生物相容性较差[42-44],鉴于此,Cui 等[45]在酵母内可控合成不同发光性能的CdSe 量子点(图1):其利用在活细胞反应器中或通过耦合不同的生物化学反应途径构建准生物体系,发展出“时-空耦合”调控新策略,从而合成纳米材料,并通过置换参与CdSe 量子点合成的关键基因的启动子,实现了可在活细胞中对CdSe 量子点的合成产率进行调节。但存在的缺点是在其合成过程包含细胞洗脱、破碎、碎片去除等一系列的操作,过程烦琐复杂。2012 年,该团队[46]基于准生物合成Ag2Se量子点并用于裸鼠近红外活体成像。Ag2Se 量子点合成关键步骤在于还原性硒前体和银前体的制备。研究人员用高效液相色谱-质谱联用技术(HPLC-MS)[47-49]监测还原性硒前体的设计过程,通过耦合Na2SeO3还原与丙氨酸和银离子结合两个生化反应过程,构建准生物反应体系完成水相合成Ag2Se的过程。这种合成策略避免了传统的高温、高压过程,无需由有机相到水相转移即可应用于生物成像。实验验证了Ag2Se光稳定性好,在体内近红外荧光穿透性可减少组织和血液自身荧光的影响,与传统量子点相比对细胞毒性更小。这种新型小尺寸的荧光纳米材料在生物成像方面具有良好的应用前景。但目前,对于量子点生物与准生物合成,特别是对于小尺寸的近红外量子点,其尺寸的精确控制及修饰可操作性还比较低。进一步对生物合成量子点的核形成与生长过程的控制以及对其表面进行修饰研究,将更有利于其在生物成像中的应用。

图1 CdSe在酵母细胞中的可控合成[45]Fig.1 Controllable CdSe synthesis in yeast cells[45]
此外,Höckner 等[50]将蚯蚓置于含CdCl2和Na2TeO3的土壤环境中,利用蚯蚓对重金属的解毒能力生产出可产生绿色荧光的量子点。蚯蚓通过金属依赖性硫蛋白,将重金属转移到嗜氯细胞组织合成纳米晶体CdTe量子点,并在CdTe量子点表面添加聚乙二醇可进一步应用于巨噬细胞成像(图2)。利用蚯蚓作为反应器在嗜氯细胞中合成CdTe 存在的问题是形成的量子点的成像存在自荧光干扰,在不同种类蚯蚓中背景干扰程度不同,并且生长形成的CdTe 量子点粒径大小单一不可调控。Sweeney 等[51]用含氯化铬和硫化钠的培养基培育大肠杆菌可高效生产出CdS 纳米晶体。该团队证明纳米晶体的合成是在细菌内部,CdS纳米晶体主要在静止期开始合成,在对数生长期前中期形成大量的CdS 纳米晶体沉淀,而非在外部合成之后运输到细菌内部,表明了大肠杆菌具有诱导纳米晶体合成的能力,这对于研究细菌生长阶段对晶体合成的可控调节具有重要作用。其不足是CdS纳米晶体合成过程与沉淀积累对于细菌的生长阶段具有极高依赖性。Bao 等[52]也通过大肠杆菌成功合成CdTe 量子点。与前述生物合成方法不同的是,该CdTe 量子点的合成并不直接依赖于细菌本身,而是通过细菌分泌蛋白辅助合成,分泌蛋白与可溶解的纳米晶体核结合,进一步促进小的纳米晶体溶解,之后结合蛋白可以与更大的纳米晶体结合,将CdTe 成分传递从而促进了纳米晶体的成长。这种环境友好的细菌胞外合成方法可以在不需要细胞洗涤与破碎的前提下合成出光稳定性较好的量子,可应用于体外细胞成像。

图2 在蚯蚓细胞中CdTe的生物合成[50]Fig.2 CdTe biosynthesis in earthworm cells[50]
这些纳米材料的合成新策略能够提供更多的机会调控纳米材料的结构,实现对纳米材料性能的控制。目前利用活细胞及准生物体系的“时-空耦合”调控策略,除了可以合成CdSe[53-54]、CdTe[55]、CdS[56-57]量子点之外,还可控合成出生物相容性好的金纳米粒子、金团簇、近红外量子点等荧光纳
米材料,并且将此原理扩展已成功构建了由酶、辅酶、氨基酸、肽等组成的“准生物体系”[58]。通过该方法得到的近红外Ⅱ区[59-61]Ag2S[62-63]、Ag2Se和AgAuSe 量子点[64-65],具有低毒性、耐光漂白、荧光穿透深等特点,可用于对病毒感染过程的活体成像等研究。此外,相较于传统的修饰方法,活细胞内原位合成纳米杂合材料的方法依赖于特定的生物学过程,可以通过基因技术、生化反应等对活性材料的合成进行精准调控。如Park 等[66]利用基因编辑技术将其他物种的植物螯合肽合成酶和金属硫蛋白的基因引入大肠杆菌,构建了合成金属纳米颗粒的微生物工厂,将各种金属与大肠杆菌混合培养,合成了多种量子点和其他金属纳米颗粒(图3),并且通过改变金属离子浓度能够对纳米颗粒大小进行可控调节。当然,虽然已在重组微生物体内合成了多种金属纳米颗粒,但是微生物合成金属钠米粒子的机制仍有待深入解析。

图3 大肠杆菌细胞中合成CdSeZn、PrGd纳米晶体[66]Fig.3 CdSeZn and PrGd nanocrystals synthesized in E.coil cells[66]
2 合成生物学与生物分子标记技术
胞内活性材料的合成和后续的荧光成像过程都需要生物分子修饰,利用合成生物学技术,可以对特定靶标分子进行高效、特异或多重标记,为分子标记提供新的技术和方法。本文将主要介绍蛋白和核酸分子的荧光标记。
2.1 合成生物学应用于蛋白分子标记
荧光分子或荧光纳米材料需要通过特定策略与目标蛋白分子进行链接,从而实现荧光标记与成像。这些策略中,链接过程主要采用:荧光非天然氨基酸的掺入而合成荧光基团、自标记酶衍生物与荧光基团结合后与目标蛋白融合、利用外源性酶催化小肽连接荧光基团与目标蛋白融合等[67-69]。非天然氨基酸可以将不同功能基团通过遗传密码扩展而引入到需要标记蛋白的特定位置[70]。例如荧光氨基酸:2-氨基酸-3-[5-(二甲氨基)萘-1-磺酰胺]-丙酸,可以利用琥珀色标签密码子和相应的正交tRNA/氨基酰基tRNA 合成酶对基因进行编码。Summerer等[71]将这种荧光氨基酸用来标记超氧化物歧化酶,用于观察在盐酸胍存在下的蛋白质折叠过程。这类荧光氨基酸可以直接用于配体-抗体结合的相互作用,酶催化或调控中涉及的蛋白质构象变化,以及蛋白质翻译后修饰等研究。此外有可能将这种方法扩展到荧光量子产率更高或发射波长更长的氨基酸,并将这种方法直接应用于细胞成像等。病毒蛋白的荧光标记也是合成生物学在蛋白分子标记技术方面的重要组成部分。AP标签是一段由15个氨基酸GLNDIFEAQKIEWHE组成的短肽,它能够被生物素连接酶(BirA)催化发生生物素修饰。Li 等[72]利用链霉亲和素与生物素间的特异性相互作用将链酶亲和素偶联的QD特异性地结合到HIV-1 病毒的Vpr 上,实现了Vpr在胞内的量子点偶联,获得了病毒锥形核心内包装量子点的艾滋病毒颗粒。进一步,他们还建立了通过细胞辅助的病毒包装量子点的新技术[73],将基质蛋白与BAP 标签融合,用量子点对病毒基质蛋白MA 进行了荧光标记。利用量子点的高亮度、耐漂白等优点(见表1中几种不同荧光材料的性能参数比较),可以实现对单分子或单个病毒进行标记和长时间实时动态示踪。2019年,Qin等[77]利用量子点纳米材料,结合合成生物学技术,设计合成了含有特异识别病毒基因组或者病毒外壳蛋白标签的荧光量子点,通过生物素链霉素亲和素反应,对病毒颗粒的遗传物质或者蛋白外壳进行定点改造,获得了量子点标记的多色流感病毒颗粒,用于解析单个流感病毒颗粒IAV的脱壳过程。

表1 不同荧光材料性能参数[74-76]Tab.1 Properties of different fluorescent materials[74-76]
基于合成生物学技术,可以对荧光蛋白进行拆分改造,构建新型荧光蛋白分子标记技术:如荧光互补系统[78-79]。该系统是对荧光蛋白分子在特定位点进行切割,之后产生2 个或3 个不能发射荧光的蛋白片段,将此蛋白分子片段分别与不同的目标分子连接,如果目标分子间能够发生相互作用,那么蛋白-蛋白片段在空间上互补发出荧光。该系统可以在细胞和活体水平研究蛋白-蛋白、蛋白-核酸的相互作用过程,包括蛋白质寡聚化过程、G 蛋白偶联受体与信号转导以及病毒-宿主的相互作用过程等[80]。Outeiro 等[81]利用荧光蛋白GFP,拆分构建了双分子荧光互补系统,利用该系统他们研究并发现Hsp70蛋白可以降低α-synuclein寡聚物产生的细胞毒性。近来,Chen 等[82]用iRFP 构建双分子荧光互补系统用于HIV-1 病毒IN 蛋白和LEDGF/p75 的相互作用。此外,他们还构建了荧光蛋白Venus 的三片段互补(TFFC)系统[83],研究了HIV-1 病毒IN 蛋白、BAF 和LEDGF/p75 的三分子相互作用过程。首先,将可发光的Venus蛋白切割成3个不能发射荧光的片段构建质粒,分别和NFAT1、bJun 和bFos 编码序列相连构建了三分子互补系统。随后,用切割的3个荧光蛋白片段和与HIV-1 病毒侵入细胞整合相关的IN、BAF 和LEDGF/p75 相连接形成三元复合物,通过IN、BAF 和LEDGF/p75 的相互作用可产生黄色荧光在细胞中定位[图4(a)],研究了IN、BAF和LEDGF/p75与宿主染色体相互作用并发现了BAF 与HIV-1的潜伏期有重要关系。Chen等[84]还利用具有光激活和光转换特性的mIrisFP 荧光蛋白,成功构建了三片段荧光互补系统,实现了蛋白-蛋白以及蛋白三聚体亚基间相互作用的超高分辨成像[图4(b)],空间分辨率可精确到40 nm,并且实现了G 蛋白三聚体亚基间相互作用的单分子水平成像,解析了G 蛋白αβγ异源三聚体与βγ异源二聚体在亚衍射细胞空间的不同分布模式,揭示了霍乱毒素导致αs亚基与βγ 异源二聚体亚基解离的动态过程。当然,目前双/三分子荧光互补系统还存在一些缺点有待改进,譬如常需要低温处理、荧光亮度低信号弱、荧光互补难以实时反映分子间相互作用的动态变化等。

图4 Venus-TFFC系统与mIrisFP-TFFC系统成像[83-84]Fig.4 Schematic diagram for imaging of Venus-TFFC(a)and mIrisFP-TFFC systems(b)[83-84]
2.2 合成生物学应用于核酸分子标记
2012 年,Paige 等[85]开 发 了 名 为“Spinach”的RNA-荧光基团复合物用于示踪细胞内的各种RNA。Spinach 是一类可以与GFP 蛋白媲美的新型荧光工具,它是根据荧光蛋白的形状合成化学分子,通过插入人工RNA 序列构建出发射不同荧光波长的“RNA-荧光基团”复合物。该荧光基团复合物具有荧光强度高、特异性强、无背景荧光干扰且无细胞毒性等特点。进一步,Song 等[86]基于Spinach,开发出在结合目标蛋白时可产生荧光的RNA 传感器:将RNA 代谢物与Spinach 结合、折叠,在空间上茎链接近,折叠后的结构与3,5-二氟-4-羟基苯亚甲基咪唑啉酮(DFHBI)结合使DFHBI 产生荧光,这种荧光复合体具有荧光强、可选择性结合靶蛋白的能力,用于成像单个大肠杆菌中蛋白表达能力变化的动态过程。但是DFHBI 的适配子存在光不稳定的问题,会出现光诱导的异构化产生荧光漂白,从而终止荧光反应。该团队[87]在2017 年开发了新型核酸适配子“Corn”可以与3,5-二氟-4-羟基苯亚甲基咪唑啉酮-2-肟(DFHO)结合,并且Corn 具有抑制激发态DFHO 荧光衰变的能力,产生的荧光具有亮度高且光稳定性强的特点,可用于直接测量细胞内核糖核酸聚合酶反应转录速率和调控机制动态变化。Corn 与DFHO 结合的荧光基团还可用于研究mTOR 抑制剂对细胞中RNA 聚合酶Ⅲ的抑制作用(图5)。但需要注意的是,如果DFHO 浓度过高,会出现未结合的DFHO产生背景荧光干扰现象。
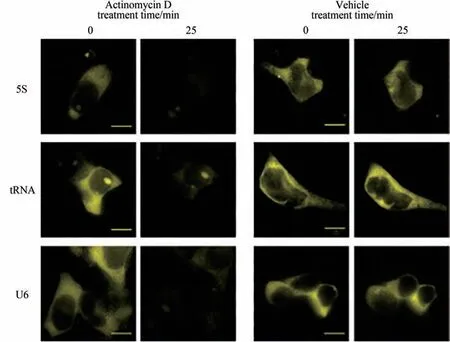
图5 核酸适配子Corn荧光基团成像单细胞中聚合酶活性示意图[87](转入报告质粒的HEK293T细胞加入荧光团DFHO可产生黄色荧光,加入RNA聚合酶抑制剂放线菌素D后荧光消失)Fig.5 Schematic diagram for imaging polymerase activity in single cells through aptamer corn fluorophore[87](HEK293T cells transformed with the reporter plasmid could produce yellow fluorescence by adding fluorophore DFHO,but the fluorescence disappears after adding the RNA polymerase inhibitor actinomycin D.)
Filonov 等[88]开发了可快速生成荧光RNA 复合体的方法。利用荧光基团选择合适的适配子,然后将大量适配子在大肠杆菌中表达,再结合流式分选技术进一步优化合适的适配子。该方法改进了传统方法中需大量诱变和有效折叠的步骤,减少适配子筛选时间,并且能够避免核酸荧光复合物的丢失,可快速鉴定出与细胞环境兼容、在细胞表达中折叠优化的成像适配子。该团队用SELEX-FACS 方法鉴定出几种新型高性能的荧光核糖核酸适配子,其中Broccoli具有分子量小、高折叠效率、热稳定性好等优点,适用于哺乳动物细胞内RNA 荧光成像。此外,其折叠过程不依赖tRNA,成像功能不依赖镁,避免了外源镁对成像功能的干扰。但Broccoli 和Spinach 这些RNA 荧光材料不能用于探测RNA 聚合酶Ⅱ依赖的mRNA 转录。Braselmann 等[89]开发了一种新的应用于哺乳动物细胞RNA 成像的方法,以细菌核糖开关作为RNA 标签在结合一系列分子探针时可产生荧光,利用核糖开关与其天然配体钴胺素(CbI)的选择性和高亲和性,与荧光基团结合发生荧光猝灭的分子标记技术(图6)。该成像系统核糖核酸分子量小,光稳定性较好,并且可以用于不同的核糖核酸分子成像,具有广泛适用性。

图6 细菌核糖开关原理及结构[89]Fig.6 Diagram for the working principle and structure diagram of bacterial riboswitch[89]
近来,华东理工大学杨弋团队[76]基于合成生物学的基本原理,结合分子共同定向进化技术,成功构建出了一系列稳定、亮度较高、背景干扰较低的高性能荧光RNA。这些荧光RNA是仅含40 余个核苷酸的RNA 分子片段,特异结合不发光的合成染料HBC 的一系列类似物,进而产生强烈荧光。它们具有蓝绿色、黄色、橙、红等不同颜色,与五颜六色的辣椒相似,因此被命名为“Pepper”。通过基因编辑等手段,Pepper 可以插入到不同的天然非编码RNA 与编码RNA 分子序列中,进而在活细胞内对各种RNA 进行荧光标记和实时成像,而不影响它们的转录、定位、翻译、降解等正常代谢。利用该技术他们实现了在哺乳动物细胞对不同种类RNA 的标记和无背景成像(图7)。

图7 荧光RNA复合体在HeLa细胞成像[76](合成染料HBC的一系列类似物与Pepper结合可发出不同颜色荧光,HBC类似物与Pepper结合物在HeLa细胞中进行成像,用空载细胞做对比,在共聚焦显微镜和双光子显微镜下进行成像观察)Fig.7 Schematic diagram for imaging of the fluorescent RNA complex in HeLa cells[76](A series of analogues of synthetic dye HBC combined with Pepper can emit fluorescence with different colors.HBC analogues were combined with Pepper in HeLa cells and are compared with mock cells.Imaging observation is carried out with confocal microscope and two-photon microscope.)
3 合成生物学技术用于病毒荧光成像和示踪
利用合成生物学技术对病毒颗粒特定蛋白或核酸进行定点、定量标记和多色多组分同时标记,可以精细示踪病毒颗粒的侵染过程与行为,揭示病毒感染宿主细胞的机制。例如,对于已造成近3300 万人死亡的艾滋病毒[90],病毒致病机制研究和药物研发等仍有待突破。为了更好地研究病毒侵染机制,科研人员结合合成生物学与单病毒示踪技术[91],建立了多种病毒荧光标记技术,用于病毒的标记和示踪,从而深入研究和阐明病毒侵染宿主细胞的时空动态过程和机制。2009 年,Miyauchi 等[92]利用有机染料分子DID 和荧光蛋白GFP 对艾滋病毒粒子进行双标记,发现HIV 通过受体介导的胞吞作用进入细胞并释放遗传物质的过程。2016 年,Ma 等[93]动态追踪了HIV 病毒颗粒脱壳过程。利用金属钌有机配合物([Ru(phen)2(dppz)]2+)、双砷染料(FlAsH/ReAsH)和荧光蛋白分别对病毒的基因组RNA、衣壳蛋白CA和基质蛋白MA进行荧光标记,构建了具有良好侵染能力的双色和三色荧光标记病毒颗粒。结合活细胞内单颗粒示踪技术,对单个艾滋病毒入侵宿主细胞时的解离脱壳过程进行实时、动态、可视化分析,揭示了艾滋病毒在侵染宿主细胞后60~120 min,病毒基因组、衣壳蛋白和基质蛋白以一个类似于“火箭升空逐级分离”的模式动态顺序解离。
荧光蛋白发光强弱依赖于蛋白的数量和其肽链的折叠结构,单个荧光蛋白分子发光能力较弱,而大量荧光蛋白的掺入也会影响目标分子和病毒的功能。同时,荧光蛋白也易光漂白,在进行长时间标记过程中荧光强度容易发生变化,且荧光蛋白具有较宽的发射光谱,不同荧光蛋白间发射光谱容易重叠,不利于对目标分子进行多重荧光标记。
鉴于此,科研人员通过合成生物学方法,建立了多种基于量子点纳米材料的病毒荧光标记技术,用于病毒颗粒的示踪和检测。Li等[77]对HIV-1-QDs的结构特征分析表明QDs标记的HIV-1 感染性相比于没有标记的HIV-1 感染性并无下降,表明QDs适合用于对HIV-1进行标记成像和示踪。人巨噬细胞是HIV-1 的主要靶点,Li 等[77]用前述量子点标记病毒颗粒的Vpr 蛋白,对HIV-1 在巨噬细胞中运动过程进行示踪,追踪了病毒颗粒与细胞内体膜融合过程,发现了HIV-1通过网格蛋白介导的内吞作用进入巨噬细胞(图8),和肌动蛋白细胞骨架有助于HIV-1病毒在细胞内运动过程。为了进一步了解HIV-1入侵人巨噬细胞机制,该团队利用量子点标记基质蛋白的病毒,实现在较长时间内对病毒颗粒进行实时示踪,研究了病毒入侵巨噬细胞,基质蛋白与Vpr 蛋白动态解离等过程[74],研究还发现病毒感染巨噬细胞初期促进了细胞内线粒体的分裂。Yin 等[94]结合病毒囊膜以及细胞组分等多重荧光标记,实时动态成像了单个HIV-1病毒颗粒膜融合入侵人原代CD4 T 淋巴细胞和跨越皮质肌动蛋白屏障的时空过程。

图8 HIV-1通过内吞作用进入细胞[77]Fig.8 Schematic diagram for HIV-1 entering cell through endocytosis[77]
量子点标记技术也可以应用于流感病毒的示踪成像,Qin 等[75]首次解析了单个流感病毒颗粒IAV脱壳过程(图9)。他们利用不同量子点标记的流感病毒,实时示踪病毒入侵细胞、病毒脱壳以及遗传物质在细胞核内的运动过程,揭示了流感病毒IAV 的8 个vRNP 复合物是通过分开的方式释放到胞质后分别进入细胞核。但是利用量子点标记病毒颗粒存在的问题是标记效率较低,需要改进或发展新的标记技术提高其标记效率。
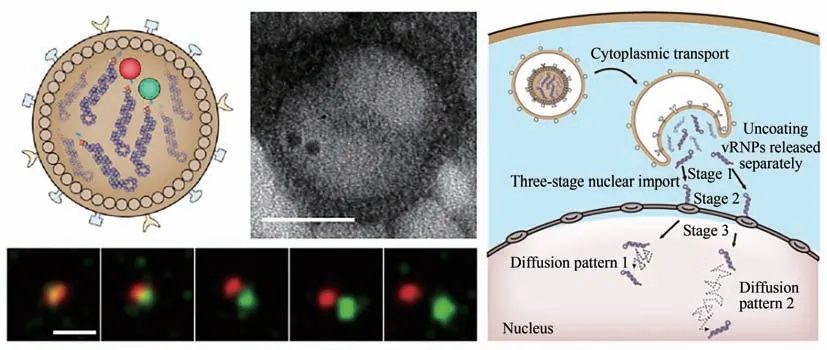
图9 流感病毒脱壳过程实时动态示踪[75][QD625在电镜下又黑又圆,而QD705为三角形。用QD625(红色)和QD705(绿色)分别标记vRNP,结果显示在脱壳过程中QD625和QD705分别释放。IAV释放的vRNPs经过三阶段的主动运输到达细胞核,在细胞核经历两种扩散模式]Fig.9 Real-time dynamic tracking of the influenza virus unpacking process[75][Under electron microscope,QD625 is black and round,while QD705 is triangular.vRNP is labeled with QD625(red)and QD705(green)respectively,and the results showed that QD625 and QD705 are released during the hulling process,respectively.IAV-released vRNPs released from IAV reach the nucleus through a three-stage active transport,where they undergo two diffusion modes.]
2017年,Ma等[95]通过设计筛选了特异性识别HIV-1 前病毒基因的TALE 识别元件,基于反式环辛烯与四嗪的生物正交反应,建立活细胞内针对TALEs 的量子点标记技术。通过时-空耦合设计,采用不同类型的生物正交反应,获得不同颜色量子点标记的TALE 探针。量子点-TALE 探针特异性标记整合在染色体上的HIV 前病毒DNA,实现了活细胞内单拷贝整合态前病毒DNA 原位标记与成像(图10)。在整合HIV 前病毒DNA 的潜伏感染细胞中,可以对HIV 前病毒进行实时动态示踪和3D 定位分析,并测定单细胞内整合态前病毒的数目。但TALEs 探针的优化筛选和操作过程比较烦琐复杂,靶向效率稍低。之后,他们又设计了特异识别病毒基因组的CRISPR 识别体系,通过与两种颜色量子点结合形成双色CRISPR 探针对HIV-1 进行标记成像,与TALEs 探针相比,CRISPR 设计更简单方便,并获得了高效靶向标记效果[96]。

图10 量子点标记的TALEs用于基因组成像[95]Fig.10 TALEs labeled with quantum dots are used for gene composition imaging[95]
通过构建一端修饰CdTe:Zn2+量子点[97],另一端修饰猝灭基团的发夹型DNA 信标探针,实现了量子点表面DNA 分子个数(价态)的精准控制。研究发现量子点表面只偶联一条DNA 的纳米信标与靶标结合后,荧光恢复效果最好、信噪比高、可用于单个RNA 的标记与检测。该探针用于活细胞内HIV-1 潜伏-激活研究,实现了单个RNA 的荧光成像与检测。该技术还可用于其他核酸序列的荧光标记与检测,实现低浓度核酸的高灵敏检测和活细胞内核酸标记与成像分析。
生物发光和荧光成像技术也可以用于病毒颗粒活体成像研究,例如,Wang 等[98]将Nluc基因插入寨卡病毒C 基因编码区构建了寨卡病毒-Nluc报告病毒,实现了对寨卡病毒侵染小鼠活体动物运动过程的实时示踪成像。除了对病毒进行荧光动态示踪研究,利用特异性标记和合成改造病毒,还可以将其应用于活体成像和疾病筛查:猴病毒40(SV40)主要衣壳蛋白VP1 在体外可自组装形成多态性的结构,这种多态性为SV40 病毒纳米颗粒作为合成生物学多功能纳米结构的构建平台提供了多种可能性。Li 等[99]通过将SV40 包装近红外二区荧光Ag2S 量子点,显著提高了活体成像的穿透深度,实现了SV40 病毒样颗粒活体行为的实时动态追踪。另外,Sun 等[100]利用SV40 病毒样颗粒的组装和负载能力等优点,包裹QDs 形成多功能纳米颗粒,通过可控组装设计构建了装载治疗药物的可靶向治疗的荧光成像材料,实现了在小鼠活体内对动脉粥样斑块荧光的成像检测,对动脉粥样硬化的早期筛查和靶向治疗研究等提供了理想的平台(图11)。

图11 SV40-QD VLPs用于靶向小鼠动脉粥样硬化斑块成像[100](SV40 VLPs 含有动脉粥样硬化靶向单元,凝血酶抑制剂并可以封装QD800。FH-QDs可靶向集中在小鼠体内动脉粥样硬化形成区)Fig.11 SV40-QD VLPs for imaging atherosclerotic plaques in targeted mice[100](SV40 VLPs contain atherosclerotic targeting units,thrombin inhibitors,which can encapsulate as QD800.FH-QDS can target atherosclerotic areas in mice.)
4 问题与展望
基于合成生物学原理与技术,设计合成生物相容性好的荧光材料与探针、对关键生物分子进行可控标记,能更好地应用于细胞或活体内重要生命过程实时成像。该研究领域逐渐显示其优势和潜力,但也存在一些问题亟待解决。
在荧光材料方面,如何获得性能更加优异的荧光纳米探针,如何进一步提高荧光材料分子对特定目标的靶向性和特异性、实现荧光信号的绝对定量、避免多种荧光材料分子荧光信号的相互干扰等都需要进一步的解决。荧光纳米材料的性能难以精确控制,且通常尺寸较大,易影响生物体的结构和生物活性。而且,探针的性能不但受到识别单元和信号单元各自性能的影响,还取决于二者间的有效连接和耦合,但至今还没能很好地实现识别单元和信号单元连接的定向、定量调控,导致探针的重现性、特异性等性能不理想。因此,亟需发展新的合成生物学体系,构建适用于不同生物体生物分子跨尺度研究的高特异、高亲和、高灵敏、小尺寸、低活性扰动的生物探针。传统的合成生物学多是以“基因操纵”为主,荧光产物基本为有机物,而在活细胞内合成无机材料及纳米杂合功能材料,将拓展合成生物学的技术理念,并推动生物功能材料的创新合成及其生物医学应用。然而,基于杂合荧光生物材料的合成,可以利用的生化反应途径依然较少,较难实现材料的组装及进一步功能化。在此过程中,如何更好地实现细胞间的交流,利用活细胞这个强有力的化学工厂,实现多样化的材料的可控制备,依然是极大的挑战。
在荧光标记技术方面,生物分子在细胞和活体的成像示踪不仅需要性能优异的生物探针,还需针对生物分子的结构和动态过程特征,建立高效、特异、定点、定量的原位标记方法,并尽可能减少对生物分子活性的影响,从而获取真实可靠、高灵敏的测量信号。目前,常规生物分子组分的标记方法主要包括:①利用亲和靶向识别细胞或生物体的特定组分;②通过生物化学反应将标记物直接与生物分子偶联;③将荧光蛋白与目标蛋白融合表达。然而上述方法存在一定的局限性,如亲和标记在复杂体系中易受干扰物及基质影响,导致结合能力和特异性较低;生物化学标记方法难以实现定量控制,而大量标记物的结合易破坏原有生物体生物分子的结构与功能,影响其活性;荧光蛋白的可标记组分有限,且荧光蛋白的大量表达也会影响生物分子活性。另一方面,以病原微生物为例,为了准确详细地获取病原微生物感染全过程的多元信息,需要对细菌细胞膜和细胞壁,病毒囊膜、衣壳、核酸等不同结构和组分分别进行标记,而已有的病原微生物双标或多重标记标方法普遍存在烦琐、低效的问题。因此,生物分子标记技术仍是制约其感染过程研究的难点,迫切需要发展新的合成生物学标记体系,创建和发展对不同生物分子不同结构和组分的特异、原位标记方法。
总之,基于合成生物学新原理新技术,针对蛋白、核酸、病原体等不同类型标志物为靶标,从物质基础、能量驱动和调控机制三方面作为突破口,创建功能性荧光材料体内原位合成新方法,合成性能优异的荧光材料和探针,发展生物分子定点、定量、特异、精准标记新技术,结合实时动态、高灵敏高分辨影像新技术解析重要生命活动过程与机制,将极大促进合成生物学技术在荧光生物成像技术方面的应用。合成生物学与荧光成像技术的交叉融合,也将拓展合成生物学的研究领域,并推动荧光成像技术发展和进步。
