绵羊TNF-α基因启动子克隆、活性分析及转录因子预测
2022-05-12张彦兵魏立翔解艺璇李增强李珂儿孙延鸣
张彦兵,魏立翔,解艺璇,李增强,李珂儿,孙延鸣
(石河子大学动物科技学院,新疆 石河子 832003)
绵羊肺炎支原体(Mycoplasma ovipneumoniae,MO)感染致病的主要原因是,引发肺部强烈的炎性反应导致肺部病理损伤[1-2];肿瘤坏死因子α (Tumor necrosis factor α,TNF-α)等炎性细胞因子在MO感染的绵羊肺部高水平表达[3],则促进绵羊肺部炎症反应。TNF-α是一种多功能的细胞因子,参与众多生物学过程,因其可以杀伤体内的肿瘤细胞而被发现[4-5],在病原菌以及LPS刺激时,绵羊体内的TNF-α表达量显著增加[6]。先前的文献报道,猪TNF-α表达与其启动子激活密切相关[7-8]。但是,绵羊TNF-α高水平产生的机理不明,以及绵羊TNF-α启动子尚未克隆,需要进一步研究。
绵羊TNF-α定位于20号染色体上,其高表达于绵羊肺泡巨噬细胞[9]。已知,人源TNF-α启动子上含有转录因子:AP-1、AP-2、NF-κB和cAMP响应元件(CRE)等,上述转录因子对于炎性刺激物脂多糖(LPS)也较为敏感[10-12]。另外TNF-α启动子甲基化水平与其表达相关,如LPS刺激猪源、鸡源细胞会降低TNF-α启动子甲基化水平,易于TNF-α高水平转录表达[8,13]。然而,绵羊TNF-α启动子上具有哪些转录因子还不清楚,有待于进一步研究。
本文旨在克隆绵羊TNF-α启动子,构建启动子荧光素酶报告载体并验证活性,预测绵羊TNF-α启动子上潜在的转录因子;这将弄清楚绵羊TNF-α启动子的基本特征,为后续从转录调控、基因启动子甲基化等方面,解析MO感染绵羊诱导产生高水平TNF-α的机理打下基础。
1 材料与方法
1.1 实验试剂和细胞
DMEM培养基(HyClone)、胎牛血清(GibcoTM)、PBS(HyClone)、DMSO(二甲基亚砜,Sigma)、双荧光素酶活性检测试剂盒(Promega)、DNA提取试剂盒(诺唯赞生物公司)、大肠杆菌DH5α(生工生物公司)、LA taq酶和限制性内切酶(TaKaRa中国);pGL3-Basic质粒(中国农业科学院上海兽医研究所邱亚峰研究员馈赠),引物由生工生物公司(郑州)合成。
细胞系:HEK293T细胞用含有10%胎牛血清(FBS)和抗生素(100 U·mL-1青霉素和100 U·mL-1链霉素)的DMEM培养基培养。
1.2 启动子预测及其活性区域预测
利用UCSC(https://genome.ucsc.edu/)数据库和National Center for Biotechnology Information(https://www.ncbi.nlm.nih.gov/)数据库分别预测绵羊TNF-α基因启动子,利用DNASTAR Version7.1.0(44)软件比对预测的TNF-α启动子序列,分析序列同源性。利用在线软件Berkeley Drosophila Genome Project(https://www.fruitfly.org/seq_tools/promoter.html)预测启动子可能的活性区域,进一步设计引物(表1)。

表1 引物序列
1.3 绵羊TNF-α基因启动子克隆及载体构建
利用试剂盒提取萨福克绵羊肺组织DNA作为模板,扩增TNF-α基因启动子,扩增反应体系及程序如下:LA taq 0.3 μL,dNTPs(2.5mM)4.5 μL,10×LA buffer(Mg2+)5 μL,DNA 2 μL,上下游引物各2 μL,ddH2O 34.7 μL;反应程序:预变性95 ℃ 3 min,进入35个循环95 ℃ 15 s,62 ℃ 15 s,72 ℃ 1 min;最后72 ℃ 7 min。PCR产物琼脂糖凝胶分析,按照试剂盒说明回收DNA并测序,测序确认扩增的序列正确后,并将其连接到pGL3-Basic载体中得到报告质粒pGL3-TNF-α。具体操作如下,首先用KpnI和XhoI线性化pGL3-Basic载体,胶回收备用;连接体系:T4 DNA连接酶 1 μL,线性化载体 2 μL,纯化的片段 15 μL,10×buffer 2 μL,16 ℃水浴反应过夜,直接转化大肠杆菌DH5α感受态细胞,挑选阳性克隆大量培养提取质粒。酶切验证后,在生工生物工程(上海)股份有限公司测序,以pGL3-Basic载体的通用引物RVprimer3和GLprimer2双端测序验证。
1.4 绵羊TNF-α基因启动子活性验证
将较高转染效率的HEK293T细胞接种24孔板,过夜培养后,将300 ng pGL3-TNF-α质粒及6ng 的pRL-TK质粒共转染细胞,同时共转染300ng的pGL3-Basic和6ng 的pRL-TK质粒作为对照组,转染24 h后利用Promega双荧光素酶报告基因系统检测启动子活性;具体检测过程如下:用蒸馏水将5×Passive Lysis Buffer(PLB)稀释为1×PLB备用;LAR II:根据试剂盒说明利用Luciferase Assay Buffer II将Luciferase Assay Substrate粉末溶解,充分混匀后分装冻存在-70°C;根据所需用Stop & Glo®Buffer将50×Stop & Glo®Substrate稀释为1×Stop & Glo®Substrate备用;用室温PBS洗涤细胞三遍,24孔板每孔加入100 μL 1×PLB,室温震荡裂解30 min,将裂解液转移至离心管,离心后取上清备用;萤火虫荧光素酶和海肾荧光素酶的活性检测:1.5 mL离心管中加入50 μL LAR II,加入细胞裂解上清液10 μL混匀立即读数,结束后迅速加入50 μL 1×Stop & Glo®Substrate混匀并读数,记录数值,计算萤火虫荧光素酶与海肾荧光素酶活性的比值。
1.5 绵羊TNF-α基因启动子转录因子预测
分析预测绵羊TNF-α基因启动子上潜在的转录结合因子,打开AliBaba2.1软件(http://gene-regulation.com/pub/programs/alibaba2/index.html),将分析序列粘贴至对话框,点击分析按钮,预测完毕后会显示转录因子结合序列的区域及其名称等信息,选择评分较高排名较前的转录因子,标注出具体区域及转录因子名称。
2 结果
2.1 绵羊TNF-α基因启动子及其活性预测、克隆及报告载体构建
从不同数据库预测所得绵羊TNF-α基因启动子同源性高达99.9%,仅存在1个碱基的差异(图1)。

图1 不同数据库预测的绵羊TNF-α启动子序列比对
通过对启动子活性区域预测,发现绵羊TNF-α基因启动子含有2个潜在的活性区域,启动子活性区域中含有典型的TATA-box元件(图2)。
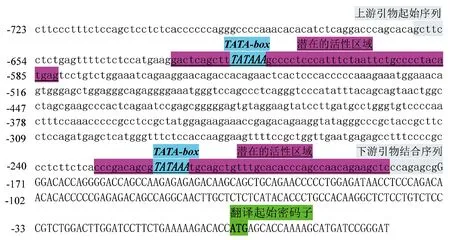
图2 绵羊TNF-α基因启动子及其活性区域预测
基于对绵羊TNF-α启动子预测和活性区域分析,确定扩增在翻译起始位点上游-659到-172的区域。以绵羊肺组织DNA为模板成功扩增到目的片段(488 bp)(图3A);将扩增的DNA片段和pGL3-Basic质粒双酶切、连接,构建pGL3-TNF-α荧光素酶报告质粒;KpnI和XhoI双酶切质粒后,出现线性化的pGL3-Basic载体条带和绵羊TNF-α启动子条带(图3B),双酶切验证后将质粒送生物公司测序,测序获得序列与预测的一致。成功克隆了绵羊TNF-α基因启动子,并构建了pGL3-TNF-α荧光素酶报告质粒。

图3 绵羊TNF-α启动子扩增(A),荧光素酶报告载体双酶切验证(B)
2.2 绵羊TNF-α启动子活性验证
将荧光素酶报告质粒pGL3-TNF-α转染HEK293T细胞,转染24 h后,按照Promage公司双荧光素酶检测说明分析TNF-α启动子活性,结果显示,克隆的绵羊TNF-α启动子活性是basic活性的16倍左右(图4),说明克隆的绵羊TNF-α启动子具有较强的活性。

**代表p<0.01,差异显著图4 绵羊TNF-α启动子活性测定
2.3 预测绵羊TNF-α启动子上潜在的转录因子
绵羊TNF-α启动子的激活需要转录因子结合,因此,进一步利用转录因子软件分析可能的结合靶点。本文只选择了评分较高、排名靠前的转录因子,Sp1(Specificity protein 1,Sp1)、C/EBPalp、c-Jun等;Sp1有6个结合区域,C/EBPalp有2个结合区域,c-Jun有1个结合区域;转录因子及其结合序列用不同的颜色进行了标注,如图5所示。

图5 绵羊TNF-α基因启动子转录因子预测结果
3 讨论
绵羊肺炎支原体感染往往会诱导产生高水平的炎性因子TNF-α等,高水平分泌的炎性因子则促进炎性反应。强烈的炎性反应是导致绵羊肺部损伤的重要原因之一,然而激活绵羊TNF-α高水平产生的机制不清楚。已有的文献报道,人和猪的TNF-α高水平产生与其启动子激活密切相关[8]。因此,本文开展绵羊TNF-α启动子的研究,我们成功克隆了绵羊TNF-α启动子,验证了启动子活性,初步分析预测了启动子上潜在的转录因子。
克隆的绵羊TNF-α启动子位于翻译起始位点上游-659至-172的区域,启动子含有两个典型的TATA-box;TATA结合蛋白(TATA-binding protein,TBP)识别启动子区域TATA-box并结合RNA聚合酶II开始转录。此外,预测的绵羊TNF-α启动子活性区域正好包含TATA-box。通过构建绵羊TNF-α启动子报告质粒,以及质粒转染细胞检测双荧光素酶活性,证明绵羊TNF-α启动子具有很高的活性,证明绵羊TNF-α启动子被成功鉴定,绵羊TNF-α启动子的成功克隆为深入研究启动子激活调控其表达的机制打下初步基础。
基因启动子区域DNA甲基化水平升高会抑制基因转录表达,反之则利于基因转录表达[15-16]。据报道,人、猪等物种TNF-α启动子甲基化参与调控其表达[17-19],如LPS刺激猪肺泡巨噬细胞可诱导TNF-α启动子区域去甲基化[8],有利于TNF-α转录表达;那么,在MO感染中,绵羊TNF-α启动子区域甲基化水平是否会发生变化,以及绵羊TNF-α启动子区域甲基化是否与其表达相关,均不清楚。目前,已成功克隆和鉴定绵羊TNF-α启动子,为进一步探索绵羊TNF-α启动子区域甲基化调控其转录表达奠定基础。
新近的研究表明猪源TNF-α转录激活与其启动子上的转录因子表达相关,例如,IRF1通过结合猪源TNF-α启动子反应元件促进启动子活性[7]。然而,激活绵羊TNF-α启动子的转录因子还不清楚,通过对克隆的绵羊TNF-α启动子序列预测分析,预测获得了9个转录因子,分为Sp1、C/EBPalp和c-Jun三种。先前的研究证实,人源Sp1可以结合Fbxl2 基因启动子激活其高水平表达[20],Sp1还参与激活HIV-1 LTR(long terminal repeat,LTR)的转录表达[21]。因此,转录因子Sp1在结合促进启动子激活方面发挥重要作用。另外,鲈鱼TRAIL(Tumor necrosis factor related apoptosis-inducing ligand,TRAIL)启动子区域上也含有Sp1、C/EBPalp和NF-kappaB等转录因子[22]。与之类似的是,绵羊TNF-α启动子上也预测到了Sp1和C/EBPalp等转录因子。然而,绵羊TNF-α启动子上潜在的上述3种转录因子,是否可以激活TNF-α的表达还需要进一步研究。我们会在将来克隆Sp1、C/EBPalp、c-Jun等基因并构建真核表达载体,利用转录因子与启动子共转实验验证转录因子的作用。
总之,本文成功克隆了绵羊TNF-α启动子,利用报告基因系统验证绵羊TNF-α启动子具有很高的活性,初步鉴定了绵羊TNF-α启动子;进一步,预测了绵羊TNF-α启动子上潜在的转录因子。本文为将来探讨转录因子调控绵羊TNF-α基因启动子的激活,以及研究DNA甲基化调节绵羊TNF-α启动子激活等研究打下基础;最终可为将来解析MO等病原微生物感染导致绵羊TNF-α上调表达的机制奠定基础,也为绵羊TNF-α启动子其他相关研究提供数据参考。
