山西大豆根瘤菌的分离、鉴定及共生匹配性筛选
2022-05-10王晓丽秦杰王敏王利祥杜维俊
王晓丽 秦杰 王敏 王利祥 杜维俊
(山西农业大学农学院,太谷 030801)
根瘤菌可以与豆科植物形成不同类型的根瘤,如和大豆、百脉根形成有限型根瘤;和苜蓿、豌豆形成无限型根瘤两种类型,从而能够共生固氮,其固氮量占生物固氮量的60%[1]。大豆作为重要的共生固氮经济作物,其生长所需氮素营养的50%-90%是由根瘤菌-大豆共生体系固定的,因此对大豆根瘤菌的研究具有巨大的实际应用价值[2]。前人研究表明根瘤菌具有明显的生物地理分布特征。Zhang等[3]利用种水平的rpoB高通量测序方法,在对我国东北、黄淮海和南方3个大豆种植区土壤中的土著大豆根瘤菌的研究中,发现黄淮海的土著大豆根瘤菌多样性最高,包括,Bradyrhizobium sp.III、B. japonicum、B. elkanii、B. huanghuaihaiense、Sinorhizobium fredii和S. sojae。而位于黄淮海的山西作为大豆的起源地之一,大豆种质资源丰富。根瘤菌与大豆在进化过程中是相互影响,共同进化的,因此与其共生的根瘤菌资源也相当丰富。但是,针对山西大豆根瘤菌的大量分离却报道很少。
目前,对于根瘤菌的分类采用表型和基因型相结合的多相分类办法。根瘤菌的表型分析,包括在不同碳源和氮源培养基上的生长情况、在YMA上生长的单菌落形态、耐盐性、对不同生长温度、pH的耐受性,BTB产酸或产碱判定等。表型的测定,不仅工作量大,易受环境影响,重复性还差,但仍是根瘤菌分类鉴定及发表新种的必需条件[4]。根瘤菌的基因型分析,包括DNA(G+C)mol%的测定及DNA-DNA杂交、16S/23S rRNA同源性分析、共生基因(NodA、NodC、NifH)同源性分析、限制性片段长度的多型性、随机扩增DNA片段的多型性、REPBOX/BOX-PCR/ERIC-PCR指纹图谱分析、持家基因MLSA(多位点序列分析)等[5-7]。相较于表型的鉴定,随着测序技术的高速发展,基因型的测定快速、准确,已成为分类的主要依据。16S rRNA进化树确定其系统发育地位构成多相分类的主干,通常被用来确定属、种的分类。然而,16S rRNA基因较高的保守性,使其在种以下水平的分类应用中受到限制[8]。指纹图谱中,尤其是BOX-PCR,操作简便,不需要菌株的特异性 DNA 探针,对基因组的要求不高,容易获得较为丰富的扩增条带,这说明BOX-PCR指纹图谱分析更适合于种及种以下水平上遗传多样性研究[9]。
根瘤菌的利用在减少氮肥的使用、增加产量和农业可持续发展等方面有巨大的潜能。但国内的根瘤菌使用情况与巴西、美国、阿根廷等几个大豆主产国还相距甚远[10]。由于土壤中土著根瘤菌的存在,使得菌剂的竞争性较差,接种效果差强人意[11]。通过对土著根瘤菌的分离,筛选出既适应当地土壤条件,竞争效果好,又与地方大豆品种共生固氮效果好的根瘤菌,可以有效避免这一问题。因此,本研究通过分离、鉴定山西的土著根瘤菌,并将其分类,以期通过苗期筛选出与山西主栽大豆品种的匹配性好的土著根瘤菌。
1 材料与方法
1.1 材料
1.1.1 菌株和大豆品种:菌株分离自山西大同广灵(黄壤沙土)、太原清徐(盐碱土)、晋中太谷(黄壤土)。大豆品种为晋大88号黄豆,科丰1号黑豆。
1.1.2 培养基与试剂 YMA培养基:甘露醇10 g,酵母粉3 g,NaCl 0.1 g,MgSO4·7H2O 0.2 g,K2HPO40.25 g,KH2PO40.25 g,琼脂粉15 g,蒸馏水1 000 mL,pH值6.8-7.0。
刚果红培养基:1 000 mL YMA培养基中加入0.5%的刚果红溶液5 mL。
BTB培养基:YMA液体培养基中加入溴百里香酚蓝(BTB)使其终浓度为0.01%,调节溶液pH,使溶液呈淡黄绿色。
EasyTaq@ DNA Polymerase购自北京全式金生物技术有限公司。
1.2 方法
1.2.1 根瘤菌的分离与纯化 选取不同大豆品种根上个大、饱满的根瘤,当天进行分离培养。分离、纯化方法参照Peter等[6]方法,具体如下:在超净工作台中,依次放置6个灭菌的培养皿,依次倒入无菌水、75%乙醇、5%的NaClO溶液,后3个倒入无菌水。将取下的根瘤依次放入培养皿中,每个浸泡的时间为5 min,然后用镊子将根瘤夹破,将创伤面贴在含有刚果红的YMA培养基上。在28℃下倒置培养3-4 d。挑取未被染色的乳白色、粘稠状的菌落在刚果红YMA培养基上多次划线,获得单菌落。之后在培养基上划线扩繁,制备成25%的甘油菌并保存-80℃冰箱。
1.2.2 根瘤菌的表型与基因鉴定 将分离的菌株进行关键结瘤固氮基因nifH、nodA PCR扩增,对能扩增出目标条带的菌株,再进行16S rDNA扩增,以及BTB染色鉴别。引物如表1所示。菌液PCR反应体系为 :EasyTaq DNA Polymerase 0.3 μL,10×EasyTaq@ Buffer 1.5 μL,2.5 mmol/L dNTPs 1.3 μL,F 引物 0.3 μL,R引物0.3 μL,菌液培养3 d左右(OD600=0.5),DNA模板(煮沸的菌液)1 μL,最后ddH2O补足到15 μL。nifH PCR 反应程序为 :94℃预变性 4 min,94℃ 变性 30 s,56℃ 复性 30 s,72℃延伸 1 min,30个 循 环 ;72℃ 延 伸 10 min。nodA和 16S rDNA PCR反应程序分别是57℃复性30 s,72℃延伸36 s,55℃复性30 s,72℃延伸1.5 min,其余与nifH PCR反应程序一样。将PCR产物在1%的琼脂糖凝胶电泳检测后,送去北京六合华大进行测序。测序结果在NCBI数据库Blast后,选择相似性较高的序列,在MEGA7.0软件利用邻接法构建系统发育树来确定根瘤菌的分类地位。
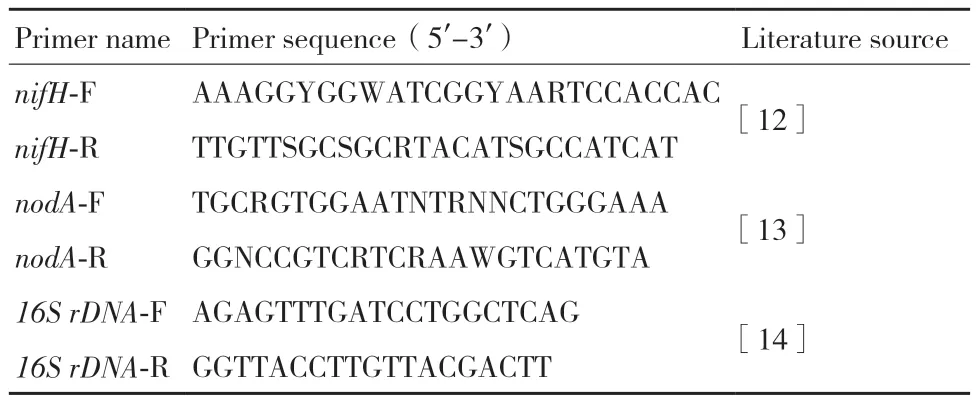
表1 根瘤菌基因鉴定所用引物Table 1 Primers for gene identification of rhizobia
1.2.3 BOX-PCR 用 BOX A1R(5′-CTACGGCAAGGCGACGCTGACG-3′)扩增根瘤菌DNA的重复区域。PCR 反应体系与1.2.2一样。反应程序为:94℃预变性 4 min,94℃ 变性 1 min,53℃ 复性 1 min,72℃延伸5 min,30个循环;72℃延伸10 min。80 V电压下,BOX-PCR扩增产物在1.5%琼脂糖凝胶上电泳2.5 h,将照胶图片保存为TIFF格式。所有跑胶图片在Bionumerics@7.5软件采用UPGMA算法和Cosine系数进行聚类分析,以70%的相似性进行根瘤菌的分类。
1.2.4 根瘤菌与大豆的匹配性筛选 将蛭石灭菌,装入10 cm×10 cm的双层营养钵中。晋大88号黄豆和科丰1号黑豆用70%的无水乙醇进行表面消毒5 min,再用5%的NaClO消毒5 min,之后用无菌水清洗3次,播种到营养钵中并在温室中培养。一周后间苗,每个营养钵留一株长势一致的大豆。用无氮营养液浇灌,待第一片真叶展开时,每株大豆接3 mL菌液(OD600=0.5),以不接菌作对照,接种USDA110做参照菌株,3次重复。4周后进行指标测定。测定指标包括株高、地上部分鲜重、根鲜重、植株干重、根瘤数、根瘤鲜重。
1.2.5 数 据 分 析 采 用 Excel、DPS(V9.01)、MEGA7.0、Bionumerics@7.5对数据进行分析。
2 结果
2.1 根瘤菌的分离、纯化
对菌落形态进行观察后,发现菌株的单菌落一般为圆形或椭圆形,表面湿润,不透明,乳白色,微微凸起,这些生长表型均同根瘤菌的基本特征相符(图1-A)。快生型根瘤菌一般生长3 d,菌落直径就可达到2 mm,慢生型根瘤菌生长5-7 d,菌落直径为1 mm。共分离出203株菌,并用分离地将其命名,分离自山西大同广灵、太原清徐和晋中太谷分别命名为GL、QX和TG(附表1)。

图1 根瘤菌表型鉴定Fig.1 Phenotype identification of rhizobia
2.2 根瘤菌的表型与分子鉴定
nifH固氮基因扩增条带为460 bp,nodA结瘤基因扩增条带为660 bp,16S rDNA扩增条带为1 400 bp(图2)。203株菌中有187株可以扩增出nifH和nodA基因,基本确定为根瘤菌。对这187株根瘤菌进行BTB染色鉴别,其中172株快生型根瘤菌,产酸变黄(图1-B);15株慢生型根瘤菌,产碱变蓝(图1-C)。将前25株快生型根瘤菌的nifH、nodA、16S rDNA的PCR产物测序,3个基因都属于中华根瘤菌属(Sinorhizobium),且与S. fredii有99%以上的相似性,但是并不能很好将菌株区分。因此,不再依次对后续菌株进行测序。挑选BOX-PCR三类带型不同的慢生型根瘤菌,进行16S rDNA测序鉴定种属,均属于慢生型根瘤菌(Bradyrhizobium),以能与大豆结瘤的慢生型根瘤菌Bradyrhizobium japonicum,B. diazoefficiens,B. liaoningense,B. elkanii,B.yuanmingense,B. canariense,B. huanghuaihaiense,B. daqingense做参比菌株构建进化树,表明QX13和B. diazoefficiens同源性较高,GL67、GL76和B.daqingense同源性较高(图3-6)。
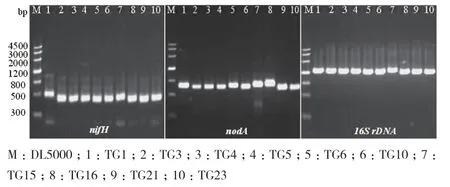
图2 根瘤菌基因鉴定结果Fig. 2 Gene identification results of rhizobia

图3 nifH 进化树Fig. 3 nifH phylogenetic tree
图4 nodA 进化树Fig. 4 nodA phylogenetic tree

图5 16S rDNA 进化树Fig. 5 16S rDNA phylogenetic tree

图6 慢生型根瘤菌16S rDNA 进化树Fig. 6 16S rDNA phylogenetic tree of Bradyrhizobium
2.3 根瘤菌BOX-PCR结果
将全部根瘤菌进行BOX-PCR,其电泳条带的大小在5 000 bp-400 bp之间,条带数目在25-3条不等(图7)。将187株根瘤菌进行BOX-PCR,去除条带完全一致的菌株,用剩余的85株菌和两个参比菌株USDA110、S. fredii 45436进行聚类分析。这87株根瘤菌以45%的相似性可分为A、B、C、D和E五大类群,其中B类群为慢生型根瘤菌,A、C、D和E类群为快生型根瘤菌,根瘤菌QX13表型和16S rDNA鉴定为慢生型,却聚类到快生型根瘤菌中的A群中,GL19表型鉴定为快生型,却聚类到慢生型根瘤菌中的B群中(图8)。以70%的相似性可将这87株菌共分为18类,两个参比菌株各为一类,本实验分离的根瘤菌分为16类,表明分离的土著根瘤菌与两个参比菌株相似性差距较大。16个聚类中11个由少于3个分离株组成,3个由4个分离株组成,1个由5个分离株组成,剩下的第一、三、六类聚到的根瘤菌株较多。第一类的9株菌株全部分离自广灵;第三类32株菌株,除1株分离自广灵外,其余全部分离自太谷;第六类14株菌株,7株分离自太谷,7株分离自广灵,但其在72%的水平上便各自分为一类,第一小类9株中,7株分离自太谷,2株分离自广灵,第二小类5株,全部分离自广灵。表明根瘤菌具有较强的地域性。

图7 BOX-PCR电泳结果Fig. 7 Results of BOX-PCR electrophoresis

图8 BOX-PCR聚类结果Fig.8 Results of BOX-PCR clustering
2.4 根瘤菌与大豆的匹配性筛选
从分离的16类土著根瘤菌中,每一类挑选出一株代表性菌株进行共生匹配性筛选。接种根瘤菌的晋大88号植株长势整体要比CK好,接种效果明显(图9-A),接种根瘤菌的科丰1号植株长势和CK差别不大,接种效果不明显(图9-B)。接种不同根瘤菌的2个大豆品种都在株高、地上干重和根干重存在显著差异(表2)。接种不同根瘤菌的2个大豆品种也都在根瘤鲜重、根瘤干重和根瘤数有显著差异(表3)。表明接种的菌株都可以和这两个大豆品种结瘤,但不同的根瘤菌与大豆品种匹配性存在较大差异。接种同一根瘤菌的晋大88号的根瘤性状要好于科丰1号,除接种GL16和QX13外,其根瘤大小没有太大区别(图10),表明科丰1号较晋大88号对根瘤菌的识别更加严格。通过对根瘤生物量和植株生物量的比较,发现其并非严格对应,根瘤性状最好的,地上性状并非最好,可能与根瘤菌的固氮能力有关。筛选出与晋大88号匹配性较好的根瘤菌GL11和GL43,其地上部分干重较接种USDA100分别增加38.5%、30.1%;根干重分别增加22.2%、27.8%;根瘤鲜重分别增加24.3%、41.5%;根瘤干重分别增加36.6%、31.0%。筛选出与科丰1号大豆匹配性较好的根瘤菌TG37,其株高、地上干重、根干重、根瘤鲜重、根瘤干重和根瘤数较接种USDA110分别增加8.1%、15.0%、28.6%、27.3%、44.3%和60.6%。

图9 根瘤菌与大豆的匹配性实验的大豆植株长势Fig.9 Soybean plant growth in the matching experiment between rhizobia and soybean

表2 根瘤菌与大豆匹配性筛选中植株性状的比较Table 2 Comparison of plant traits between rhizobium and soybean in matching screening

表3 根瘤菌与大豆匹配性筛选中根瘤性状的比较Table 3 Comparison of nodule traits between rhizobium and soybean in matching screening
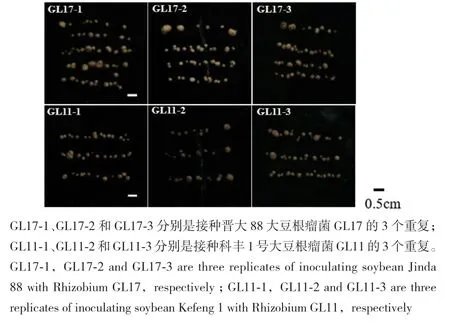
图10 根瘤菌与大豆的匹配性实验的根瘤图片Fig. 10 Picture of nodules in the matching experiment between rhizobia and soybean
3 讨论
本研究从山西大同广灵、晋中太谷、太原清徐共分离203株菌,其中187株根瘤菌可以扩增出nifH和nodA基因。通过BTB染色鉴定172株为快生型根瘤菌,包括了大同广灵的84株,晋中太谷的88株;15株为慢生型根瘤菌,包括了大同广灵的12株,太原清徐的3株。挑选前25株快生型根瘤菌进行nifH、nodA和16S rDNA测序,与相似菌株建立进化树,表明其都属于S. fredii。挑选3株慢生型根瘤菌进行16S rDNA测序,构建的进化树显示从大同广灵的分离的两株GL67、GL76属于 B. daqingense,在山西广灵是首次分离出来。B.daqingense最先从黑龙江省大庆市分离鉴定出来[15]。在酸性和中性土壤中一般以慢生根瘤菌为主,在碱性土壤中一般以费氏中华根瘤菌(S. fredii)为主[16]。QX13属于B. diazoefficiens,是从清徐的盐碱土中分离。葛诚等[17]从辽宁铁岭地区的盐碱土中分离出超慢型大豆根瘤菌。在新疆盐碱地中,S. fredii(45%)和B. liaoningense(43%)是大豆根瘤菌的优势类群,B. japonicum、B. yuanmingense和Rhizobium为次要类群[18]。同为盐碱土,因地理位置和气候条件的不同,大豆根瘤菌种类也有较大差异。
不同的生态区,大豆主栽品种和农家品种各不相同。张璐等[19]通过宏基因组高通量测序分析了大豆根区、根际到根瘤的细菌群落构成,发现大豆对细菌群落的构成有明显的逐层过滤和富集作用,其中在大豆非根际土壤中的优势根瘤菌是Bradyrhizobium属和 Mesorhizobium属,在根际土壤中主要的优势根瘤菌是Sinorhizobium属和 Rhizobium属,根瘤内部的优势菌是Sinorhizobium属。大豆对根瘤菌的特异性识别,使得大豆根瘤菌的分布差异更加明显。史清亮等[20]从山西分离根瘤菌情况表明S.fredii为优势菌,占分离菌总数的90%,而且菌株类型也较多,分布最广,抗逆性较强,是山西省大豆侵染结瘤的主要类群和优势资源菌株,与此次的分离结果相同。张红侠等[21]还从山西分离出两类慢生型根瘤菌为B. liaoningense和B. japonicum。
利用BOX-PCR以45%的相似性将87株根瘤菌分为A、B、C、D和E五大类群,其中B类群为慢生型根瘤菌,A、C、D和E类群为快生型根瘤菌,根瘤菌QX13表型和16S rDNA鉴定为慢生型,却聚类到快生型根瘤菌中的A群中,GL19表型鉴定为快生型,却聚类到慢生型根瘤菌中的B群中,QX13和GL19在59%的相似性上都独自聚为一类,可能是不同的PCR仪、不同批次电泳、电泳液对结果的影响[22]。在BOX-PCR分析中,相似度低于70%的菌株被认为是差异较大的,并被单独分为一类[23]。以70%的相似性可将这87株菌分为18类,两个参比菌株各为一类,本实验分离的根瘤菌分为16类,分离的土著菌株在基因组上差别较大,而且,从同一地区分离的菌株一般聚到一类,与王卫卫等[24]对东北三省的根瘤菌分类情况相似,说明根瘤菌具有较强的地域性分布。综合表明山西的根瘤菌资源丰富、种类多样。
根瘤菌与大豆复杂识别调控机制,造就了两者极强的种属特异性,环境也对共生固氮的效果产生较大影响,筛选出适应性广,竞争力强的根瘤菌一直是科研工作者努力的目标。张红侠等[25]通过蛭石初筛和土壤复筛获得的优良耐旱菌株 B.liaoningense4345和 S. fredii4338,接种晋豆25可显著提高植株生物量、全氮量和产量,具有较强竞争能力,具有良好的应用前景。蒋攀等[26]综合水培实验、盆栽和大田小区试验筛选出了与四川大豆主栽品种贡选号、南豆号匹配性最佳的菌株S65。本研究结果显示两个大豆品种都可以和16种根瘤菌结瘤,晋大88的结瘤效果整体比科丰1号的好,不管是接种分离的土著根瘤菌,还是USDA110。表明科丰1号对根瘤形成调控更加严格。结瘤固氮是高耗能的过程,豆科植物通过结瘤自调控(AON)来调控碳氮平衡,通过地上部分生物量的比较,也可以部分解释科丰1号的结瘤效果更差。通过匹配性筛选,表明不同的根瘤菌与大豆品种匹配性存在较大差异,这与伍慧等用8株优良大豆根瘤菌与不同地区27个大豆主栽品种的匹配性研究结果相似[27]。与晋大88号匹配性较好的是GL11和GL43,其地上部分、根和根瘤的生物量都较接种USDA110增加在22.2%-41.5%。与科丰1号匹配性较好的是TG37,其地上干重、根干重、根瘤鲜重、根瘤干重和根瘤数较接种USDA110增加在15.0%-60.6%之间。为后续研究提供候选菌株。
4 结论
本研究从山西分离出187株根瘤菌,172株为快生型根瘤菌,属S. fredii,是当地的优势菌,15株为慢生型根瘤菌,分别属B. diazoefficiens和 B.daqingense。BOX-PCR聚类表明,此次分离的根瘤菌分为16类,表明山西根瘤菌资源丰富,种类繁多。通过苗期的匹配性筛选,能够与晋大88号共生固氮效果好的是GL11和GL43,与科丰1号共生固氮效果好的是TG37。为后续的大田实验提供菌剂资源。
文章所有附图附表数据请到本刊官网下载(http://biotech.aiijournal.com/CN/1002-5464/home.shtml)。
