肝癌细胞周期调控相关基因预后模型构建与评估
2022-04-29孙东旭朱文静金志朋刘华元朱朋程史光军
孙东旭,朱文静,金志朋,刘华元,朱朋程,史光军
(1 大连医科大学研究生院,辽宁 大连 116000; 2 青岛大学附属青岛市市立医院肝胆外科; 3 青岛大学青岛医学院)
肝细胞癌是世界范围内发病率较高的恶性肿瘤,约占肝癌病人的90%[1]。尽管肝细胞癌的治疗取得了一些进展,但肝细胞癌病人的预后仍然很差[2]。既往生物信息学综合性研究所构建的肝癌预后模型等研究结果十分广泛,包括基于免疫相关编码基因集合[3]、p53相关的microRNA集合[4]等。但由于预后肿瘤标志物和治疗靶点尚未得到充分研究和临床应用,肝细胞癌病人的预后判断和个体化诊疗仍是一大挑战。本研究的目的是构建预后模型,为肝癌病人的预后预测和个体化治疗提供分子标志物和新的方向。
1 资料和方法
1.1 肝癌转录表达数据的获取和差异表达基因的筛选
从TCGA数据库(https://portal.gdc.cancer.gov/)下载TCGA-LIHC肝癌数据集。TCGA数据库肝癌数据集包含374例肝细胞癌肿瘤组织样本和50例癌旁正常肝组织样本的表达数据以及临床数据。使用统计学软件R软件(3.6.1版)[5]和Bioconductor ‘edge’软件包分析肝细胞癌样本与正常组织间差异表达基因的表达差异[6-7]。|Log2FC|>2和校正后P值<0.05的基因被定义为差异表达基因。
从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)[8]GPL10558平台(Illumina HumanHT-12 V4.0 expression beadchip)下载肝癌数据集GSE36376。GSE36376数据集包含240例肝细胞癌组织样本和193例癌旁组织样本的表达数据和临床数据。|Log2FC|>1和校正后P值<0.05的基因被鉴定为差异表达基因。使用维恩图在线工具(http://bioinformatics.psb.ugent.be/webtools/Venn/) 绘制韦恩图鉴定共同上调和下调基因。
从ICGC数据库(https://dcc.icgc.org/projects/LIRI-JP/)LIRI-JP肝癌数据集下载231例肝癌样本的表达数据和临床数据。这些样本主要来自日本乙型肝炎病毒(HBV)或丙型肝炎病毒(HCV)感染人群[9]。样本数据使用了标准化的计数值。
1.2 肝癌差异表达基因的通路和功能富集分析
利用Metascape网站[10]对差异表达基因进行通路和功能富集分析。基因GO功能注释及基因参与通路来源于以下数据库的并集:Kyoto Encyclopaedia of Genes and Genomes (KEGG) Pathway, Gene Ontology (GO) Biological Processes, Reactome Gene Sets, Canonical Pathways, CORUM, TRRUST, DisGeNET, PaGenBase, Transcription Factor Targets, COVID。将基因组中的所有基因作为富集背景。P值的计算基于累积超几何分布,q值的计算采用Benjamin-Hochberg (BH)进行多重检验[11]。最后使用Cytoscape可视化网络[12]。
1.3 肝癌细胞周期调控相关基因预后模型的构建和验证
采用单因素Cox回归分析细胞周期调控相关差异表达基因的预后价值。根据表达量的中位值将病人分为高表达组和低表达组,通过在线Kaplan-Meier plotter (http://kmplot.com/analysis/)进行Kaplan-Meier生存曲线验证[13]。使用Lasso Cox回归分析方法建立预后模型[14-15]。采用‘glmnet R’包使用LASSO算法进行选择和收缩自变量。根据中位风险评分将病人分为高风险组和低风险组。基于模型中的基因表达,采用‘stats’R包的‘prcomp’程序进行主成分分析(PCA);同样基于模型中的基因表达,采用‘Rtsne’R包中的t-分布随机相邻嵌入分析(t-SNE)方法,分析不同风险组的分布,确定各风险组的区分显著性。采用‘survminer’ R包的‘sur_cutpoint’程序来确定最佳截断表达值,进行Kaplan-Meier生存分析确定高低风险组的病人生存情况差异。使用单因素和多因素Cox回归分析确定模型风险评分是否为总生存期(OS)的独立预后因素。应用‘survival ROC’R包进行时间依赖性受试者工作特征(ROC)曲线分析,以评估模型基因集的预测能力。生成用于模型可视化和临床应用的列线图(Nomogram),应用校准曲线(Calibration curve)评价列线图的校准度,应用决策曲线分析(DCA)评价临床适用度。
1.4 样品采集和标准化处理
收集青岛大学附属青岛市市立医院肝胆外科3例确诊为肝细胞癌病人的肝癌组织和癌旁组织,样本采集和存储采用标准化的方法。对组织样本进行基因转录水平二代测序(NGS),对数据进行标准化处理,统计方法采用Mann-WhitneyU检验。
1.5 统计学分析
所有统计分析均使用R软件。除特殊标注外,计量资料比较采用t检验,计数资料比较采用χ2检验。应用Cox回归估计危险比(HR)和95%置信区间(CI)。生存分析采用Kaplan-Meier法,采用logrank检验确定差异是否有统计学意义。使用BH法校正P值。采用双侧检验,P<0.05为差异有统计学意义。
2 结 果
2.1 肝癌肿瘤组织和正常肝脏组织差异表达基因的筛选
TCGA数据库TCGA-LIHC肝癌数据集共筛选出3 619个差异表达基因 (|log2FC|>2, FDR<0.05),差异表达基因的热图和火山图见图1 A、B。 GEO数据库GSE36376肝癌数据集共筛选出687个差异表达基因 (|log2FC|>1, FDR<0.05)。应用韦恩图共同鉴定了141个差异表达基因,其中70个基因表达显著上调,71个基因表达显著下调。见图1 C、D和表1。
2.2 肝癌细胞周期调控相关预后基因的确定
通路及功能富集分析显示,肝癌差异表达基因共参与了409个重要功能及通路(图1),其中有95个通路和功能与肝癌细胞周期调控密切相关,通过统计归纳,最后确定了28个与肝癌细胞周期调控相关基因。见表2。单因素Cox回归分析显示,与肝癌预后相关的细胞周期调控基因有24个,其中包括CDC20、AURKA、NUSAP1、HMMR、TP2A和MDK等(HR>1,FDR<0.05)(图2A);基因表达热图显示了这些基因的表达水平(FDR<0.05)(图2B)。应用在线Kaplan-Meier Plotter分析验证肝癌病人细胞周期调控相关基因的预后价值,最终确定这24个细胞周期调控相关基因均与肝癌病人的预后显著相关(图2C)。
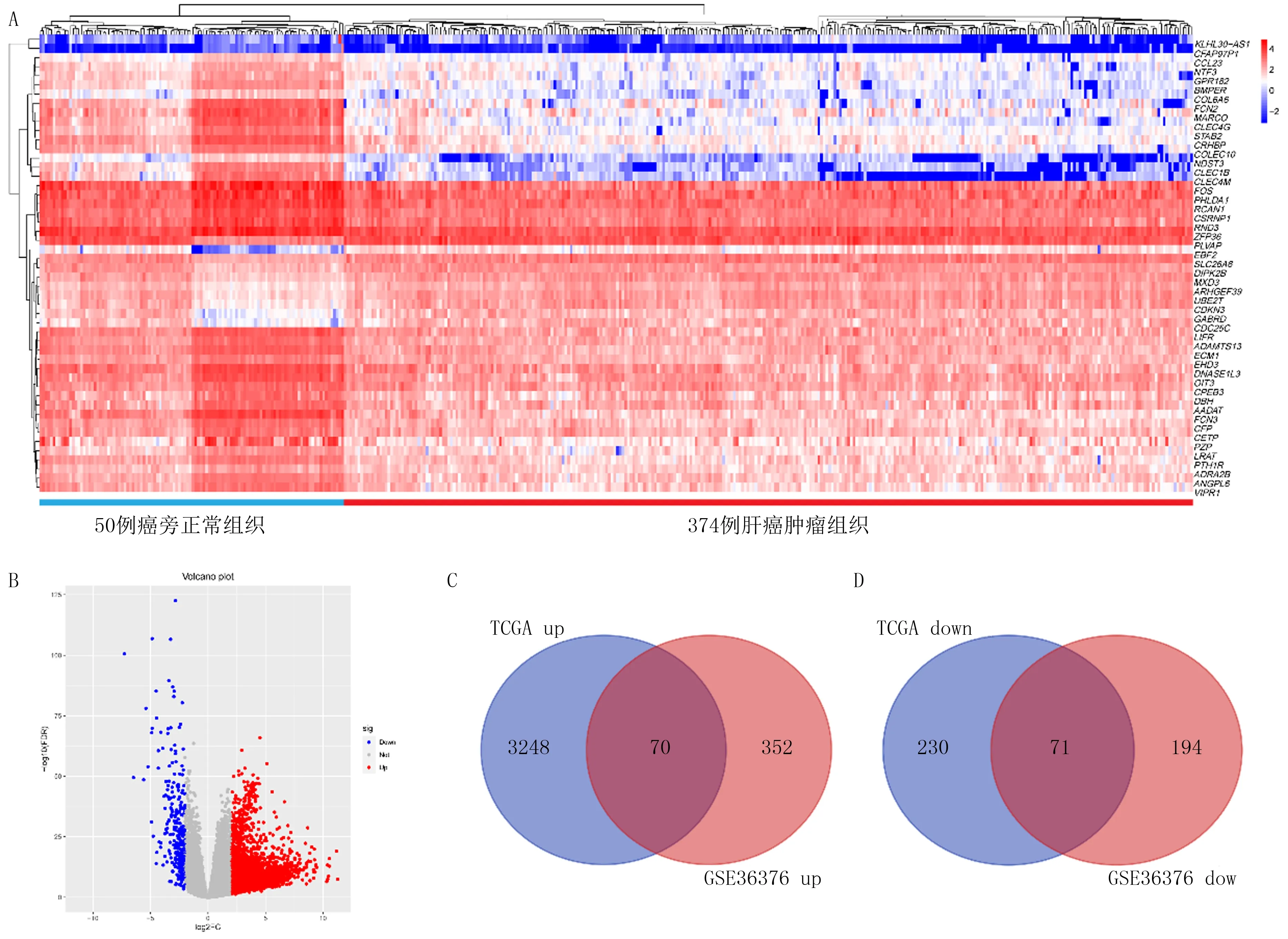
A、B:TCGA-LIHC肝癌数据集374例肝癌组织与50例癌旁正常组织差异表达基因热图和火山图;其中行代表基因,列代表样本;蓝色代表低表达,红色代表高表达。C、D:TCGA-LIHC肝癌数据集和GSE36376肝癌数据集中肝癌组织和癌旁组织差异表达基因的交集,其中70个基因表达上调,71个基因表达下调。
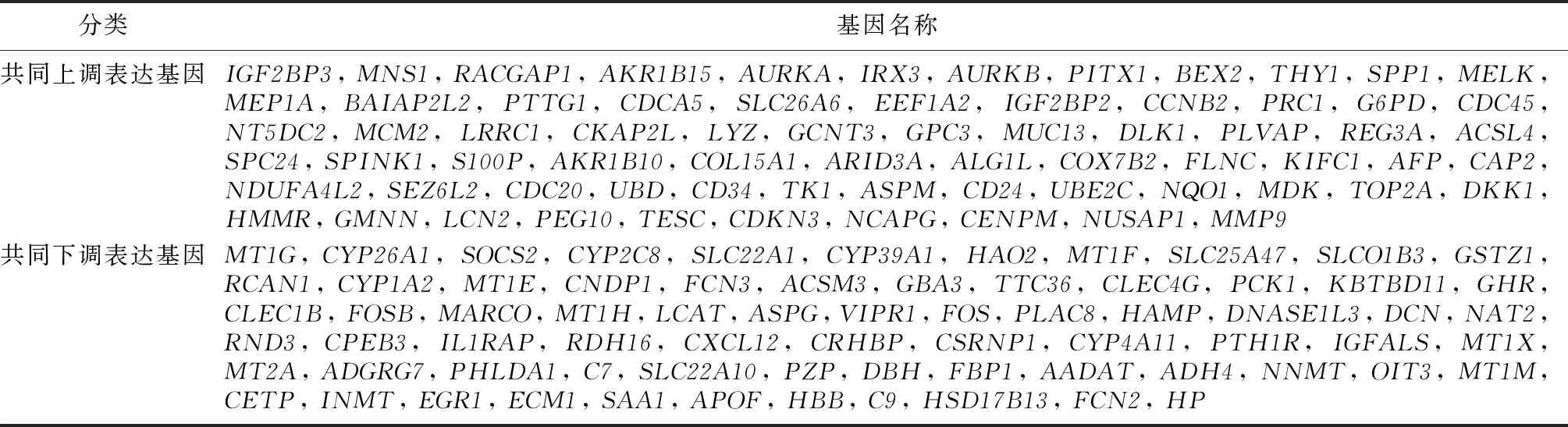
表1 TCGA-LIHC数据集与GSE36376数据集交集差异表达基因
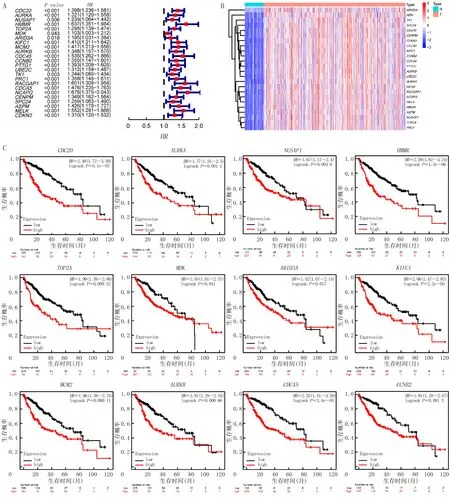
A:单因素Cox回归分析确定影响肝癌病人预后的细胞周期调控相关基因;B:影响肝癌病人预后的细胞周期调控相关基因在肝癌肿瘤组织和癌旁正常组织中表达热图;C:Kaplan-Meier Plotter在线分析验证细胞周期调控相关基因的预后价值。
2.3 肝癌细胞周期调控基因预后模型的构建
基于TCGA数据库TCGA-LIHC肝癌病人队列,用Lasso Cox回归分析建立预后模型。基于惩罚参数的最优值λ,确定了一个8个基因的基因集(图3)。风险评分计算方法如下:风险评分=e(0.319×CDC20表达量-0.393×NUSAP1表达量+0.438×HMMR表达量+0.066×ARID3A表达量+0.068×RACGAP1表达量+0.123×NCAPG表达量-0.141×SPC24表达量+0.004×MELK表达量)。根据其中位截断值,将病人分为高风险组(n=182)和低风险组(n=183)(图3A)。PCA和t-SNE分析显示,高风险组和低风险组病人离散方向不同(图3B、C),高风险病人早期死亡的可能性高于低风险病人(图3D)。Kaplan-Meier曲线分析显示,高风险组的OS明显低于低风险组(图3E,P<0.001),低风险评分的肝癌病人较高风险评分者有更好的预后。应用ROC曲线评估模型的预测能力,生存时间1年的ROC曲线下面积(AUC)为0.800(95%CI=0.737~0.863),2年为0.750(95%CI=0.687~0.813),3年AUC为0.731(95%CI=0.659~0.804),表明本文建立的预后模型具有良好的预后预测准确度和特异度(图3F)。利用TCGA队列中多因素Cox回归模型生成的系数,将风险评分与分期、分级、年龄和性别等重要的临床变量整合在一起,以进一步提高预后预测的准确性,建立了模型可视化和临床应用的列线图(图4A)。校准曲线检测出列线图预测与实际观测之间的最佳预测阈值(图4B)。最后,通过1、2和3年的DCA比较风险评分与其他临床指标的临床净效益(图4C~E),结果显示,在上述阈值概率的大部分范围内,风险评分显示出更大的净收益,表明风险评分在预测肝癌病人预后方面具有较好的临床应用价值。
2.4 肝癌细胞周期调控基因预后模型的验证
为了检验肝癌病人队列模型的稳健性,按照与TCGA数据库TCGA-LIHC肝癌病人队列构建模型的相同公式,将ICGC数据库LIRI-JP肝癌病人队列分为高风险组(n=182)和低风险组(n=78)(图5A)。PCA分析和t-SNE分析确定了病人在两个亚组中离散方向的分布,见图5B、C。与低风险组相比,高风险组病人早期死亡可能性更高(图5D),生存时间更短(图5E,P<0.001)。ROC曲线分析显示,生存时间1年的AUC为0.722(95%CI=0.584~0.861),2年为0.739(95%CI=0.633~0.845),3年为0.733(95%CI=0.627~0.839),预后模型具有良好的预测准确度和特异度(图5F)。
2.5 肝癌细胞周期调控基因预后模型风险评分的独立预后价值
单因素Cox回归分析显示,TCGA-LIHC肝癌病人队列(构建队列)和LIRI-JP肝癌病人队列(验证队列)的风险评分与OS之间存在显著相关性(构建队列:HR=3.767,95%CI=2.661~5.333,P<0.001;验证队列:HR=3.752,95%CI=2.240~6.266,P<0.001)。多因素Cox回归分析显示,风险评分是OS的独立预测因子(TCGA数据库肝癌病人队列:HR=3.436,95%CI=2.402~4.916,P<0.001;ICGC数据库肝癌病人队列:HR=3.264,95%CI=1.920~5.549,P<0.001)。见图6。
2.6 肝癌细胞周期调控相关预后基因的转录表达水平鉴定
本文NGS结果显示,包括CDC20、AURKA和NUSAP1等在内的16个细胞周期调控相关预后基因在肝癌中表达显著上调(图7)。
3 讨 论
肝癌等恶性肿瘤细胞的特点是无限增殖,这与细胞周期调控密切相关。尽管细胞周期调控的机制已经成为肿瘤研究的核心领域,但其具体机制仍不明确,细胞周期调控的机制以及相关基因对肝癌病人预后的预测价值也尚不清楚。既往的研究结果表明,基于p53相关的microRNA集合[5]、免疫相关编码基因集合[4]、CpG岛甲基化表型(CIMP)相关基因[16]、控制胚胎发育的claudin基因家族[17]等构建的肝癌预后模型显示了优秀的预测能力。与这些研究相比,本研究1、2、3年的ROC曲线及DCA曲线等结果均显示本文构建的预后模型具有良好的准确性、特异性及临床适用性,能够准确预测肝癌病人的预后。
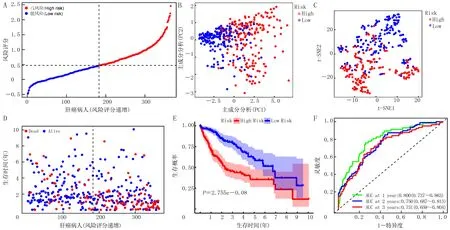
A:病人风险评分分布及中位值;B:PCA确定高风险组和低风险组病人离散方向的分布;C:t-SNE确定高风险组和低风险组病人离散方向的分布;D:高风险组与低风险组病人生存时间、生存状态散点图;E:高风险组和低风险组病人OS的Kaplan-Meier生存曲线分析;F:ROC曲线评估模型的预后预测能力。
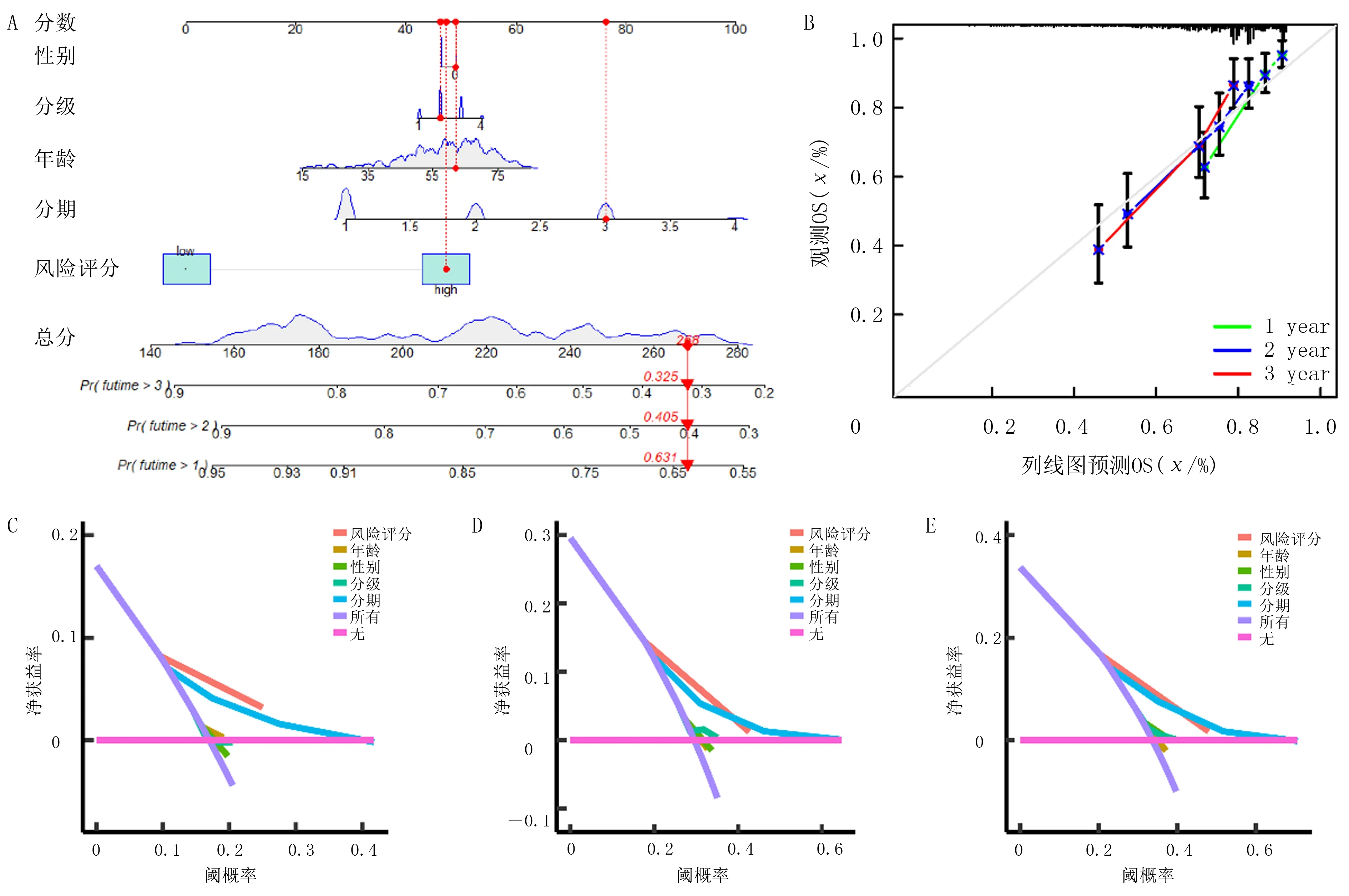
A:TCGA-LIHC肝癌病人队列风险评分及临床变量指标1、2和3年OS的复合列线图预测;B:1、2、3年观测OS概率和复合列线图预测OS概率的校准曲线;C~E:TCGA-LIHC肝癌病人队列风险评分及临床变量指标1、2、3年的DCA。
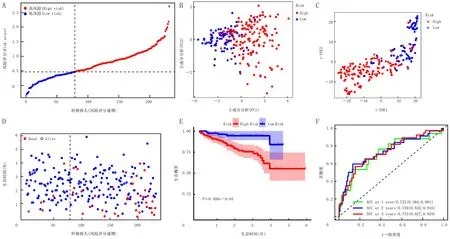
A:病人风险评分分布及中位值;B:PCA确定高风险组和低风险组离散方向的分布;C:t-SNE确定高风险组和低风险组两个亚组中离散方向的分布;D:高风险组与低风险组病人生存时间、生存状态散点图;E:高风险组和低风险组病人OS的Kaplan-Meier生存曲线分析;F:ROC曲线评估模型的预后预测能力。

A:构建队列风险评分单因素Cox回归分析;B:验证队列风险评分单因素Cox回归分析;C:构建队列风险评分多因素Cox回归分析;D:验证队列风险评分多因素Cox回归分析。

表2 28个肝癌细胞周期调控相关基因缩写及英文全称
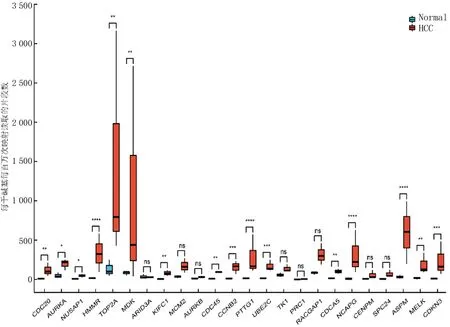
ns:P>0.05;*P<0.05;**P<0.01;***P<0.001;****P<0.000 1。
本文构建的预后模型中,参与模型的共有8个细胞周期调控相关基因,分别为RACGAP1、CDC20、NUSAP1、HMMR、ARID3A、NCAPG、SPC24和MELK。迄今为止的研究显示,其中6个致癌基因CDC20[18]、NUSAP1[19]、RACGAP1[20-21]、NCAPG[22]、MELK[23]和SPC24[24]已经在肝癌中被确定具有重要作用,但HMMR和ARID3A在肝癌中的作用尚不清楚。有生物信息学研究结果表明,HMMR可能是肝癌中较高表达的致癌基因[25]。本文研究表明,HMMR可能通过调控肝癌细胞周期影响病人的预后。此外,ARID3A基因在肿瘤中作用研究甚少,本文研究显示ARID3A可能通过调控细胞周期影响肝癌病人的预后。为了验证本文筛选出的预后基因的表达水平,我们使用NGS技术检测3例肝癌组织与癌旁组织基因表达,结果显示16个细胞周期调控相关预后基因在肝癌中表达显著上调,在转录水平上证明了细胞周期调控相关预后基因的作用。
综上所述,本研究成功构建了细胞周期调控相关基因的预后模型,为肝癌病人的预后及治疗提供新的方向。本文测序分析为后续的模型验证提供了转录水平表达数据基础,但仍需检测更多的组织样本进行验证,并进行更加深入的基础实验研究。
