基于SSR-HRM技术的向日葵DNA指纹图谱构建
2022-04-27刘苡含牟青山陈珊宇阮关海关亚静
刘苡含,牟青山,陈珊宇,阮关海,胡 晋,关亚静,*
(1.浙江大学 农业与生物技术学院,浙江 杭州 310058; 2.海南浙江大学研究院,海南 三亚 572025; 3.浙江省农业科学院 作物与核技术利用研究所,浙江 杭州 310021)
向日葵(L.)属菊科向日葵属,一年或多年生,是仅次于大豆、低芥酸菜籽和花生的世界第四大油料作物,目前在世界各地广泛种植。同时,它还是一种重要的观赏性植物,其花穗、种子皮壳和茎秆可作为饲料或工业原料,花穗亦可供药用,种子可供食用。我国有丰富的向日葵种质资源,主要种植区分布于东北、华北和西北的半干旱或轻盐碱地区。近几年,向日葵作为“美好作物”被江浙一带引进,在美丽乡村建设中发挥重要作用。但随着我国新审定和从国外引进的向日葵品种越来越多,市场上品种混杂、假冒伪劣种子频现和品种侵权的现象屡见不鲜,且很多品种间存在性状相似、命名混乱的状况。因此,为有效区分不同品种与真伪种子,有必要对向日葵品种进行遗传多样性分析并开展DNA指纹图谱构建工作。
种质鉴定的方法有很多,传统的鉴定方法一般采用田间种植鉴定,占地面积大、费时费工、受种植地光温条件限制,而且向日葵的表型性状复杂,田间表型性状鉴定品种难以在短时间内完成。DNA指纹图谱鉴定技术是目前较为先进的遗传标记系统,它能直接从DNA水平上反映生物个体之间的差异,具有简单稳定、变异性高、位点丰富、专一性高、特异性强等优点。简单重复序列(simple sequence repeat,SSR)标记因具有共显性、多态性丰富、稳定性高、操作简单可靠等优点,已广泛应用于水稻、花生、玉米、棉花、小麦、甘蔗等作物的品种真实性检测、遗传多样性分析和DNA指纹图谱构建中。目前,SSR扩增产物的检测方法主要采用聚丙烯酰胺-银染法,虽然此方法灵敏度和分辨率高、操作简单,但难以读取扩增片段大小,且在分析的材料和位点较多、片段差异较小时,数据的准确性会降低。高分辨率熔解曲线分析技术(high-resolution melting curve analysis,HRM)依靠核酸分子片段大小、GC含量等物理性质能够准确有效地区分SSR位点与不同基因型,识别产物片段中极小的碱基差异,是一种分辨率高且成本相对较低的DNA检测技术。本研究将SSR分子标记和HRM技术相结合,对向日葵的基因型进行快速鉴定,并使用非加权组平均法(unweighted pair group method with arithmetic averaging,UPGMA)对SSR-HRM原始数据进行聚类分析计算,依据分析结果构建DNA指纹图谱,以期为建立浙江省向日葵主栽品种的真实性和纯度鉴定体系,以及向日葵育种研究提供参考。
1 材料与方法
1.1 材料
供试的32份向日葵种质资源搜集于浙江省各县市,由浙江省农业科学院提供。种质编号、名称、类型、特性与来源信息见表1。
1.2 方法
1.2.1 DNA提取与纯化
从每个向日葵样品中随机取10颗种子播种,在合适的光温条件下土培至长出真叶,时间约2周。取真叶约0.5 g,液氮环境下使用组织磨样机研磨。采用CTAB法提取样品中总DNA。经琼脂糖凝胶电泳检测后,用NanoDrop1000分光光度计检测所提取DNA的纯度和浓度,原液置于-20 ℃保存,工作液DNA调整终浓度为100 ng·μL,作为模板用于后续试验。
1.2.2 SSR引物
查阅已公开发表文献中引物的多态性和扩增情况,结合位点的DNA序列,设计并合成了95对候选SSR引物。
1.2.3 SSR-HRM引物筛选
根据材料特性和来源随机抽取5份向日葵DNA材料(编号分别为2、7、15、24、31)进行引物筛选。PCR反应体系为20 μL:10 μL 2×PCRMasterMix (Vazyme),2 μL模板DNA,上、下游引物(10 μmol·L)各1 μL,6 μLddHO;反应程序为95 ℃ 5 min;95 ℃ 15 s,58 ℃ 15 s,72 ℃ 20 s,35个循环;72 ℃ 10 min,4 ℃保存;采用6%聚丙烯酰胺凝胶电泳,设置电压为200 V,电流为160 mA,电泳2 h左右,经快速银染显影后读取条带。选取条带清晰、多态性稳定的引物用于SSR-HRM分析与DNA指纹图谱构建。
1.2.4 SSR-HRM的反应体系和程序
SSR-HRM采用Touchdown快速PCR程序连同高分辨率熔解曲线的分析方法,以提取的32份向日葵DNA为模板,利用筛选出的17对核心SSR标记引物,在Light Cycler 480Ⅱ型实时荧光定量PCR仪上进行PCR扩增与熔解曲线分析。PCR反应选用LightCycler480HighResolution Melting Master Mix,体系为20 μL:Mix10μL,模板DNA 2 μL,上、下游引物各0.4 μL(10 μmol·L),dd HO 7.2 μL。PCR扩增程序为:95 ℃预变性10 min;95 ℃(20 s),62~50 ℃ 20 s,72 ℃ 20 s,55个循环,退火温度从第一个循环62 ℃开始递减,每个循环减1 ℃直到50 ℃不再变化;HRM程序为:95 ℃变性60 s,立刻冷却至40 ℃保持60 s,接着从65 ℃开始以0.02 ℃·s的速度升至97 ℃。每板跑32个DNA样品,用1对引物,每个样品3次生物学重复。
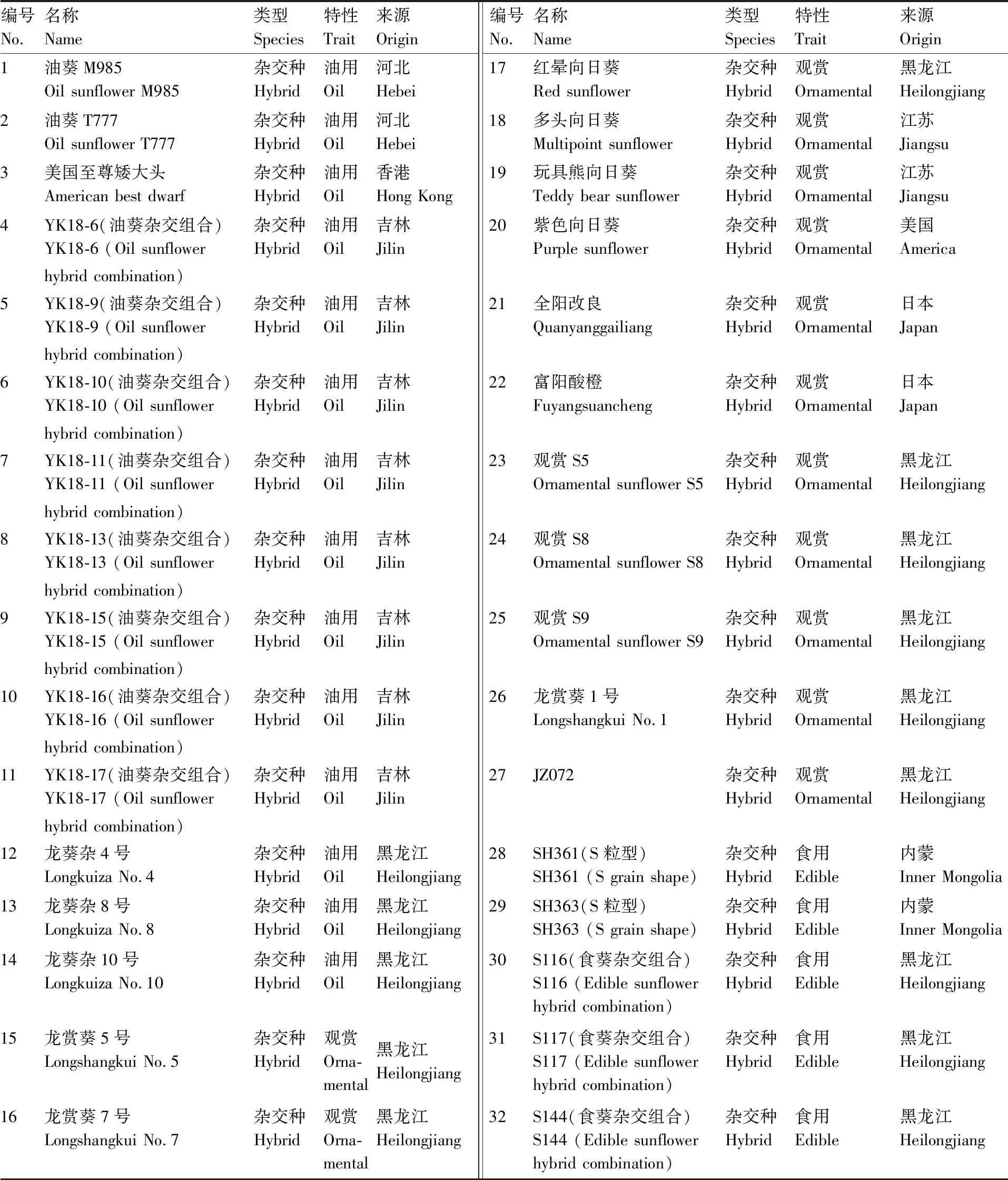
表1 三十二份向日葵种质资源信息
1.2.5 SSR-HRM数据分析
待反应程序结束,使用LightCycler96(Version 1.1.0.1320 Roche Applied Science,Mannheim,Germany)分析原始数据,分析时将delta Tm discrimination和curve shape sensitivity参数分别调至50%,得到每对引物反应下32份向日葵材料的基因分型结果并记录,重复性差的样品记为“0”。
1.2.6 向日葵亲缘关系分析
分析17对SSR引物对应的高分辨率熔解曲线,得出基因分型结果,然后导入NT-SYS 2.10e软件,采用非加权组平均法分析所有样品的亲缘关系,构建聚类树。
2 结果与分析
2.1 引物筛选结果
大部分引物均能扩增出清晰整齐、多态性高的条带,仅8对引物无扩增产物。根据引物筛选结果,选用条带清晰、多态性稳定的17对引物用于32份材料的SSR-HRM分析与DNA指纹图谱构建。筛选出的17对核心引物的扩增结果见图1,引物信息见表2。
2.2 基于SSR-HRM的遗传关系分析
32份向日葵种质的高分辨率熔解曲线分析结果表明,标记ORS543、ORS665、ORS229和ORS1114的多态性较好。以标记ORS665的SSR-HRM分析结果为例,32份向日葵样品的高分辨率熔解曲线均有差异,根据熔点等的不同,样品被分成7个基因型组别,每组的高分辨率熔解曲线在形状上明显不同(图2-A);结合差异图(图2-C)得到32份向日葵样品的基因分型结果(图3)。7组的代表品种分别是YK18-10、YK18-13、龙赏葵5号、YK18-9、S117、S116和S144,将它们的熔解曲线和差异图单独列出,可以更加清晰地观察到各基因型组别间的差异(图2-B和图2-D)。其余16对SSR标记的分析结果与标记ORS665类似,且均可有效区分供试的32份向日葵品种。根据17对SSR标记对32份向日葵样品的基因分型结果,构建了DNA数字指纹图谱(表3)。
2.3 基于SSR-HRM的聚类分析结果

M,DNA 2000 marker;ORS543、ORS456等为引物名称;图片中品种从左至右依次为油葵T777、YK18-11、龙赏葵5号、观赏S8、S117。M, DNA 2000 marker; ORS543, ORS456, etc. were the name of primers. The order from left to right represented oil sunflower T777, YK18-11, Longshangkui No.5, Ornamental sunflower S8 and S117.图1 核心引物筛选结果Fig.1 Screening result of core primers

表2 十七对SSR引物信息

A,32份向日葵种质资源的差异图,颜色相同的线为同一基因型组;B,7个基因型分组中各代表品种的差异图;C,32份向日葵种质资源标准化高分辨率熔解曲线图;D,7个基因型分组中各代表品种的标准化高分辨率熔解曲线图。A, Difference plots of 32 sunflower germplasm resources, and the same line color indicates the same genotype calculated by HRM analysis; B, Difference plots of the seven distinguished sunflower germplasm resources; C, Normalized HRM melting curve of 32 sunflower germplasm resources; D, Normalized HRM melting curve of the seven distinguished sunflower germplasm resources.图2 ORS665分子标记对32份向日葵种质资源的SSR-HRM分析结果Fig.2 SSR-HRM analysis of 32 sunflower germplasm resources with the SSR marker ORS665

图3 ORS665对32份向日葵样品的基因分型结果Fig.3 Genotype group of 32 sunflower germplasm resources with SSR marker ORS665
采用UPGMA法对SSR-HRM分型结果进行聚类分析,结果表明,17对引物可有效区分32份向日葵材料(图4),32份向日葵样品在遗传相似系数为0.20处被分为两大类群,分别记为A、B。其中,来自美国的观赏型紫色向日葵(Purple sunflower)独立组成B类群,它与A类群中同为观赏型的红晕向日葵(Red sunflower)和多头向日葵(Multipoint sunflower)亲缘关系最近,与来自河北的油用型品种油葵M985(Oil sunflower M985)亲缘关系最远。A类群分为多个亚类群,同一类型或相同地理来源的向日葵材料多聚类在一簇;不同类型和地理来源的材料间聚类相对较分散。14种油用向日葵材料中,来自吉林、黑龙江的大部分材料亲缘关系较为接近,说明其在育种上很可能存在性状相似、亲本来源较为狭窄的问题;13种观赏型向日葵材料间遗传多样性最高,其亲缘关系与地理来源无明显相关,说明国内外向日葵种质资源间具有丰富的遗传多样性;5种食用型向日葵材料亲缘关系与地理来源相关性一般,遗传多样性相对较高,但可能存在样本量小、不具有代表性的问题。

表3 三十二份向日葵材料的数字化指纹图谱

图4 三十二份向日葵品种基于SSR标记的聚类图Fig.4 Dendrogram of 32 sunflower cultivars based on SSR analysis
3 结论与讨论
我国存在向日葵传统地方品种老化、种植良种混杂、育成品种稀缺等问题,亟需进行种质筛选鉴定与创制工作。一个准确、快速、高效的分子鉴定技术对向日葵的品种鉴定具有重要意义。近年来,随着现代生物技术的不断发展,DNA指纹图谱在种质鉴定中发挥越来越重要的作用。SSR分子标记是构建DNA指纹图谱的首要选择,HRM技术在构建遗传指纹图谱等领域的应用也有较大的拓展。国内外已有大量学者将SSR与HRM技术融合应用于植物的品种鉴定。Pereira等将HRM技术应用于橄榄油和葡萄酒的真实性鉴定。An等将自主开发的丝瓜基因组SSR分子标记与高分辨率熔解曲线融合,成功应用于丝瓜的基因型分析。殷豪等在西洋梨中的研究也证明,HRM技术是SSR标记分型的有效手段。陈媞颖等应用HRM技术对已知的7对SSR引物进行筛选,为金银花种质鉴定做出贡献。赵均良等验证了HRM技术应用于水稻SSR、InDel和SNP标记基因型分析的可行性和可靠性。
本文基于SSR分子标记技术,对32份向日葵种质资源进行高分辨率熔解曲线分析和聚类分析。筛选出的17对可以有效区分32份向日葵样品的SSR引物,且聚类结果与样品的地域来源、表型特征、亲缘关系基本一致,这说明SSR-HRM技术在向日葵基因分型和种质资源的鉴定中是可靠的。32份向日葵种质资源中,同类型和同来源的材料间亲缘关系较为接近,也有部分不同类型、不同来源的材料间遗传相似系数较大,说明它们之间没有明显界限,推测其亲本来源可能较为狭窄,也间接说明SSR分子标记辅助向日葵育种是可行的。
在向日葵品种鉴定上,可以利用本研究筛选出的核心引物开展向日葵DNA指纹库的深入研究,建立完整的向日葵品种真实性和纯度鉴定体系。在向日葵核心种质创制方面,可以辅助利用SSR-HRM技术,避免重复无效育种,多拓宽亲本来源,多利用亲缘关系较远的野生种质和国外种质资源如紫色向日葵(观赏,美国)、富阳酸橙(观赏,日本)等,以提高向日葵育成品种的遗传多样性。
