龙眼SNP高密度遗传图谱的构建及单果质量QTL定位
2022-04-15张晨晨王佳卉刘丽琴王一承周建男胡小文石胜友
张晨晨,王佳卉,刘丽琴,王一承,周建男,胡小文,石胜友
(1 华中农业大学园艺林学学院,武汉,430070;2 中国热带农业科学院南亚热带作物研究所/农业部热带果树生物学重点实验室,广东湛江,524091)
单果质量是龙眼重要的果实品质性状之一,对龙眼单果质量性状进行精确的遗传解析,对于快速培育大果优质龙眼具有重要意义。龙眼单果质量是多基因控制的数量性状遗传[1],基于系谱的连锁作图是解析植物数量性状遗传机制的主要方法[2]。目前报道的龙眼遗传图谱均是由我国完成的。龙眼上构建的多为父、母本单个连锁遗传图谱[3-5],仅有1个整合图谱,总图距为2 060.6 cM,标记间的平均距离为4.5 cM,包含20个连锁群,定位到6个与单果质量相关的QTL位点[1]。前人构建图谱所用的作图群体规模较小(仅94株),使用RAPD、SRAP、AFLP、ISSR等传统分子标记进行图谱构建和QTL定位研究,此类标记本身存在缺陷[6],且连锁群与实际染色体数目不对应,致使图谱精度不高,无法准确、高效的鉴定QTL区段的候选基因。高密度遗传图谱与利用传统的分子标记构成的图谱相比,具有高通量,定位区间小等优势,有利于精准定位和候选基因挖掘的研究。本研究以龙眼优质品种“凤梨朵”和大果主栽品种“大乌圆”为亲本,杂交产生的200株杂交后代为作图群体,基于RAD-seq技术开发SNP标记构建高密度遗传图谱,并对单果质量性状进行定位研究,以期为未来龙眼单果质量等重要性状进行精细定位、图位克隆以及功能基因挖掘等研究提供有力的依据。
1 材料与方法
1.1 材料
以优质品种“凤梨朵”为母本,大果品种“大乌圆”为父本杂交所获得的200株F1(FD群体)及亲本为作图群体。于2002年杂交,实体材料均保存在中国热带农业科学院南亚热带作物研究所龙眼种质资源圃中。
采集龙眼自然转绿叶片放于锡箔纸中,随即置于液氮中,采集完毕后放于-80 ℃超低温冰箱中保存,提取群体DNA,使用0.8%琼脂糖凝胶电泳检测DNA质量,使用Nanodrop ND 2000分光光度计检测DNA浓度。
1.2 RAD文库的构建
RAD文库构建方法参考文献[7]。父母本及FD群体的每个样本取基因组DNA 1 μg用限制性内切酶EcoRI进行酶切,从而得到适合的marker密度,在酶切后的片段两端加P1 Adapter,把DNA片段pooling随机打断到一定的片段,电泳回收200~400 bp的龙眼片段,末端平化后加A,加入局部为双链分叉Y型DNA的P2 Adapter,PCR扩增两端分别含有P1和P2接头的tag序列,Cluster制备,上机测序。
1.3 RAD序列的分析和SNP分子标记的开发
将父母本和200个子代文库测序获得的原始数据进行过滤,过滤掉含有接头序列的reads pair,单端测序read中N含量超过该条read长度比例的10%的paired reads,及单端测序read中含有低质量(质量值Q ≤5)碱基数超过该条read长度比例50%的paired reads。将测序数据与参考基因组进行比对,参考基因组的大小为495 Mb。
1.4 龙眼高密度高精度遗传图谱的构建
利用高质量SNP标记,构建龙眼高密度高精度分子遗传图谱。使用Joinmap 4.1划分连锁群,对划分好的连锁群采用最大释然法进行排序,对排序结果进行校正,然后再使用回归算法进行排序,父本与母本图谱完成后,使用mergemap进行整合获得整合图谱。
1.5 龙眼单果质量QTL定位
6月中下旬果实成熟时,从植株树冠外围选取生长发育正常并有代表性的果实,每植株选取果实10个,用电子天平称重,取平均值,精确到0.1 g,对单果质量进行QTL分析,采用复合区间作图法,获得龙眼单果质量的QTL位点数量、位置等信息。
2 结果与分析
2.1 测序结果分析及龙眼SNP标记的开发
利用RAD-seq测序技术对“凤梨朵”ד大乌圆”200株杂交后代及父母本进行测序,202个样本中总测序量为232.3 Gb,平均每个个体的原始数据的碱基数量为1.15 Gb,碱基序列为150 bp。母本“凤梨朵”和父本“大乌圆”的标准文库分别含有原始数据的碱基数量3.08 Gb和3.21 Gb,鸟嘌呤胞嘧啶(GC)含量分别为35%和34.54%。经过过滤和筛选,“凤梨朵”和“大乌圆”分别保留高质量碱基数量3.04 Gb和3.14 Gb ,数据的有效利用率分别能够达到98.64%和97.88%(见表1)。
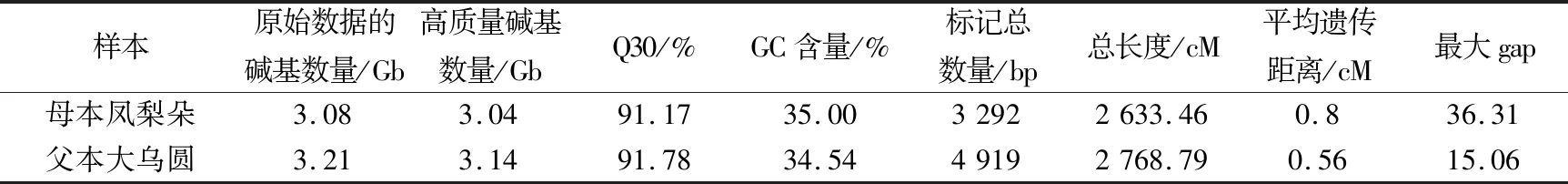
表1 龙眼样本测序结果和父母本遗传信息
经过数据过滤,获得SNP位点698 615个,包括lm×ll标记301 235个,占比43.12%,nn×np标记184 735个,占比26.44%,hk×hk标记123 178个,占比17.63%。在完整性过滤,卡方检验后,对30 844个SNP标记进一步分析。在这些标记中,18 663个杂合SNPs存在于“凤梨朵”lm×ll中,11 942个杂合SNPs存在于“大乌圆”nn×np中,239个杂合SNPs存在于双亲hk×hk中(见表2)。
在使用Joinmap 4.0测试标记分离率时,22 830个标记被排除在进一步分析之外,最后保留SNP标记8 014个,由4 722个lm×ll标记、3 095个nn×np标记和197个hk×hk标记组成,用于连锁测定后的遗传图谱构建。
2.2 龙眼高密度SNP遗传图谱构建
利用检测到的8 014个高质量SNP位点构建遗传图谱。构图时,分群LOD阈值设置为6.0,采用回归算法和Kosambi’s函数计算遗传距离。母本“凤梨朵”被划为15个连锁群,总遗传距离为2 768.79 cM,平均遗传距离为0.56 cM,包含SNP标记3 292个(见表1和图1);父本“大乌圆”被划分为15个连锁群,总遗传距离为2 633.46 cM,平均遗传距离为0.8 cM,包含SNP标记4 919个(见表1和图2)。使用MergeMap软件对父母本连锁群进行整合,成功构建了由15个连锁群组成的龙眼高密度遗传图谱(见图3),该图谱总的遗传距离为2 873.39 cM,每个连锁群的长度为113.93~442.99 cM,该图谱中长度最大的连锁群为LG5,长度最小的连锁群为LG12;该连锁群中共含有8 014个上图标记,每个连锁群的上图标记数量为312~1 075个不等,15个连锁群中上图标记数量最多的为LG5,上图标记数量最少的为LG12;该连锁群的平均遗传距离为0.36 cM,每个连锁群的平均遗传图距为0.27~0.41 cM,标记距离最小的连锁群为LG8,标记距离较大的连锁群为LG2、LG3和LG5。该图谱是目前龙眼上密度最高的龙眼遗传图谱。

图2 父本“大乌圆”龙眼遗传连锁图谱

图3 龙眼高密度SNP遗传图谱
2.3 单果质量性状表型分析
“凤梨朵”单果质量为9.15 g,“大乌圆”单果质量为15.27 g,FD群体单果质量最大值15.89 g,最小值4.68 g,平均(9.76±2.35)g,变异系数24.11%,偏度-0.52,峰度-0.04。FD群体的平均单果质量在母父本之间,表现为中亲优势。单果质量性状偏度和峰值的绝对值都小于1,符合正态分布规律,可用于单果质量的QTL定位。
2.4 单果质量QTL定位
本研究采用MapQTL 6.0中的区间映射(IM)模型进行QTL分析。基于200个的单果质量表型数据,结合SNP高密度遗传图谱进行QTL作图。结果可以看出,总共定位到QTL位点65个,分布于7个连锁群上,即LG1、LG3、LG4、LG5、LG8、LG10和LG14,其中,LG3和LG5上各检测出QTL位点1个,LG1、LG4、LG8、LG14、LG10上分别检测出QTL位点11、3、4、5和40个(见图4和表3)。

表3 单果质量在“凤梨朵”ד大乌圆”F1代中的QTL定位

图4 “凤梨朵”ד大乌圆”龙眼F1代单果质量QTL定位
3 结论与讨论
遗传图谱作为阐明数量性状遗传结构的重要工具,其密度对QTL位点的发现和精准定位至关重要[8]。研究表明,作图群体的大小会对遗传图谱的构建、QTL定位的准确性等造成一定的影响,且随着作图群体的增大,其准确性也随之上升[9]。当作图群体的规模超过200时,群体规模对遗传图谱构建和QTL分析准确性的影响较小[9-10]。此外,以现有的遗传图谱的间距(≥4.5 cM)实现图位克隆的难度较大[3]。因此,构建图谱群体数量大,密度高的龙眼高密度遗传图谱,对于实现龙眼目标性状的精准定位和分子标记辅助育种至关重要。

注:LG1—LG15表示15个连锁群。图1至图3同。图1 母本“凤梨朵”龙眼遗传连锁图谱
简化基因组测序技术在一定程度上增加了分子标记的数量和遗传图谱的密度,对于分析目标性状的遗传机制是有效的[11]。该技术目前广泛应用于非模式生物[12-13],如枣树[14],茶树[15],木本竹[16],鹅掌秋[17],杜鹃花[18],烟草[19]等。王中堂[20]利用GBS技术,成功地完成枣高密度遗传图谱构建,并定位到与6个叶片表型性状、1个针刺性状和 10个果实性状相关的235个QTL位点;孙磊[21]基于群体基因组重测序,开发大量SNP位点,构建了总图距为1 442.638 cM,平均遗传距离为0.928 cM的葡萄高密度遗传图谱,成功定位到与葡萄果实颜色和花色苷合成调控相关的10个QTL位点。本研究利用开发的SNP标记,成功构建了总图距为2 873.39 cM,平均遗传距离为0.36 cM的遗传图谱,与之前[1]构建的遗传图谱相比,我们构建的遗传图谱密度高,平均遗传距离更小,可用于龙眼单果质量等重要数量性状精细定位、图位克隆以及功能基因挖掘等。
