桃品种遗传多样性SSR分析
2022-04-07魏姗姗杨敏生梁海永
魏姗姗, 杨敏生, 梁海永
(1.衡水学院, 河北 衡水 053000; 2.河北农业大学, 河北 保定 071000)
桃(Prunuspersica)是蔷薇科(Rosaceae) 李属(Prunus)桃亚属植物,起源于中国的西部地区[1],在长期的人工选育过程中形成了适应不同生态环境、满足不同市场需求的品种,现分布在欧、亚、美、澳、非等五大洲,是一种在全球范围内广泛种植的落叶类核果果树[2-3]。相关研究表明,国内各种桃品种种质资源圃共收集保存了2 000余份桃种质资源[4],了解这些资源是我们更好利用桃种质资源的前提。从20世纪80年代初开始,国内已有不少科研工作者对桃的种质资源进行分类[5]。现在,已经可以从形态学、细胞学、分子标记等方面对桃品种进行分类和鉴定[6]。但上述方法不适合一些杂交的桃品种分类。因此研究桃品种的分子标记辅助分类具有重要的意义[7]。
简单序列重复(Simple sequence repeat,SSR) 具有标记多态性好、通用性强且稳定易重复等特点,广泛用于鉴定果树种质资源、分析果树亲缘关系[8]。但是,桃品种中已报道的SSR引物较少[9]。2013年,Verde等[10]将80 797条桃品种的ESTs 序列上传至 NCBI,为以后人们利用SSR标记对桃品种进行遗传多样性分析提供了宝贵的资源[11]。凌世鹏等[12]分析了53份桃资源的遗传多样性。包文泉等[13]对长柄扁桃10个野生群体遗传多样性和遗传结构进行聚类分析。本研究通过查找分布在桃品种8个连锁群中的SSR位点,利用Popgene软件对95个桃品种进行遗传多样性分析,以期更好地揭示桃品种中不同的生物学性状与连锁群间的遗传关系。
1 材料与方法
1.1 试验材料
95份供试材料主要来源于保定满城县林业局分院果树种子苗木站,所取材料及编号见表1。采集桃幼嫩叶片,以保鲜袋包装避免高温,编号后带回实验室,于-80 ℃冰箱中保存备用。
试剂及设备:2×TaqMaster Mix(是由TaqDNA Polymerase、PCR buffer、Mg2+、dNTPs以及PCR稳定剂和增强剂组成的预混体系)购自北京世纪生物科技有限公司;相关的PCR引物序列是由上海生工生物工程有限公司负责合成的;PCR反应由Biometer公司的TG型PCR仪完成。引物分别从桃的8对染色体上各选取4个SSR位点,从中筛选出18对引物。其序列如表2所示[14]。
1.2 试验方法
1.2.1DNA提取及纯化
采用改良的CTAB法提取桃的基因组DNA,进行PCR之前,用微量分光光度计检测DNA的质量与浓度。并将DNA稀释至30 ng/μL以备用。
1.2.2PCR扩增反应
PCR反应总体积为10 μL,包括:4 μL 2×TaqMaster Mix引物0.5 μmol/L,1 μL模板DNA。SSR反应程序为:95 ℃预变形5 min,94 ℃变形50 s,53~60 ℃退火50 s,72 ℃复性50 s,30个循环,72 ℃延伸7 min,最后置于4 ℃冰箱保存。
1.2.3电泳检测
将PCR产物与1/2体积的6×Loading Buffer充分混匀,取1.5 μL上样在8%的变性聚丙烯酰胺凝胶上,之后用230 V电压进行60 min电泳。结束后,取下凝胶,放在0.1% AgNO3中银染10 min;之后倒掉液体,用水洗去残液。再将凝胶放在含有1.5% NaOH和0.4%甲醛的显色液中直至条带清晰为止。取出凝胶,并对其拍照记录[15]。
1.3 数据分析
假定凝胶上相同位置的条带是来自于同一连锁群上的同一个等位基因。按基因型统计,从下往上,条带依次记作A、B、C、D、E、F、G、H。对每个样品的扩增电泳谱带进行统计,构成基因型的原始数据矩阵。假定桃品种中各个等位基因的频率与哈迪-温伯格平衡相符。利用Popgene软件,计算桃品种间的观测杂合值、观测纯和值、Nei’s基因多样度、Shannon信息指数、期望杂合值、期望纯和值、等位基因频率及群体间遗传距离。
2 结果与分析
2.1 连锁群遗传分析
如表3所示,桃品种8个连锁群观测到的等位基因数在4~9之间,有效等位基因数从2.2~5.5。第7连锁群的观测纯合值最高,为0.89;其有效等位基因数最少,为2.2。第6连锁群的观测纯合值最低,为0.47;其有效等位基因数最高,为5.45。说明桃品种的7号连锁群上的等位基因较少且纯合较多,表明其连锁群的保守性较高,不易突变。而桃品种的6号连锁群上的等位基因较多且宜杂合,表明其连锁群的保守性较差,易发生突变。从表2~表10中可以看出,所含有的有效等位基因数目从小到大依次为:7=2<4<8<1<5<3<6;桃品种8个连锁群的纯合度依次为:7>2>1>3>8>5>4>6。其中,第4连锁群和第5连锁群较为特殊。第4连锁群有效等位基因数较多,其有效等位基因数较少,其纯合度较低,即杂合度较高;第5连锁群恰好相反,其有效等位基因数较多,其纯合度较高,即杂合度较低。

表1 试验材料

表2 试验SSR引物

表3 不同连锁群桃品种的遗传多样性
从表4可以看出,等位基因A、B、C和D是桃品种中分布最广的四种基因,在桃的8个连锁群中均有分布。等位基因E仅次于上述四种等位基因,分布在除第7连锁群外的其余连锁群上。等位基因H和I为6号连锁群特有基因。从分布较广的四种基因来看,等位基因C的频率较高,为0.27,等位基因A的频率较低,为0.11。从基因在连锁群上的分布情况来看,等位基因A在第6连锁群的频率最高,为0.31,在第7和第8连锁群的频率最低,为0.03;等位基因B在第4连锁群的频率最高,为0.55,在第7连锁群的频率最低,为0.03;等位基因C在第2连锁群的频率最高,为0.63,在第6连锁群的频率最低,为0.02;等位基因D在第7连锁群的频率最高,为0.49,在第2和第6连锁群的频率最低,为0.06;等位基因E在第8连锁群的频率最高,为0.36,在第2连锁群的频率最低,为0.01;等位基因F在第5连锁群的频率最高,为0.22,在第3连锁群的频率最低,为0.09;等位基因G在第6连锁群频率较高,而等位基因H和I仅在6号连锁群出现。

表4 不同连锁群桃品种等位基因频率
2.2 果实形状类群遗传分析
表5所示,球形和扁球形两个类群观测到的等位基因数在4~5之间,有效等位基因数在2.7左右。两个类群从杂合值和纯合值可以看出,球形果实品种纯合子较多,而扁球形果实品种杂合子较多。基于桃果形性状的类群遗传分析中,扁球形类群的Nei’s基因多样度高于球形类群,说明扁球形类群遗传多样性更为丰富。

表5 不同果实形状桃品种的遗传多样性
表6列出了球形和扁球形两个桃类群的平均等位基因频率。通过分析比较可以看出,扁球形类群等位基因频率分布较球形类群更为均衡,说明扁球形类群杂合性偏高。等位基因B的频率在两个类群中同为最高。通过对表中数据分析可知,球形类群等位基因频率顺序与扁球形类群差异较大,这说明两者亲缘关系较远。

表6 不同果实形状桃品种等位基因频率
从遗传一致度(表7)看,两个类群的遗传一致度很高,为0.96,反映出连续分布类群间有较小的遗传分化特点。

表7 两类群间的Nei氏遗传一致度的无偏估计值
2.3 果实表皮有无毛类群遗传分析
表8所示,有毛和无毛两个类群观测到的等位基因数在4左右,有效等位基因数都是2.8。综合表中数据可知,两类群纯合子较多,所占比例接近。基于桃有无毛性状的类群遗传分析中,有毛类群的Nei’s基因多样度和Shannons遗传表型指数均与无毛类群接近,说明两者遗传多样性相似。
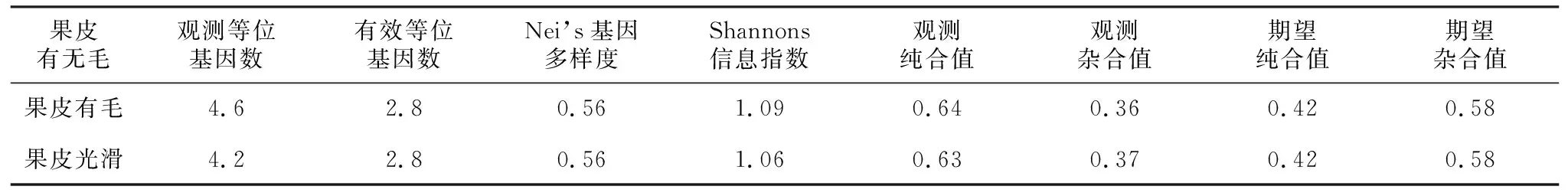
表8 果皮有无毛桃品种类群的遗传多样性
如表9,等位基因C的频率在两个类群中同为最高。等位基因E与H,B与D,A与F在两类群中的等位基因频率顺序恰好相反,据此推测等位基因E,B和A可能与有毛性状相关,等位基因H、D和F可能与无毛性状相关。

表9 果皮有无毛桃品种类群的等位基因频率
如表10,两种群的遗传一致度很高,为0.93,反映出连续分布种群间有较小的遗传分化特点。

表10 两类群间的Nei氏遗传一致度的无偏估计值
3 讨 论
SSR分子标记技术由于其共显性强、检测方便且便于重复等特点,已成为果树育种中最重要的分子标记之一[15]。目前,SSR分子标记技术已成功用于桃的遗传分析及品种鉴定[16]。本研究采用来自桃8条染色体18对核心引物,对95份桃品种的基因连锁群和遗传性状进行遗传分析。更好地区分桃已有的不同性状,为桃种群分类提供更好的分子鉴定体系。
遗传多样性是物种长期进化的结果,是种群生存和发展的前提[17]。通过Nei’s基因多样度、Shannons信息指数不难看出,桃品种群体的8个连锁群中7号连锁群上的等位基因较少且纯合较多,表明其连锁群的保守性较高,不易突变。而桃品种的6号连锁群上的等位基因较多且易杂合,表明其连锁群的保守性较差,易发生突变。等位基因A、B、C和D是桃品种中分布最广的三种基因,在桃的8个连锁群中均有分布,为未来桃品种的连锁群基因分析奠定了基础。
基于桃果形性状的类群遗传分析中,扁球形类群的Nei’s基因多样度略高于球形类群,Shannons遗传表型指数极为接近,球形的为1.06,扁球形的为1.05,说明扁球形与球形的品种类群遗传多样性比较均衡,果实的形状在选种时并没有作为重要的指标。
基于桃有无毛性状的类群遗传分析中,有毛类群的Nei’s基因多样度和Shannons遗传表型指数均高于无毛类群,说明有毛类群的遗传多样性和基因杂合度高于无毛类群。SSR分析中最重要的环节是引物的筛选,只要能开发出合适的引物,能够广泛地覆盖不同的染色体,并对分析体系进行优化,就能挖掘SSR在种质资源研究中的应用潜力。
