基于神经网络的LncRNA 与蛋白质互作关系预测算法*
2022-04-07李巧君李江岱王爱菊
李巧君 李江岱 王爱菊
(1.河南工业职业技术学院电子信息工程学院 南阳 473000)(2.郑州工程技术学院信息工程学院 郑州 450000)
1 引言
长链非编码RNA(Long non-coding RNA,LncRNA)是一种不具有显著开放性读码框而长度大于200 个核苷酸的非编码功能细胞内源性RNA[1]。与信使RNA(mRNA)相比,由于LncRNA 拼接效率较低常被认为是转录噪声,然而,实验证明LncRNA 在植物的发育、激素依赖性信号传导和胁迫反应中具有不可或缺的作用[2],特别是LncRNA 与蛋白相互作用与基因表达调控和植物抗病等细胞过程有关。LncRNA 均是通过与相应的RNA 结合蛋白的相互作用而发挥作用的,RNA 结合蛋白也可以与不同的LncRNA 相互作用,调节不同的细胞过程[3]。因此,识别潜在的LncRNA 与蛋白质相互作用对于理解LncRNA功能至关重要。
目前,对于LncRNA 和蛋白质相互调控机制的研究大多集中在动物和人类癌症方面,在植物中还没有广泛的研究,为深入探索LncRNA 和蛋白质的相互作用,本文借鉴PLRPIM[4]方法,使用K-mer 和One-hot 分别提取LncRNA 和蛋白质的数字向量,利用栈式自编码器(Autoencoder,AE)[5]和融合神经网络分别提取特征向量,对特征向量进行点乘方法形成整体特征的融合矩阵,最后通过训练以整体特征为输入并且融合了注意力机制[6]的深层网络结构,获得了具有期望功能的预测模型。该模型结合卷积神经网络(ConvoLutionaL NeuraL Networks,CNN)[7]和长短期记忆网络(Long Short-Term Memory,LSTM)[8]的不同优势,充分获得具有时间依赖和参数共享特点的更加高级的特征,实现了对LncRNA和蛋白质互作关系的关联预测。通过以玉米和拟南芥为样本的试验,可以看出本方法具有较为稳定且良好的表现。
2 相关工作
预测LncRNA与蛋白质的相互作用研究一般分为实验法和计算预测两种方法。2015年Marinbejar和Huarte 提出RNA 下拉法(RNA-puLLdown)[9],2016 年GagLiardi 和Matarazzo 提出RNA 结 合蛋 白免疫共沉淀技术(RIP)[10]等,这些均是通过实验方法获取相互作用,传统的湿实验方法不仅耗时费力,在实验过程中仅有少量的LncRNA 与蛋白质相互作用关系被证实,所以使用计算预测的方法来作为LncRNA-蛋白质互作研究的补充机制显得尤为重要。
深度学习(Deep Learning,DL)方法已被研究人员广泛应用于人类和植物疾病中的分子机制[11]。2011 年,MuppiraLa 等提出了一种名为RPISeq 的方法,该方法提取了3-mer 和4-mer 序列特征来训练RF和SVM模型,用于预测蛋白质-RNA相互作用[12]。2013年,王等基于朴素贝叶斯(NB)和扩展的NB分类器,提出了一种预测蛋白质和RNA 之间相互作用的模型[13]。2016 年,Pan 等开发了一种基于序列的方法IPMiner,基于堆叠式自动编码器预测LncRNA-蛋白质相互作用[14]。2018 年Yi等提出了基于堆叠式自动编码器和RF 的RPI-SAN 用于LncRNA-蛋白质相互作用的方法[15],同年,Hu 等提出了一种新的工具HLPI-EnsembLe,该工具基于SVM、极端梯度增强(XGB)和RF 来预测人类LncRNA-蛋白质相互作用[16]。
以上的方法均与序列的生物学或理化性质有关,但是通常不同物种中的生物性质和特点会有所不同,因此,利用生物特性作为特征用于预测是蛋白质和LncRNA 否具有关联性的方法可能在不同物种中的性能会有较大差异,所以寻找一个以大部分物种共性为特征的新方法,可能有助于预测模型获得更好的泛化性能。本文提出了一种基于学习的混合方法,使用融合神经网络预测LncRNA 和蛋白质关联作用,称为PIPAFNN,在拟南芥和玉米两个数据集上的实验结果表明,我们的方法优于RPISeq-RF、RPI-SAN和IPMiner方法。
3 数据预处理
3.1 数据集与实验环境
本模型在Python 3.7.3 环境下利用Keras 2.3.1实现,选取拟南芥和玉米的LncRNA 及其结合蛋白质的序列数据作为样本数据集。数据集源自植物LncRNA 数据库(PLncRNADB),网站:http://bis.zju.edu.cn/PLncRNADB。拟南芥拥有390 个LncRNA和163 个RNA 结合蛋白,包含948 个阳性样本(互动对),玉米拥有1107 个LncRNA 和190 个RNA 结合蛋白,包含22,133 个阳性样本。通过将蛋白质与LncRNA 随机配对并进一步去除现有的阳性对,拟南芥包含2867 个阴性样本,玉米包含24361 个阴性样本。
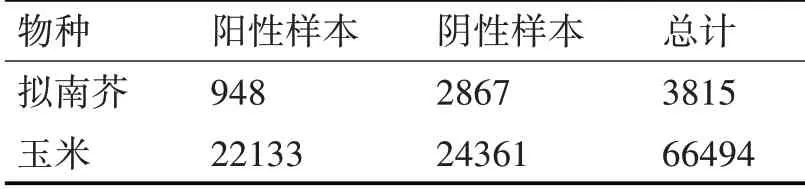
表1 拟南芥和玉米样本数据集统计
3.2 数据预处理及特征编码
1)K-mer 矩阵
特征是LncRNA 和蛋白质的基于序列的整合属性,这些属性编码为用于预测的数字载体。本文选择k-mer 模型从LncRNA 和蛋白质中提取特征,其中遗传序列子集S的长度用一个整数k表示。为了获得高效的特征,我们从由LncRNAs 和蛋白质的各种性质编码的特征向量中提取了一组599 个描述子。从LncRNA 序列中共获得256 个特征,从蛋白质序列中获得343个氨基酸描述符。
我们通过从左到右搜索每个序列提取RNA 序列(A,C,G,T)的4聚体稀疏矩阵,得到256(4×4×4×4)特征图。对于蛋白质序列,我们根据它们的化学相似性来划分氨基酸组成。根据偶极矩(<1.0,<1.0,(1.0,2.0),(2.0,3.0),>3.0,>3.0,and<1.0)和链体积(<50,>50,>50,>50,>50,>50,>50,>50和<50)对蛋白质序列的7 组物理化学性质{VaL,GLy,ALa},{Phe,Pro,Leu,ILe},{Ser,Tyr,Met,Thr},{His,Asn,Tpr,GLn},{Arg,Lys},{GLu,Asp}和{Cys}进行编号,提取3聚体标记,形成343个(7×7×7)稀疏矩阵特征图。
2)One-hot 编码
本文除K-mer 矩阵外,还使用One-hot 方法来获取序列的可计算特征。One-hot 就是每个位点只具有一个热点的信息提取方法。本文的每个LncRNA 和蛋白质样本数据,在One-hot 编码后可分别得到大小为4 × L 和20 × L 的特征矩阵。由于相互作用的LncRNA 和蛋白质片段均为不定长的序列,这给后续的模型计算和预测研究造成了很大阻力,我们通过利用K-mer 和One-hot 补0 的方法对序列文本信息进行编码,即可将变长的序列转化为定长的特征矩阵,以便输入到后续的特征提取和模型学习。
4 PIPAFNN模型
本文提出的PIPAFNN 模型由特征提取、特征融合、注意力机制和评分预测四个阶段组成。模型的整体结构如图1所示。
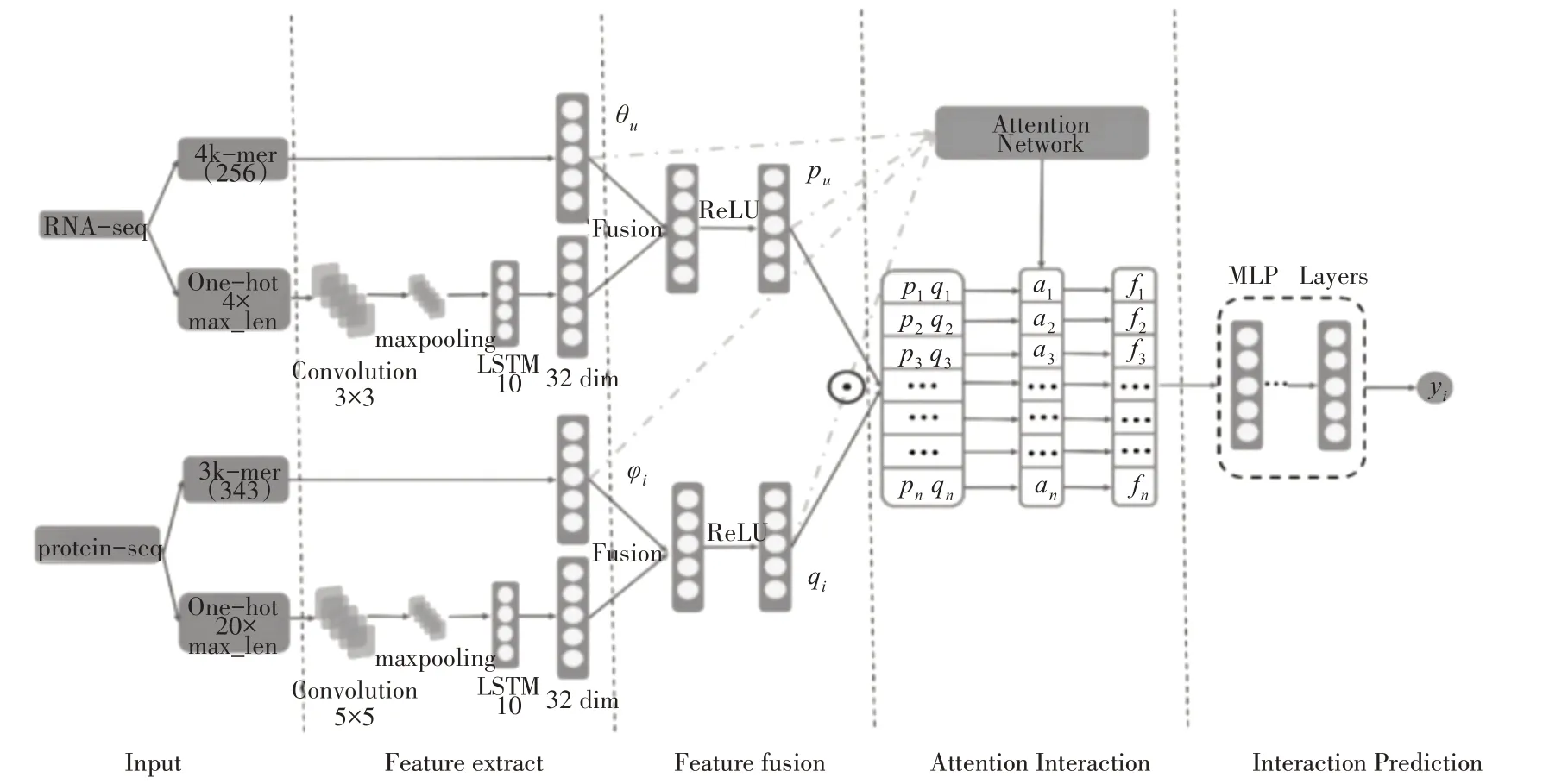
图1 模型整体结构图
4.1 特征提取阶段
本文使用栈式自编码器和融合神经网络分别对两种特征向量进行特征提取。我们采用单层栈式自编码器将LncRNA 和蛋白质由K-mer 特征编码得到的稀疏矩阵进行压缩,得到大小为32 维的特征矩阵。为了便于区分,此处将压缩LncRNA 得到的特征矩阵记为θu,而对于蛋白质得到的特征矩阵记为φi。其中θu代表样本中第u 条LncRNA 的自编码器特征矩阵,φi代表样本中第i 条蛋白质经自编码器提取出的特征矩阵。
本文运用CNN-LSTM 融合神经网络对经过One-hot 处理的特征矩阵进行特征提取,结合CNN和LSTM 的不同优势,获得具有时间依赖和参数共享特点的更加高级的特征。在模型中,对LncRNA用大小为3×3,步长为1 的卷积核进行卷积,并用最大池化对数据降维,一共经过三次卷积层和池化层交替得到更加显著的深层信息,并且在经过展开后接入到到LSTM 层中,进行以ReLU 为激活函数的更加精确学习,最后再加入全连接层将其展开为32 维,以对应用自编码器提取出的特征大小,便于后续的特征融合。对于蛋白质也采用同样的流程,有所不同的是蛋白质中对应的卷积核大小为5×5。
4.2 特征融合阶段
特征融合部分将嵌入的特征和基于回顾的特征进行融合,以便更好地进行表征学习。在以往的研究中,将基于评分和基于评论的特征相结合的策略被广泛采用来提高推荐性能。加法融合方法已经在RBLT 和ITLFM 中得到应用,为了获取更佳的预测效果,我们在加法融合之后直接添加一个全连接神经层,全连接层采用非线性ReLU 激活函数。在实验过程中,我们发现附加层可以有效地提高性能。
在经过两种不同的管道分别对LncRNA 和蛋白进行特征提取后,它们均得到两个类别的特征。分别将两者的两个特征进行融合,得到LncRNA 的整体特征pu以及蛋白质的组合特征qi,pu代表第u个LncRNA 样本的特征矩阵,qi代表第i 个蛋白质样本的特征矩阵。最后再将LncRNA 和蛋白质的特征矩阵都结合起来,形成一个总体的样本特征矩阵。
4.3 注意力机制阶段
Mnih 等在2014 年提出了注意力机制,以观察使用者在其关注项目中更加注重的特征,同时对关注度有所差异的属性赋予不同的关注向量。
本文将注意力机制应用于LncRNA 与蛋白质互作的预测模型中,通过将在历史学习中得到的信息添加到模型里,以识别在预测中对于不同样本具有突出贡献的特征空间中的不同主要属性,并对其赋予不同的关注度,形成具有特征偏好的模型,获得更优的预测效果。注意向量是在将自编码器得到的LncRNA 和蛋白质特征加上融合后的特征矩阵作为注意向量的输入后,经过权重和偏置运算,在经过激活层后被赋予输出权重得到的,详见式(1)。其中au,i即为期望的注意向量,θu、φi、pu、qi四者的联合向量是输入层的输入,Wa为输入层的权重矩阵,ba则为偏置向量,激活函数为ReLU,vT为输出权重。而含有栈式自编码器特征和含有历史信息的CNN-LSTM 提取特征的样本融合特征矩阵也作为感知器的输入,将学习到的注意力加权到样本的特征属性中去,最终得到模型的预测打分,见式(2)。F 为互作特征,由注意向量点乘对应样本的LncRNA 和蛋白质融合特征向量得到。
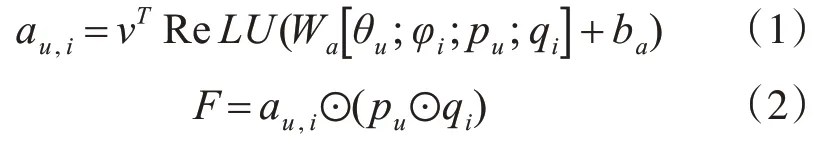
4.4 评分预测阶段
评分预测部分本质上是一个多层感知机(MuLti-Layer Perceptorn,MLP)。该部分将得到的交互特征向量F按如下方式馈入全连接层。
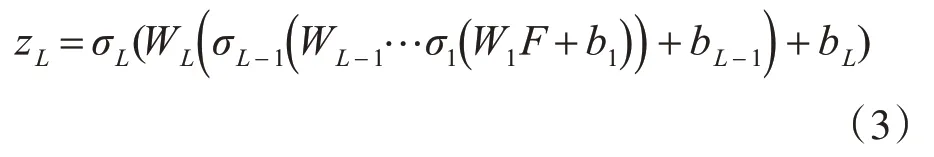
L为隐藏层数,WL,bL和σL分别是第L层的权值矩阵、偏置向量和激活函数。我们对所有层采用ReLU激活函数。预测等级r̂u,i通过回归层得到。

其中W和b分别为权值矩阵和偏差向量。
4.5 模型实现
PIPAFNN 模型首先将K-mer 的向量矩阵输入到栈式自编码器中进行特征提取,获得一个大小为32 维的特征矩阵,而One-hot 矩阵则运用CNN-LSTM 融合神经网络来获得特征向量,对LncRNA 用大小为3 × 3,步长为1 的卷积核进行卷积,经过3 次卷积层和池化层交替得到更加显著的深层信息,展开后接入到LSTM 层中,进行以ReLU为激活函数的更加精确学习,再加入全连接层将其展开为32 维,对蛋白质设置卷积核大小为5 × 5。将LncRNA 和蛋白质分别通过两个途径获得的特征进行融合,经过ReLU激活层后,把LncRNA 和蛋白质的特征向量进行点乘,得到一个包含LncRNA和蛋白质整体特征的融合矩阵,最后通过训练以整体特征为输入且融合注意力机制的深层网络结构,获得具有期望功能的预测模型。
5 结果分析及对比
为了验证模型预测的结果是否准确可靠,本算法运用五折交叉验证方法:通过随机函数得到互不相交的5 个子数据集,将其中4 个子集用于模型训练,而剩余未用于训练的一个集合,即为常说的测试集,用于预测模型的运行结果,此过程重复五次,最终得到五次验证结果的平均值,即可视为是较为稳定且可靠的评估数据。通过多次重复实验,模型对拟南芥和玉米正负样本比按照1∶1 的比例进行实验并得到相应结果,选取准确率(ACC)、精确率(PRE)、召回率(RecaLL)、特效度(SPE)、接受者操作特征曲线(ROC)下的面积(AUC)作为评价指标。
我们将PIPAFNN 模型与另外三种基于序列的计算模型RPISeq-RF,RPI-SAN 和IPMiner 进行比较,比较各种模型在准确率、精确率、召回率、特效度和AUC 方面的表现,见表2。在准确率方面,PIPLPFNN 表现较好,对两种植物的准确率分别为91.61%和85.72%。如图2(a)所示,拟南芥在PIPLPFNN,IPMiner,RPISeq-RF 和RPI-SAN 的AUC 值分别为0.9582,0.8823,0.8761 和0.8164。对于玉米数据集,AUC 值分别为0.9251,0.9034,0.8980和0.8792,如图2(b)所示。
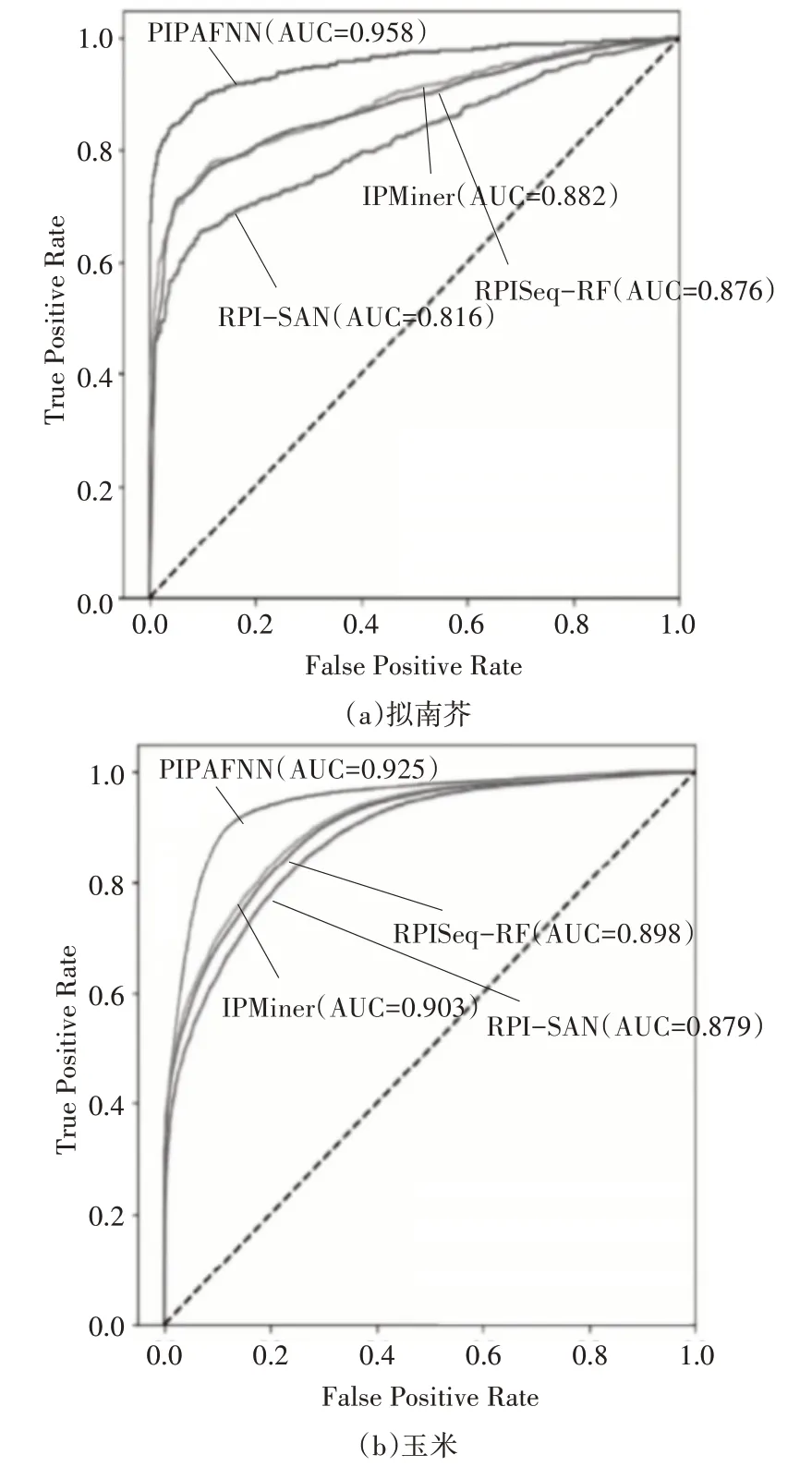
图2 不同方法在拟南芥和玉米数据集上的ROC曲线
通过利用稀疏约束的性能优势,PIPAFNN 模型学习了最丰富的序列特征信息。在表2 中,本方法在拟南芥和玉米数据集的准确率、精确率、召回率、特效度和曲线下面积(AUC)方面都优于其他方法。
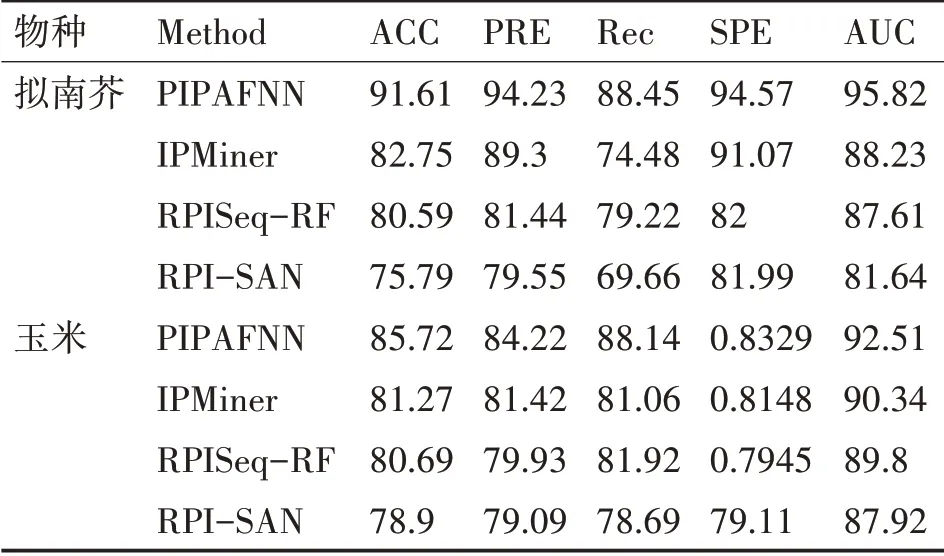
表2 其他方法和PIPAFNN方法的预测性能(%)
图2(a)显示本方法在拟南芥数据集上的AUC方面有更好的性能,与其他方法相比,AUC 提升了7%。图2(b)显示我们的方法在玉米数据集上AUC方面具有更好的性能,与其他方法相比,该方法的AUC提高了2%,表明模型的分类效果十分显著。
6 结语
本文提出了一种预测LncRNA 和蛋白质相互作用的新方法PIPAFNN,该方法利用CNN-LSTM融合神经网络应用于特征提取,将注意力机制应用于模型预测,提升了模型的学习性能,与其他方法相比,预测性能得到明显提升。通过充分利用多个分类器,该方法对基于基因组序列的LncRNA-蛋白质相互作用预测具有很高的成功率。但是,该方法仍有一些潜在的限制需要解决,首先,由于已知LncRNA-蛋白质互作关系稀疏,因此不同物种的植物LncRNA 相关蛋白的研究程度受到限制;其次,数据集数据的偏差可能会影响植物中LncRNA 与蛋白质之间相互作用概率的测量,因此,掌握具有更多经过实验验证的数据源会进一步提高模型性能。
