中华按蚊几丁质酶基因家族的全基因组鉴定与分析
2022-04-02刘燕邱品品刘蒨莹向凯杨小林乔梁
刘燕 邱品品 刘蒨莹 向凯 杨小林 乔梁
(重庆师范大学生命科学学院, 重庆 401331)
几丁质是一种线性多糖,由N-乙酰-D-葡萄糖胺通过β-1,4-糖苷键聚合而成,主要以α-几丁质、β-几丁质、γ-几丁质3种形式存在,是仅次于纤维素的第二丰富的生物多聚体[1-6]。几丁质是昆虫表皮与围食膜的重要组成成分。昆虫在其生长发育周期中会经历多次蜕皮,即蜕下旧表皮的同时合成新的初生表皮,旧表皮中的几丁质会被降解、吸收、重新利用[7-8],而这些过程无不依赖于具有生物催化水解活性的几丁质酶[5,9-10],特别是昆虫体内某些几丁质酶家族成员的功能是独一无二不可替代的。因此,昆虫体内几丁质酶的表达与合成影响着昆虫的蜕皮、化蛹、羽化及围食膜再生等重要生理活动。探究几丁质酶基因的功能和作用机制,可为害虫防治和经济昆虫育种提供理论参考或重要的靶标分子。
已有研究发现,有的几丁质酶基因虽然在昆虫整个生长发育时期均有表达,但只在羽化阶段起作用[5,7,9,11];而有的几丁质酶基因则在昆虫整个生长发育时期都有表达且发挥重要作用[12]。几丁质酶的生理活性主要受pH值、温度、金属离子、作用底物等影响,除此之外,昆虫不同组织部位中的几丁质酶活性也有所不同[10]。对几丁质酶的催化作用研究也已经相当深入,因此目前对几丁质酶的研究主要集中在几丁质酶及类似蛋白质的结构与功能方面。
中华按蚊(Anophelessinensis)在我国除新疆和青海以外的省份均有分布,是我国平原地区疟疾传播的重要媒介昆虫。因此,中华按蚊的防控对维护人类健康具有重要意义。本项研究针对中华按蚊的几丁质酶基因家族进行全基因组鉴定分析,并将中华按蚊几丁质酶基因与冈比亚按蚊(Anophelesgambiae)、黑腹果蝇(Drosophilamelanogaster)、赤拟谷盗(Triboliumcastaneum)、埃及伊蚊(Aedesaegypti)、家蚕(Bombyxmori)等昆虫的几丁质酶基因进行系统进化分析,为下一步对中华按蚊几丁质酶基因的表达及功能研究打下基础。
1 材料与方法
1.1 数据来源
中华按蚊基因组和蛋白质数据来自于重庆师范大学昆虫与分子生物学实验室,冈比亚按蚊、赤拟谷盗、黑腹果蝇和埃及伊蚊4种昆虫的几丁质酶的氨基酸序列来源于NCBI数据库(https://www.ncbi.nlm.nih.gov/genbank/),家蚕的几丁质酶的氨基酸序列来源于SGID数据库(http://sgid.popgenetics.net/)。
1.2 目的基因的筛选与鉴定
根据已报道的代表性昆虫的几丁质酶基因编号,在 NCBI数据库中下载其氨基酸序列和DNA序列。以冈比亚按蚊的几丁质酶氨基酸序列为种子序列在中华按蚊蛋白质数据库进行本地序列对比,并对其氨基酸序列进行结构域预测,根据包含 Glyco hydro 18这一几丁质酶特征结构域,初步判定几丁质酶基因,从而初步得到中华按蚊中候选的几丁质酶基因的编码产物。此外,综合NCBI多序列比对结果、HMMER3.0同源建模搜索结果,以及FGENESH在线预测结果(http://www.softberry.com/berry.phtml),最终确定中华按蚊中预测的几丁质酶基因。
1.3 基因家族成员的基本性质和进化分析
中华按蚊几丁质酶蛋白的基本理化性质在ExPASyProtParam Server(http://web.expasy. org/protparam/)网站上预测得到。利用NCBI的在线程序Batch CD-Search(https://www.ncbi.nlm.nih.gov/Structure/ bwrpsb/bwrpsb.cgi)对几丁质酶蛋白的结构域进行预测,根据预测位置提取出所有结构域序列(多个结构域记为AsCht10-dm-a、AsCht10-dm-b、AsCht10-dm-c等)。
结合中华按蚊的gff注释文件或者预测结果绘制几丁质酶基因的外显子和内含子的位置结构图以及几丁质酶基因在染色体上的分布图,通过结构域预测结果绘制几丁质酶基因的结构域图。
联合构树序列文件是所有比对物种几丁质酶蛋白的结构域序列集合,并使用MAFFTT Alignment(https://mafft.cbrc.jp/alignment/server/)进行多重序列比对,随后使用IQtree 2.1.3版本对对齐序列文件进行系统发育树的构建,各分支重复检验次数为1 000次。
1.4 基因选择压力分析
将DNAsp用于分析几丁质酶家族基因的替换率以计算选择压力,将单个中华按蚊几丁质酶基因与冈比亚按蚊中的直系同源基因作为一个组合进行选择压力的分析。
2 结果与分析
2.1 中华按蚊的几丁质酶基因家族成员
通过与冈比亚按蚊和黑腹果蝇的几丁质酶氨基酸序列比对,筛选出22个预测的中华按蚊几丁质酶基因。进一步通过FGENESH程序在中华按蚊基因组中发现1个未被注释的区域,也可能包含有几丁质酶基因。氨基酸序列分析结果显示:这些预测基因的编码产物均包含几丁质酶的保守基序FDGXDLDWEYP、KXXXXXGGW、MXYDXXG、GXXXWXXDXD中的1条或多条。另外,预测蛋白质中均含有几丁质酶中的DWEYP特征基序;预测蛋白质中均存在 Glyco hydro 18这一几丁质酶特征结构域。因此,我们初步推测中华按蚊的基因组中存在23个几丁质酶基因。
2.2 中华按蚊23个几丁质酶的基础性质和结构域
从表1可见,预测的几丁质酶参考蛋白质序列长度为271~2 361 aa,理论分子质量为30.49~264.06 kDa,等电点为5.05~8.61。
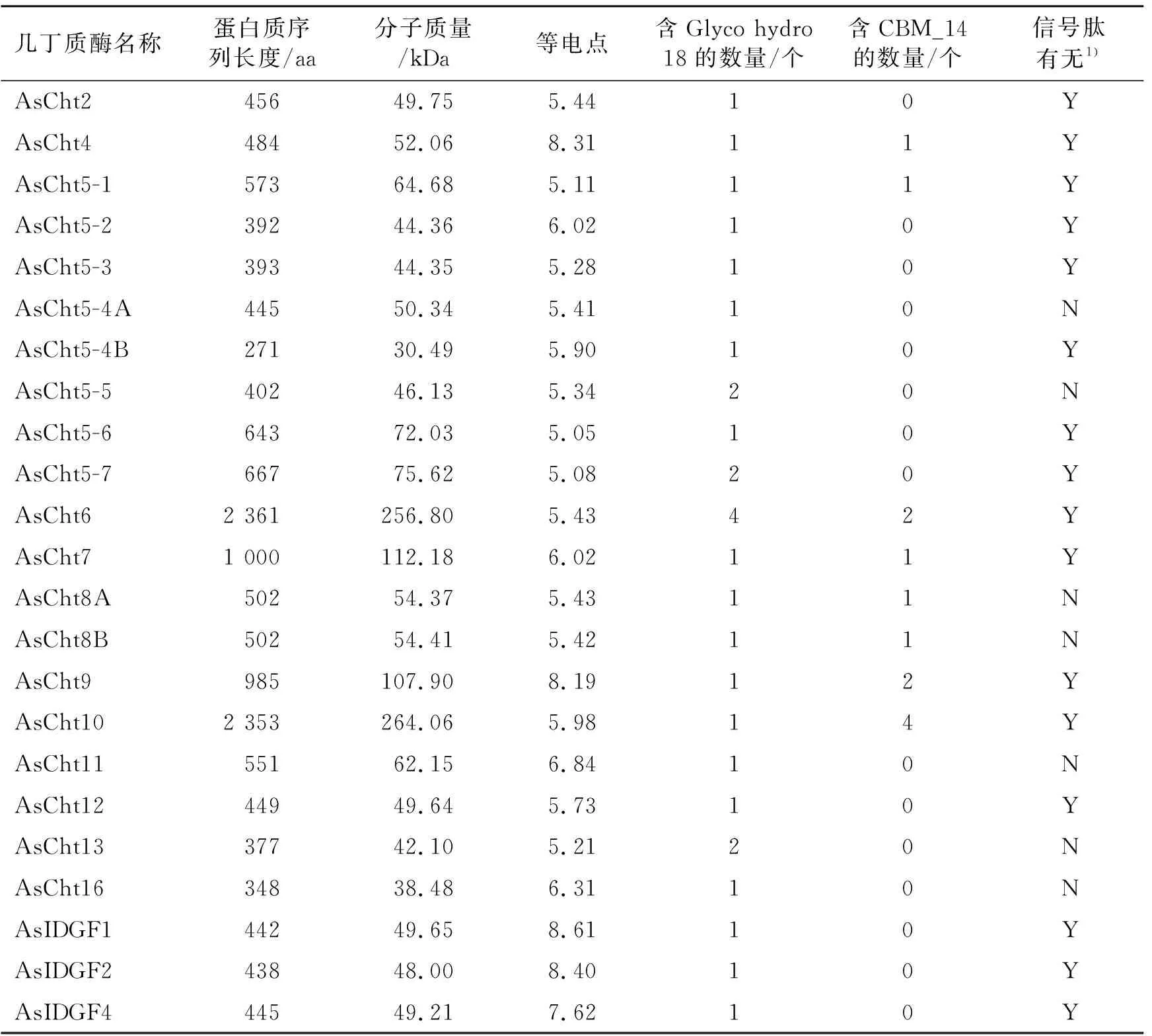
表1 中华按蚊23个几丁质酶的基本特性
不同几丁质酶的序列长度差异较大。结构域分析表明,几丁质酶AsCht5-4A、AsCht5-5、AsCht8、AsCht11、AsCht13、AsCht16不含信号肽;而其余的几丁质酶均含有信号肽。据此初步判断中华按蚊中的大多数几丁质酶属于分泌型蛋白质,会被分泌到胞外,后续可能参与表皮中几丁质的降解代谢。
2.3 中华按蚊23个几丁质酶基因的结构及分布
中华按蚊的23个几丁质酶编码基因的外显子数目差异较大,其分布数量为2~13个,其中AsCht6、AsCht11分别拥有13和8个外显子(图1)。
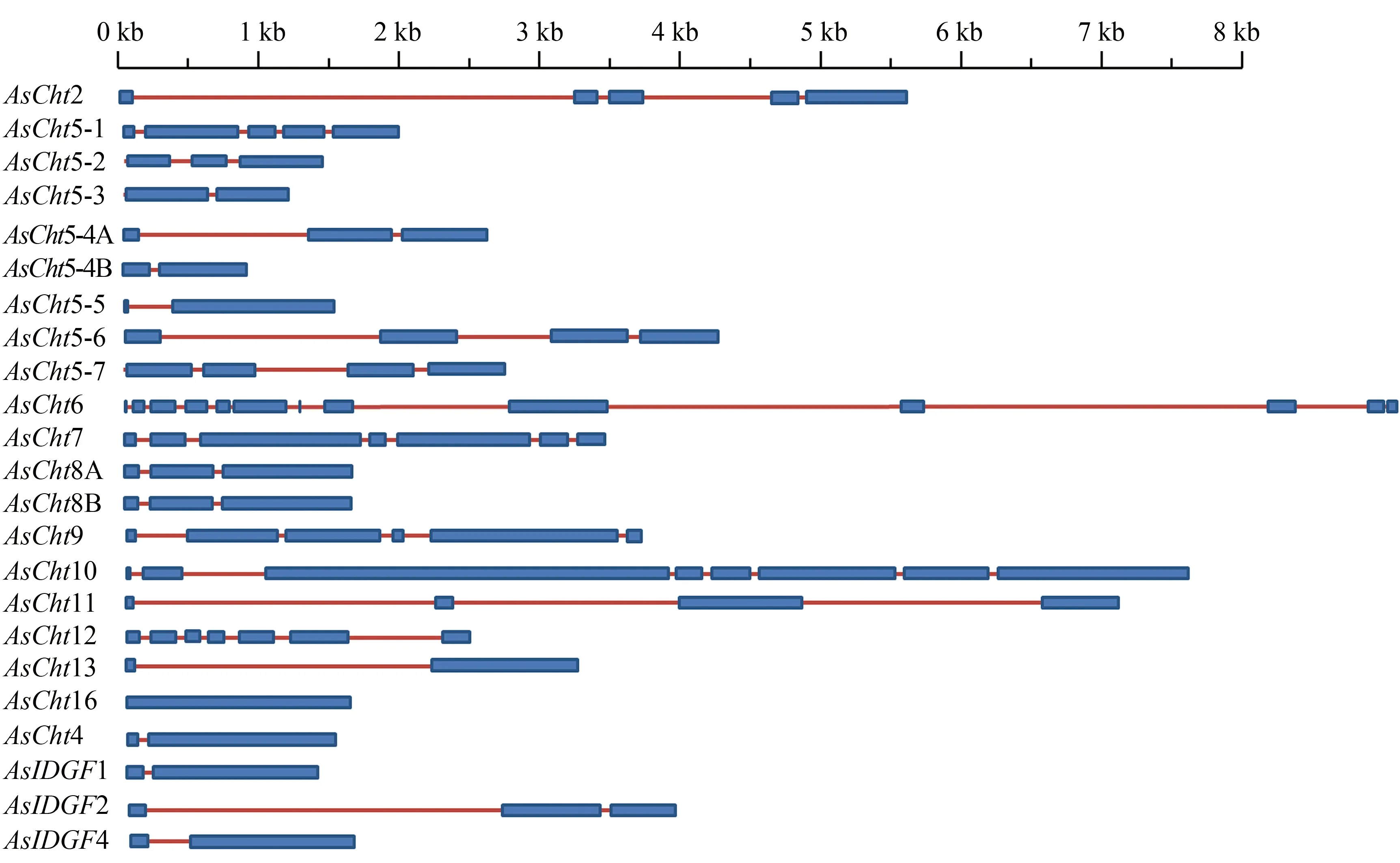
蓝色为外显子,红色为内含子。
中华按蚊的大多数几丁质酶基因包含1个或2个编码Glyco hydro 18结构域的特征序列;而AsCht10有4个编码Glyco hydro 18结构域的特征序列,这与冈比亚按蚊、黑腹果蝇等昆虫Cht10基因编码多结构域的情况相一致(图2)。
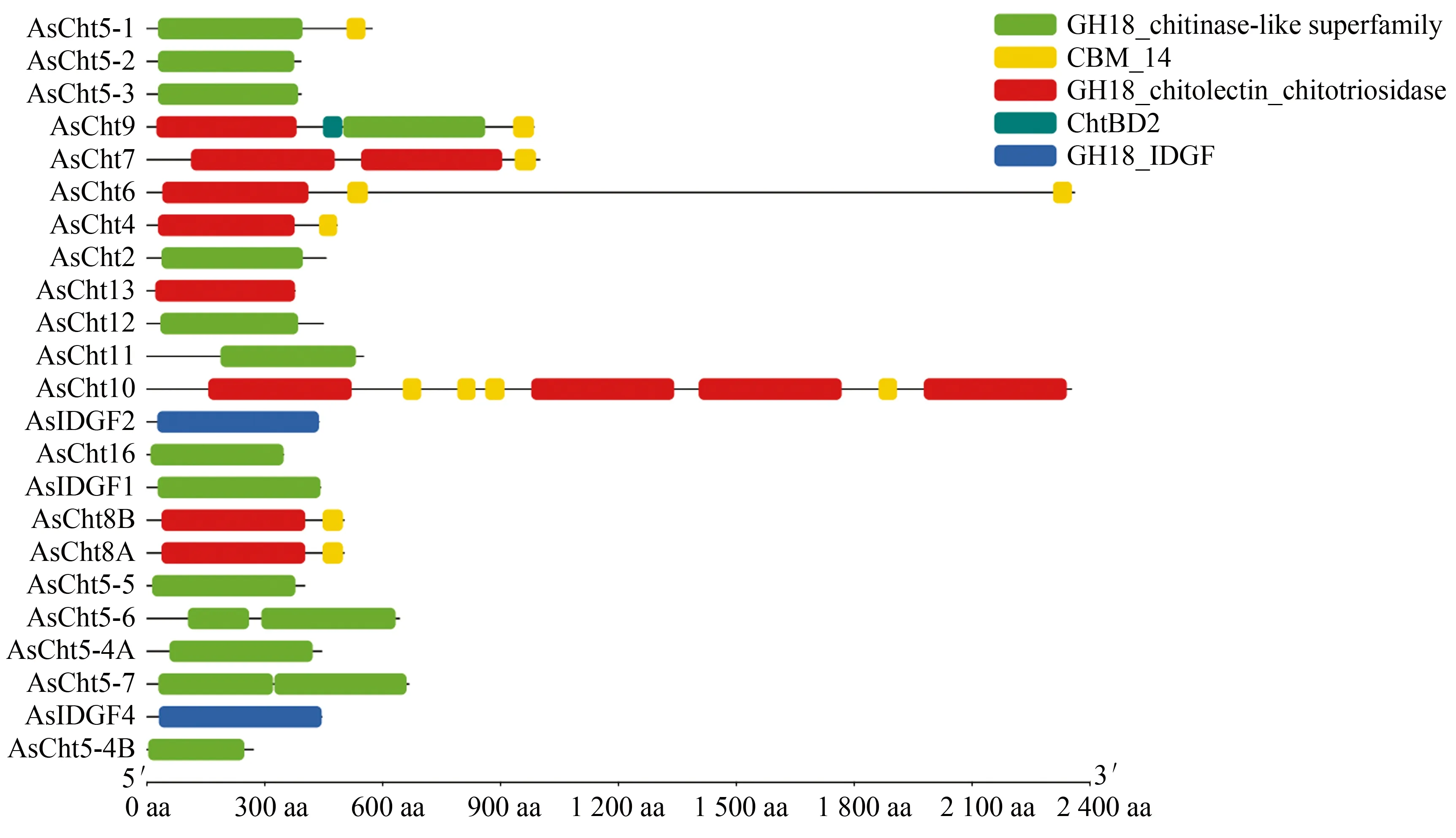
图2 中华按蚊23个几丁质酶基因编码蛋白质的结构域示意图
23个几丁质酶基因分别位于14个scaffold上(图3)。冈比亚按蚊的Cht6、Cht11位于X染色体上,我们推测这2个基因在中华按蚊中的直系同源基因AsCht6和AsCht11可能也位于X染色体上。Cht5亚家族成员在冈比亚按蚊和埃及伊蚊中均存在不少于3个拷贝的形式,且串联成簇排列。中华按蚊中存在3个IDGF基因,且成簇分布;而埃及伊蚊和冈比亚按蚊中,均只存在其中的2个IDGF基因(表2)。
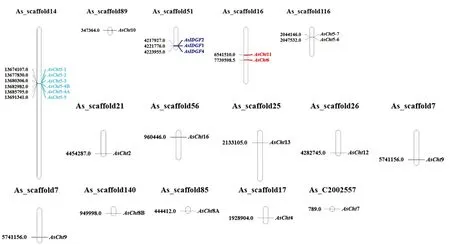
图中每个scaffold的右边为基因名称,左边为该基因的起始位置。
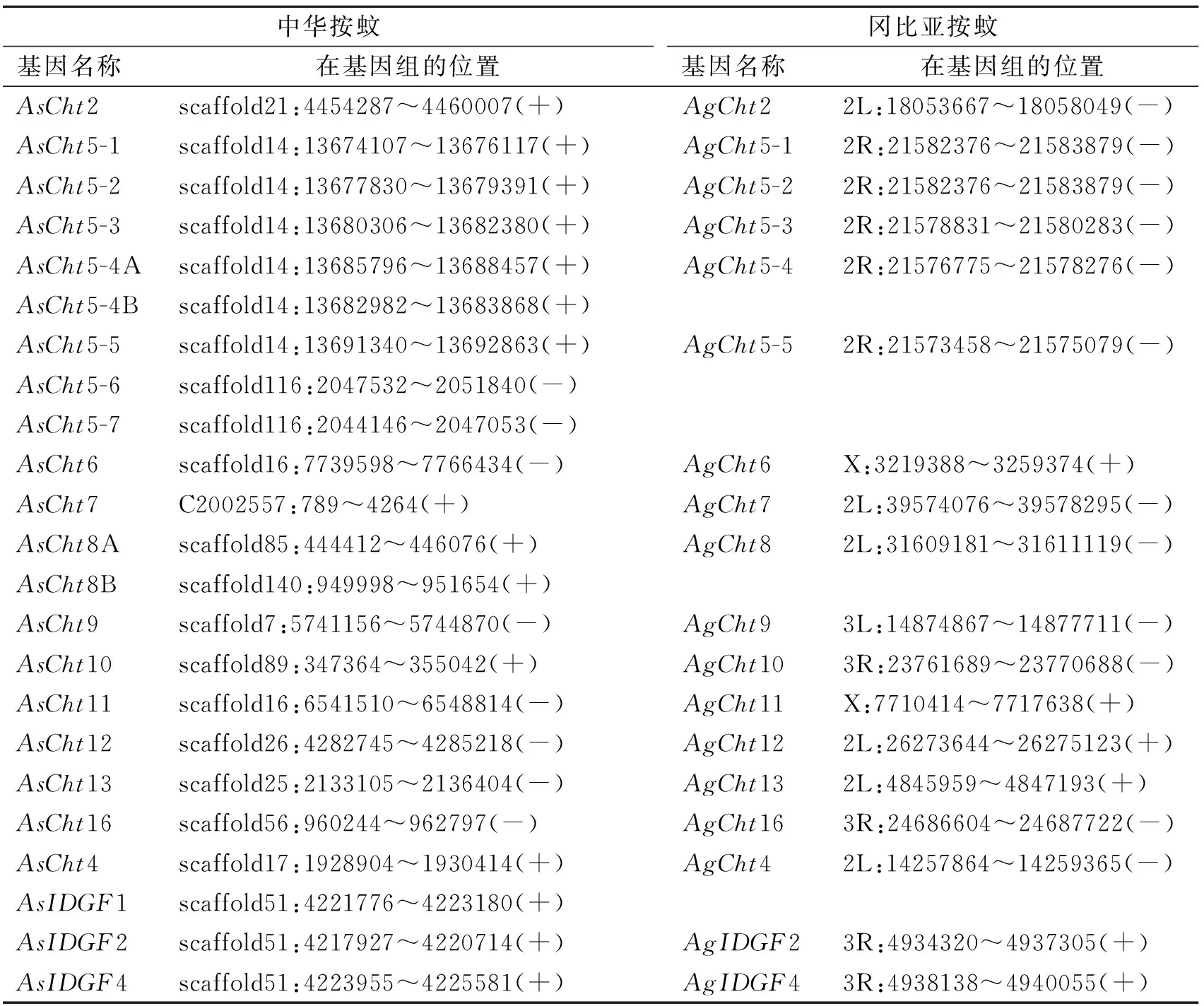
表2 中华按蚊23个几丁质酶基因及其对应的冈比亚按蚊直系同源基因的位置信息比对
2.4 中华按蚊几丁质酶的系统进化分析
根据中华按蚊与冈比亚按蚊、黑腹果蝇、赤拟谷盗、埃及伊蚊和家蚕等昆虫的几丁质酶Glyco hydro 18结构域序列构建的ML系统发生树如图4所示,据此将该类几丁质酶的编码基因划分为9个亚家族。
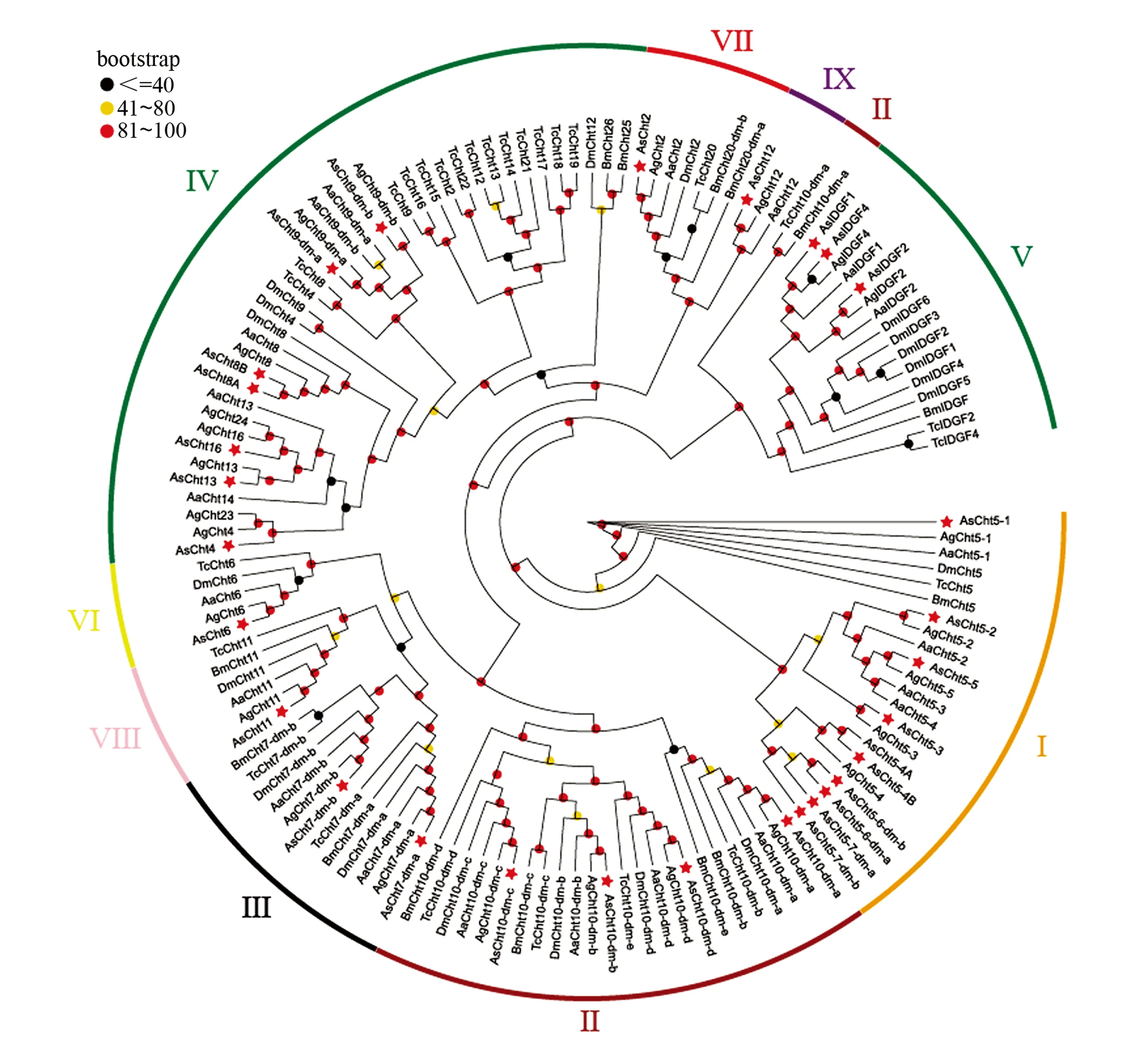
As—中华按蚊Anopheles sinensis,Aa—埃及伊蚊Aedes aegypti,Ag—冈比亚按蚊Anopheles gambiae, Dm—黑腹果蝇Drosophila melanogaster,Bm—家蚕Bombyx mori, Tc—赤拟谷盗Tribolium castaneum;利用最大似然法进行构建,各分支上的数字表示bootstrap值。
亚家族Ⅰ主要包含中华按蚊的AsCht5,其中AsCht5-4A、AsCht5-4B与冈比亚按蚊AgCht5-4的bootstrap值>80,支持其与AgCht5-4同源,故中华按蚊中存在2个Cht5-4基因,推测其可能经历了基因重复而形成;AsCht5-6和AsCht5-7则聚类在此家族的另一分支上,且bootstrap值较高,推测二者可能为此家族独立演化出的旁系基因。
亚家族Ⅱ、Ⅲ、Ⅵ、Ⅶ、Ⅷ和Ⅸ均只包含1种几丁质酶基因,且分别独立聚类为一支。其中,亚家族Ⅱ仅包括Cht10,亚家族Ⅲ仅包括Cht7,亚家族Ⅵ仅包括Cht6,亚家族Ⅶ仅包括Cht2,亚家族Ⅷ仅包括Cht11,亚家族Ⅸ仅包括Cht12。值得一提的是亚家族Ⅱ中TcCht10和BmCht10的第一个Glyco hydro 18结构域编码序列出现分离现象并与亚家族Ⅴ聚在一起,此现象还曾出现在Ye Pan等[13]构建的系统发育树中。
亚家族Ⅳ包含的几丁质酶基因数目众多,包括AsCht8、AsCht9、AsCht4、AsCht13、AsCht16。值得一提的是在中华按蚊中AsCht4和AsCht16仅有1个拷贝,而冈比亚按蚊中的AgCht4与AgCht23,以及AgCht16与AgCht24高度同源(bootstrap值>80),为高度相似基因,我们推测这些基因的形成可能与基因重复事件相关。
亚家族Ⅴ主要包括几丁质酶类似蛋白编码基因,包括:IDGF1、IDGF2、IDGF4。系统发生树显示:AsIDGF1与AsIDGF2分别与埃及伊蚊中的直系同源基因聚类,AsIDGF4与冈比亚按蚊中的AgIDGF4聚在一起,且AsIDGF1与AsIDGF4所在支的bootstrap值仅为65,暗示二者间在演化过程中产生了较大的分化。
2.5 中华按蚊几丁质酶基因的选择压力分析
以冈比亚按蚊作为参照,对中华按蚊中Cht5、Cht8、Cht9这3个几丁质酶基因家族成员进行选择压力分析。这些基因的Ka/Ks值均<1,暗示它们受到纯化选择(表3),推测这些几丁质酶基因家族成员可能在中华按蚊的重要生理过程中有不可或缺的作用。
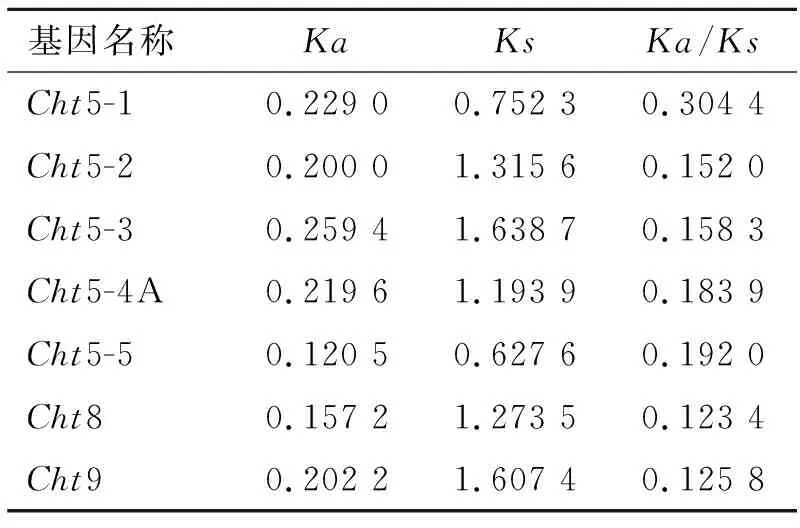
表3 中华按蚊3个几丁质酶基因家族成员的 选择压力
3 讨论
本项研究采用生物信息学方法鉴定了中华按蚊的23个几丁质酶基因。对几丁质酶基因的分布进行分析发现,Cht6、Cht11同时存在于冈比亚按蚊和果蝇的性染色体上,此外在中华按蚊、淡色按蚊(Anophelesalbimanus)、黑小按蚊(Anophelesatroparvus)中的分布位置均是如此(未发表数据)。推测这2个基因间以及其与性染色体间可能存在连锁关系,对此需进一步通过实验验证,这可以为后续针对性别特异性基因的研究提供基础数据。在前期的研究中发现,中华按蚊某些几丁质酶基因的表达具有雌雄特异性(未发表数据),这预示着对其表达的性别特异性进行更加深入的研究将为害虫的靶向防治提供新的思路。同时,或可为研究其他昆虫中类似基因在性别二态型中所扮演的角色起推动作用。
我们在研究中还发现,不同昆虫的几丁质酶种类尽管大致相似,但其数量却呈现较大差异。有的昆虫可以通过基因重复这一方式,增加类似功能的几丁质酶基因拷贝数量来调控其表达水平,进而行使其相应的生理功能。
