哈维弧菌flhF和CU052_26825缺失株的构建及其生物学特性
2022-03-31邓益琴张亚秋林梓阳程长洪马红玲刘广鑫苏友禄
邓益琴,张亚秋,3,林梓阳,4,冯 娟,程长洪,马红玲,刘广鑫,苏友禄
哈维弧菌和缺失株的构建及其生物学特性
邓益琴1,张亚秋1,3,林梓阳1,4,冯 娟1,程长洪1,马红玲1,刘广鑫1,苏友禄2
(1.中国水产科学研究院南海水产研究所 / 农业农村部南海渔业资源开发利用重点实验室,广东 广州 510300;2.广东省水环境与水产品安全工程技术研究中心 / 仲恺农业工程学院动物科技学院,广东 广州 510225;3.上海海洋大学水产与生命学院,上海 201306;4.海南大学海洋学院,海南 海口 570228)
【目的】研究哈维弧菌()345的GTP结合蛋白编码基因和未知功能基因的生物学功能。【方法】利用同源重组技术构建345的和的缺失突变株,比较野生株和突变株的生长、运动性、胞外酶活性、耐药性等生物学特性以及对花鲈()的毒性。【结果】基因和的缺失均不影响菌株的生长、运动性、胞外蛋白酶活性、对H2O2和Cu2+的压力感应、对铁的吸收利用、对大多数被测抗生素的抗性以及对花鲈的毒性;但和缺失后对氯霉素和氟苯尼考两种氯霉素类抗生素均更加敏感。此外,缺失后,细菌生物膜形成能力显著增强。【结论】和均参与调控哈维弧菌对氯霉素类抗生素的耐药性,还参与调控哈维弧菌生物膜形成。
哈维弧菌;基因敲除;;;生物学特性;氯霉素类耐药性
弧菌病是一种流行范围广、危害极为严重的水产动物传染性疾病,极大地阻碍了水产养殖业的健康发展。哈维弧菌()是导致中国华南沿岸海水鱼感染和死亡最主要的病原弧菌,是养殖业主要影响因素之一[1-2]。由于疫苗和免疫增强剂等其他防控方法的局限性,抗生素广泛用于预治水产养殖细菌性疾病[3];但抗生素的过度使用会导致耐药和多重耐药细菌产生和积累[4]。Deng等[5]发现,华南沿岸分离的弧菌对所测试15种抗生素的耐药指数可达0.60。Sadat等[6]从埃及鱼贝中分离的副溶血弧菌()和溶藻弧菌()的多重耐药率分别为69.04%和38%。这些均说明细菌高耐药的严峻形势。因此,深入认识生物耐药性的发生与发展机制,是保证药物正确使用的前提和基础。
GTP结合蛋白编码基因与细菌运动性、生物膜形成和毒力相关[7]。本课题组发现,对数期哈维弧菌于40℃下热击60 min后,未知功能基因表达量下降约30倍,而温度对细菌致病性有潜在影响[8];因此,可能与哈维弧菌毒力相关。本研究利用同源重组技术构建致病性哈维弧菌 () 345的染色体上基因和未知功能基因[9]的缺失突变株,比较野生株与突变株的生长、运动性、胞外酶活性、耐药性等多种生物学特性以及对花鲈()的毒性,为哈维弧菌和的基因功能研究奠定基础,为防治哈维弧菌病提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌株、质粒和引物 哈维弧菌345由中国水产科学研究院南海水产研究所渔业生物病害防治研究室(下称“本实验室”)分离自中国南部沿海患病石斑鱼()[9];自杀质粒pSW7848[10]、表达质粒pMMB207[11]、大肠杆菌Π3813[12]和GEB883[13]分别来自Didier Mazel、Zhao Zhe、Didier Mazel和Annick Jacq教授,并保存于本实验室。所用引物见表1。
1.1.2 主要试剂 高保真酶PrimeSTAR®Max DNA polymerase、普通PCR酶Premix™(TaKaRa™ Version 2.0)及限制性内切酶I购自Takara公司;琼脂糖凝胶DNA回收试剂盒、DNA产物纯化试剂盒购自天根生物科技(北京)有限公司;-葡萄糖、-阿拉伯糖、氯霉素、2′-脱氧胸苷(2′-Deoxythymidine,Thy)和2-2′-联吡啶(2,2′-Dipyridyl,DIP)购自生工生物工程(上海)股份有限公司;2,6-二氨基丙烯酸(2,6-Diaminopimelic acid,DAP)购自Sigma公司;等温组装试剂盒ClonExpressMultiS One Step Cloning Kit购自Vazyme公司;抗生素药敏纸片购自杭州天和微生物试剂有限公司。
1.1.3 主要培养基 LB(luria-bertani)培养基、MH肉汤培养基和技术琼脂粉购自广东环凯生物科技有限公司。1 L LB含10 g胰蛋白胨、5 g酵母提取物、10 g氯化钠。1 L固体培养基含15 g琼脂粉。将LB培养基氯化钠质量浓度提高至30 g·L−1,即制得LBS(luria-bertani-salt)培养基。
1.1.4 实验用鱼 花鲈()购自深圳一养殖场。

表1 本研究所用引物
1.2 方法
1.2.1和缺失株的构建 参考Zhang等[14]方法并稍作修改。用引物对pSW7848-F/R线性化自杀质粒pSW7848。用引物对-UP-F/R和-DOWN-F/R扩增上、下游同源臂片段,并用引物对-UP-F/R、-DOWN-F/R扩增上、下游同源臂片段。使用等温组装试剂盒等温组装线性化质粒pSW7848和上下游同源臂片段,以及线性化质粒pSW7848和上下游同源臂片段。将等温组装液转化进入接合作用中间宿主菌Π3813,用含有20 μg/mL氯霉素和0.3 mmol/L Thy的LB平板筛选可能成功的重组子,并用质粒检测引物对Del-check-pSW7848-F/R对重组自杀质粒进行PCR和测序检测,组装成功的质粒PCR片段大小均约为2.3 kb,分别命名为pSW7848-Δ和pSW7848-Δ。进一步将重组自杀质粒pSW7848-Δ和pSW7848-Δ转化进入接合作用供体菌GEB883。通过接合作用分别将重组自杀质粒pSW7848-Δ和pSW7848-Δ送入受体菌哈维弧菌野生株345。基于自杀质粒pSW7848在哈维弧菌中不能自主复制、有氯霉素抗性、毒性基因的表达受葡萄糖抑制而受阿拉伯糖诱导的特点[10],用含34 μg/mL氯霉素和2 g/L-葡萄糖的LBS平板进行第1次筛选,用含34 μg/mL氯霉素和2 g/L-阿拉伯糖的LBS平板进行第2次反向筛选,重组自杀质粒上基因的上下游同源臂片段分别与哈维弧菌基因组和基因的上下游同源臂片段发生两次同源重组,得到哈维弧菌和缺失株,分别命名为345Δ和345Δ。
1.2.2和回补株的构建 参考Liu等[11]方法并稍作修改。用引物对pMMB207-F/R线性化回补质粒pMMB207。用引物对-F/R和-F/R分别扩增和基因回补片段(包括启动子+ORF序列+终止子)。用等温组装试剂盒等温组装线性化质粒pMMB207和回补片段,以及线性化质粒pMMB207和回补片段。将等温组装液转化入接合作用中间宿主菌Π3813,用含20 μg/mL氯霉素和0.3 mmol/L Thy的LB平板筛选可能成功的重组子,进一步用质粒检测引物对com-check-pMMB207-F/R对重组质粒进行PCR和测序,组装的PCR片段约为2.5、2.7 kb,分别命名为pMMB207-compMMB207-com。将重组回补质粒pMMB207-com和pMMB207-com转化进入接合作用供体菌GEB883。通过接合作用将重组回补质粒pMMB207-com送入受体菌哈维弧菌的缺失株345Δ,并将重组回补质粒pMMB207-com送入受体菌哈维弧菌CU052_26825的缺失株345Δ。基于回补质粒pMMB207在哈维弧菌中能自主复制,有氯霉素抗性[11],用含34 μg/mL氯霉素的LBS平板筛选并纯化2次,得到哈维弧菌和回补株,分别命名为345Δ-pMMB207-com-和345ΔpMMB207-com。同时,通过接合作用分别将回补质粒pMMB207送入受体菌哈维弧菌野生株345、的缺失株345Δ以及的缺失株345Δ,得到哈维弧菌345-pMMB207、345Δ-pMMB207及345Δ-pMMB207。
1.2.3 菌株生长分析 菌液制备:将野生株345,缺失突变株345Δ345Δ划线于LBS平板,于28℃培养16 h,挑取单克隆分别接种至LBS液体培养基,于28℃、200 r·min–1条件下振摇培养16 ~ 24 h。用新鲜的LBS液体培养基将菌液稀释至光密度(600 nm)= 0.001,按100 μL/孔分装于96孔板,并用Spark微孔板检测仪(TECA)测定不同时间点细菌光密度(600 nm),绘制生长曲线。实验重复3次,由于3次重复结果相同,选择1次代表性结果呈现,并使用IBM SPSS Statistics 19软件进行单因素协方差分析(ANCOVA)[以野生株和缺失突变株为固定因子,以时间为协变量,以(600 nm)值为因变量]。
1.2.4 细菌游动性(Swimming)和涌动性(Swarming)测试 参考Zhang等[14]方法进行。按1.2.3制备菌液,用新鲜的LBS液体培养基将菌液稀释至(600 nm) = 3.0,每个菌株取3 μL平行3次点样到含3 g/L琼脂的LBS半固体平板上,于28℃下静置培养16 h,测量菌斑直径,测试其游动性;同时取3 μL平行3次点样到含3 g/L琼脂的LBS固体平板上,于28℃下静置培养24 h,测菌斑直径,测试其涌动性。实验重复3次。用IBM SPSS Statistics 19软件进行Student’s检验,=0.05作为统计学显著性阈值。
1.2.5 细菌生物膜形成能力分析 参考Zhang等[14]方法进行。按1.2.3制备菌液,用新鲜LBS液体培养基将菌液稀释至(600 nm) = 3.0,取50 μL菌液接种到装有5 mL LBS液体培养基的无菌玻璃试管,于28 ℃下静置培养48 h,加入200 μL 26 g/L的结晶紫(终质量浓度为1 g/L)染色5 min,弃菌液,并用水轻轻冲洗几次,加入200 μL 330 mL/L冰乙酸充分溶解结晶紫,测定570 nm处光密度值(570 nm)。重复实验3次,数据处理同1.2.4。
1.2.6 细菌胞外蛋白酶活性测试 参考Zhang等[14]方法进行。制备1.2.3的菌液,用新鲜LBS液体培养基稀释至(600 nm) = 3.0,取3 μL平行3次点样到含15 g/L脱脂奶粉的LBS固体平板上,于28℃下静置培养24 h,测量蛋白质透明分解圈直径,以指示细菌蛋白酶活性。实验重复3次,数据处理同1.2.4。
1.2.7 菌株对H2O2、Cu2+抗性以及对铁离子的吸收能力测试 参考张亚秋[15]方法进行。按1.2.3制备菌液,用新鲜LBS液体培养基稀释至(600 nm) = 3.0,并以10倍梯度稀释各菌株菌液至最高稀释倍数107,将稀释的菌液分别取3 μL平行3次点样到LBS平板,以及添加有0.002 mL/L H2O2或0.2 mmol/L CuSO4或120 μmol/L DIP的LBS平板上,于28℃下静置培养24 h,观察各菌株生长情况,实验重复3次。
1.2.8 菌株对不同抗生素的敏感性测试 按1.2.3制备菌液,吸取600 μL菌液于10 mL无菌9 g/L生理盐水中,倾入宽13 cm的正方形LBS平板,将培养基表面全部润湿,弃去培养皿内多余菌液,静置约10 min,待培养基表面菌液全部晾干,将各抗生素纸片置于平板上,于28℃下静置培养24 h,观察并测量抑菌圈直径。重复实验3次,数据处理同1.2.4。
参考吴金军[16]方法分析菌株对抗生素的最低抑菌浓度(Minimum inhibitory concentration,MIC):用MH肉汤培养基将培养16 ~ 24 h菌液浓度调至1×106cfu/mL得上样菌悬液。制备质量浓度为128 μg/mL抗生素母液,在无菌细胞培养板第1 ~ 10孔中加入100 μL无菌MH肉汤培养基,在第1孔中加入100 μL抗生素母液,反复吹吸,混匀,吸取100 μL至第2孔,反复吹吸,混匀,吸取100 μL至第3孔,以此稀释至第10孔,每孔终体积为100 μL。向第1 ~ 10孔中加入上样菌悬液,反复吹吸,充分混匀,每孔终体积为200 μL,在第11、12孔中分别加入200 μL无菌MH肉汤培养基(阴性对照)和待测菌株悬液(阳性对照),每个样品设置3个平行。上样后置于28℃恒温培养箱中培养20 h,根据美国临床和实验室标准协会CLSI文件判读实验结果[17]。
1.2.9 菌株毒力测试 取花鲈240尾,于容积100 L的塑料桶中暂养2周,用气石不间断充气。将菌株345-pMMB207、345Δ-pMMB207、345Δ-pMMB207、345Δ-pMMB207-com和345Δ-pMMB207-com划线于LBS平板,于28℃下培养16 ~ 24 h,挑取单克隆分别接种至LBS玻璃斜面的固体培养基,于28℃下培养16 h,每个斜面用3 mL生理盐水洗下菌体。野生株345-pMMB207对花鲈的半致死剂量LD50为4.49×104cfu/g[18],将345-pMMB207、345Δ-pMMB207、345Δ-pMMB207-com稀释至8×LD50,而将345-pMMB207、345Δ-pMMB207、345Δ-pMMB207- com稀释至10×LD50,按100 μL/尾对花鲈腹腔注射菌液,每株菌注射30尾,按10尾/桶分别养殖于3个100 L塑料桶,用气石不间断充气。同时设3桶注射同剂量生理盐水作为对照组。观察7 d内花鲈死亡情况,比较各组花鲈死亡率差异。
2 结果
2.1 flhF和CU052_26825缺失突变株构建
扩增所得线性化自杀质粒pSW7848片段约3.3 kb(图1(a)),上下游同源臂片段均约为1 kb(图1(b-c)),与预期片段大小相同。重组自杀质粒PCR扩增检测到约2.3 kb的片段(图1(d–e)),与预期片段大小相同,表明重组自杀质粒pSW7848-Δ和pSW7848-Δcom构建成功。重组自杀质粒上的和上下游同源臂片段通过接合作用分别与哈维弧菌野生株345的和上下游同源臂片段发生2次同源交换,缺失株PCR检测结果表明,对于基因,野生株可扩增得1 604 bp的片段,而缺失株可扩增出589 bp的片段(图1(f)),获得缺失突变株;对于基因,野生株可扩增得1 792 bp的片段,而缺失株可扩增出341 bp的片段(图1(g)),获得缺失突变株。
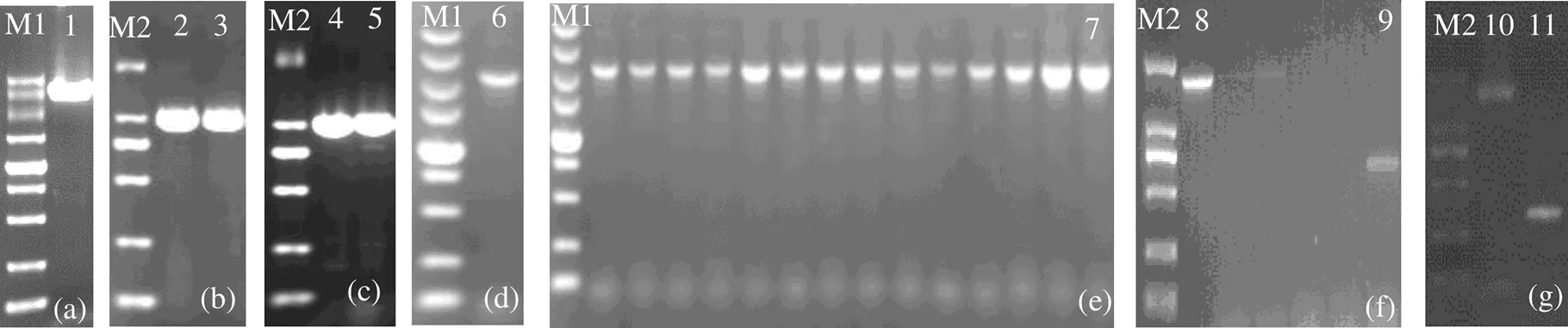
M1,DNA分子标准DL5000,各条带依次为5 000、3 000、2 000、1 500、1 000、750、500、250、100 bp;M2,DNA分子标准为 DL2000,各带依次为2 000、1 000、750、500、250、100 bp;1,pSW7848线性片段;2-3,分别为上、下游同源臂片段;4-5,分别为上、下游同源臂片段;6-7,分别为重组子pSW7848-ΔpSW7848-Δ检测片段;8,野生株345检测片段;9,候选突变株检测片段;10,野生株检测片段;11,候选突变株检测片段;其他未标注泳道为同时跑胶的其他实验结果。
M1, DNA Marker DL5000, and the segment size are 5 000, 3 000, 2 000, 1 500, 1 000, 750, 500, 250, 100 bp, respectively; M2, DNA Marker DL2000, and the segment size are 2 000, 1 000, 750, 500, 250, 100 bp, respectively; 1, Linearized segment of pSW7848; 2-3, upstream and downstream homologous segments of; 4-5, upstream and downstream homologous segments of; 6-7, segment of recombinant suicide vector pSW7848-Δ, pSW7848-Δ; 8, segment of345; 9, segment ofdeletion mutant; 10, segment of; 11, segment ofdeletion mutant; The rest unlabeled lanes are the results of other experiment.
图1和缺失突变株的PCR鉴定
Fig.1 PCR identification for deletion mutants with deletedand
2.2 flhF和CU052_26825回补株构建
扩增得约7.5 kb的线性表达质粒pMMB207片段(图2(a)),与预期片段大小相同。扩增得和的回补片段,分别为2 164、2 359 bp(图2(b-c))。引物对com-PMMB207-check-F/R对二回补株分别扩增得2 469、2 664 bp的片段(图2(d))。
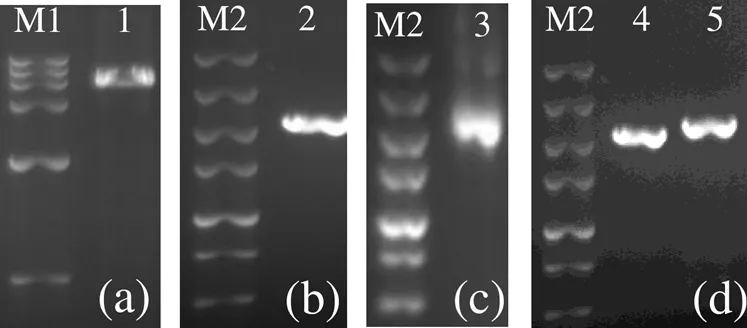
M1,DNA 分子标准DL15 000,从上到下条带依次为15 000、10 000、7 500、5 000、2 500、1 000 bp;M2,DNA 分子标准DL5 000,从上到下条带依次为5 000、3 000、2 000、1 500、1 000、750、500 bp;1,pMMB207线性化片段;2,基因回补片段;3,基因回补片段;4-5,回补重组子pMMB207-compMMB207-com的检测片段。
M1, DNA Marker DL15000, and the segment size are 15 000, 10 000, 7 500, 5 000, 2 500, 1 000 bp, respectively; M2, DNA Marker DL5 000, and the segment size are 5 000, 3 000, 2 000, 1 500, 1 000, 750, 500 bp, respectively; 1, pMMB207 linearization segment; 2, the complemented segment of; 3, the complemented segment of; 4-5, the complemented recombinant segments ofand.
图2和的回补株PCR鉴定
Fig.2 PCR identification for complemented strain ofand
2.3 哈维弧菌flhF和CU052_26825缺失株的生长
因3次重复结果相同,选择1次代表性结果进行分析。图3可见,在LBS培养基中,缺失株345Δ345Δ的生长与野生株均无显著差异(> 0.05),表明和均不影响哈维弧菌的生长。
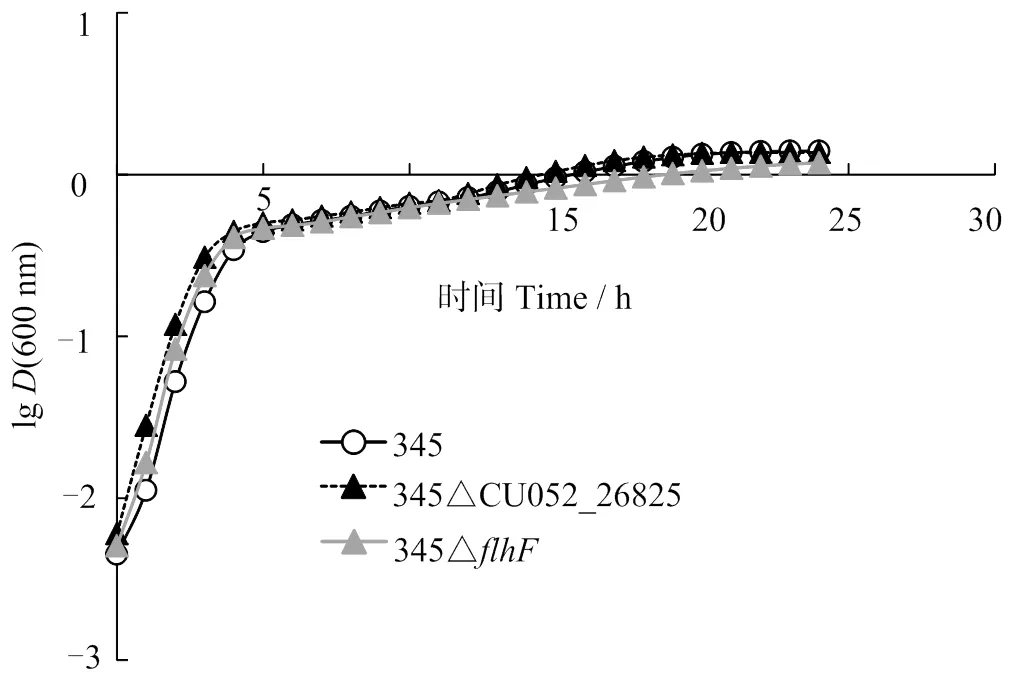
图3 哈维弧菌345、345ΔflhF和345ΔCU052_26825在LBS培养基中的生长曲线
2.4 哈维弧菌flhF和CU052_26825缺失株的运动性
图4(a)可见,野生株345在15 g/L琼脂LBS平板上无涌动性,在3 g/L琼脂LBS平板上也无游动性,且和缺失株在这两种平板上也均未表现出涌动性和游动性。3次重复实验的结果一致。二缺失株运动性相对野生株均无显著差异(> 0.05,图4(b)),表明和均不影响哈维弧菌的运动性。
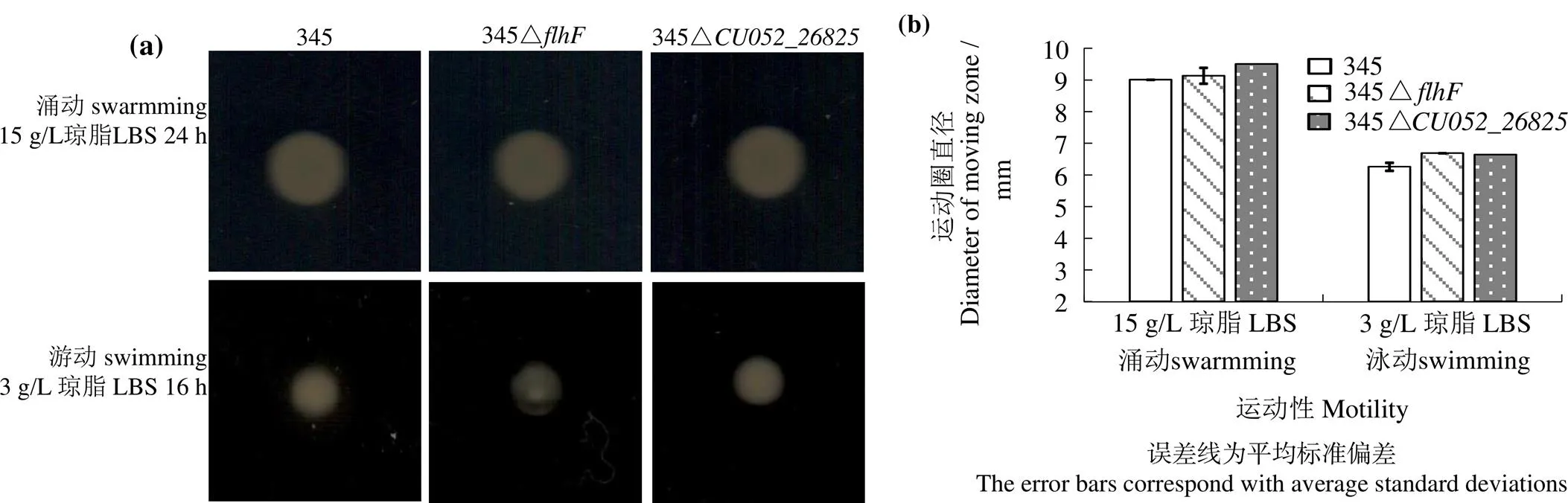
图4 哈维弧菌345、345ΔflhF和345ΔCU052_26825在15、3 g/L琼脂LBS平板上的运动性
2.5 flhF和CU052_26825缺失对哈维弧菌胞外蛋白酶活性的影响
和缺失后,在含15 g/L脱脂奶粉LBS平板上,胞外蛋白酶分解圈直径与野生株几无差异(图5(b)),3次重复实验结果一致。统计分析表明,缺失株胞外蛋白酶活性相对野生株均无显著差异(> 0.05,图5(b)),表明和均不影响哈维弧菌胞外蛋白酶活性。
2.6flhF和CU052_26825缺失对哈维弧菌抗H2O2、Cu2+及吸收铁离子的影响
在LBS平板,添加0.002 mL/L H2O2、0.2 mmol/L CuSO4、120 µmol/L DIP的LBS平板上,随稀释倍数增加,突变株和野生株菌落数均减少,稀释倍数为107时,菌落数明显变少。相对于野生株,突变株对H2O2、CuSO4抗性,铁离子的吸收均无显著差异(图6)。3次重复结果一致,表明和均不影响哈维弧菌对H2O2、Cu2+的抗性,对铁离子的吸收。
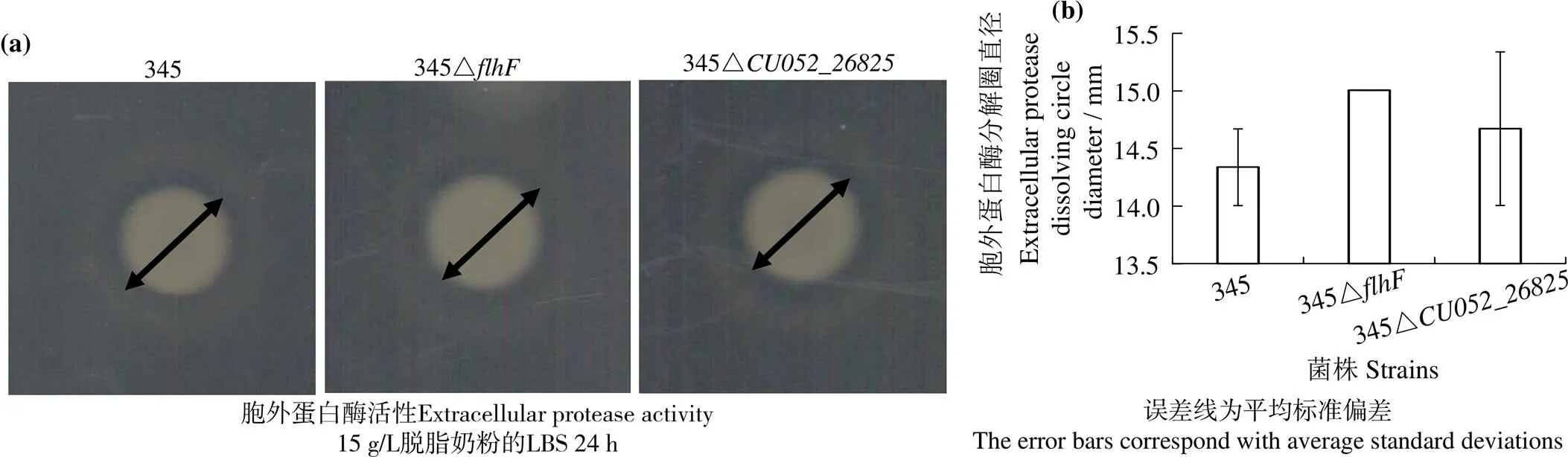
图5 哈维弧菌345、345ΔflhF和345ΔCU052_26825在LBS+15 g/L脱脂奶粉平板上的胞外蛋白酶活性

1,345;2,345ΔflhF;3,345ΔCU052_26825
2.7 flhF和CU052_26825对哈维弧菌生物膜形成的影响
图7(a)可见,在玻璃试管中静置培养后,哈维弧菌野生株和突变株在管壁上仅形成极少量生物膜,而突变株在管壁上形成一层厚厚的生物膜,结晶紫染色后,管壁上呈清晰的染色圈。3次重复实验结果一致。图7(b)可见,突变株与野生株生物膜形成能力无显著差异,而突变株相对野生株,生物膜形成能力极显著增强(< 0.001)。
回补后,与哈维弧菌野生株345-pMMB207和回补株345Δ-pMMB207-com相比,突变株345Δ-pMMB207形成大量生物膜,并出现较明显的染色圈(图7(c))。统计分析表明,生物膜形成能力极显著增强(< 0.001)(图7(d)),回补株345Δ-pMMB207-com生物膜形成能力与野生株相似,无显著差异(图7(c、d)),表明回补株表型与野生株相似。
2.8 flhF和CU052_26825对哈维弧菌抗生素敏感性的影响
哈维弧菌野生株,和突变株均对利福平、多西环素、万古霉素、复方新诺明和四环素等5种抗生素耐药,对呋喃唑酮、阿莫西林、氯霉素、环丙沙星和诺氟沙星等5种抗生素敏感。虽然野生株和2个突变株对妥布霉素、庆大霉素、红霉素和麦迪霉素的抗性不同,但是2个突变株相对野生株的抑菌圈直径无显著差异。而2个突变株对氟苯尼考和氯霉素更加敏感,抑菌圈直径相对野生株菌显著增大(< 0.05,表2)。
分析发现,野生株345,缺失株345Δ和345Δ对氯霉素MIC分别为0.5、0.125、0.125 μg/mL,对氟苯尼考的MIC分别为16、2、0.5 μg/mL(表3)。
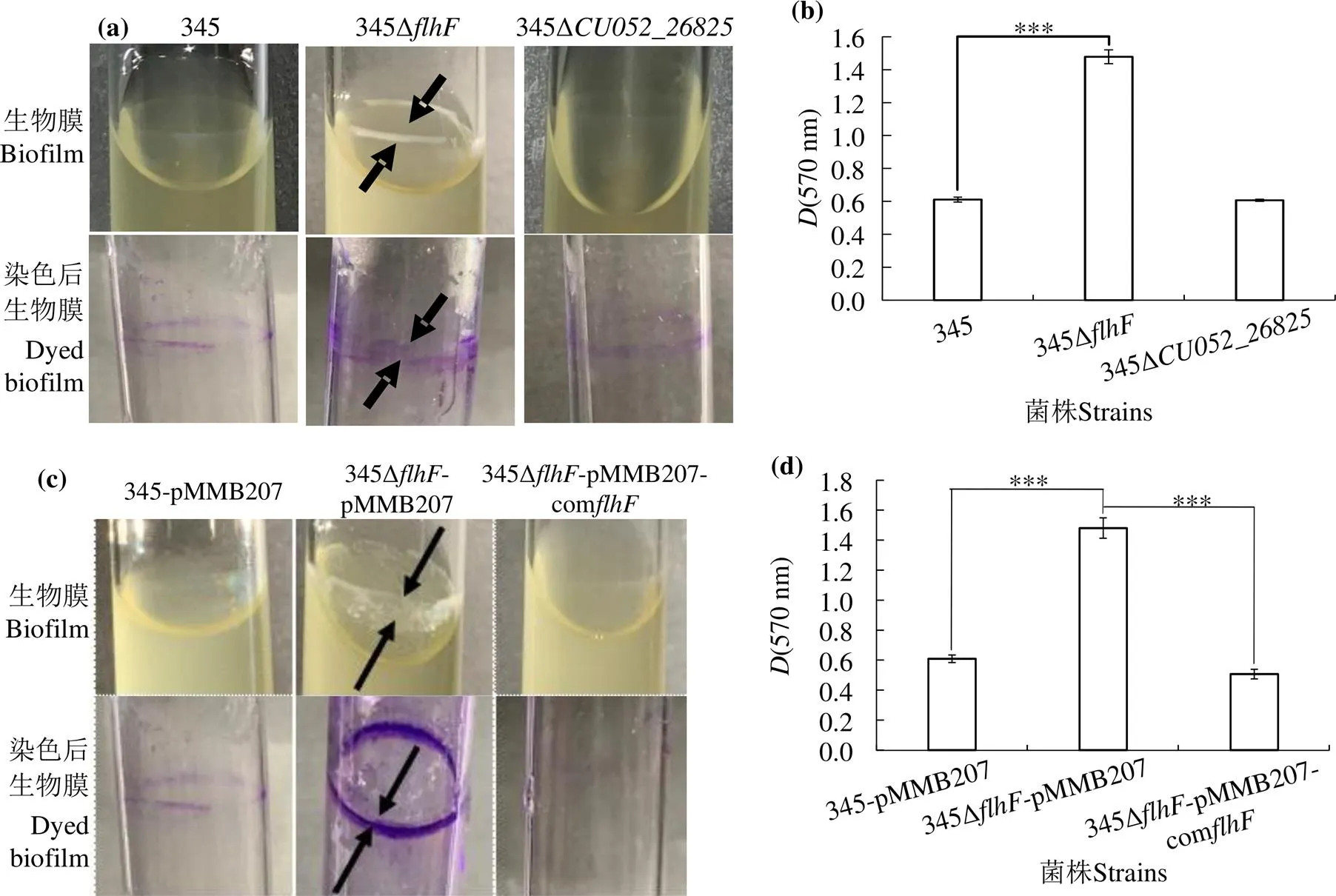
***,差异极显著the highly significant difference(P < 0.05)

表2 哈维弧菌345、345ΔflhF以及345ΔCU052_26825对各抗生素抗性
说明:R,抗性;S,敏感;I,中介;*,与野生株345差异显著(< 0.05)。
Notes: R, Resistance; S, Sensitivity; I, Intermediary; *, the significant difference from345 at level of 0.05.
由于回补质粒pMMB207有氯霉素抗性,因此,仅用哈维弧菌菌株345-pMMB207、345Δ-pMMB207、345Δ-pMMB207-com345Δ-pMMB207和345Δ-pMMB207-com分析氟苯尼考耐药性的回补情况,发现这5株菌对氟苯尼考的最低抑菌浓度(MIC)分别为16、1、8、1、32 μg/mL(表3)。
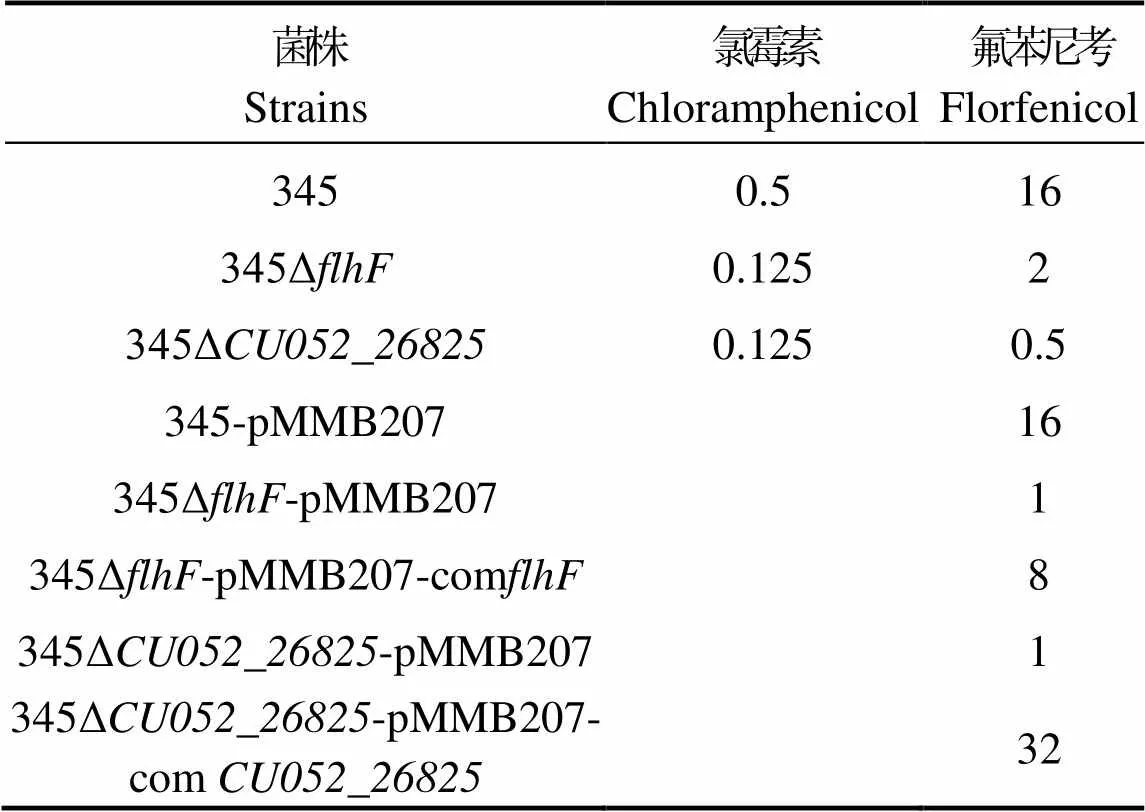
表3 各菌株的最低抑菌浓度
2.9 flhF和CU052_26825对哈维弧菌毒力的影响
缺失组感染剂量为8 LD50(3.59×105cfu/g),生理盐水组实验期间未死鱼,其他组在注射12 h后死亡率即达到8%以上,随着注射时间的延长,死亡率逐渐升高,注射7 d后,345-pMMB207、345Δ-pMMB207和345Δ-pMMB207-com组的累积死亡率分别稳定在58.33%、62.50%、66.67%(图8(a))。
缺失组感染剂量为10 LD50(4.49×105cfu/g),生理盐水组实验期间未出现死鱼,其他组在注射12 h后死亡率即达到70%以上,随着注射时间延长,死亡率逐渐升高,注射4 d后,345-pMMB207、345Δ-pMMB207、345Δ-pMMB207-com组的累积死亡率分别稳定在100.00%、96.67%、100.00%(图8(b))。

图8 各菌株的累积死亡率
3 讨论
哈维弧菌是近十多年水产养殖业重要致病菌,可感染虾(如对虾的发光病)、鱼(如鲈、大菱鲆的溃疡病)和贝(如鲍溃烂病)等[19]。哈维弧菌的致病性与蛋白酶、溶血素、脂肪酶和脂多糖等胞外产物的分泌,以及发光、群体效应、形成生物膜、噬菌体感染、结合铁的能力以及温盐等因素有关[19-20]。如孙铂光[21]发现,哈维弧菌突变的VHH溶血素在大肠杆菌中重组表达后,突变的VHH溶血素S153G失去溶血活性和对大菱鲆的致病性,说明哈维弧菌的溶血素与致病性有关。郝贵杰等[22]研究大黄鱼()致病株哈维弧菌GYC1108-1并证实哈维弧菌胞外蛋白酶为半胱氨酸蛋白酶,攻毒试验显示,该蛋白酶对大黄鱼有致死作用,若该酶被热灭活则失去毒性。因此,深入挖掘哈维弧菌潜在毒力调控基因,并研究其毒力调控机制对于哈维弧菌病害防治有重要意义。本研究表明,和基因的缺失均不影响哈维弧菌致病菌株345的生长能力,运动能力,胞外蛋白酶分泌活性,对抗H2O2、Cu2+和限铁环境胁迫的能力,对大多数被测抗生素的抗性以及对花鲈的毒性,但是和缺失后对氯霉素和氟苯尼考两种氯霉素类抗生素均更为敏感,缺失后生物膜形成能力显著增强,表明和均参与正调控哈维弧菌对氯霉素类抗生素的耐药性,还参与负调控哈维弧菌生物膜形成。这些结果为哈维弧菌基因功能研究奠定基础。
生物膜是细菌黏附于接触表面,分泌多糖基质、纤维蛋白、脂质蛋白等,将自身包绕其中而形成的大量细菌聚集膜样物[23],除有助于细菌黏附宿主细胞外,还调控细菌的耐药性、帮助细菌逃避宿主的免疫反应等[24]。鞭毛运动与细菌生物膜形成,尤其是生物膜形成早期在接触面的定位与结合密切相关[25]。如空肠弯曲菌()鞭毛基因的插入突变显著降低细菌运动性和食物表面形成生物膜的能力[26]。FlhF是一种信号识别粒子型的GTP酶,与弧菌(sp.)、希瓦式菌(sp.)、弯曲杆菌(sp.)、螺杆菌(sp.)和铜绿假单胞菌()鞭毛极性定位相关[27-31]。此外,FlhF还调控细菌的鞭毛数量[27]。López-Sánchez等[32]和Navarrete等[33]发现,恶臭假单胞菌()的转座插入突变体,在生物膜形成、鞭毛合成和鞭毛运动方面均显示重大缺陷。空肠弯曲菌()FlhF调节鞭毛合成,其缺失影响细菌定植、黏附侵袭和趋化基因的表达,进而改变细菌对宿主毒力效应[34]。溶藻弧菌()中,FlhF对极性鞭毛数量和细菌涌动群集运动有正调节作用[35]。本研究中,缺失后,哈维弧菌运动性并无显著变化,但生物膜形成显著增加。因此,缺失可能并不影响哈维弧菌鞭毛数量,而更多影响哈维弧菌鞭毛的定位,从而影响哈维弧菌对宿主表面的黏附和结合以及生物膜的形成。
抗生素广泛用于预防或治疗水产养殖中的细菌性疾病[3,36]。抗生素的过度使用会导致耐药菌产生及食品安全问题。解析细菌耐药机制是保证抗生素正确使用的前提和基础。本研究中,和缺失后对氯霉素和氟苯尼考这两种氯霉素类抗生素菌更为敏感,MIC分别是野生株的1/4、1/4和1/8、1/32,表明和均参与氯霉素类耐药性的调控。
大多数临床分离株中,细菌对氯霉素的耐药性由质粒编码的氯霉素乙酰转移酶(CAT) 介导。CAT催化了氯霉素C-3位羟基的乙酰化,生成了3-乙酰氯霉素,而3-乙酰氯霉素不能与细菌的核糖体结合,从而缺乏抗菌活性[37]。基因广泛存在于革兰阴性菌和大多数革兰阳性菌,可位于染色体、R质粒,甚至在可转移因子中[37]。还发现其他与氟苯尼考和/或氯霉素耐药相关基因。如位于质粒上的非酶抗性基因[38-39]通过主动泵出使菌体内氯霉素含量明显减少而实现细菌耐药[40];耐药基因和均位于质粒上,是氯霉素和甲砜霉素共同的耐药机制,但与氟苯尼考耐药性均无直接关系[41];Kim等[42]从鱼的巴氏杆菌质粒中发现耐氟苯尼考基因并命名为[37],随后在沙门氏菌、大肠杆菌等病原菌中克隆到的同源基因[37]。此外,与抗生素泵出相关的基因、多重耐药基因、外排泵基因转录调节因子RamA[43-44]与氯霉素和/或氟苯尼考耐药相关。除基因位于细菌染色体外,绝大多数已知的氟苯尼考和/或氯霉素耐药相关基因位于可移动遗传元件。本研究发现2个位于染色体上的基因[9]——信号识别粒子型的GTP酶编码基因和未知功能基因,二者均正调控氟苯尼考和氯霉素的耐药性,且可能通过影响哈维弧菌的鞭毛定位,影响哈维弧菌生物膜的形成。细菌生物膜可调控细菌的耐药性,通过渗透限制、营养限制和耐药表型机制导致细菌耐药[45]。渗透限制表现为生物膜中多聚糖所构成的分子屏障和电荷屏障阻止或延缓某些抗生素渗入[45]。营养限制机制与渗透限制密切相关,由于生物膜渗透限制的存在,营养物质不易通过生物膜,而使生物膜内营养缺乏,内层细菌生长减慢,处于饥饿状态,导致细菌对抗生素的敏感性降低[46]。耐药表型机制主要是指细菌耐药性的形成与某些生物膜表型相关基因表达差异有关[47]。因此,可能通过其中某一机制影响细菌对氟苯尼考和氯霉素的耐药性。而基因可能存在类似RamA转录调节因子的作用,通过调控其他与氟苯尼考和氯霉素耐药直接相关基因的表达而间接影响细菌的耐药性。但具体调控机制还需进一步研究。
4 结论
本研究成功构建哈维弧菌和缺失株,与野生株相比,二缺失株生长、运动性、胞外酶活性、抗H2O2和Cu2+压力、吸收铁的能力以及致病性无显著影响,对氯霉素和氟苯尼考两种氯霉素类抗生素更加敏感,缺失株生物膜形成能力显著增强,表明和均参与调控哈维弧菌对氯霉素类抗生素的耐药性,还参与调控哈维弧菌生物膜形成。本研究丰富了哈维弧菌氯霉素类耐药性的研究,对防止氯霉素类药物耐药菌的产生和指导氯霉素类药物的合理应用有重要意义。
[1] SHEN G M, SHI C Y, FAN C, et al.Isolation, identification and pathogenicity of, the causal agent of skin ulcer disease in juvenile hybrid groupers×[J].Journal of Fish Diseases, 2017, 40(10): 1351-1362.
[2] XU X D, LIU K F, WANG S F, et al.Identification of pathogenicity, investigation of virulent gene distribution and development of a virulent strain-specific detection PCR method forisolated from Hainan Province and Guangdong Province, China[J].Aquaculture, 2017, 468: 226-234.
[3] SANTOS L, RAMOS F.Antimicrobial resistance in aquaculture: Current knowledge and alternatives to tackle the problem[J].International Journal of Antimicrobial Agents, 2018, 52(2): 135-143.
[4] HE Y, JIN L L, SUN F J, et al.Antibiotic and heavy-metal resistance ofisolated from fresh shrimps in Shanghai fish markets, China[J].Environmental Science and Pollution Research, 2016, 23(15): 15033-15040.
[5] DENG Y Q, XU L W, CHEN H X, et al.Prevalence, virulence genes, and antimicrobial resistance ofspecies isolated from diseased marine fish in South China[J].Scientific Reports, 2020, 10: 14329.
[6] SADAT A, EL-SHERBINY H, ZAKARIA A, et al.Prevalence, antibiogram and virulence characterization ofisolates from fish and shellfish in Egypt: a possible zoonotic hazard to humans[J].Journal of Applied Microbiology, 2021, 131(1): 485-498.
[7] MAZZANTINI D, CELANDRONI F, SALVETTI S, et al.FlhF is required for swarming motility and full pathogenicity of[J].Frontiers in Microbiology, 2016, 7: 1644.
[8] KONKEL M E, TILLY K.Temperature-regulated expression of bacterial virulence genes[J].Microbes and Infection, 2000, 2(2): 157-166.
[9] DENG Y Q, XU H D, SU Y L, et al.Horizontal gene transfer contributes to virulence and antibiotic resistance of345 based on complete genome sequence analysis[J].BMC Genomics, 2019, 20(1): 761.
[10] VAL M E, SKOVGAARD O, DUCOS-GALAND M, et al.Genome engineering in: a feasible approach to address biological issues[J].PLoS Genetics, 2012, 8(1): e1002472.
[11] LIU J X, ZHAO Z, DENG Y Q, et al.Complete genome sequence ofLMB 29 isolated from red drum with four native megaplasmids[J].Frontiers in Microbiology, 2017, 8: 2035.
[12] LE ROUX F, BINESSE J, SAULNIER D, et al.Construction of amutant lacking the metalloprotease geneby use of a novel counterselectable suicide vector[J].Applied and Environmental Microbiology, 2007, 73(3): 777-784.
[13] NGUYEN A N, DISCONZI E, CHARRIÈRE G M, et al.gene duplication drives the evolution of redundant regulatory pathways controlling expression of the major toxic secreted metalloproteases inLGP32[J].mSphere, 2018, 3(6): e00582-18.DOI:10.1128/msphere.00582-18
[14] ZHANG Y Q, DENG Y Q, FENG J, et al.CqsA inhibits the virulence ofto the pearl Gentian grouper (♀×♂)[J].Aquaculture, 2021, 535: 736346.
[15] 张亚秋, 邓益琴, 冯娟, 等.哈维弧菌基因缺失株的构建及其相关生物学特性研究[J].南方水产科学, 2020, 16(2): 43-53.
[16] 吴金军.华南海水鱼类网箱养殖区耐药菌和耐药基因研究[D].上海: 上海海洋大学, 2019.
[17] CLSI.Performance standards for antimicrobial susceptibility testing[R].CLSI supplement M100.Clinical and Laboratory Standards Institute, 2019.
[18] 张亚秋.哈维弧菌假定毒力基因的筛选及功能研究[D].上海: 上海海洋大学, 2020.
[19] 李洋, 李强, 张显昱.哈维弧菌及其主要致病因子的研究进展[J].中国农业科技导报, 2014, 16(4): 159-166.
[20] 贝蕾, 苏友禄, 赵超, 等.哈维弧菌基因的克隆与表达[J].南方水产科学, 2018, 14(2): 75-82.
[21] 孙铂光.哈维氏弧菌VHH溶血素作用机理的研究[D].青岛: 中国海洋大学, 2009.
[22] 郝贵杰, 沈锦玉, 潘晓艺, 等.哈维氏弧菌GYC1108-1胞外蛋白酶的制备及免疫原性研究[J].渔业科学进展, 2010, 31(3): 107-112.
[23] GU H, REN D C.Materials and surface engineering to control bacterial adhesion and biofilm formation: a review of recent advances[J].Frontiers of Chemical Science and Engineering, 2014, 8(1): 20-33.
[24] MAH T F C, O’TOOLE G A.Mechanisms of biofilm resistance to antimicrobial agents[J].Trends in Microbiology, 2001, 9(1): 34-39.
[25] GUTTENPLAN S B, KEARNS D B.Regulation of flagellar motility during biofilm formation[J].FEMS Microbiology Reviews, 2013, 37(6): 849-871.
[26] KIM J S, PARK C, KIM Y J.Role of flgA for flagellar biosynthesis and biofilm formation ofNCTC11168[J].Journal of Microbiology and Biotechnology, 2015, 25(11): 1871-1879.
[27] CORREA N E, PENG F, KLOSE K E.Roles of the regulatory proteins FlhF and FlhG in theflagellar transcription hierarchy[J].Journal of Bacteriology, 2005, 187(18): 6324-6332.
[28] MURRAY T S, KAZMIERCZAK B I.FlhF is required for swimming and swarming in[J].Journal of Bacteriology, 2006, 188(19): 6995-7004.
[29] GREEN J C D, KAHRAMANOGLOU C, RAHMAN A, et al.Recruitment of the earliest component of the bacterial flagellum to the old cell division pole by a membrane-associated signal recognition particle family GTP-binding protein[J].Journal of Molecular Biology, 2009, 391(4): 679-690.
[30] GAO T, SHI M M, JU L L, et al.Investigation into FlhFG reveals distinct features of FlhF in regulating flagellum polarity in[J].Molecular Microbiology, 2015, 98(3): 571-585.
[31] BALABAN M, JOSLIN S N, HENDRIXSON D R.FlhF and its GTPase activity are required for distinct processes in flagellar gene regulation and biosynthesis in[J].Journal of Bacteriology, 2009, 191(21): 6602-6611.
[42] LÓPEZ-SÁNCHEZ A, LEAL-MORALES A, JIMÉNEZ-DÍAZ L, et al.Biofilm formation-defective mutants in[J].FEMS Microbiology Letters, 2016, 363(13): fnw127.
[33] NAVARRETE B, LEAL-MORALES A, SERRANO-RON L, et al.Transcriptional organization, regulation and functional analysis of flhF and fleN in[J].PLoS One, 2019, 14(3): e0214166.DOI:10.1371/journal.pone.0214166
[34] 任方哲.空肠弯曲菌FlhF调节鞭毛合成分子机制及其对细菌毒力的影响分析[D].扬州: 扬州大学, 2019.
[35] KUSUMOTO A, KAMISAKA K, YAKUSHI T, et al.Regulation of polar flagellar number by the flhF and flhG genes in[J].Journal of Biochemistry, 2006, 139(1): 113-121.
[36] MO W Y, CHEN Z T, LEUNG H M, et al.Application of veterinary antibiotics in China’s aquaculture industry and their potential human health risks[J].Environmental Science and Pollution Research, 2017, 24(10): 8978-8989.
[37] 杜向党, 阎若潜, 沈建忠.氯霉素类药物耐药机制的研究进展[J].动物医学进展, 2004, 25(2): 27-29.
[38] DORMAN C J, FOSTER T J, SHAW W V.Nucleotide sequence of the R26 chloramphenicol resistance determinant and identification of its gene product[J].Gene, 1986, 41(2/3): 349-353.
[39] BISSONNETTE L, CHAMPETIER S, BUISSON J P, et al.Characterization of the nonenzymatic chloramphenicol resistance () gene of the In4 integron of Tn1696: similarity of the product to transmembrane transport proteins[J].Journal of Bacteriology, 1991, 173(14): 4493-4502.
[40] GEORGE A M, HALL R M.Efflux of chloramphenicol by the CmlA1 protein[J].FEMS Microbiology Letters, 2002, 209(2): 209-213.
[41] SAMS R A.Florfenicol: Chemistry and metabolism of a novel broad-spectrum antibiotic[J].Tieraerztliche Umschau, 1995, 50(10): 703-707.
[42] KIM E, AOKI T.Sequence analysis of the florfenicol resistance gene encoded in the transferable R-plasmid of a fish pathogen,[J].Microbiology and Immunology, 1996, 40(9): 665-669.
[43] KEHRENBERG C, SCHWARZ S.fexA, a novelgene encoding resistance to florfenicol and chloramphenicol[J].Antimicrobial Agents and Chemotherapy, 2004, 48(2): 615-618.
[44] DAI L, WU C M, WANG M G, et al.First report of the multidrug resistance gene cfr and the phenicol resistance genein astrain from swine feces[J].Antimicrobial Agents and Chemotherapy, 2010, 54(9): 3953-3955.
[45] DAVIES D.Understanding biofilm resistance to antibacterial agents[J].Nature Reviews Drug Discovery, 2003, 2(2): 114-122.
[46] WALTERS M C III, ROE F, BUGNICOURT A, et al.Contributions of antibiotic penetration, oxygen limitation, and low metabolic activity to tolerance ofbiofilms to ciprofloxacin and tobramycin[J].Antimicrobial Agents and Chemotherapy, 2003, 47(1): 317-323.
[47] MIKKELSEN H, BALL G, GIRAUD C, et al.Expression ofCupD fimbrial genes is antagonistically controlled by RcsB and the EAL-containing PvrR response regulators[J].PLoS One, 2009, 4(6): e6018.DOI:10.1371/journal.pone.0006018.
Deletion Mutant Construction and Biological Characteristics of Geneandin
DENG Yi-qin1, ZHANG Ya-qiu1,3, LIN Zi-yang1,4, FENG Juan1, CHENG Chang-chang1, MA Hong-ling1, LIU Guang-xin1, SU You-lu2
(1,,,510300,; 2.,,,510225,; 3,201306,; 4.,570228,)
【Objective】 To study the effect of gene deletion of GTP binding protein geneand an unknowngeneon the biological functions of345【Method】Deletion mutant ofandof345 were constructed by homologous recombination technology.The biological characteristics including growth, motility, extracellular protease activity, and drug resistance and toxicity towere compared to the wild type strain【Result】 Neither the deletion ofnoraffected the bacterial growth, motility, extracellular protease activity, pressure sensing to H2O2and Cu2+, absorption and utilization of iron, resistance to most tested antibiotics and toxicity to.However, the deletion mutant ofandwere more sensitive to chloramphenicol and florfenicol.In addition, the ability of bacterial biofilm formation was significantly enhanced with the deletion of.【Conclusion】Theandare involved in regulating the resistance ofto chloramphenicol antibiotics, andis also involved in regulating the biofilm formation of.
;gene knockout;;; biological characteristics; chloramphenicol resistance
S94;Q933
A
1673-9159(2022)02-0001-12
10.3969/j.issn.1673-9159.2022.02.001
2021-10-07
国家自然科学基金(31902415);中国水产科学研究院中央级公益性科研院所基本科研业务费专项资金资助(2022GH03);国家重点研发计划资助(2019YFD0900105);广东省自然科学基金(2019A1515011833);中国水产科学研究院基本科研业务费专项资金(2020XT0407);中国水产科学研究院南海水产研究所中央级公益性科研院所基本科研业务费专项资金资助(2020TS04)
邓益琴(1990―),女,博士,副研究员,从事鱼类细菌病及防治技术研究。E-mail: yiqindd@126.com
邓益琴,张亚秋,林梓阳,等.哈维弧菌和缺失株的构建及其生物学特性[J].广东海洋大学学报,2022,42(2):1-12.
(责任编辑:刘庆颖)
