基于3D卷积网络和多模态MRI的脑胶质瘤自动分割
2022-03-29王瑞齐崇孟蓝熙刘志强李少武
王瑞,齐崇,孟蓝熙,刘志强,李少武
1.北京市神经外科研究所/首都医科大学附属北京天坛医院,北京 100070;2.国家癌症中心/国家肿瘤临床医学研究中心/中国医学科学院北京协和医学院肿瘤医院放疗科,北京 100021
前言
脑胶质瘤是最常见的由大脑和脊髓胶质细胞癌变所产生的原发性脑肿瘤,本质上是浸润性的,大多数在白质纤维附近增长,但是可以扩散到大脑的任何部分,使它们很难被发现[1]。世界卫生组织通常将脑胶质瘤分为4级,其中I和II级被称为低级脑胶质瘤(LGG),III和IV级被称为高级脑胶质瘤(HGG),其中,IV级脑胶质瘤(GBM)进展尤其迅速、生存率很低。
磁共振成像技术(MRI)是脑胶质瘤诊断和分析的重要成像方式,具有软组织对比度好、参数多、无电离辐射等优势。相比于单模态MRI 成像,多模态MRI 可以为脑胶质瘤提供更多的肿瘤形态信息,为肿瘤精确分割提供基础。这些模态包括T1加权像、T1对比增强加权像、T2加权像、T2液体衰减反转恢复像和弥散张量像等。
在多模态MRI 图像中准确分割脑肿瘤、病变组织的准确定位和量化计算对脑胶质瘤的诊断分级、治疗策略选择、监测肿瘤生长和预后评估等十分重要[2]。目前,脑胶质瘤分割的金标准是通过放射科医生的手动分割,但是存在耗时、耗力、不同医生间存在差异等问题,发展脑胶质瘤的自动分割技术尤为重要。然而,由于不同脑胶质瘤患者的肿瘤位置、形状和结构是高度变化的,因此,如何利用好多模态MRI 获取的丰富的脑胶质瘤形态信息,实现精确自动分割技术是具有挑战并非常有必要的。
利用多模态MRI 数据的脑胶质瘤自动分割算法主要包括生成模型和分类模型[3]。生成模型需要先验的MRI 解剖结构知识,通过图像配准使用后验概率对解剖图像上每个像素进行分类,如基于atlas 方法等[4]。分类模型不需要先验的解剖结构知识,通过提取MRI 图像特征而不是直接利用原始MRI 图像,对解剖图像上每个像素进行分类,如基于机器学习的支持向量机[5]、随机森林[6]等方法。这类方法在训练分类模型前需要进行降维和图像特征提取,这个过程耗时、耗力、依赖经验,并且会因提取的特征不全面而影响分割精度。基于卷积神经网络的深度学习方法可以在训练模型的过程中实现特征的自动提取,因此在肿瘤自动分割方面应用非常有潜力[7-8]。
U-net卷积神经网络在生物医学图像分割方面有很好的效果,它是由同样数量的下采样层和上采样层组成的网络框架,融合不同尺度特征,经过多次下采样后的低分辨率信息可以提供分割目标在整个图像中的上下文信息,而高分辨率信息能够为分割提供更加精细的特征,为精准分割提供定位依据[9]。医学图像的相邻层具有序列信息,使用二维(2D)网络进行分割会丢弃掉部分信息,而3D 卷积神经网络可以更好地捕获序列信息和空间维度特征。本研究提出了一种基于U-net 结构的3D 卷积神经网络模型,从脑胶质瘤患者多模态MRI 图像中自动提取特征,实现脑胶质瘤的自动分割。
1 资料与方法
脑胶质瘤自动分割工作流程(图1)主要包括:(1)多模态MRI 进行预处理;(2)预处理后的多模态MRI和肿瘤分割标注输入3D卷积网络模型进行训练和验证;(3)新患者的多模态MRI 进行预处理;(4)预处理后的多模态MRI 输入到已经训练好的3D 卷积网络模型得到分割输出结果。

图1 基于3D卷积网络和多模态MRI的脑胶质瘤自动分割流程图Figure 1 Flowchart of automatic segmentation of glioma based on 3D convolutional network and multimodal MRI
1.1 数据
本研究选取BraTS2020 公共训练数据集[3,10-11]对模型进行训练和测试,数据集包括369 例脑胶质瘤,其中76 例LGG 和293 例HGG。这些数据是术前MRI 图像,包括T1加权(T1)、T1对比增强加权(T1C)、T2加权(T2)和T2液体衰减反转恢复(FLAIR)4个模态图像(图2)。这些多模态MRI 数据来自于多家单位的不同临床方案和扫描设备,所有图像数据由1~4名医生手动分割,并按照相同标注模板进行标注,这些标注结果进一步由经验丰富的神经放射科医生确认。标注区域(图2)包括增强肿瘤区域(ET-红色标注4)、瘤周水肿区域(ED-绿色标注2)、坏死和非增强肿瘤核心区域(NCR/NET-蓝色标注1)、其他非肿瘤区域(NT-标注0),这些标注被作为金标准(Ground Truth, GT)用于模型训练和测试。这些MRI 数据已经与标准空间的参考图像进行配准,将颅骨剥离分离出大脑区域,并将大脑图像重新采样至1 mm×1 mm×1 mm体素分辨率。

图2 脑胶质瘤多模态MRI和肿瘤标注区域Figure 2 Multimodal MRI of glioma and labeled tumor regions
1.2 数据预处理
为了充分利用多模态MRI数据的3D空间维度,按照3D脑组织的边界轮廓对MRI和标注图像(mask)进行3D裁剪,并重新取样为128×128×128的体像素。
由于运动和磁场不均匀性,会产生多模态MRI伪影,主要包括背景强度不均性和非标准MRI 强度值两种伪影。应用磁场偏差修正技术修正MRI 图像的背景强度不均性伪影,利用N4ITK 方法[12]进行修正。对于非标准MRI 强度值伪影,采用归一化的方法如式(1)所示进行修正:

其中,x表示每个模态MRI的强度值,μ和σ表示MRI中整个大脑区域强度值的平均值和标准差。归一化后的强度值截断在[-5,5]的区间,并最终归一到[0,1]之间。
1.3 3D卷积网络模型
本研究采用基于U-net 的3D 卷积网络结构[13],如图3所示。相比于U-net 网络,本研究采用的网络在不同维度上结合了多个分割特征,应用元素求和将分割特征从网络结构的一个阶段传递到另一个阶段,在下采样过程中采用残差卷积网络的形式,经过多次下采样后的低分辨率信息,可以提供分割目标在整个图像中的上下文信息,这个特征有助于物体的类别判断,上采样过程中结合不同尺度的特征为分割提供了更加精细的定位依据,整合不同尺度的分割层连接网络输出实现深度监督。

图3 基于3D卷积网络的脑胶质瘤自动分割网络模型Figure 3 Automatic segmentation network model of glioma based on 3D convolution network
网络模型结构解释如下:3DConv 包括3D 卷积+归一化InstanceNormalization+激活函数LeakyReLU,其中卷积核是3×3×3。Strided 3DConv 表示步长是(2, 2, 2)的3DConv。Context module 包括3DConv+随机关闭部分训练节点SptaialDropout3D+3DConv。Upsampling module 包括3D 上采用UpSampling3D+3DConv。Localization module 包括3DConv+卷积核是1×1×1 的3DConv。Segmentation layer 包括卷积核是1×1×1 的3DConv。3DUpscale 包括3D 上采用UpSampling3D。Sigmoid是分类器函数。
1.4 训练和测试
随机选取64%的训练样本(training)、16%的验证样本(validation)和20%的测试样本(testing)训练模型。考虑4个MRI模态的整个脑部区域,网络模型的输入数据维度是4×128×128×128,模型训练参数batchsize设置为2,选择adam优化器沿着梯度下降的方向优化模型,每迭代2个训练样本更新一次优化权值,最大迭代次数epochs 设置为200,初始学习率(learning rate,lr)设置为5e-4。
本研究采用多类相似系数Dice 作为损失函数,如公式(2)所示,此损失函数具有可微分性,可以整合到模型的训练框架里。

其中,x是经过网络分类器输出的肿瘤分割,y是编码的金标准GT 肿瘤分割标记,i是x和y包括的像素数量,c∈C是分类的种类,C是分类数量。
训练过程中随着迭代次数增加,自动调整学习率lr,即训练迭代10 次以后,验证数据集上的损失函数不再减小,学习率lr减小为原来的一半。考虑有限的数据样本可能出现过拟合现象,训练过程中应用数据扩增和Early-Stopping技术。数据扩增是训练过程中将数据进行随机3D 翻转增加训练数据的多样性,Early-Stopping 是训练迭代20 次后,验证数据集上的损失函数不再减小,停止继续迭代,保留在验证数据上损失函数最小的模型作为最终的模型。
对于新测试例,将患者的多模态MRI 经过1.2 中的预处理后输入已经训练好的3D 卷积网络模型,便可以得到肿瘤的自动分割结果。
1.5 评价指标
分割结果的评价基于3 个指标,即相似性系数(Dice Similarity Coefficient,DSC)、召回率(Recall)和精确率(Precision)。对整体肿瘤区域(包括ET、ED和NCR/NET)、核心肿瘤区(包括NCR/NET 和ET)和增强肿瘤区(ET)分别计算3个评价指标。
DSC 表示对象间的相似程度,主要用于评价分割出的肿瘤区域O 与专家标记的金标准实际肿瘤区域G的空间重叠情况,如公式(3)所示:

召回率是指分割正确的肿瘤区域占实际肿瘤区域的比例,评价的是分割算法分割出脑胶质瘤的能力,如公式(4)所示:

精确率是指分割正确的肿瘤区域占分割肿瘤区域的比例,评价的是分割算法正确分割出脑胶质瘤的能力,如公式(5)所示:

2 结果
本研究的测试数据集包括随机选出的74例脑胶质瘤样本,其中HGG 为57 例,LGG 为17 例。图4是3D 卷积网络模型分割出的HGG 与LGG 的定性结果,整个分割区域中绿色区域为瘤周水肿区(ED),红色区域为增强肿瘤区域(ET),蓝色区域为坏死和非增强肿瘤核心区域(NCR/NET),可以看出网络模型的自动分割能力很强,自动分割的结果与专家分割的GT一致性很好。

图4 基于3D卷积网络的脑胶质瘤自动分割结果实例Figure 4 Examples of automatic segmentation results of glioma based on 3D convolutional network
图5是在测试数据集上脑胶质瘤不同区域的分割结果的箱式图。整体而言,测试数据集上整体肿瘤区域、核心肿瘤区域和增强肿瘤区域的DSC 平均值分别为0.88、0.77 和0.73,Recall 平均值分别为0.88、0.78 和0.78,Precision 平均值分别为0.89、0.83 和0.75;测试数据集上整体肿瘤区域、核心肿瘤区域和增强肿瘤区域的DSC 中位值分别为0.90、0.84 和0.81,Recall 中位值分别为0.90、0.84 和0.84,Precision中位值分别为0.91、0.89和0.79。
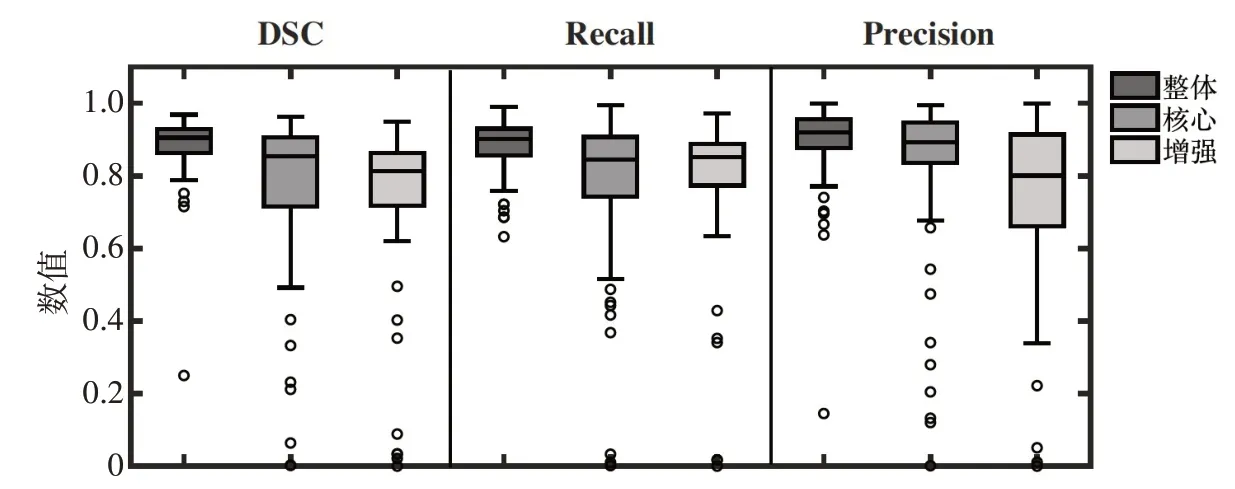
图5 脑胶质瘤不同区域分割结果箱式图Figure 5 Box plots of the segmentation results of different regions of glioma
3 讨论
脑胶质瘤的分割为诊断分级和治疗策略选择提供重要信息,自动分割技术的发展可以解决脑胶质瘤分割耗时耗力和依赖医生经验的问题。相比于传统的生成模型和分类模型自动分割技术,基于卷积神经网络的深度学习方法是近几年的研究热点,非常具有潜力。本研究将基于U-net的3D卷积神经网络应用在多模态MRI数据上实现了脑胶质瘤的自动分割,具有对HGG、LGG同时进行自动分割的能力。本研究利用的BraTS2020数据集是目前公开可以利用的最新的多模态MRI数据样本,包括的数据最多,是测试自动分割算法的优质数据库。本研究应用的网络模型在形式上看类似于U-net网络结构,但从细微结构上看使用了大量的残差网络微结构ResNet,即在下采样过程中使用了残差网络结构,可以更好地保留肿瘤区域空间结构特征,并且应用3D网络模型不但融合了不同尺度的多模态MRI特征,而且捕获了更多空间维度特征,模型优化迭代过程中利用多类相似系数Dice作为损失函数,其直接与最后的评价指标相关。
不同研究者利用深度学习网络在脑胶质瘤自动分割方面进行了不同的探索。Naser等[14]利用2D的U-net网络,在110例5折交叉验证数据集上,考虑FLAIR模态作为输入,实现对整体肿瘤区域的分割结果DSC平均值为0.84。Buda等[15]同样利用2D的U-net网络,在110例22折交叉验证数据集上,考虑3个模态MRI图像数据作为输入,实现对整体肿瘤区域的分割结果DSC平均值为0.82。Hussain等[16]利用2D深度卷积网络,在BraTS2013和BraTS2015数据集上,考虑4个模态MRI数据作为输入,实现对整体肿瘤区域的最好分割结果DSC平均值为0.87。Pereira等[17]利用2D卷积网络,在BraTS2013和BraTS2015数据集上,考虑4个模态MRI数据作为输入,实现对整体肿瘤区域的分割结果DSC平均值分别为0.84和0.78。与前述研究相比,本研究在BraTS2020数据集上,考虑4个模态MRI数据作为模型输入,实现对整体肿瘤区域的分割结果DSC平均值为0.88。本研究用的数据是目前公开可以利用的最新数据,虽然与其他研究者用的数据不同,但存在部分重叠,初步结果表明与其他研究者的结果相近甚至更好。
本研究利用最新的BraTS2020公开数据集,应用基于U-net的3D卷积神经网络在多模态MRI上实现了对HGG、LGG的自动分割。网络模型不局限于对脑胶质瘤的自动分割,如果有可以利用的训练数据,同样可以实现对其他不同肿瘤的自动分割,同时可以应用在更多的医学图像处理领域,如应用在肿瘤放射治疗中基于多模态MRI的肿瘤靶区、危及器官的自动分割等[18]。
