苎麻kinesin基因家族全基因组鉴定与表达分析
2022-03-25于娜陈晓蓉石亚亮牛娟陈静钟意成陈建华孟超敏栾明宝
于娜,陈晓蓉,石亚亮,牛娟,陈静,钟意成,陈建华,孟超敏*,栾明宝*
(1.河南科技大学农学院,河南 洛阳 471000;(2.中国农业科学院麻类研究所/农业农村部麻类生物学与加工重点实验室,湖南 长沙 410205;(3.江西省麻类研究所,江西,宜春 336000)
驱动蛋白(Kinesin)属于马达蛋白的一种,参与生物体的生命活动过程[1-4]。该家族基因包含由两个头部构成的马达结构域(motor domain),其上具有ATP结合位点和微管结合位点[5],驱动蛋白的头部依附在微管上,利用ATP水解释放的能量来驱动自身及其携带的货物分子运动。茎部主要由缠绕螺旋结构(coiled coil)构成,对驱动蛋白的运动方向或活动调节等特性至关重要[6]。尾部与包含货物的囊泡等膜性细胞器相连接[7]。
驱动蛋白是一类蛋白质超级家族,在动物、植物、人和真菌等生物体中普遍存在[8-10]。1985年从鱿鱼的轴突细胞中首次发现了驱动蛋白,随后被广泛研究[11-12],伴随基因组测序技术的普遍应用,相继从人[8]、小鼠[13]、拟南芥[14]、水稻[15]、毛果杨[16]、秀丽隐杆线虫[16]、酿酒酵母[16]等生物体中鉴定出来。Kinesin蛋白沿微管运输细胞货物,并调控微管的生长和稳定性,在细胞分裂、细胞伸长和细胞器的细胞内转运等细胞生物学过程发挥重要作用[17-18]。拟南芥AtFRA1,主要参与纤维素微纤丝的定向沉积[19]。Kinesin-like-钙调素结合蛋白通过直接与皮质微管相互作用,在棉纤维发育过程中对皮层微管的重组发挥作用[20]。新型驱动蛋白KCH1,在棉纤维中以点状方式修饰皮层微管,其可能在介导棉纤维中微管和肌动蛋白微丝之间的动态相互作用中发挥作用,进而影响棉花纤维细胞的发育[21]。GhKCH2是一种植物特异性微管依赖性运动蛋白,不仅与微管相互作用,且与微丝强结合,与两种细胞骨架元素相互作用,在棉纤维发育过程中发挥重要作用[22]。
苎麻是麻类作物中纤维性状最优良的韧皮纤维作物之一,是重要的纺织工业原料。研究苎麻kinesin基因对于揭示韧皮纤维发育分子机制,提高苎麻产量和品质等具有重要意义。本研究基于苎麻基因组数据,通过生物信息学方法,在苎麻中鉴定kinesin家族基因,并对其基本理化性质、蛋白结构、顺式作用元件和表达进行了分析,为进一步研究苎麻kinesin基因的功能和纤维发育分子机制提供依据。
1 材料和方法
1.1 试验材料
试验材料为成熟期苎麻品种“中苎1号”,取自国家种质麻类资源圃。参照Xie等[23]的方法并略加修改,对苎麻的顶端茎皮(TOP)、非完全伸长节间(ER)和完全伸长节间(FER)进行区分和收集。样品立即在液氮中冷冻,并储存在-80℃冰箱备用。
1.2 苎麻kinesin基因家族的鉴定
通过SRAtoolkit软件下载登录号为SRR9112643-SRR9112651的SRA数据,该数据是包含3个生物学重复的苎麻3个不同部位RNA-seq原始数据[23]。从Pfam数据(http://www.pfam.org/)中下载kinesin家族蛋白序列的隐马尔科夫模型文件(HMM,PF00225),使用HMMER3.0软件将kinesin HMM文件与本地蛋白数据库进行比对,搜索含有kinesin蛋白保守结构域的蛋白及核酸序列,筛选出kinesin基因家族成员。利用在线网站ExPaSy(http://www.expasy.org/tools/)预测苎麻kinesin蛋白的氨基酸数(aa)、分子量(MW)和等电点(PI),基于pLoc-mPlant在线预测苎麻kinesin蛋白序列的亚细胞定位。
1.3 BnKIN基因系统进化树的构建
从Swiss-Prot数据库下载拟南芥和水稻的kinesin家族基因蛋白质序列,使用分析软件MEGA7的ClustalW程序对kinesin蛋白进行多重序列比对,采用邻接法(NJ,Neighbor-Joining)构建系统进化树,校验参数Bootstrap重复500次。
1.4 BnKIN基因的结构及蛋白保守结构域分析
通过TBtools软件确定苎麻kinesin家族基因内含子与外显子的结构,并绘制基因结构图。利用SMART(http://smart.embl-heidelberg.de/)软件在线分析苎麻的 kinesin蛋白结构域,并利用TBtools软件可视化结果。
1.5 BnKIN家族基因启动子区顺式作用元件分析
选取BnKIN基因转录起始位置上游2.5 kb作为启动子区段,利用在线分析网站PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析BnKIN基因启动子顺式作用元件,并利用TBtools软件绘制启动子中顺式调控元件图。
1.6 BnKIN家族基因GO富集分析
通过在线软件 PANNZER(Protein ANNotation with Z-scoRE)(http://ekhidna2.biocenter.helsinki.fi/sanspanz/)对BnKIN进行基因本体分析,利用TBtools将预测结果进行可视化。
1.7 BnKIN基因家族表达分析
利用Xie等[23]测序完成中苎1号苎麻3个不同发育部位茎皮转录组测序数据,获得BnKIN家族基因的FPKM值(Fragments Per Kilobase of transcript per Million fragmentsmapped),通过TBtools软件对FPKM值进行log2转换并制作表达热图。
1.8 RNA的提取和cDNA反转录
使用艾德莱EASYspin Plus Plant RNA Kit试剂盒提取苎麻茎皮RNA,于Thermo Scientific NanoDrop 2000和琼脂糖凝胶电泳进行RNA浓度和质量检测,检测合格后储存在 -80℃冰箱备用。利用Thermo Scientific RevertAid First Strand cDNA Synthesis Kit试剂盒合成cDNA第一条链,新合成的cDNA用ddH2O稀释一倍,并保存于-20℃冰箱备用。
1.9 荧光定量PCR(qRT-PCR)分析
根据BnKIN家族基因序列,利用Premier 5.0软件设计特异性结合引物(表1),以苎麻18S基因作为内参基因,使用Vazyme公司的ChamQ Universal SYBR qPCR Master Mix试剂,于BIO-RAD CFX96实时荧光定量PCR仪,进行RT-qPCR扩增。反应体系:cDNA模板0.75μL,上下游引物各0.75μL,SYBR Mix 5μL,ddH2O 3.5μL,充分混匀后瞬时离心。反应程序:95℃ 3min;95℃ 30 s,60℃30 s,信号采集,40个循环,反应结束后分析熔解曲线。每个样品设置3个重复处理,使用2-ΔΔCt方法计算基因的相对表达量。

表1 用于RT-qPCR分析的BnKIN基因和18S基因的引物列表Table 1 The primer of BnKIN gene and 18S gene for qRT-PCR analysis

续表1
2 结果与分析
2.1 苎麻kinesin基因家族的鉴定及理化性质与亚细胞定位分析
通过HMMER3.0程序,检索苎麻蛋白组数据中的BnKIN基因家族成员,经过Pfam保守结构域检测,共鉴定出57个基因家族成员。根据苎麻基因组蛋白数据库注释结果,对基因家族成员进行命名。对其蛋白理化性质进行分析发现,苎麻kinesin蛋白的分子量分布范围在15 893.8 Da(BnKIN-7O_2)~325 534.9 Da(BnKIN-12D),氨基酸在142~2857 aa之间,理论等电点范围在4.79(BnKIN-12C)~10.44(BnKIN-8A),其中理论等电点小于7的蛋白占61%。亚细胞定位预测结果显示,35个蛋白位于细胞核,3个蛋白位于细胞质。此外,19种蛋白质位于细胞核和细胞质,表明他们可能具有特定的生物功能。
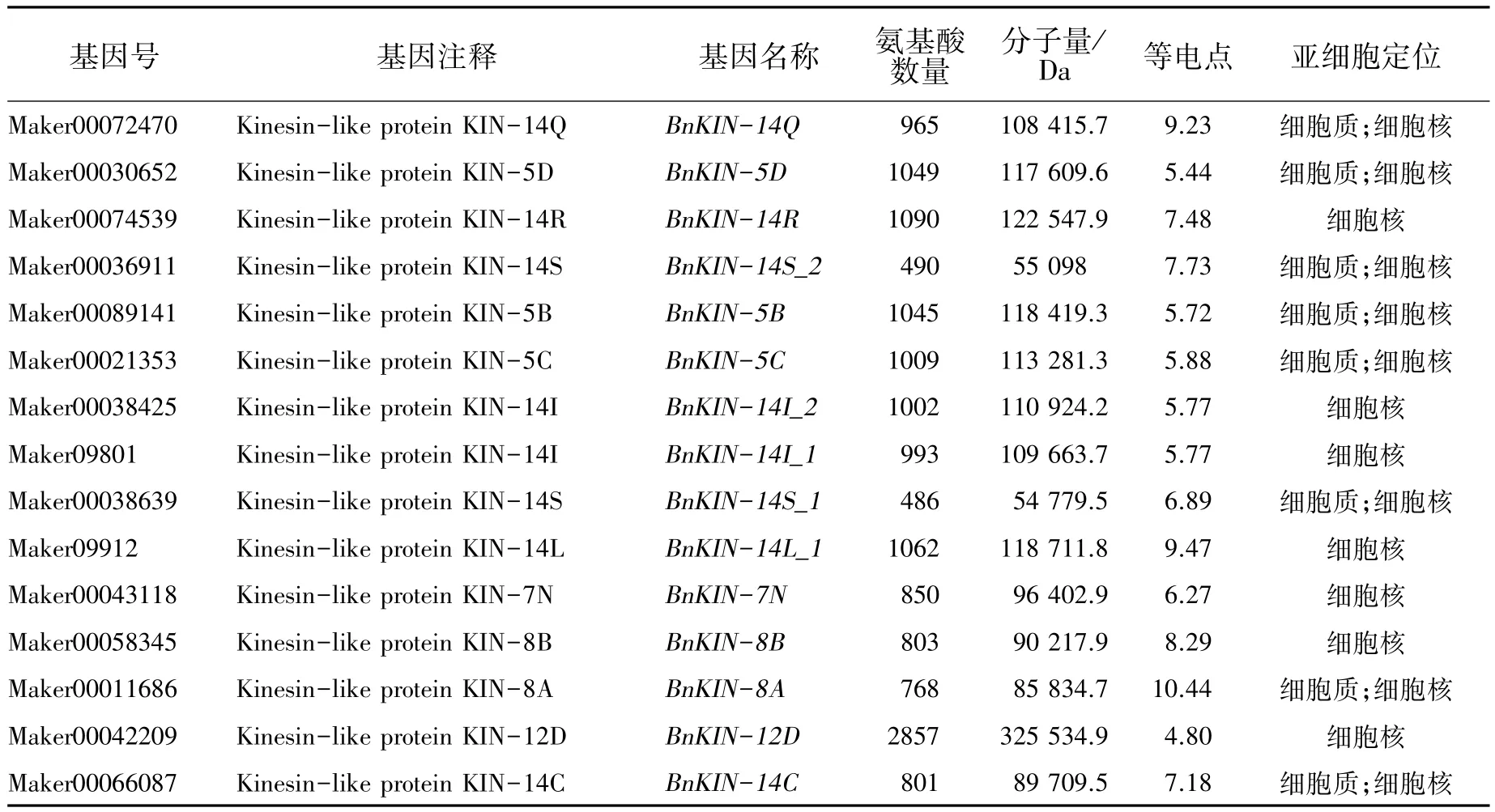
表2 苎麻kinesin家族蛋白理化性质分析Table 2 Analysis of physicochemical properties of BnKIN family

续表2
2.2 苎麻BnKIN家族系统进化分析
在前人[24-26]构建kinesin系统进化树基础上,根据61个拟南芥、52个水稻和57个苎麻的kinesin蛋白序列,构建系统进化树。结果显示,苎麻kinesin家族基因被分成13个类群,其中由于个别亚家族的亲缘关系较远,BnKIN-14亚族基因被分为3个类群,BnKIN-12亚族基因被分为2个类群,在同组内同一个系统进化树分支中的亚族成员具有高度相似的基因结构。13个类群均包含来自拟南芥、水稻和苎麻的kinesin家族基因,且苎麻与水稻和拟南芥的kinesin家族基因大多数聚集在一起,说明该家族在进化的过程中具有一定的保守性。

图1 苎麻、拟南芥和水稻的kinesin家族成员的系统发育树Fig.1 Phylogenetic tree of kinesin familymembers among Boehmeria nivea,Arabidopsis thaliana,and Oryza sativa
2.3 BnKIN基因的结构及蛋白保守结构域分析
根据kinesin基因结构特点,将鉴定出的57个BnKIN家族基因分为9个亚族,进行蛋白保守结构域分析。结果显示,同一亚族的BnKIN基因的保守结构域具有相似性。57个苎麻kinesin基因均具有一个高度保守的kinesin结构域(KISC),44个苎麻kinesin基因具有α-螺旋组成的卷曲的螺旋结构域(α-helical coiled-coil domain),8个BnKIN-14亚家族的基因(BnKIN-14L_1、BnKIN-14L_2、BnKIN-14J、BnKIN-14F、BnKIN-14I_1、BnKIN-14I_2、BnKIN-14I_3和BnKIN-14I_4)具有CH结构域,该结构域可能使BnKIN基因具备特殊的生物学功能[9-10]。基因结构分析显示:BnKIN家族基因具有3~42个外显子,BnKIN-12E的外显子的个数最多,且其内含子/外显子区与非编码区长于其他基因;BnKIN-12D的coiled-coil蛋白保守结构域最多,表明BnKIN-12亚家族的基因成员具有较多的结构域,可能具有复杂的生物学功能;BnKIN-7O_2的外显子个数最少,且其内含子/外显子区与非编码区也短于其他基因。
2.4 BnKIN家族基因启动子区顺式作用元件分析

图2 BnKIN的蛋白保守结构域Fig.2 Conservative domain of BnKIN proteins
为探究苎麻kinesin家族基因功能,对BnKIN启动子区进行顺式作用元件预测。结果显示:BnKIN基因家族成员启动子区含有大量顺式作用元件,可分为激素类、生物和非生物胁迫类和光响应类。响应激素信号的顺式作用元件中,主要含有:茉莉酸甲酯(methyl jasmonate,MeJA)、赤霉素(gibberellin,GA)、脱落酸(abscisic acid,ABA)、生长素(auxin,IAA)、水杨酸(salicylic acid,SA)。此外,还发现了4个与MYB转录因子相关的顺式作用元件。57个BnKIN基因均含有与植物激素有关的顺式作用元件,47个BnKIN家族基因具有MYB转录因子相关的顺式作用元件。
2.5 BnKIN家族基因GO富集分析
对苎麻kinesin家族基因进行GO富集分析,了解其在苎麻纤维发育过程中的作用,结果显示差异表达基因的功能包含18个生物过程(GO:0008150)、15个细胞组分(GO:0005575)和5个分子功能(GO:0003674)。在分子功能中全部kinesin基因具有催化活性和结合功能,少数具有结构分子活性等。在生物过程类别中,kinesin基因主要参与生物调节、细胞进程、发育进程、信号转导及运动等;部分kinesin基因还参与了代谢过程、反应及生长等。在细胞组成类别中,全部kinesin基因是细胞、膜及细胞器等组分。
2.6 BnKINs基因家族表达分析
随着苎麻的生长,韧皮部组织纤维也处于不同发育和成熟阶段,完全伸长节间(FER)韧皮部组织的纤维发育成熟程度最高,非完全伸长节间(ER)次之,顶端茎皮部(TOP)最弱。根据Xie等获得的[23]中苎1号苎麻3个不同部位RNA-seq原始数据,绘制kinesin家族基因表达热图(图6),进而筛选与苎麻韧皮纤维伸长、增厚相关的候选基因。结果显示:kinesin基因家族的不同成员在苎麻不同发育阶段的韧皮部组织(TOP、ER和FER)中表达量存在较大差异。BnKIN-5D等44个基因在TOP中表达较高,BnKIN-14I_2和BnKIN-7H在ER中表达较高,BnKIN-UC_1和BnKIN-1在FER中表达较高。进一步筛选出|log2|>1且Padj值小于0.05的基因,log2FC值大于1的基因为上调表达,小于-1的为下调表达。BnKIN-5D等29个基因在以TOP为对照的ER中下调表达,而BnKIN-14I_2、BnKIN-7H在以TOP为对照的ER中上调表达;BnKIN-12D、BnKIN-14I_3、BnKIN-14I_4、BnKIN-14F在以ER为对照的FER中上调表达(表3)。

表3 中苎一号苎麻纤维发育相关的差异表达基因Table 3 Differentially expressed genes associated with fiber development of Zhongzhu No.1
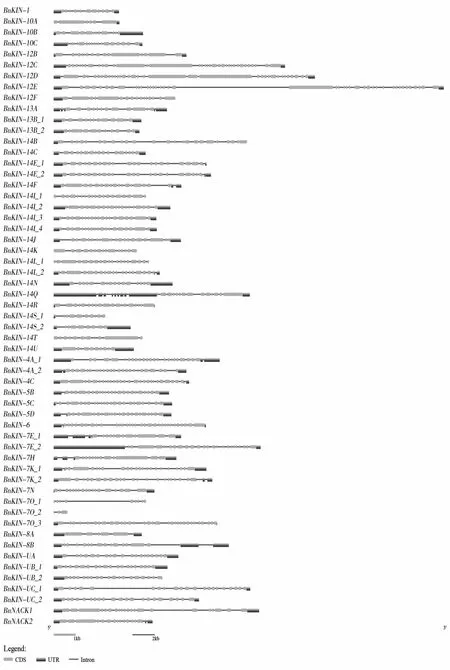
图3 BnKIN的基因结构Fig.3 The gene structures of BnKIN

图4 BnKIN基因启动子顺式作用元件分布图Fig.4 Distributionmap of promoter cis-acting element of BnKIN genes
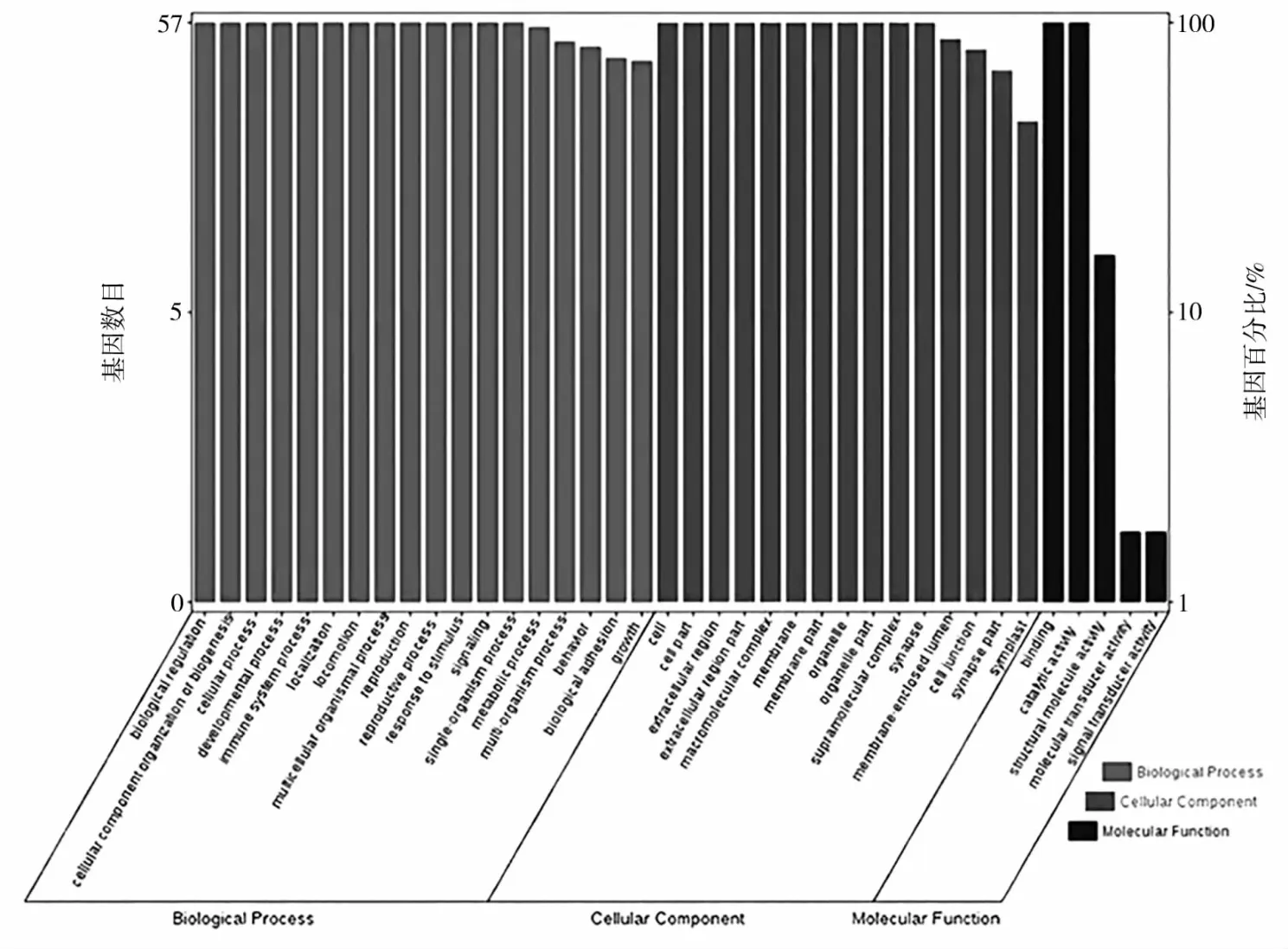
图5 BnKIN的GO富集分析Fig.5 The GO enrichment analysis of Kinesin proteins

图6 中苎一号苎麻kinesin家族基因表达热图Fig.6 Expression profiles of kinesin family genes in different tissues of Zhongzhu No.1
2.7 荧光定量PCR分析BnKIN基因家族不同发育部位的表达量变化
为得到苎麻韧皮纤维发育的候选基因,依据筛选出的苎麻不同部位的差异表达基因,利用荧光定量PCR检测其表达水平。结果显示,30个基因在顶端、节间伸长区和节间完全伸长区的茎皮中的表达存在差异,其中14个基因有显著表达。BnKIN-5D、BnKIN-14S_2、BnKIN-5B、BnKIN-14S_1、BnKIN-7N、BnKIN-12D、BnKIN-14C、BnKIN-14N、BnKIN-4A_2、BnKIN-12C、BnKINUA、BnKIN-7K_1、BnKIN-7H、BnKIN-14I_3、BnKIN-14I_4、BnKIN-12B、BnKIN-7E_1、BnKIN-4C、BnKIN-12F、BnKIN-14F和BnKIN-10C在完全伸长节间的茎皮中表达量最高,BnKIN-8A和BnKIN-6在未完全伸长节间的茎皮中表达量最高,BnKIN-5C、BnKIN-14I_2、BnKIN-UC_2、BnKIN-4A_1、BnKIN-7O_3、BnKIN-14L_2和BnKIN-14J在顶端的茎皮中表达量最高。为消除苎麻顶部其他组织对筛选韧皮纤维发育候选基因的影响,与转录组测序取用顶芽不同,本研究实际取样为苎麻顶端茎皮,这可能是转录组测序数据与荧光定量PCR基因表达量不一致的原因。
3 讨论
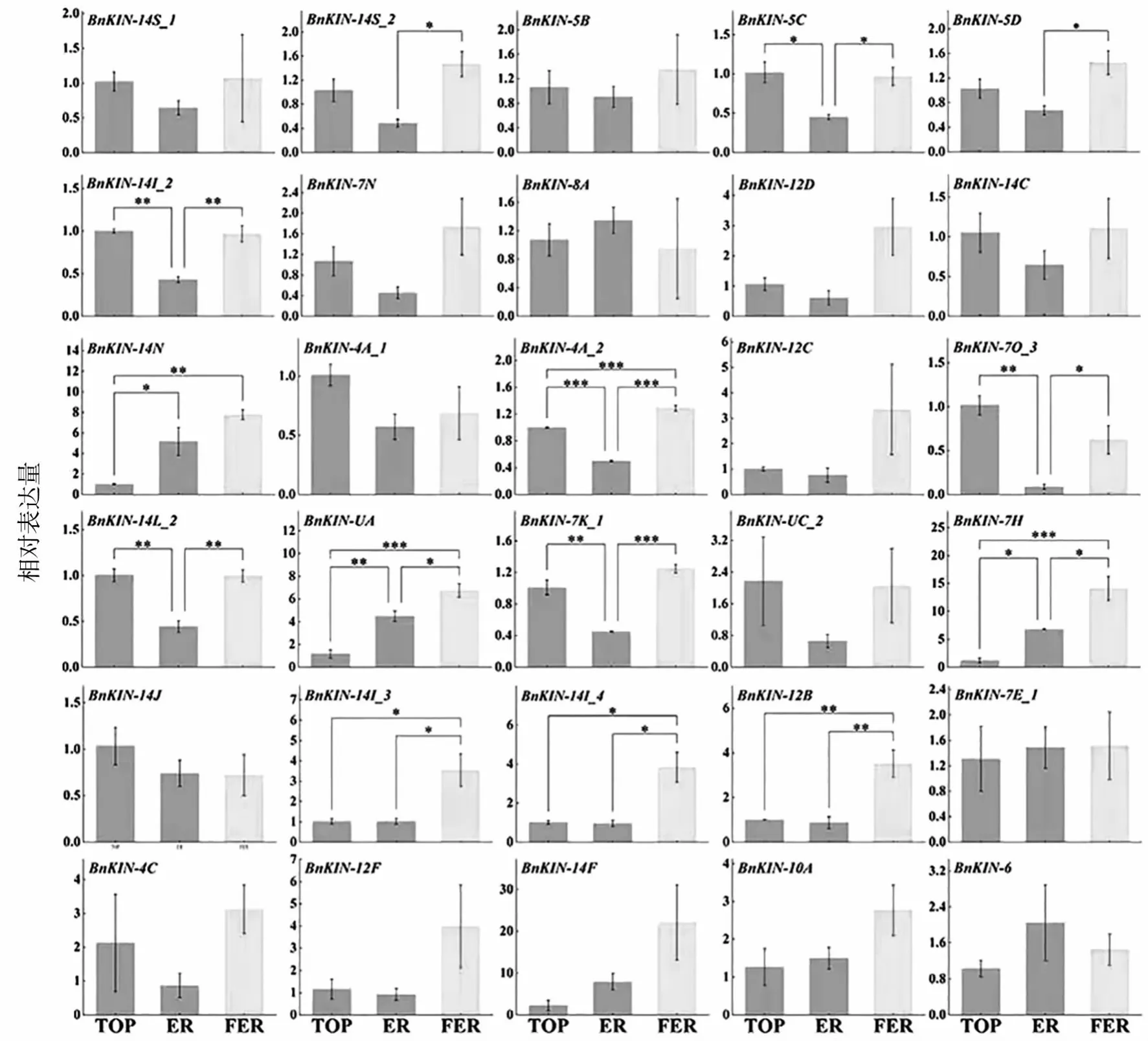
图7 中苎一号苎麻纤维发育相关的基因表达分析Fig.7 Expression profile of genes associated with fiber development of Zhongzhu No.1
Kinesin是真核生物中普遍存在的蛋白,在植物细胞有丝分裂、信号转导、细胞器运输方面发挥重要作用[26-29]。Kinesin家族基因广泛存在于生物体中,目前研究结果表明,拟南芥有61个kinesin基因家族成员[17],毛果杨有52个[9],水稻有 52个[8],但尚未有对苎麻kinesin基因家族进行鉴定与分析的报道。伴随苎麻全基因组测序的完成,本研究首次从苎麻中鉴定出57个kinesin基因家族成员,根据kinesin家族基因的同源关系,将其分为9个亚族,对其结构域进行分析,结果显示,9个亚族的所有成员均包含一个高度保守的KISC结构域,44个BnKIN家族成员具有α-helical coiled-coil结构域,部分BnKIN-14亚家族成员具有CH结构域。CH结构域与微丝和细胞骨架结合,和发育中的棉纤维中的肌动蛋白丝相互作用[14-15],表明具有CH结构域的部分BnKIN-14亚族成员可能在韧皮纤维发育过程中发挥重要作用。进化树分析表明,每一组均含有苎麻、拟南芥和水稻家族基因,表明这三种作物的亲缘关系较近。每一组均包含来自单子叶和双子叶植物的kinesin蛋白,表明该家族基因基本特征在单、双子叶植物分化之前已经形成。
前人[30-31]认为生长素、茉莉酸甲酯、赤霉素对纤维发育起积极作用,而脱落酸抑制纤维生长。有学者发现,MYB转录因子调节一系列下游基因,从而激活次生壁生物合成,进而影响纤维发育[32-33]。通过苎麻BnKIN基因家族成员的顺式作用元件分析,发现BnKIN基因家族成员启动子区含有许多响应激素信号、光和转录因子相关的元件,包括茉莉酸甲酯、赤霉素、脱落酸、生长素、水杨酸和MYB转录因子等。表明BnKIN的上游启动子可能受到植物激素和MYB转录因子的调控,从而在纤维生长发育中发挥重要作用。
转录组测序数据显示kinesin基因家族的不同成员在苎麻不同发育阶段的韧皮部组织中表达量存在较大差异。本研究采用qRT-PCR技术对中苎一号苎麻不同部位的kinesin家族基因成员表达变化进行分析,结果发现不同BnKIN家族成员在苎麻不同组织中的表达量存在差异,大多数基因在成熟程度较高的茎皮中表达量较高,揭示这些基因可能参与次生细胞壁生物合成,在苎麻韧皮纤维的生长发育过程中起重要的作用,后续有待于进一步对这些基因的功能进行验证。对苎麻kinesin家族的鉴定及分析,可为进一步研究BnKIN的生物学功能和韧皮纤维发育的分子机制奠定基础[34-36]。
4 结果
苎麻是我国重要的韧皮纤维作物,苎麻纤维及其织物、织品是我国重要工业原料和传统的出口创汇产品,纤维品质的优劣决定着纤维的可纺性能和织物的使用性能。分子生物学技术的完善,使得在常规育种的基础上,研究纤维相关基因是培育新品种及创制优异种质资源的关键环节,对纤维作物而言,这些基因的研究对改良纤维品质具有重要意义。因此,培育纤维发育优异品质苎麻种质资源迫在眉睫,这既满足日常人们生活需要,又对农业工业生产都具有重要意义。本研究通过对苎麻kinesin基因家族全基因组鉴定及表达分析,筛选到苎麻纤维发育的候选基因BnKIN-5D、BnKIN-14S_2、BnKIN-5B、BnKIN-14S_1、BnKIN-7N、BnKIN-12D、BnKIN-14C、BnKIN-14N、BnKIN-4A_2、BnKIN-12C、BnKIN-UA、BnKIN-7K_1、BnKIN-7H、BnKIN-14I_3、BnKIN-14I_4、BnKIN-12B、BnKIN-7E_1、BnKIN-4C、BnKIN-12F、BnKIN-14F和BnKIN-10C,为后续苎麻 kinesin家族的功能验证奠定基础,并为筛选苎麻纤维发育候选基因提供依据。
