陆地棉GELP家族基因鉴定及其响应胁迫的表达分析
2022-03-23默韶京王志城王省芬刘正文吴立强张桂寅马峙英张艳段会军
默韶京,王志城,王省芬,刘正文,吴立强,张桂寅,马峙英,张艳,段会军
(河北农业大学农学院,华北作物改良与调控国家重点实验室,河北省作物种质资源重点实验室,河北 保定 071000)
脂肪酶是一种脂质水解酶,广泛分布于动物、植物及微生物中。脂肪酶家族成员包含3个高度保守的氨基酸残基(Ser、Asp和His),可形成催化三联体[1]。根据基因保守结构域特点将脂肪酶分为GxSxG-motif和GDSL-motif脂肪酶。GxSxG型脂肪酶的活性位点位于蛋白的中心附近,而GDSL型脂肪酶(GDSL-type esterase/lipase proteins,GELPs)的活性位点Ser位于蛋白的N端附近,在保守区Ⅰ中表现为Gly-Asp-Ser-(Leu)[GDS(L)]基序[2-3]。所有GELP脂肪酶蛋白结构中都包括5个保守域(Ⅰ~Ⅴ),其中Ⅰ、Ⅱ、Ⅲ和Ⅴ保守域内分别存在严格保守的Ser(S)、Gly(G)、Asn(N)和His(H)催化残基,因此GELP脂肪酶也属于SGNH水解酶超家族。GELP脂肪酶的活性位点非常灵活,因此该脂肪酶家族具有广泛的底物特异性,能水解硫酯、芳酯、磷脂和氨基酸等多种底物[3-5]。
GELP脂肪酶家族成员众多,对其功能的报道主要聚焦在调节植物次生代谢、生长发育、形态建成等,GELP脂肪酶参与植物防御反应逐渐引起研究者的注意,目前这方面的研究主要集中在模式植物拟南芥中。拟南芥GLIP1(GDSL LIPASE-LIKE 1)具有抗菌活性,可通过对乙烯信号进行正、负反馈调节来调控植物免疫[6-8];拟南芥GLIP2通过负调控植物生长素信号通路,参与了对胡萝卜软腐菌的抗性[9]。最新研究表明,拟南芥脂肪酶基因(At1g06990)的突变降低了植株对黑斑病的抗性[10]。此外,过表达拟南芥AtGDSL1基因可引起脂质前体磷脂酸水平的升高,激活下游ROS/SA防御相关通路,进而提高植株对菌核病的抗性[11]。在非模式植物中,辣椒CaGLIP(GDSL-type lipase)可受水杨酸、乙烯和茉莉酸的诱导,对油菜黄单胞菌(Xanthomonascampestrispv.Vesicatoria,Xcv)具有抗性,此外,辣椒不同脂肪酶基因也参与了对非生物胁迫的反应[12-14]。
为了深入发掘GELP脂肪酶基因家族在植物中的生物学功能,研究者对不同物种的GELP基因家族进行了系统的分析,分别从拟南芥、水稻、白菜、葡萄、大豆中鉴定到105、114、121、83和194个GELP家族成员[5,15-18]。作为异源四倍体,陆地棉拥有庞大而复杂的基因组,关于棉花GELP脂肪酶家族成员在棉花基因组中的鉴定和分析尚未见报道。
棉花是重要的天然纤维作物,由土传真菌大丽轮枝菌引起的黄萎病是影响棉花产量和品质较严重的限制因素之一。黄萎病菌寄主范围极广,其产生的微菌核可在土壤中存活数十年,使黄萎病的防治难度大。因此,通过基因工程手段增强植物的免疫系统是提高植物对抗病原菌有效、环保的策略[19-22]。本研究基于已公布的陆地棉TM-1三代基因组对棉花GELP家族成员进行全基因组鉴定,并对家族成员特性进行系统分析。结合黄萎病菌、干旱、高温、冷害和盐胁迫处理后的转录组数据库,对参与黄萎病抗性反应和非生物胁迫的GhGELP基因进行筛选,为陆地棉GELP基因家族成员的功能研究奠定重要基础。
1 材料与方法
1.1 数据来源
陆地棉TM-1三代基因组数据下载自CottonFGD(http://www.cottonfgd.org/),拟南芥AtGELP氨基酸序列下载自TAIR(http://arabidopsis.org/),陆地棉4种逆境胁迫(冷、热、干旱和盐)处理后的转录组数据下载自NCBI SRA(sequence read archive)数据库(genome sequencing project accession:PRJNA248163)。
1.2 陆地棉GELP家族基因的鉴定及理化性质
基于GhGELP基因家族特有保守结构域(PF00657),从Pfam数据库(http://pfam.xfam.org/)下载结构域文件,HMMER3.0软件建立隐马尔可夫模型并鉴定候选基因,利用SMART(http://smart.embl-heidelberg.de)和 CDD (http://www.ncbi.nlm.nih.gov/cdd)分析候选蛋白序列,去除不含完整保守结构域的序列。得到的候选基因在CottonFGD进一步确认保守结构域。通过PSORT(https://www.genscript.com/psort.html)进行亚细胞定位分析。蛋白长度、分子量、等电点利用本地Perl脚本批量计算。
1.3 陆地棉GELP家族基因的系统进化分析
通过ClustalW功能对陆地棉GhGELP家族基因编码的蛋白序列进行多序列比对,MEGA7.0软件构建系统进化树,采用邻接法(NJ,Neighborjoining),Bootstrap设置为1 000次。
1.4 陆地棉GELP基因的染色体定位、基因结构、保守结构域和顺式元件预测
基因结构信息和染色体位置信息提取自基因组结构注释文件,并用TBtools软件展示基因结构图,用MapChart软件绘制染色体定位图。MEME(http://meme-suite.org/tools/meme)分析GhGELP家族的保守结构域,motif数量设置为20。利用棉花基因组数据库CottonFGD提取陆地棉GELP基因序列起始密码子ATG上游2 000 bp序列,在线网站PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测可能存在的顺式作用元件,TBtools软件制图。
1.5 陆地棉GELP基因在黄萎病胁迫下的表达量热图绘制
利用本实验室构建的陆地棉栽培品种农大601受黄萎病菌胁迫处理2、6、12、24、48 h根部的转录组数据库(未发表),FPKM(fragments per kilobase of transcript per million fragments mapped)用于评估基因表达量。将GELP家族基因的表达量进行log2(fold change)标准化(fold change代表处理FPKM与对照FPKM的比值),使用HemI(Heatmap Illustrator version 1.0)软件绘制GELP基因家族各成员表达量热图。
1.6 陆地棉GELP基因在非生物胁迫下的表达量分析
利用从NCBI SRA数据库下载的陆地棉低温、高温、盐和PEG6000模拟干旱逆境胁迫处理下的不同时期的转录组数据,对棉花GELP基因在各种非生物胁迫中的全基因组表达进行了分析。差异表达倍数FC(fold change)代表胁迫处理FPKM与对照FPKM的比值。选取差异表达基因的标准为至少在一个处理时间点差异表达倍数FC>2(上调)或FC<0.5(下调)。
2 结果与分析
2.1 陆地棉GELP家族成员分析
通过HMM搜索,在陆地棉基因组中共鉴定出210个GELP的候选基因,并根据它们在相应的染色体中从上到下的顺序和位置,将其命名为GhGELP1~GhGELP210。陆地棉GhGELP基因编码蛋白的氨基酸数目在149(GhGELP42)~804(GhGELP3)之间,平均蛋白序列长度为356个氨基酸。基因组中GhGELP基因的长度则在1 056~18 365 bp之间;蛋白质的分子量分布在18.76~82.87 kD之间,等电点则在4.48(GhGELP210)~9.8(GhGELP95)之间。对棉花GELP家族成员进行亚细胞定位分析,定位结果显示,分别有33、44、6、53、27、2、45个基因定位到细胞质、内质网、高尔基体、线粒体、细胞核、液泡和胞外。亚细胞定位结果在一定程度上印证了GhGELP家族成员在植物生长发育等众多生物学过程中发挥着关键的作用。
2.2 陆地棉GELP家族基因的系统发育分析
为了更好地阐明陆地棉与拟南芥GELP基因的系统发育关系,对210个GhGELP和111个AtGELP蛋白构建系统发育树(图1)。基于最新的TAIR数据库,拟南芥GELP蛋白序列与之前报道[15]的略有不同(拟南芥中不存在AtGELP29和AtGELP57),新增的8个AtGELP均以基因ID表示。依据系统发育树,将321个GELP成员分为10个亚家族(A~J),每个亚族包含3~89个成员。大多数亚族同时包含棉花GELP和拟南芥GELP,表明这些成员可能在双子叶植物中功能保守。此外,Ⅰ亚族7个成员都来自棉花,表明Ⅰ亚族的GhGELP出现于棉花和拟南芥分化之后,可能在棉花生长发育和防御反应中起着独特的作用。拟南芥中AtGLIP1(AT5G40990、AtGLP97)、AtGLIP2(At1g53940、AtGLP20)、AtGDSL1(At1g2967 0、AtGLP16)、AtGLP1(At1g06990)等基因参与了拟南芥对各种病原菌的防御[6-7,9-11,23]。另外,拟南芥中过表达AtLTL1(AT3G04290、AtGLP60)可增加转基因拟南芥的耐盐性[13]。棉花中与AtGLIP1(AtGLP97)和AtGLIP2(AtGLP20)同位于C亚族且同源性最高的GhGELP128、GhGELP28、GhGELP127、GhGELP27,与AtGLP1(At1g06990)同位于E亚族且同源性最高的GhGELP121、GhGELP39、GhGELP11,与AtGDSL1(AtGLP16)同位于F亚族且同源性最高的GhGELP175、GhGELP69、GhGELP130、GhGELP30、GhGELP148、GhGELP46等基因,可能参与了棉花对抗病原菌的防御反应。而与AtLTL1同位于G亚族且同源性最高的GhGELP52、GhGELP154、GhGELP59、GhGELP163则有可能参与了棉花对非生物胁迫的响应。
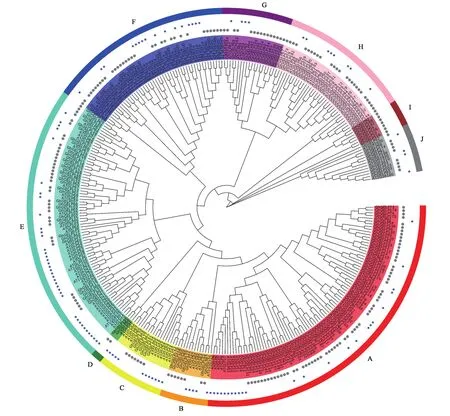
图1 陆地棉和拟南芥GELP家族成员进化分析Fig.1 Phylogenetic analysis of GELP proteins in cotton and Arabidopsis
2.3 陆地棉GELPs染色体定位、基因结构分析
如图2所示,陆地棉210个GhGELP基因不均匀地分布在26条染色体上(GhGELP63位于大片段上,未标注),且主要分布在染色体两端,这与之前报道的GELP基因在大豆基因组中的分布特征相似[18]。其中,在A5和D5染色体上分布比较集中,分别有17和21个成员;A2、D3、D4和D6染色体上分布较少,都仅有4个成员(图2)。

图2 陆地棉GELP家族成员的染色体分布Fig.2 Chromosomal location of the 210 GhGELP genes in G.hirsutum L.
植物GELP基因的显著特征是有5个外显子和4个内含子的结构。例如,在水稻和大豆中,大约49.1%和74.2%的GELP成员含有4个内含子[5,18]。对陆地棉中GELP基因的内含子数目进行统计(未列出),发现陆地棉GhGELP基因内含子数目从1到12不等,所有基因都含有至少1个内含子,除GhGELP30和GhGELP41分别含有7个和12个内含子外,其他成员内含子数目都不多于6个。高达66.7%的GhGELP基因有4个内含子,与水稻和大豆中情况一致。
2.4 陆地棉GELPs保守域和顺式元件分析
保守序列和基序在酶的功能中起着重要作用。210个GhGELP蛋白中共发现了20个保守基序。其中保守基序4、5、9和1分别代表了GELP家族的保守域Ⅰ、Ⅱ、Ⅲ和Ⅴ(图3),这4个保守基序几乎存在于所有GhGELP中。此外,在超过60%的GhGELP蛋白中检测到另外8个保守基序(motif 2、3、6、7、8、11、12和14),其中,高达90%的成员中包含保守基序2和3。其他的保守基序仅存在于系统发育树个别的亚组中,如motif 17、18、20仅存在于A亚族,motif 19仅存在于A、B、D和H亚族。

图3 陆地棉GELP基因家族成员保守基序分析Fig.3 Motif logos of four conservative blocks detected in GhGELP proteins
对GhGELP基因启动子区上游2 000 bp的序列进行分析发现,顺式作用元件包括与激素信号相关的ABA反应元件、GA反应元件、auxin反应元件、水杨酸(SA)应答元件、MeJA应答元件及与环境胁迫相关的低温应答元件和干旱应答元件。其中,ABA反应元件是最丰富的顺式作用元件,128个GhGELP成员含有ABA反应元件。另外,含有GA反应元件、auxin反应元件、SA应答元件、MeJA应答元件、低温应答元件和干旱应答元件的GhGELP基因数目分别为 102、87、89、124、92和111。SA应答元件、JA应答元件、低温应答元件和干旱应答元件的富集表明,棉花GELP基因可能参与了植物对各种病原菌及环境胁迫的响应。
2.5 黄萎病菌胁迫下GhGELP基因表达模式分析
基于黄萎病菌胁迫下陆地棉栽培品种农大601的转录组数据库[24],利用接种处理后不同时间点的表达数据构建GhGELP基因家族成员的表达模式图(未列出),发现有将近1/3的GhGELP基因受黄萎病菌诱导。将这些基因提取出来重新构建表达模式图(图4),进行进一步分析。根据受黄萎病菌诱导后表达模式的不同,将69个GhGELP基因分成3个亚组,第1亚组(Group 1)的成员受黄萎病菌诱导后表达量降低,且该类型基因在响应黄萎病菌诱导的GELP脂肪酶基因中占绝大多数。其中,预测与黄萎病抗性有关的GhGELP175位于该亚组。第2亚组(Group 2)的成员受黄萎病菌诱导后表达持续上调。其中,预测与黄萎病抗性有关的GhGELP128、GhGELP28和GhGELP27位于该亚组。第3亚组(Group 3)的成员受黄萎病菌诱导后表达先下调后上调,该类型基因仅有2个成员。
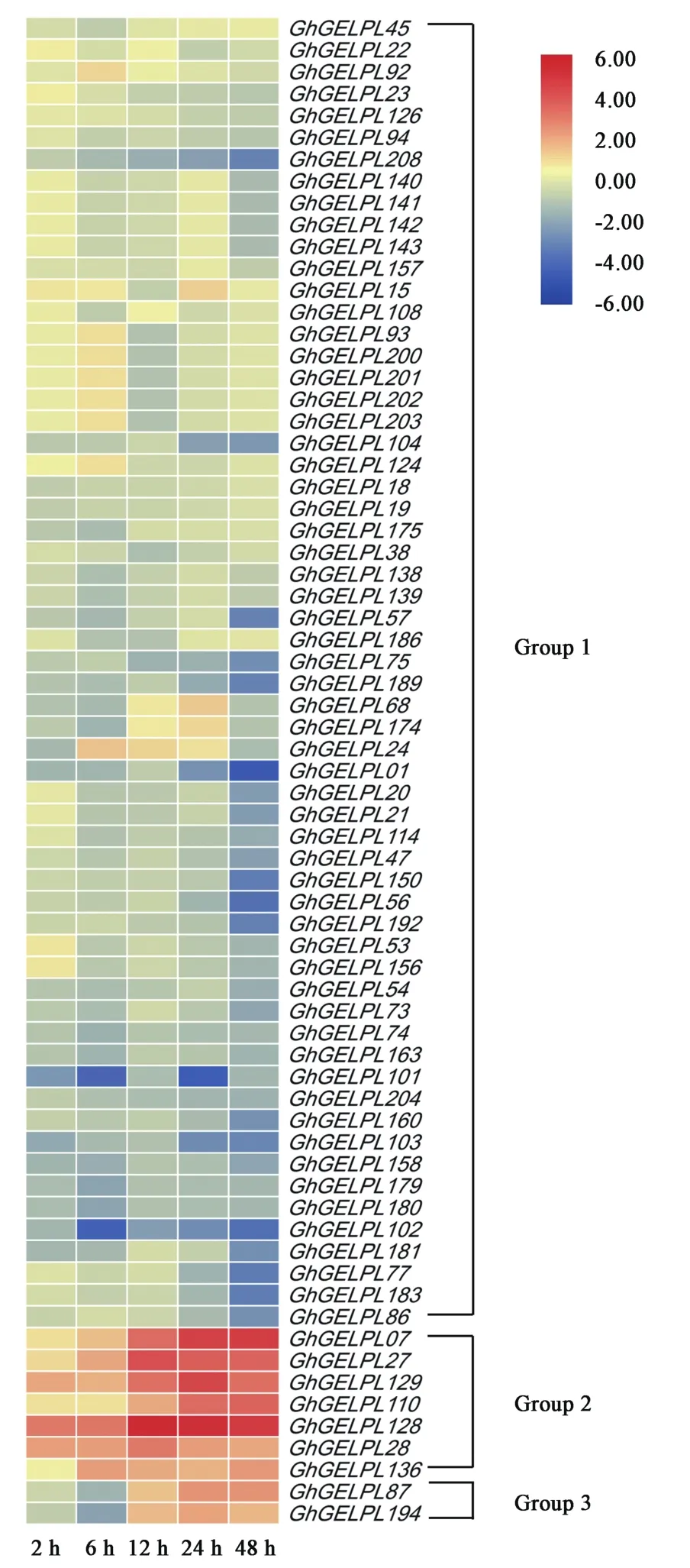
图4 陆地棉GELP基因在黄萎病胁迫下的表达模式Fig.4 Expression profiles ofGhGELPsfrom upland cotton inoculated withV.dahliae
2.6 陆地棉GELP基因家族成员参与非生物胁迫表达分析
基于陆地棉TM-1受到非生物胁迫的转录组数据库,分析GhGELP基因家族响应干旱、高温、冷害和高盐胁迫的表达模式。将受非生物胁迫诱导后显著差异表达的GhGELP基因提取出来构建表达模式图(图5),进行进一步分析。在盐胁迫处理中,大部分的基因在胁迫处理后6和12 h表现出显著下调表达。在PEG处理中,大部分的基因则在胁迫处理后12 h表现出显著下调表达。在热处理中,有将近1/2的基因在胁迫处理后1 h表现出显著下调表达。在冷处理中,有将近1/3的基因在胁迫处理后12 h显著下调表达。在各种胁迫处理中,都只有少数基因在个别时间点显著上调表达。这些表达模式结果表明,陆地棉GELP家族基因受非生物胁迫诱导后呈下调表达趋势。此外,GhGELP2、GhGELP5、GhGELP29和GhGELP37等14个基因表达量同时在4种非生物胁迫处理下发生显著变化。GhGELP26、GhGELP30、GhGELP32和GhGELP42等18个基因表达量同时在3种非生物胁迫下发生显著变化(表1)。这些结果表明,GELP基因在棉花应对各种复杂的环境胁迫时发挥着重要作用。

表1 GhGELP基因对不同胁迫处理的响应Table 1 GhGELP genes response to different abiotic stress

续表Continued

图5 陆地棉GELP基因在各种非生物胁迫下的表达模式Fig.5 Expression profiles of GhGELPs in response to different abiotic stress
3 讨论
GELP基因家族已在拟南芥[15]、水稻[5]和大豆[18]等植物中进行了系统分析。陆地棉属于异源四倍体,基因组庞大而复杂,目前只有极少数GhGELP基因被鉴定[25-26],且主要集中在纤维、种子发育等棉花生长发育进程中。本研究利用高质量的陆地棉三代测序基因组系统鉴定了GELP脂肪酶基因家族,并结合系统发育、染色体分布和序列特征等信息对GELP基因家族进行了系统分析。通过对棉花基因组中GELP基因的搜索,鉴定了210个成员。亚细胞定位结果显示,这些成员可分别定位到细胞质、内质网、高尔基体、线粒体、细胞核、液泡和胞外,与该家族复杂多样的功能相一致[18]。根据系统发育关系将这210个成员分为10个亚家族。染色体定位结果显示,这210个成员大多分布在染色体的两端。5个外显子和4个内含子的结构是植物GELP基因的显著特征,棉花中66.7%的GhGELP基因有4个内含子。该系统分析是了解陆地棉中GELP作用的第1步,为预测GELP在棉花中可能的作用奠定了坚实的基础。
GELP家族的脂肪酶具有灵活的活性位点,可以随着不同底物的存在和结合而改变构象使其拥有更加广泛的底物结合性,因此,该酶底物特异性广泛且具有多重功能[3]。该特性一方面导致GELP基因可在植物生长发育等多种生物过程中发挥作用,如在种子油脂代谢、抗逆性、角质层形成、组织形态发生发育和雄性育性等方面起重要作用[16,25,27-32];另一方面,本研究发现,在响应非生物胁迫的78个GhGELP基因中,有32个基因同时在3种以上胁迫处理下表达量发生显著变化。这可能是由其底物特异性广泛造成的,同一GhGELP蛋白可降解不同的底物以应对不同的环境胁迫。与该结果一致的是,辣椒中CaGLIP1同时介导了对病菌的感病性和对干旱、ABA、葡萄糖等非生物胁迫的抗性[12]。拟南芥AtLTL1受到SA诱导表达上调,在增强转基因植株耐盐性的同时可能也参与了对病原菌的防御反应[13]。
脂肪酶是一类具有脂质代谢功能的酶,脂质及其代谢产物在植物生长和对环境刺激的反应中均发挥着不同的作用,越来越多的证据表明,脂质可作为信号分子介导植物免疫[33-34]。水稻中病原菌感染和水杨酸处理可抑制OsGLIP1和OsGLIP2的表达,同时下调OsGLIP1和OsGLIP2可以提高植物对细菌和真菌病原体的抗性,而过表达OsGLIP1和OsGLIP2则显著降低植物的抗病性,表明这两个基因均是介导水稻抗病性的负调节因子[28]。本研究分析了陆地棉脂肪酶GELP基因家族对黄萎病胁迫的响应,发现棉花接种黄萎病菌后,大多数GhGELP基因呈现下调表达模式,表明棉花与水稻中情况相似,可通过负调控一部分GELP基因来增强植物的抗病性。而AtGDSL1则正调控拟南芥对核盘菌的抗性,其功能缺失导致拟南芥对菌核病易感性增强,油菜中过表达AtGDSL1可增强油菜对菌核病的抗性[11]。棉花中有9个GhGELP基因受黄萎病菌诱导后表达量显著上调,其可能为介导棉花对黄萎病抗性的正调控因子。这些GELP家族基因是否在响应黄萎病胁迫中具有重要作用,还需要进一步研究。
